大麦SERK基因家族cDNA克隆及生物信息学分析
2016-07-02杨沙沙李颖波谭何新郭桂梅陈志伟刘成洪黄剑华
杨沙沙,李颖波,谭何新,郭桂梅,何 婷,陈志伟,刘成洪*,黄剑华*
(1上海海洋大学水产与生命学院,上海201306;2上海市农业科学院生物技术研究所,上海市农业遗传育种重点实验室,上海201106;3第二军医大学药学院,上海200433)
大麦SERK基因家族cDNA克隆及生物信息学分析
杨沙沙1,2,3,李颖波2,谭何新3,郭桂梅2,何 婷2,陈志伟2,刘成洪2*,黄剑华2*
(1上海海洋大学水产与生命学院,上海201306;2上海市农业科学院生物技术研究所,上海市农业遗传育种重点实验室,上海201106;3第二军医大学药学院,上海200433)
摘 要:通过对大麦体细胞胚胎发生受体类蛋白激酶SERK(Somatic embryogenesis receptor-like kinases)基因家族进行注释和基因克隆,得到3条大麦序列,即HvSERK1、HvSERK2和HvSERK3,其中基因HvSERK3与相应已有参考序列(Accession:AK374641)相比,编码氨基酸有所改变。生物信息学分析表明:3条大麦SERK基因都具有1 870 bp左右的开放阅读框,620 aa左右的氨基酸残基数,69 kD左右的相对分子质量大小,平均理论等电点5.5,说明SERK为酸性蛋白;大麦SERK蛋白具有强烈的亲水性,且存在N端信号肽和跨膜螺旋区,二级结构组件主要是环(loop)。本研究为进一步探讨大麦中SERK基因家族的功能奠定了基础。
关键词:大麦;SERK;注释;克隆;生物信息学分析
体细胞胚胎发生受体类蛋白激酶(Somatic embryogenesis receptor-like kinases,SERKs)广泛存在于植物体内,参与调节植物生长发育和各类胁迫响应过程[1]。有关SERK基因的功能已在模式植物拟南芥[2-8]和一些重要禾本科作物如水稻[9-11]、小麦[12]和玉米[13]中都有研究报道,但大麦中有关SERK基因家族的信息还有待研究。
大麦(Hordeum vulgare L.)是仅次于水稻、小麦和玉米的第四大谷类作物,普遍用作麦芽制造、啤酒酿造、饲料生产和食品加工,其适应性广泛,具有较强的耐旱、耐寒和耐盐特性,近年来已成为重要的遗传和生理模式作物[14]。因此,对大麦中的耐逆性相关的基因开展深入研究,将有助于揭示大麦对环境较强适应性的分子机制,为禾谷类作物的耐逆性育种研究提供基础。
在前期研究工作中,Gao等[15]利用Affymetrix基因芯片,对大麦盐胁迫响应基因的表达水平进行了转录谱分析,获得了3 326个差异表达基因,其中探针contig5422_at在大麦耐盐性较强的品种中检测到,注释为Brassinosteroid insensitive 1-associated receptor kinase 1 precursor,进一步序列比对分析确认该基因属于SERKs基因家族。本研究在此基础上对大麦中SERKs基因家族的cDNA进行了全长克隆、验证和生物信息学分析,以便进一步开展大麦中SERKs基因功能研究。
1 材料与方法
1.1材料
供试材料为大麦品种‘花30’,由上海市农业科学院生物技术研究所提供。
用湿润纱布包裹种子于28℃恒温条件下催芽,至种子露白后移种到营养土中,人工气候室中培养至两叶一心期时,取新鲜叶片液氮速冻于-70℃,用于总RNA提取。
1.2方法
1.2.1大麦SERK家族基因的注释及同源性分析
在PLEXdb网站(http://www.plexdb.org/modules/PD_probeset/annotation.php)中检索获得contig5422_ at探针序列;以contig5422_at探针序列为参照,利用大麦全基因组数据库IPK(http://webblast.ipk-gatersleben.de/barley/viroblast.php)进行BLAST分析[16],Database设为“full length cDNA”;利用ClustalX2软件进行多序列比对并用GeneDoc软件对比对结果进行编辑,从而显示序列间相似性和特征性保守区段;利用MEGA 5.0软件,对部分禾本科植物的SERK家族基因及8个大麦序列构建系统进化树;禾本科植物SERK蛋白序列同源比对采用NCBI上的Blastp程序完成。
1.2.2总RNA提取和cDNA合成
利用TRNzol试剂(北京天根生化科技有限公司)提取大麦‘花30’叶片的总RNA;RNA质量检测采用0.8%琼脂糖凝胶电泳的方法,RNA浓度[C(μg/μL)= A260×40×稀释倍数/1 000]采用紫外分光光度计来测定;按照反转录试剂盒产品说明书进行反转录。
1.2.3基因克隆及测序验证
根据目标研究基因在GeneBank中已知的cDNA序列,利用Vector NTI软件设计特异性引物(表1),确保引物无回文结构(Palindromes)和重复序列(Repeats)等。以RNA反转录合成的cDNA为模板进行RT-PCR反应,用于扩增基因全长并测序验证。

表1 引物设计Table 1 Design of primers
1.2.4生物信息学分析
利用网站PredictProtein(https://www.predictprotein.org/)在线预测SERK蛋白二级结构;其他所用生物信息学软件和网站参照参考文献[17]。
2 结果与分析
2.1大麦SERK家族基因的注释及同源性分析
在IPK数据库中比对后,得到8个与探针contig5422_at相似性较高的全长cDNA序列,即AK372118 (99%)、AK252995(83%)、AK374641(78%)、AK371411(68%)、AK251794(68%)、AK358689(68%)、AK363516(68%)和AK360261(69%)。其中前3个序列的同源性明显高于其他5个基因,且E-value值为0.0,本研究将这3个大麦序列分别暂命名为HvSERK1(AK372118)、HvSERK2(AK252995)和HvSERK3 (AK374641)。

图1 8个大麦氨基酸序列和5个拟南芥SERK蛋白序列的多重比对Fig.1 Multiple alignment of 8 barley amino acid sequences and 5 Arabidopsis SERK protein sequences
上述8个大麦氨基酸序列和5个拟南芥SERK蛋白序列(AtSERK1—5)的多重比对,结果显示(图1):Leucine zipper结构域中,拟南芥AtSERK1—3和大麦HvSERK1—3都具有完整的“Leu-X6-Leu-X6-Leu-X6-Leu”共识序列,而拟南芥AtSERK4、AtSERK5和其他5个大麦序列只具有“Leu-X6-Leu-X6-Leu/Leu-X6-Leu”的不完整结构。除了5个大麦序列的LRR5结构域变化较大,所有参与比对的SERK序列LRR1—LRR5结构域都具有很高的一致性。SPP结构域是SERK蛋白区别于其他LRR-RLKⅡ成员的特征性结构域[18],大麦HvSERK1—3和拟南芥AtSERK1—3都具有此结构域,而拟南芥AtSERK4、AtSERK5和其他5个大麦序列缺失,且5个大麦序列与其他所有SERK基因的同源性最低。
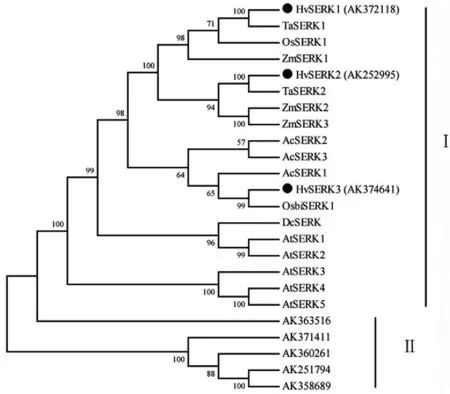
图2 大麦及其他禾本科植物SERK蛋白序列的NJ树Fig.2 NJ phylogenetic tree of SERK protein sequences in barley and other grasses
2.1.1禾本科植物SERK家族基因的进化树
为进一步确认大麦SERK基因家族成员和物种进化,该试验对常见禾本科植物水稻、小麦、玉米以及菠萝中的已知SERK蛋白[19]进行了多序列比对和系统进化树分析。
图2显示,整个进化树由2个进化分支构成:3个大麦SERK序列HvSERK1 (AK372118)、HvSERK2(AK252995)和HvSERK3(AK374641)与其他禾本科植物的SERK蛋白序列同位于1个分支,即分支Ⅰ。其中,禾本科植物的SERK又可分为3组:大麦HvSERK1与小麦TaSERK1、水稻OsSERK1、玉米ZmSERK1聚为一类,HvSERK1与TaSERK1同源性最高;大麦HvSERK2与小麦TaSERK2以及玉米ZmSERK2、ZmSERK3聚为一类,HvSERK2与TaSERK2同源性最高;大麦HvSERK3与水稻OsbiSERK1以及菠萝AcSERK1、AcSERK2和AcSERK3聚为一类,HvSERK3与OsbiSERK1同源性最高。分支Ⅱ中包含了其他的5个大麦序列(AK358689、AK363516、AK371411、AK360261和AK251794),与禾本科植物的SERK序列位于不同分支,遗传距离较远,可能属于其他类型的RLK基因。
2.1.2序列同源性分析
根据系统进化树结果,对分支Ⅰ中所有禾本科植物和大麦中的3个SERK蛋白序列进行了同源性比对(表2),结果表明:大麦HvSERK1蛋白与TaSERK1、OsSERK1和TaSERK2具有较高同源性,分别为98%、94%和92%;大麦HvSERK2蛋白与TaSERK2、OsSERK1和TaSERK1具有较高同源性,分别为99%、91%和91%;大麦HvSERK3蛋白与OsbiSERK1、AcSERK1具有较高同源性,分别为93%、91%。3条大麦SERK蛋白之间的同源性在87%—91%。

表2 大麦与其他禾本科植物SERK蛋白同源性比较Table 2 Homology comparison of SERK proteins in barley and other grasses
2.2大麦SERK基因克隆
通过PCR扩增后经测序验证得到了3条与预期片断大小(2 077 bp、1 929 bp和1 896 bp)一致的目的条带。使用Vector NTI Advance11软件包中的AlignX软件和NCBI中的BLAST程序对测序结果与已有参考序列的ORF区域及其翻译的氨基酸序列进行比对发现,与相应的已有参考序列(Accession:AK374641)相比,大麦HvSERK3基因的测序序列存在3个碱基位点(160、941和1113)的突变和3组密码子(TGC→CGC、TAC→TTC、CTT→CTC)的改变,其中密码子TGC→CGC和TAC→TTC分别导致了编码氨基酸的突变:半胱氨酸(C)→精氨酸(R),酪氨酸(Y)→苯丙氨酸(F),属于非同义突变(图3)。其他2个克隆序列与参考序列都是完全一致,未发现任何核苷酸和氨基酸的突变。

图3 大麦HvSERK3基因ORF区核苷酸及氨基酸序列Fig.3 Nucleotide sequence and amino acid sequence in ORF region of barley HvSERK3 gene
2.3大麦SERK蛋白序列理化参数的检索和分析
对大麦及模式植物拟南芥SERK蛋白序列的理化参数进行分析比较,结果显示(表3),大麦和拟南芥SERK基因的蛋白参数基本一致。理论等电点(pI)值均为5.5左右,所带氨基酸残基比例都是酸性大于碱性,表明SERK蛋白呈弱酸性。大麦和拟南芥SERK蛋白所含主要氨基酸中,Leu的含量始终最高(13.7%),亮氨酸(Leu)、甘氨酸(Gly)、缬氨酸(Val)和丝氨酸(Ser)等其次。不同的是,大麦和拟南芥SERK蛋白均为不稳定蛋白,除了大麦HvSERK1的不稳定系数小于40,为稳定蛋白。总平均疏水性均为负值,说明大麦和拟南芥SERK蛋白具有很强亲水性,均属于亲水性蛋白。

表3 SERK蛋白序列的理化参数分析Table 3 Analysis of SERK protein sequences’physicochemical parameters
2.4大麦SERK蛋白亲疏水性的预测和分析
蛋白质折叠过程中会形成疏水内核和亲水表面,其折叠情况和相应的亲疏水性序列谱(Profile)可以由ProtScale程序绘制完成[20]。大麦SERK蛋白亲疏水性预测结果显示(图4),分值(Score)越低,表明此区域亲水性越强;反之,疏水性则越强。HvSERK1蛋白在第255位氨基酸处具有最高分2.126,第399位氨基酸处具有最低分-2.089;HvSERK2在第23位具有的最高分2.142,第400位具有最低分-2.321;HvSERK3在第245位具有最高分值2.089,第393位具有最低分值-2.021;3条序列的亲水区域都要大于疏水区域,均为亲水性蛋白。对拟南芥SERK蛋白序列进行分析,结果与大麦SERK蛋白一致。结合上述ProtPram预测的结果可知,大麦SERK蛋白是亲水性蛋白,表现为亲水性。

图4 大麦SERK蛋白亲疏水性的预测Fig.4 Prediction of barley SERK proteins’hydrophobicity and hydrophilicity
2.5大麦SERK蛋白导肽的预测和分析
利用工具“TargetP 1.1 Server”在线预测大麦和拟南芥SERK蛋白序列,将置信区间设为0.95。结果显示(表4),HvSERK1、HvSERK2和HvSERK3三条大麦SERK蛋白的导肽预测可靠性分别为IV级、IV级、V级,HvSERK1可能具有分泌途径信号肽,且在蛋白序列第30位存在一个导肽分裂位点,无法确定HvSERK2、HvSERK3是否具有导肽,也未发现其导肽分裂位点。AtSERK1、AtSERK2、AtSERK3、AtSERK4 4条拟南芥SERK蛋白的导肽预测可靠性均为Ⅰ级,说明可靠性很大,很可能都具有分泌途径信号肽,且分别在蛋白序列第23位、第29位、第25位、第33位存在一个导肽分裂位点;AtSERK5蛋白的导肽预测可靠性为Ⅲ级,也可能具有分泌途径信号肽,且导肽分裂位点位于第24位氨基酸。

表4 SERK蛋白的导肽预测Table 4 Prediction of SERK proteins’leader peptide
2.6大麦SERK蛋白信号肽的预测和分析
信号肽(Signal peptide)是蛋白质肽链N端包含16—30个氨基酸的一段多肽,可以引导蛋白质跨膜转移到特定亚细胞结构或分泌到胞外[20]。为了验证和进一步确认上述导肽预测结果,利用SignalP 4.1 Server工具在线预测和分析大麦SERK蛋白信号肽(图5)。基于Y-max和S-mean值判定,3条大麦SERK蛋白N末端均存在信号肽:HvSERK1和HvSERK2剪切位点在第30位点和第31位点间,成熟蛋白启动(包括)的位点是31,信号肽区为前30个氨基酸;HvSERK3剪切位点在第22位点和第23位点间,成熟蛋白启动(包括)的位置是23,信号肽区为前22个氨基酸。对拟南芥SERK蛋白序列预测,也获得到相似结果:AtSERK1信号肽区为前27个氨基酸;AtSERK2信号肽区为前28个氨基酸;AtSERK3信号肽区为前26个氨基酸;AtSERK4信号肽区为前30个氨基酸;AtSERK5信号肽区为前25个氨基酸。基于S-mean和D值,进一步说明大麦SERK蛋白是分泌蛋白,可能被跨膜分泌到特定亚细胞结构或细胞外起作用。

图5 大麦SERK蛋白的信号肽预测分析Fig.5 Prediction and analysis of barley SERK proteins’signal peptide
2.7大麦SERK蛋白的跨膜区预测和分析
跨膜蛋白通过与胞外信号分子结合,从而转导胞外信号[20]。利用TMHMM服务器在线预测和分析大麦SERK蛋白的跨膜区(图6),结果表明:对于HvSERK1蛋白,跨膜螺旋(TMhelix)区预测位置位于243—265处的氨基酸残基处,构成了一个跨膜区;对于HvSERK2蛋白,跨膜螺旋(TMhelix)区预测位置位于10—32和244—266处的氨基酸残基处,构成了2个跨膜区;对于HvSERK3蛋白,跨膜螺旋(TMhelix)区预测位置位于237—259处的氨基酸残基处,构成了一个跨膜区。对拟南芥SERK蛋白的跨膜区进行分析,结果与大麦相似:除AtSERK1蛋白具有2个跨膜螺旋区外,AtSERK2、AtSERK3、AtSERK4和AtSERK5蛋白均具有一个跨膜螺旋区。因此可确定大麦SERK蛋白是一个跨膜蛋白,具有转导细胞信号的功能。

图6 大麦SERK蛋白的跨膜区预测Fig.6 Prediction of barley SERK proteins’membrane-spanning region
2.8大麦SERK蛋白二级结构的预测和分析
蛋白质二级结构(Secondary structure)组件包括α-螺旋(α-helix)、β-折叠(β-sheet)、无规则卷曲(coil)及模体(motif)等[20]。利用PredictProtein预测大麦SERK蛋白二级结构(表5),结果显示:环状结构(loop)是大麦SERK蛋白二级结构的主要组件;所含氨基酸残基中,暴露氨基酸和包埋氨基酸等此外全部的氨基酸的比例基本一致。对拟南芥SERK蛋白进行同样预测,结果与大麦SERK蛋白一致,即蛋白二级结构中都是富含环状结构(loop)。
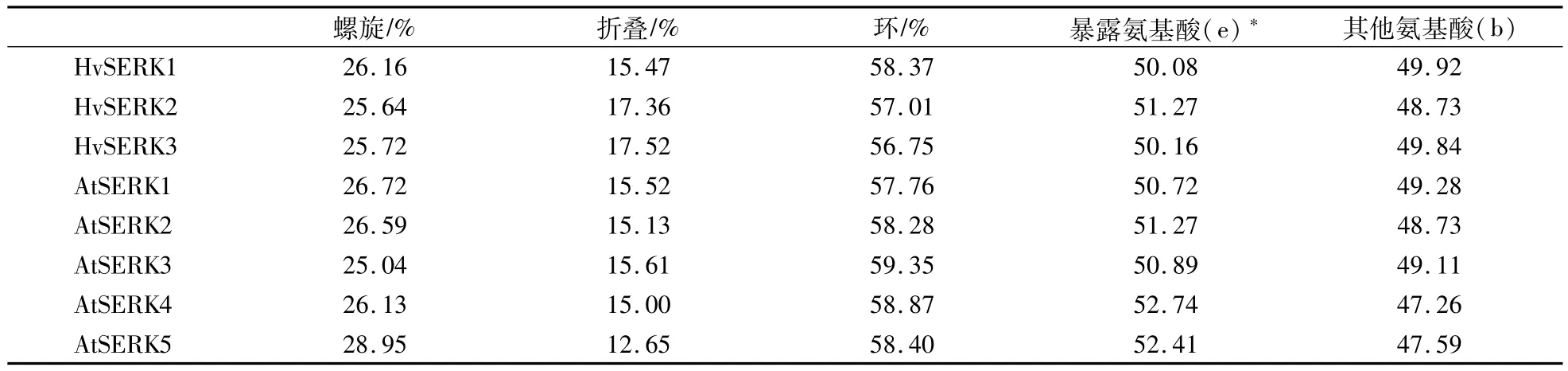
表5 SERK蛋白的二级结构和溶剂可及性预测Table 5 Prediction of SERK proteins’secondary structure and solvent accessibility
2.9大麦SERK蛋白三级结构的预测和分析
2个或多个结构域组合并折叠成的三维结构,即蛋白质三级结构(Tertiary structure)[20]。利用Phyre2线串法预测蛋白质折叠,运算模式设为“Normal”,结果如图7所示。

图7 大麦SERK蛋白三维结构预测Fig.7 Prediction of barley SERK proteins’three-dimensional structure
利用基于PROCHECK程序的在线蛋白质结构检验工具“PDBsum Generate”,从多个角度对预测蛋白模型的健康度进行评估,得到Ramachandran图(图8)和G-Factors值。大麦HvSERK1、HvSERK2和HvSERK3蛋白氨基酸残基落在最佳区(红色)的比例为85.0%、86.8%和87.2%,小于90%,说明3条大麦SERK蛋白的空间构象需要进一步优化,并没有落在最有利区;但是最佳区和次允许区(棕色)的总比例均大于90%,说明3条大麦SERK蛋白具有相对合理的空间结构;大麦HvSERK2和HvSERK3不允许区(浅色)的比例分别为0.9%和0.4%(低于1%),大麦HvSERK1不允许区比例为1.3%(高于1%),说明HvSERK2和HvSERK3的蛋白结构模型稳定性较好,HvSERK1稳定性较差。3条大麦SERK蛋白的GFactors值分别为0.05、0.06和0.04,大于-0.5,说明它们的空间结构属于正常范围内,是常见结构。

图8 大麦SERK蛋白Ramachandran图Fig.8 Ramachandran map of barley SERK proteins
3 结论与讨论
该研究以自主育成的啤酒大麦品种‘花30’为材料,通过同源克隆方法获得了大麦SERK家族3个基因的cDNA全长序列,其中2个基因的cDNA全长序列与国际大麦测序联盟(IBSC)公布的序列完全一致,而另一个基因HvSERK3与相应已有参考序列(Accession:AK374641)对比,有3个核苷酸的非同义突变以及编码氨基酸的变化。推测该序列变化可能是与克隆所用大麦材料不同有关,是否有功能上的差别还有待于深入研究。
SERKs具有多重作用,且家族成员间同时存在功能冗余和分化的现象。如,在模式植物拟南芥中,At-SERK1和AtSERK2参与植物花粉发育过程[2-3];AtSERK1、AtSERK3/BAK1和AtSERK4/BKK1参与植物BR信号通路[4,8];AtSERK3/BAK1和AtSERK4/BKK1参与植物先天免疫反应[5,7];AtSERK3/BAK1和AtSERK4/BKK1参与调节细胞死亡[4,6]。常见禾本科植物水稻的SERK基因家族中,只有OsSERK2基因参与水稻对白叶枯病[11]和稻瘟病[9-10]的免疫反应。本研究对大麦SERK家族基因的核苷酸及氨基酸序列的理化特征、结构特点和功能等预测和分析,其中可以发现大麦中的3个SERK基因在一些理化特征上仍然存在差别:如HvSERK1为稳定蛋白,而HvSERK2和HvSERK3为不稳定蛋白;HvSERK1和HvSERK3具有一个跨膜区域,HvSERK2具有2个跨膜区域等。大麦中这3个SERK基因在功能上,特别是对外界环境中生物胁迫和非生物胁迫响应是否存在差异,还有待于在本研究基础上展开深入研究。
参 考 文 献
[1]石雅丽,张锐,林芹,等.植物体细胞胚胎发生受体类蛋白激酶的生物学功能[J].遗传,2012,34(5):551-559.
[2]ALBRECHT C,RUSSINOVA E,HECHT V,et al.The Arabidopsis thaliana SOMATIC EMBRYOGENESIS RECEPTOR-LIKE KINASES1 and 2 control male sporogenesis[J].Plant Cell,2005,17:3337-3349.
[3]COLCOMBET J,BOISSON-DERNIER A,ROS-PALAU R,et al.Arabidopsis SOMATIC EMBRYOGENESIS RECEPTOR KINASES1 and 2 are essential for tapetum development and microspore maturation[J].Plant Cell,2005,17:3350-3361.
[4]HE K,GOU X P,YUAN T,et al.BAK1 and BKK1 regulate brassinosteroid-dependent growth and brassinosteroid-independent cell-death pathways[J].Curr.Biol.,2007,17:1109-1115.
[5]HEESE A,HANN D R,GIMENEZ-IBANEZ S,et al.The receptor-like kinase SERK3/BAK1 is a central regulator of innate immunity in plants [J].Proc.Natl.Acad.Sci.USA,2007,104:12217-12222.
[6]KEMMERLING B,SCHWEDT A,RODRIGUEZ P,et al.The BRI1-associated kinase1,BAK1,has a brassinolide-independent role in plant celldeath control[J].Curr.Biol.,2007,17:1116-1122.
[7]ROUX M,SCHWESSINGER B,ALBRECHT C,et al.The Arabidopsis leucine-rich repeat receptor-like kinases BAK1/SERK3 and BKK1/SERK4 are required for innate immunity to hemibiotrophic and biotrophic pathogens[J].Plant Cell,2011,23:2440-2455.
[8]GOU X P,YIN H J,HE K,et al.Genetic evidence for an indispensable role of somatic embryogenesis receptor kinases in brassinosteroid signaling[J].PLoS Genet.,2012,8(1):71-76.
[9]HU H,XIONG L,YANG Y.Rice SERK1 gene positively regulates somatic embryogenesis of cultured cell and host defense response against fungal infection[J].Planta,2005,222:107-117.
[10]PARK H S,RYU H Y,KIM B H,et al.A subset of OsSERK genes,including OsBAK1,affects normal growth and leaf development of rice[J].Mol.Cell,2011,32:561-569.
[11]CHEN X W,ZUO S M,SCHWESSINGER B,et al.An XA21-associated kinase(OsSERK2)regulates immunity mediated by the XA21 and XA3 immune receptors[J].Mol.Plant,2014,7:874-892.
[12]SINGLA B,KHURANA J P,KHURANA P.Characterization of three somatic embryogenesis receptor kinase genes from wheat(Triticum aestivum)[J].Plant Cell Rep.,2008,27:833-843.
[13]BAUDINO S,HANSEN S,BRETTSCHNEIDER R,et al.Molecular characterization of two novel maize LRR receptor-like kinases,which belong to the SERK gene family[J].Planta,2001,213:1-10.
[14]MAYER K F X,ROBBIE W,BROWN J W S,et al.A physical,genetic and functional sequence assembly of the barley genome[J].Nature,2012,491(7426):711-716.
[15]GAO R H,DUAN K,GUO G M,et al.Comparative Transcriptional Profiling of Two Contrasting Barley Genotypes under Salinity Stress during the Seedling Stage[J].International Journal of Genomics,2013,139:822-835.
[16]王文良.大麦BAK1同源基因的克隆及遗传转化[D].泰安:山东农业大学,2014.
[17]李卿,邸鹏,陆文铨,等.丹参柯巴基焦磷酸合酶的生物信息学分析[J].中草药,2015,46(6):887-894.
[18]SCHMIDT E D L,GUZZO F,TOONEN M A J,et al.A leucine-rich repeat containing receptor-like kinase marks somatic plant cells competent to form embryos[J].Development,1997,124:2049-2062.
[19]AAN DEN TOORN M,ALBRECHT C,DE VRIES S.On the origin of SERKs:Bioinformatics analysis of the somatic embryogenesis receptor kinases[J].Mol.Plant,2015,8:762-782.
[20]薛庆中.DNA和蛋白质序列数据分析工具[M].3版.北京:科学出版社,2012.
(责任编辑:程智强)
Cloning and bioinformatics analysis of SERK gene family in barley(Hordeum vulgare L.)
YANG Sha-sha1,2,3,LI Ying-bo2,TAN He-xin3,GUO Gui-mei2,HE Ting2,CHEN Zhi-wei2,LIU Cheng-hong2*,HUANG Jian-hua2*
(1College of Fisheries and Life Science,Shanghai Ocean University,Shanghai 201306,China;2Shanghai Key Laboratory of Agricultural Genetics and Breeding/Biotech Research Institute,Shanghai Academy of Agricultural Sciences,Shanghai 201106,China;3School of Pharmacy,Second Military Medical University,Shanghai 200433,China)
Abstract:By annotation and cloning of barley SERK gene family,three barley sequences(HvSERK1,HvSERK2 and HvSERK3)were obtained,and the alignment with the corresponding reference sequence(Accession:AK374641)showed that the HvSERK3 gene’s amino acid sequence somewhat varied.The bioinformatics analysis indicated that the 3 barley SERK genes have the open reading frame(ORF)of about 1 870 bp,the amino acid residues of some 620 aa and the molecular weight of approximately 69 kD,and their theoretic isoelectric point is averagely 5.5,showing that the SERK are acidic proteins;The SERK are high in hydrophilicity and have signal peptide at N-terminus and transmembrane helix region,and the predicted secondary structures are mainly “loop”.This research has laid the basis for further functional study of barley SERK gene family.
Key words:Barley(Hordeum vulgare L.);SERK;Annotation;Cloning;Bioinformatics analysis
中图分类号:S512.3
文献标识码:A
文章编号:1000-3924(2016)03-005-09
DOI:10.15955/j.issn1000-3924.2016.03.02
收稿日期:2016-03-28
基金项目:上海市自然科学基金(15ZR1436600);上海市种业发展项目[沪农科种字(2016)第1-1号];大麦青稞产业技术体系(CARS-05)
作者简介:杨沙沙(1990—),女,在读硕士,研究方向:大麦分子育种。E-mail:ayxyss0915@163.com
*通信作者:黄剑华,E-mail:sw1@saas.sh.cn;刘成洪,E-mail:liuchenghong@saas.sh.cn
