拟南芥IQM5.2的克隆、表达及其生物信息学分析
2016-06-23弓路平萧文慧周玉萍黄小玲田长恩
弓路平 萧文慧 周玉萍 黄小玲 田长恩
(广州大学生命科学学院 植物抗逆基因功能研究广州市重点实验室,广州 510006)
研究报告
拟南芥IQM5.2的克隆、表达及其生物信息学分析
弓路平 萧文慧 周玉萍 黄小玲 田长恩
(广州大学生命科学学院 植物抗逆基因功能研究广州市重点实验室,广州 510006)
为了探究拟南芥IQM5基因是否存在不同的剪切方式及其器官特异性表达,利用RT-PCR技术和T-A克隆发现了IQM5基因的一种新的剪切方式,即IQM5.2。利用生物信息学方法、半定量和定量RT-PCR分析方法分别对新的CDS序列所编码蛋白的性质以及IQM5.1和IQM5.2在不同器官中的比例进行了分析。结果表明,IQM5.2编码1个由300个氨基酸组成的多肽,其N端与豌豆重金属诱导蛋白6A(HMIP6A)具有较高同源性,含有1个IQ基序,属于钙不依赖性钙调素结合蛋白;在幼苗、莲座叶、花序叶、茎和花蕾中都存在IQM5.1和IQM5.2的转录本,且二者的比例在不同的材料中不尽相同。因此,IQM5基因存在不同的剪切方式和器官特异性表达。
IQM5.2;选择性剪切;钙调素结合蛋白;基因表达;拟南芥
钙调素(calmodulin,CaM)作为一类细胞内Ca2+的受体,绝大多数无酶活性,也无转录因子活性,更不是结构蛋白,只有通过其下游的钙调素结合蛋白(calmodulin-binding protein,CaMBP)来调节细胞的生理功能[1-3]。因此,研究CaBMP对揭示CaM的功能至关重要,将有助于解析CaM信号通路的特异性、复杂性和多样性。CaM与CaMBP的结合可分为Ca2+依赖和不依赖两种类型。其中,Ca2+不依赖型的CaMBP含有IQ基序,完整IQ基序的氨基酸序列为IQXXXRGXXXR或[I/L/V]QXXXRXXXX[R/K][4,5],但是,极少数含IQ基序的蛋白质与CaM的结合也依赖Ca2+[6,7]。在植物中已经发现了5个含IQ基序的蛋白质家族[8]:myosin家 族[9-11]、CAMTA家 族[12,13]、CNGC家族[14,15]、IQD家族[6,7,16]和IQM家族[17-19],分别由17个、6个、20个、33个和6个成员组成。在拟南芥中,IQM家族共有6个成员,均只含有1个能与钙调素结合的IQ基序[20]。IQM1(IQ motif containing 1)是IQM家族的1个成员,由At4g33050编码,IQM1在体内外均能与CaM结合,并通过蛋白截短实验证明IQ基序是其钙调素结合所必需的结构域[17];本研究组将克隆得到的全长cDNA进行生物信息学分析发现,IQM2也是1个钙调素结合蛋白[21];IQM3的启动子中存在多种光、非生物胁迫和植物激素反应的顺式作用元件,IQM3基因与种子萌发及幼苗子叶膨大有密切关系[22]。IQM5是该家族的第5个成员,由At5g57010编码,其功能尚待研究。检索生物学文献和数据库发现IQM5基因存在一种剪切方式,即IQM5.1。已知,IQM1存在4种不同的剪切方式(见www.arabidopsis.org)。鉴于此,本研究对不同生长发育时期的RNA样本进行RT-PCR扩增和测序,以探究IQM5是否也存在不同的剪切方式,并对因不同的剪切方式所产生的转录本进行定量RT-PCR分析,旨为进一步解析该基因的功能奠定必要的基础。
1 材料与方法
1.1 材料
植物材料:拟南芥(Arabidopsis thaliana)生态型为Columbia(Col);大肠杆菌菌株为E. coli DH5α;T-A克隆载体为pMD19-T Simple vector,购自TaKaRa公司;总RNA提取试剂盒(RNAiso Plus)、PrimeScriptTMRT reagent Kit with gDNA Eraser(Perfect Real Time)、SYBR®PrimeSc riptTMRT-PCR kit(Perfect Real Time)、琼脂糖凝胶回收试剂盒、DNA片段回收试剂盒、DNA Ligation Kit和PCR相关的试剂及Taq酶均为宝生物工程(大连)有限公司产品;氨苄青霉素钠、硫酸卡那霉素、蛋白胨、酵母抽提物、蔗糖、琼脂粉、各种氨基酸、NaCl等试剂均为生工生物工程上海股份有限公司产品。
1.2 方法
1.2.1 总RNA的提取 采用 Trizol 法,从7d幼苗、成熟植株不同器官中提取总 RNA。
1.2.2 不同转录本的克隆 以7 d幼苗和成熟莲座叶的总RNA为模板,以5'-atggctttatcttttggttactta-3'和5'-ttagtgccttgggcttggaagacc-3'为引物进行RT-PCR扩增,得到IQM5CDS。RT-PCR参数为:50℃反转录30 min,94℃变性2 min;94℃变性30 s,64℃退火30 s,72℃延伸2 min,共 33个循环;72℃延伸5 min。反应结束后电泳,并用凝胶成像系统(AlphamagerTM)观察、拍照,切取目的片段所在的凝胶,用胶回收试剂盒回收扩增产品,与pMD19-T载体连接,转化大肠杆菌,筛选出阳性菌落,进行菌落PCR,选择阳性菌落摇菌,菌液外送测序,并用生物信息学的方法分析测序结果。
1.2.3 基因表达分析
1.2.3.1 cDNA的获得 参考PrimeScriptTMRT reagent Kit(Perfect Real Time)的操作手册进行。先用其中的gDNA Eraser除去RNA中的基因组DNA后,再进行反转录获得cDNA,最后进行PCR 检测,以确认 DNA是否除尽。
1.2.3.2 半定量RT-PCR 以1.2.3.1所获得的cDNA为模板,进行PCR扩增。所用引物为IQM5.1-F1:5'-AAGTTTACCAGCACCGAC-3';IQM5.1-R1:5'-GCAGCAAGATTTCTACGC-3';IQM5.2-F2:5'-CGTATGTACACATGGTCTTC-3',IQM5.2-R2:5'-ACACTTCGTAAGCTTGTCTC-3';Actin2-F:5'-TGACTACGAGCAGGAGATGGAA-3',Actin2-R:5'-CAAACGAGGGCTGGAACAA-3'。扩增程序为:94℃变性2 min;94℃变性30 s,62℃(Actin2)或51.5(IQM5.1IQM5.2)退火30 s,72℃延伸10 s,共28个循环;72℃延伸5 min。反应结束后,用浓度为2%的琼脂糖凝胶进行电泳,用凝胶成像系统观察并拍照。
1.2.3.3 实时定量RT-PCR 用SYBR®PrimeSc riptTMRT-PCR kit(Perfect Real Time),相关耗材为Applied Biosystems(ABI7000)公司产品。所用基因专一引物同半定量RT-PCR,模板(1.2.3.1所获cDNA)浓度为0.5 ng·L-1,每个样品设3次重复。选用表达相对稳定的Actin2作为内参基因,以IQM5.2的相对表达量作为参照因子(即1倍)。采用△△Ct法进行基因表达的相对定量分析,即利用 2-△△Ct公式计算IQM5.1基因与IQM5.2基因的相对比例(样品的△Ct=IQM5.1基因/IQM5.2基因的Ct -Actin2Ct,△△Ct=IQM5.2基因△Ct - IQM5.1基因的△Ct)。
2 结果
2.1 IQM5.2的发现
以7 d幼苗总RNA为模板进行RT-PCR,将扩增产物克隆进测序载体,转化大肠杆菌,随机选送5个菌落直接PCR呈阳性的菌落外送测序。测序结果显示,全部为IQM5.1。而在以成熟莲座叶总RNA为模板进行的RT-PCR所获得 9个序列中,3个与IQM5.1完全匹配,另外6个序列都存在1个长75 bp的与IQM5.1不匹配的片段。进一步分析发现,这个75 bp的片段与IQM5.1的基因组序列的第6个内含子(1 345-1 419 bp)完全匹配(图1)。说明IQM5基因至少存在2种不同的剪切方式,其转录本分别为IQM5.1和IQM5.2。

图1 所得75 bp片段与IQM5基因的第6个内含子的比对
如上所述,在成熟植株的莲座叶中发现了一个新的IQM5基因的mRNA——IQM5.2。经过生物信息学分析得知,IQM5.2源于IQM5.1的第6个内含子未被剪切。该内含子两端的碱基为“GT-AG”(图1),属于第二类“GT-AG”内含子。据此,IQM5基因由IQM5.1的8个外显子拼接为IQM5.2的7个外显子(图2)。
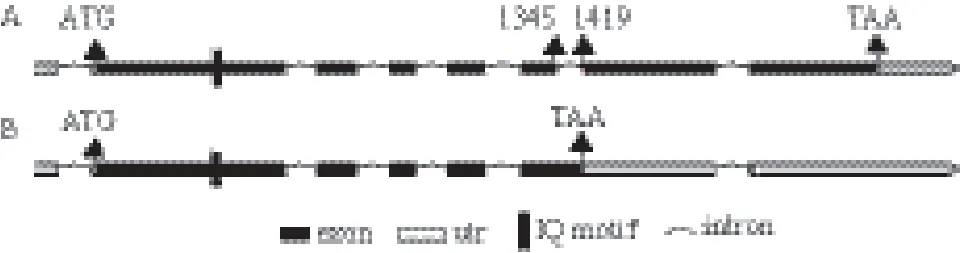
图2 IQM5.1(A)和IQM5.2(B)的剪切方式示意图
2.2 IQM5.2的cDNA及其编码蛋白
IQM5.1基因的cDNA全长1 783 bp,编码1个由495个氨基酸残基组成的多肽链(NP_200511.1)。IQM5.2 的cDNA全长1 858 bp,编码1个由300个氨基酸残基组成的多肽链(KT851542)(图3)。这是由于在多出的75 bp的第70-72位核苷酸残基为终止密码“TAA”(图1-图3)所致。
2.3 IQM5.1的基本理化性质
用ExPASy的ProtParam软件预测,IQM5.1的分子式为C2474H3925N723O736S18,相对分子质量约为56 kD,酸性氨基酸(Asp + Glu)残基58个,碱性氨基酸(Arg + Lys)残基77个,等电点(pI)为9.44,半衰期为30 h,不稳定参数(the instability index,II)是41.89,属于不稳定蛋白。该蛋白亲水性平均数(grand average of hydropathicity,GRAVY)为-0.676,预测为亲水蛋白。
2.4 IQM5.2的理化性质
用ExPASy的ProtParam软件预测,其分子式为C1528H2427N443O444S9,相对分子质量约为34 kD,酸性氨基酸(Asp + Glu)残基37个,碱性氨基酸(Arg + Lys)残基51个,等电点(pI)为9.50,半衰期为30 h,不稳定参数是46.11,属于不稳定蛋白。该蛋白亲水性平均数为-0.688,预测为亲水蛋白。
2.5 功能预测
经序列比对发现,IQM5.1的 N端与豌豆重金属诱导蛋白6A(HMIP6A)[23]、C端与栝楼天花粉素(trichosanthin)的N端[24]具有较高的同源性,第130-152位氨基酸残基为IQ基序(图4)。IQM5.2与豌豆重金属诱导蛋白6A(HMIP6A)[23]具有较高同源性,第130-152位氨基酸残基为IQ基序(图5)。HMIP6 A是在用汞、镉和铝处理的豌豆根中被发现的,具有一个含IQ基序的重复序列[23],而IQM5.1和IQM5.2均具有其中一段。提示,IQM5.2可能具有与HMIP6A相似的特性。天花粉素是从葫芦科植物栝楼(Trichosanthes kirilowii)新鲜块根中提取的植物蛋白质,属于I型核糖体失活蛋白[24]。与IQM5.1相比,IQM5.2缺少了天花粉素同源区段。

图3 IQM5.2的cDNA及其编码蛋白

图4 IQM5.1与豌豆重金属诱导蛋白6A(HMIP 6A)及栝楼天花粉蛋白(Trichosanthin)的序列比对

图5 IQM5.2与豌豆重金属诱导蛋白6A(HMIP 6A)的序列比对
2.6 AtIQM5.1和AtIQM5.2的表达分析
为了进一步分析IQM5.1和IQM5.2在不同器官中的表达比例,选取了7d幼苗、成熟植物的莲座叶、花序叶、茎和花蕾等5种不同材料进行半定量和定量RT-PCR分析。定量RT-PCR分析结果显示:若以同一材料中的IQM5.1的表达量作为1个单位,除花蕾中AtIQM5.2比IQM5.1的相对表达量低外,其他4种材料中IQM5.2比IQM5.1的表达量高(图6-B)。半定量RT-PCR分析结果(图6-A)印证了定量RT-PCR分析的结果。
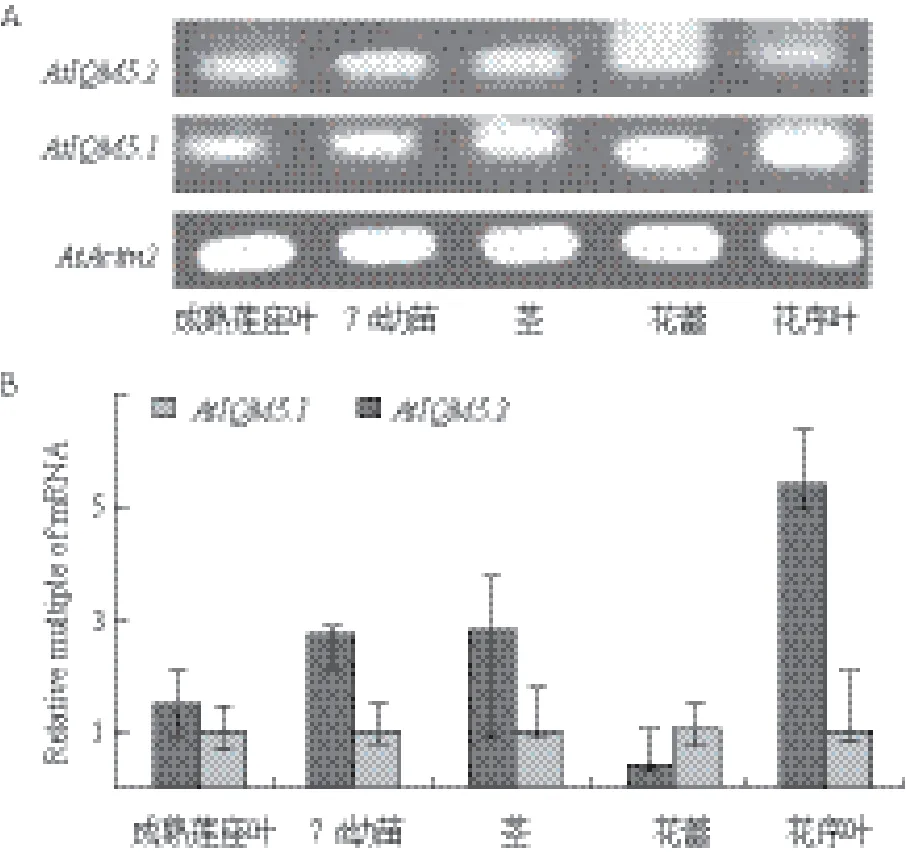
图6 AtIQM5.1和AtIQM5.2转录本的半定量(A)和定量(B)RT-PCR分析
3 讨论
本研究在生态型为Columbia(Col)的拟南芥中发现了一种IQM5基因的新的剪切方式,由IQM5.1的第6个内含子未被剪切所致,所产生的mRNA被命名为IQM5.2。与IQM5.1蛋白相比,IQM5.2蛋白缺少了天花粉素同源区段,但IQM5.2蛋白仍然保留了与HMIP6A同源的含IQ基序的区段,可能具有与HMIP6A相似但不同于IQM5.1的功能。本研究组多年从事拟南芥IQM家族的研究,利用截短蛋白所进行的实验发现IQ基序是IQM1与CaM结合的位点[17];进一步发现,若将IQ基序中的I突变成S,IQM1同样不能与CaM结合[25];在IQM2的钙调素结合活性的研究中,本研究组得到了相同的结果(待发表)。因此,推断IQM5.2蛋白可以与CaM结合,并且很可能是不依赖Ca2+的CaM结合蛋白,具体特性还有待进一步研究。半定量和定量RT-PCR分析结果显示,5种不同材料中都存在IQM5.1和IQM5.2的转录本,且二者的比例在不同的材料中不尽相同。因此推测,二者的比例可能与发育调节有关,其间的具体关系则有待深入研究。
4 结论
发现AtIQM5基因的一种新的转录本IQM5.2,并在拟南芥生态型为Columbia(Col)的幼苗、莲座叶、花序叶、茎和花蕾中均存在IQM5.1和IQM5.2两种转录本,且二者的比例在不同器官中不尽相同。
[1]McAinsh RM, Pittman JK. Shaping the calcium signature[J]. New Phytol, 2009, 181(2):275-294.
[2]Sanders D, Pelloux J, Brownlee C, et al. Calcium at the crossroads of signaling[J]. Plant Cell, 2002, 14(Suppl):S401-S417.
[3]O’Day DH. CaMBOT:profiling and characterizing calmodulinbinding protein[J]. Cell Signal, 2003, 15:347-354.
[4]Rhoads AR, Freiedberg F. Sequence motif and calmodulin recognition[J]. FASEB J, 1997, 11:331-340.
[5]Jurado LA, Chockalingam PS, Jarrett HW. Apocalmodulin[J]. Physiol Rev, 1999, 79(3):661-682.
[6]韦慧彦, 郭振清, 崔素娟. 钙不依赖性钙调素结合蛋白的研究进展[J]. 生物化学与生物物理进展, 2007, 34(2):124-131.
[7]Abel S, Savchenko T, Levy M. Genome-wide comparative analysis of the IQD gene families in Arabbidopsis thaliana and Oryza sativa[J]. BMC Evol Biol, 2005, 5:72-89.
[8]田长恩, 周玉萍. 植物具IQ基序的钙调素结合蛋白的研究进展[J]. 植物学报, 2013, 48(4):447-480.
[9]Reddy AS, Day IS. Analysis of the myosins encoded in the recently completed Arabidopsis thaliana genome sequence[J]. Genome Biol, 2001, 2(7):RESEARCH0024.
[10]Holweg C, Nick P. Arabidopsis myosin XI mutant is defective inorganelle movement and polar auxin transport[J]. Proc Natl Acad Sci USA, 2004, 101:10488-10493.
[11]Ojangu EL, Järve K, Paves H, et al. Arabidopsis thaliana myosin XIK is involved in root hair as well as trichome morphogenesis on stems and leaves[J]. Protoplasma, 2007, 230(3-4):193-202.
[12]Bouché N, Scharlat A, Snedden W, et al. A novel family of calmodulin-binding transcription activators in multicellular organisms[J]. J Biol Chem, 2002, 277(24):21851-21861.
[13]Bouché N, Yellin A, Snedden WA, et al. Plant-specific calmodulinbinding proteins[J]. Annu Rev Plant Biol, 2005, 56:435-466.
[14]Köhler C, Merkle T, Neuhaus G. Characterisation of a novel gene family of putative cyclic nucleotide and calmodulin-regulated ion channels in Arabidopsis thaliana[J]. Plant J, 1999, 18(1):97-104.
[15]Talke IN, Blaudez D, Maathuis FJ, et al. CNGCs:prime targets of plant cyclic nucleotide signalling?[J]. Trends Plant Sci, 2003, 8(6):286-293.
[16]Levy M, Wang Q, Kaspi R, et al. Arabidopsis IQD1, a novel calmodulin-binding nuclear protein, stimulates glucosinolate accumulation and plant defense[J]. Plant J, 2005, 1:79-96.
[17]Zhou YP, Duan J, Yamamoto KT, et al. AtIQM1, a novel calmodulin-binding protein, is involved in stomatal movement in Arabidopsis[J]. Plant Mol Biol, 2012, 79(4-5):333-346.
[18]Zhou YP, Fujibe T, Wang XL, et al. Initial characterization of Arabidopsis T-DNA insertion mutants of the iqm1 gene that encodes an IQ motif-containing protein[J]. Plant Cell Physiol, 2007, 48(Suppl):s197.
[19]Zhou YP, Chen YZ, Duan J, et al. Sequence and expression analysis of the Arabidopsis IQM family[J]. Acta Physiol Plant, 2010, 1:191-198.
[20]Bähler M, Rhoads A. Calmodulin signaling via the IQ motif[J]. FEBS Lett, 2002, 513:107-113.
[21]陈羽中, 周玉萍, 叶蕙, 等. 拟南芥IQM2 cDNA 的克隆与生物信息学分析[J]. 武汉植物学研究, 2010, 28(3):353-358.
[22]周玉萍, 赵军, 何滔, 等. 拟南芥IQM3基因的表达分析及其突变体的鉴定[J]. 热带亚热带植物学报, 2009, 17(4):365-370.
[23]Sävenstrand H, Strid A. Six genes strongly regulated by mercury in Pisum sativum roots[J]. Plant Physiol Biochem, 2004, 42:135-142.
[24]Chow TP, Feldman RA, Lovett M, et al. Isolation and DNA sequence of a gene encoding alpha-trichosanthin, a type I ribosomeinactivating protein[J]. J Biol Chem, 1990, 265:8670-8674.
[25]黄章科, 张艺能, 莫忠臻, 等. IQ基序突变对AtIQM1的钙调素结合活性的影响[J]. 生物技术通报, 2014, 12:128-132.
(责任编辑 狄艳红)
Cloning,Expression and Bioinformatics Analysis of IQM5.2 from Arabidopsis
GONG Lu-ping XIAO Wen-hui ZHOU Yu-ping HUANG Xiao-ling TIAN Chang-en
(Guangzhou Key Laboratory for Functional Study on Stress-resistant Genes in Plants,School of Life Sciences,Guangzhou University,Guangzhou 510006)
The Arabidopsis IQM5,the fifth member of IQM family,is encoded by At5g57010. So far,there is only one splicing mode found for the IQM5 gene in the biological literatures and databases,i.e.,At5g57010.1(IQM5.1). In this study,a new splicing mode for IQM5 gene was found by RT-PCR and T-A cloning method,designated as At5g57010.2(IQM5.2). Bioinformatics methods and semiquantitative and quantitative RT-PCR were employed to analyze the properties of proteins encoded by new CDS sequence,and the ratio of IQM5.1 and IQM5.2 in different organs,respectively. The results revealed that IQM5.2 encoded a polypeptide of 300 amino-acid residues,and its N-terminus shared sequence homology with the pea heavy metal-induced protein 6A(HMIP6A). The protein retained an IQ motif and was a calmodulin-binding protein of the calcium independent. There were transcripts of IQM5.1 and IQM5.2 in the seedling,rosette leaf,cauline leaf,stem,and flower bud,moreover,the ratios of them in the different organs varied.
IQM5.2;alternative splicing;calmodulin-binding protein;gene expression;Arabidopsis
10.13560/j.cnki.biotech.bull.1985.2016.05.009
2015-07-22
国家自然科学基金项目(31170204,31470014),羊城学者科研项目(12A001G)
弓路平,女,硕士研究生,研究方向:分子遗传学;E-mail:1537464017@qq.com
田长恩,男,教授,研究方向:分子遗传学;E-mail:changentian@aliyun.com
