2011- 2014年山东省H9亚型禽流感病毒HA基因遗传变异分析
2016-06-22陈瑞爱刘红波梁梅兰张显浩贺东生农业部动物疫病防控生物技术与制品创制重点实验室肇庆大华农生物药品有限公司广东肇庆526238华南农业大学兽医学院广东广州50642华南家禽疫病防控与产品安全协同创新中心广东广州50640
李 琳,陈瑞爱,,刘红波,梁梅兰,张显浩,贺东生(.农业部动物疫病防控生物技术与制品创制重点实验室肇庆大华农生物药品有限公司,广东肇庆526238;2.华南农业大学兽医学院,广东广州50642;3.华南家禽疫病防控与产品安全协同创新中心,广东广州50640)
2011- 2014年山东省H9亚型禽流感病毒HA基因遗传变异分析
李琳1,陈瑞爱1,2,3,刘红波1,梁梅兰1,张显浩1,贺东生2,3
(1.农业部动物疫病防控生物技术与制品创制重点实验室肇庆大华农生物药品有限公司,广东肇庆526238;2.华南农业大学兽医学院,广东广州510642;3.华南家禽疫病防控与产品安全协同创新中心,广东广州510640)
摘要:对2011-2014年山东26个H9亚型AIV毒株的HA基因进行测序分析。结果显示,所有毒株均属于h9.4.2.5亚分支,与疫苗株GD/SS/94、SD/6/96、SH/F/98的核苷酸相似性为88.0%~91.5%。HA裂解位点均只有一个碱性氨基酸(R),符合低致病性AIV的特征。所有毒株在234位发生了谷氨酸Q→亮氨酸L的突变,呈现出典型的人流感病毒受体结合特性。所有毒株在313-315位均新增1个潜在糖基化位点,且部分毒株在145-147位出现新潜在糖基化位点NGT。结果表明,山东省H9亚型AIV基因已发生较大变异,因此需要加强禽类生产贸易的监测和生物安全措施,及时更新疫苗毒株,控制H9亚型AIV的传播。
关键词:H9;AIV;HA;进化分析;变异
禽流感(Avain Influenza,AI)是A型流感病毒引起的一种传染性疾病综合征。根据病毒表面糖蛋白血凝素基因(Haemagglutinin,HA)和神经氨酸酶(Neuraminidase,NA)基因可划分17个HA亚型和9个NA亚型。H9亚型AIV为低致病性AIV的代表亚型,是我国主要的流行毒株之一,其在免疫压力下变异加快,出现了多个基因亚型[1-3]。本研究测序分析了2011-2014年山东省26个毒株的HA基因序列,旨在了解各毒株间遗传变异情况和流行规律,为该亚型AIV预防及疫苗候选株的筛选提供理论依据。
1 材料与方法
1.1病毒26个毒株均从有呼吸道症状和低死亡率的免疫鸡群发病鸡中分离鉴定。
1.2HA基因测序参照TaKaRa公司的RNA提取和RT-PCR一步法试剂盒说明书扩增HA基因。扩增引物(1.7kb):H9F:5′-CAGTCAAAATGGAGACAATATCACT-3′;H9R:5′-ATACAAATGTTGCATCTGCAAG-3′;由上海生工生物工程技术服务有限公司合成。RT-PCR扩增程序:50℃,94℃预变性3 min;94℃45 s,53℃45 s,72℃2 min,30个循环;72℃延伸10 min。PCR产物经纯化克隆后送英潍捷基公司测序。
1.3HA基因序列比对和遗传变异分析采用Lasergene 7.1软件对HA基因序列编辑翻译、核苷酸和氨基酸序列相似性、受体结合位点以及裂解位点等比较分析;将HA肽序列提交Center for Biological Sequence Analysis online server 1.0(http:// www.cbs.dtu. dk/services/NetNGlyc/),对潜在糖基化位点进行预测。利用MEGA4.0软件ClustalW方法Align比对后截齐序列,通过Neighbor-joining运算方法,进行Bootstrap 1000次重复运算,对HA基因绘制进化树。
2 结果
2.1核苷酸和氨基酸相似性分析结果显示,毒株间核苷酸最低相似性为92.6%,而与疫苗株核苷酸相似性为88.0%~91.5%。见表1。
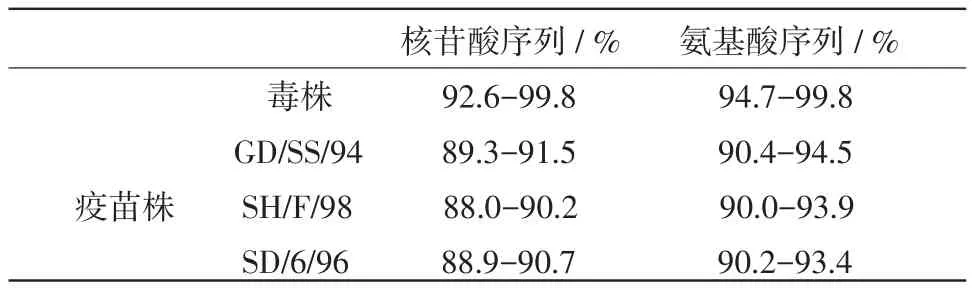
表1 26个毒株及3个疫苗株的HA基因的核苷酸和氨基酸序列相似性比较
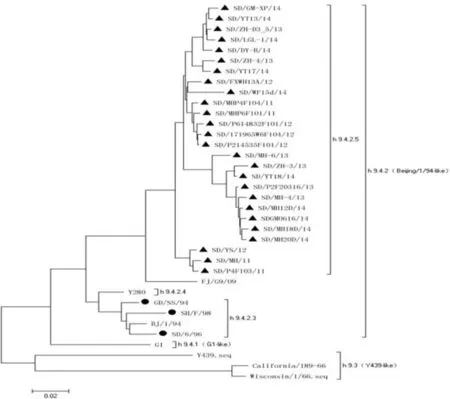
图1 H9亚型AIV毒株HA基因的遗传变异分析▲:毒株序列;●:疫苗株序列
2.2HA基因遗传变异分析见图1,本试验所有毒株均属于h9.4.2.5亚分支,是近年来中国的优势流行亚分支,疫苗株GD/SS/94、SD/6/96、SH/F/98则属于h9.4.2.3亚分支。测序毒株与疫苗株虽分属于不同的基因亚型,但均属于h9.4.2分支。未发现G1-like和Y439-like分支,表明当前的流行毒株具有同一起源[1,3],因此采用疫苗接种预防H9亚型AIV感染是有利的。
2.3HA蛋白关键位点的氨基酸残基分析见表2,所有毒株HA裂解位点的氨基酸均只有一个碱性氨基酸(R),符合低致病性禽流感病毒的特征。多数毒株抗原位点氨基酸出现了明显变异。SD/P614832F101/12出现了T206N(相当于H3亚型的T198N)的氨基酸变异。SD//ZH-3/13等10个毒株(10/26)出现了S145N(相当于H3亚型的S133N)的氨基酸变异,形成了1个潜在糖基化位点NGT。SD//ZH-3/13等8个毒株(8/26)出现了N201D(相当于H3亚型的N193D)的氨基酸变异。所有毒株均在234位(相当于H3亚型的226位)发生了谷氨酸Q→亮氨酸L的突变,呈现出典型的人流感病毒受体结合特性。所有毒株新增313-315(NCS)的潜在糖基化位点,SD//ZH-3/13 等10个毒株(10/26)在145-147(NGT)有一个新的潜在糖基化位点。SD/P614832F101/12则在206-208(NDT)有一个新的潜在糖基化位点。
3 讨论
H9亚型AIV在宿主免疫压力下不断发生基因变异,因此,选择抗原与流行毒株对应的疫苗,才能获得较好的免疫效果。本试验所有毒株均是从免疫鸡群分离得到,再次证实某些H9新毒株可导致免疫鸡感染和排毒[4],现有疫苗难以完全抵御流行毒株的感染。所有毒株均属于h9.4.2.5亚分支,因此,制备新疫苗时候选毒株应来自h9.4.2.5亚分支,并通过动物试验对疫苗的免疫保护效力进行评估。
HA蛋白上第226位等氨基酸是关键的受体结合位点,且糖基化位点在病毒传染性、与受体的结合能力及保护病毒关键蛋白表位免遭免疫攻击方面起到重要作用。本试验所有毒株谷氨酸Q均已经变异为亮氨酸L,说明226位氨基酸Q变异为亮氨酸L愈加明显,呈现出典型的人流感病毒受体结合特性。部分毒株出现了N201D的氨基酸变异,提示对禽类受体的亲和力有下降趋势。所有毒株在313-315位均新增1个潜在糖基化位点,此位点在HA蛋白裂解位点附近,可能影响到HA链的裂解,对病毒的特性产生影响。部分毒株在145-147位出现新潜在糖基化位点NGT,显示随着时间推移NGT糖基化位点愈加明显。据报道,此糖基化位点NGT可导致病毒不与抗H9亚型AIV的HA单抗发生反应[5],同时也可导致H9禽流感变异株对SPF鸡及鸡胚、细胞的致病性增强[6]。病毒HA蛋白上的抗原位点氨基酸变异以及糖基化位点增加是否对病毒致病性和抗原性有影响则需进一步的研究。
本试验结果表明,山东H9亚型AIV基因已发生较大变异,需要加强禽类生产贸易的生物安全措施,并及时更新疫苗毒株,以更好地控制H9亚型禽流感。
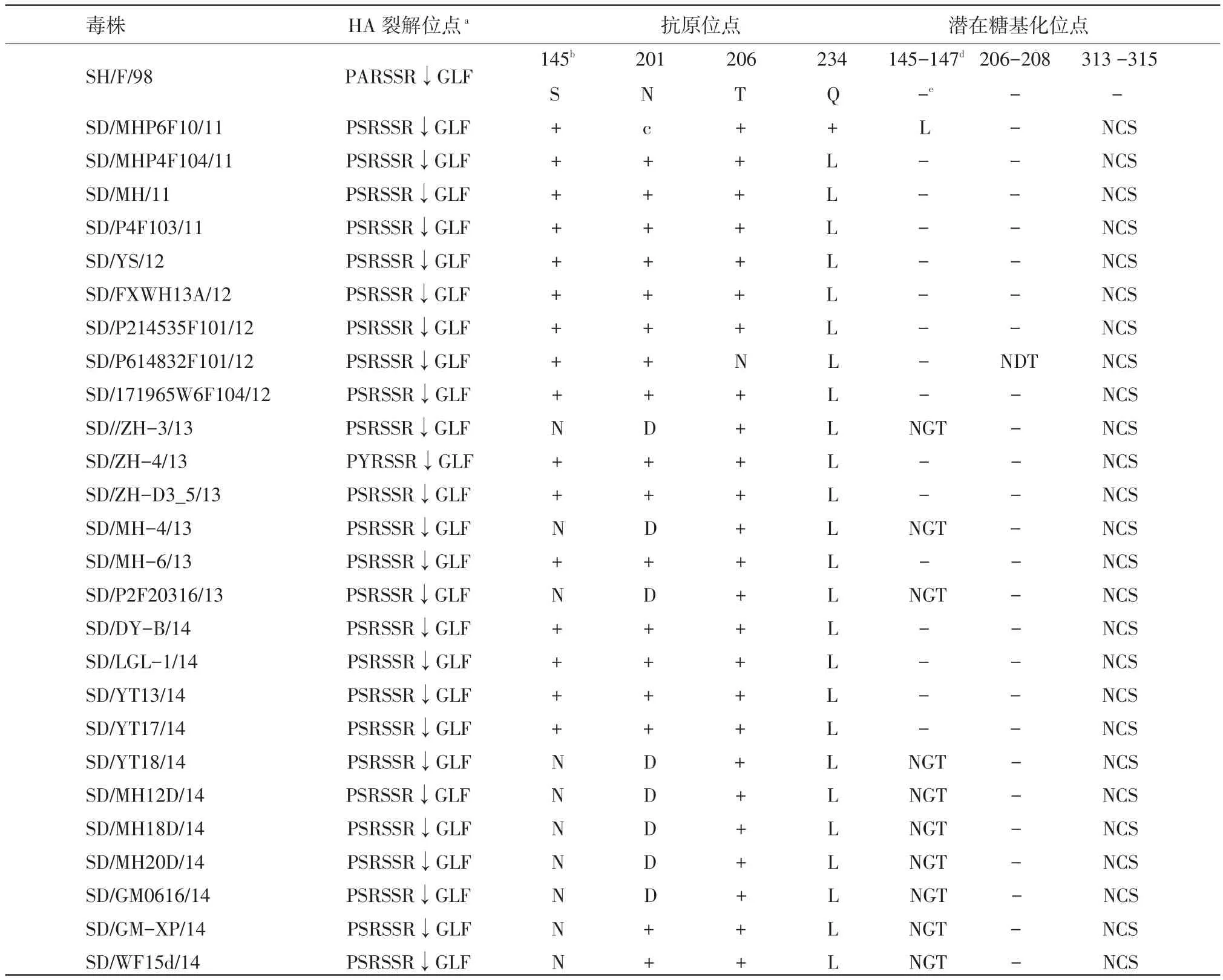
表2 H9亚型AIV的HA裂解位点、抗原位点和HA氨基酸序列潜在糖基化位点分析
参考文献:
[1]Li Chengjun,Yu Kangzhen,Tian Guobin,et al . Evolution of H9N2 influenza viruses from domestic poultry in Mainland China [J].Virology,2005,340(1):70-83.
[2]Choi Y K,Ozaki H,Webby R J,et al . Continuing evolution of H9N2 influenza viruses in Southeastern China[J]. J Virol,2004,78(16):8609-8614.
[3]Liu Hongqi,Liu Xiufan,Cheng Jian,et al . Phylogenetic analysis of the hemagglutinin genes of twenty-six avian influenza viruses of subtype H9N2 isolated from chickens in China during 1996-2001[J].Avian Dis,2003,47(1):116-127.
[4]Sun Y,Pu J,Fan L,et al.Evaluation of the protective efficacy of a commercial vaccine against different antigenic groups of H9N2 influenza viruses in chickens[J]. Vet Microbiol,2012 Apr 23;156(1-2):193-9.Epub,2011 Oct 10.
[5]Ping J,Li C,Chen H,et al.Single amino acid mutation in the HA alters the recognition of H9N2 influenza virus by a monoclonal antibody[J].Biochem Biophys Res Commun,2008,371(1):168-171.
[6]Zhao J,Chai L N,Wang Z L.Sequence and phylogenetic analysis of the haemagglutinin genes of H9N2 avian influenza[J].viruses isolated in central China during 1998-2008 Bing Du Xue Bao,2011 Mar;27 (2):122-128.
Genetic variation and phylogenetic analysis of haemagglutinin geneof avian influenza viruses in Shandong Provincefrom 2011 to 2014
LI Lin1,CHEN Rui-ai1,2,3,LIU Hong-bo1,LIANG Mei-lan1,ZHANG Xian-hao1,HE Dong-sheng2,3
(1.Key Laboratory of Biotechnology and Bioproducts Development for Animal Epidemic Prevention,Ministry of Agriculture,Zhaoqing Dahuanong Biological Medicine Co.,Ltd.,Zhaoqing 526238,China;2.College of Veterinary Medicine,South China Agricultural University,Guangzhou 510642,China;3.South China Collaborative Innovation Center for Poultry Disease Control and Product Safety,Guangzhou 510640,China)
Abstract:The HA genes were amplified and sequenced from 26 isolates of H9 subtype Avian Influenza Virus(AIV)in Shandong Province.Phylogenetic analysis indicated that all isolates belonged to the lineage h9.4.2.5.And they had 88.0~91.5%of nucleotide identity to 3 widely used vaccine strains GD/SS/94,SD/6/96 and SH/F/98.No consecutive basic amino acids insertion was found in the HA cleavage sites,showing the characteristic feature of low-pathogenic AIV . All the isolates had the substitution Q226L.Additional potential glycosylation site at residues 313-315(NCS)was found in all these isolates,and 9 of 17 strains isolated in 2013 and 2014 in 145-147 had new potential glycosylation sites NGT . Our results demonstrated that lineage h9.4.2.5 was more dominant than other lineages as it harbored more viruses that widely distributed in Shandong Province in recent years.Updated vaccine and increased veterinary biosecurity on poultry farms and trade markets need to prevent and control avian influenza.
Key words:H9;AIV;hamagglutinin;phylogenetic analysis;variation
中图分类号:S852.65
文献标志码:A
文章编号:0529- 6005(2016)03- 0010- 03
收稿日期:2014-11-14
基金项目:广东大华农动物保健品股份有限公司科研项目(03-63C201206-4)
作者简介:李琳(1979-),女,兽医师,本科,从事家禽传染病学研究,E-mail:mulian9618905@qq.com
通讯作者:贺东生,E-mail:Dhe@scau.edu.cn
Corresponding author:HE Dong-sheng
