蛋白酶高产菌株的筛选鉴定与酶学特性研究
2016-06-21赵晓艳曾志驰穆丽丽
赵晓艳,曾志驰,穆丽丽,邓 悦,王 飞
(江西农业大学 生物科学与工程学院,江西 南昌 330045)
蛋白酶高产菌株的筛选鉴定与酶学特性研究
赵晓艳,曾志驰,穆丽丽,邓 悦,王 飞*
(江西农业大学 生物科学与工程学院,江西 南昌 330045)
摘要:以酪蛋白水解圈为筛选标记,在以酪蛋白为唯一氮源的平板上,从土壤中分离筛选到3株高产蛋白酶的菌株,经形态学鉴定和16S rRNA分析,将其分别鉴定为Bacillus sp. G1、Bacillus sp. G2、Stenotrophomonas sp. V1。将这3株菌株的发酵液通过硫酸铵分级沉淀初步纯化后,其比酶活分别为22.21、19.10和16.03 U/mg。菌株Bacillus sp. G1和Bacillus sp. G2所产蛋白酶的最适反应温度和最适反应pH值均为40 ℃和7.0;菌株Stenotrophomonas sp. V1所产蛋白酶的最适反应温度为70 ℃,最适酶反应pH值为 7.5。
关键词:蛋白酶;筛选;鉴定;酶学特性; Bacillus sp. G1; Bacillus sp. G2; Stenotrophomonas sp. V1
蛋白酶(Protease, EC 3.4)属于水解酶类,是最重要的三大工业用酶之一,销售额约占全球酶制剂市场的60%[1],被广泛应用于食品、酿造、医药、纺织、皮革、日用化学、洗涤剂、饲料以及水产加工等多个行业,在我国国民经济的发展中起着重要的作用[2]。产生蛋白酶的微生物种类繁多,细菌、病毒、真菌、原生动物等皆有报道[3-4]。目前,用于蛋白酶工业生产的菌株多为地衣芽孢杆菌、枯草芽孢杆菌、短小芽孢杆菌等[4-5]。在已知的3000多种蛋白酶中,绝大多数来源于常温生物,其作用的温度、pH值、离子强度范围均较窄,从而在应用上受到限制。因此,耐受低温、高温、盐、酸、碱的蛋白酶越来越受到关注[6-10]。在尊重生物多样性的基础上,寻找和开发新型微生物蛋白酶资源,仍然具有重要的科研、应用及商业价值。
本研究以酪蛋白为底物,从土壤中分离筛选到3株产蛋白酶的菌株,通过形态和16S rRNA序列测定对这3株菌株进行了鉴定,并对3株菌所产蛋白酶的酶学特性进行了初步研究,以期为开发新的微生物蛋白酶制剂提供菌株资源。
1材料与方法
1.1材料与试剂
Foline-phenol试剂、酪氨酸、酪蛋白、Tris-平衡酚、葡萄糖、胰蛋白胨、酵母提取物均购于国药集团化学试剂有限公司。牛血清蛋白、考马斯亮蓝G250、溶菌酶、蛋白酶K、Taq DNA聚合酶购于上海博彩生物科技有限公司。
筛选培养基:可溶性淀粉2 g、酪蛋白1.0 g、K2HPO40.05 g、MgSO4· 7H2O 0.05 g、NaCl 0.05 g、FeSO4· 7H2O 0.001 g、琼脂2 g、水100 mL, pH 7.2~7.4。
产酶培养基:牛肉膏0.3 g、酪蛋白1.0 g、琼脂2.0 g、NaCl 0.5 g、蒸馏水100 mL, pH 7.6~7.8。
LB培养基(L):酵母粉 5.0 g、胰蛋白胨10.0 g、NaCl 10.0 g, pH 7.0~7.2。
1.2主要仪器与设备
SHIMADZU UV-2401紫外可见分光光度计,日本SHIMADZU公司; Beckman台式高速冷冻离心机(Allegra X-22R),美国Beckman公司; DYCP-31C水平核酸电泳仪,北京六一公司。
1.3试验方法
1.3.1菌株筛选从江西南昌、上高、铜鼓等地采集土样,稀释涂布于筛选培养基,在30 ℃下培养2 d,将有水解圈的菌株反复在筛选培养基上划线纯化。
1.3.2菌株鉴定 将菌株划线纯化,挑取单菌落进行革兰氏染色,镜检。用高盐法[11]提取各产酶菌株的总DNA;以DNA作模板,利用16S rRNA基因的通用引物27f:5’-AGAGTTTGATCCTGGCTCAG-3’(E.colibases 8 to 27)和1492r:5’-TACCTTGTTACGACT T-3’(E.colibases 1507 to 1492)[12]进行PCR扩增,扩增反应体系为:10×Taq聚合酶反应缓冲液5L、dNTP(20 mmol/L)4L、引物(25 pmol/L)各1L、Mg2+(25 mmol/L)4L、菌体DNA(约50 ng/L)1L、Taq DNA聚合酶(5 U/L)0.5L,用灭菌双蒸水补至50L。反应条件:95 ℃变性5 min,95 ℃ 30 s,55 ℃ 30 s,72 ℃ 1.5 min,30个循环;最后72 ℃延伸10 min。
PCR产物经0.75%琼脂糖凝胶电泳纯化后,将其送至南京金斯瑞生物科技有限公司测序。对测得的16S rRNA基因序列在GenBank中进行Blast比对,下载高同源性菌株的16S rRNA基因序列,并加入大肠杆菌E.coliATCC 11775T的16S rRNA基因序列作为外参,用MEGA version 5.05软件包中的Cluster W进行同源聚类分析。然后利用邻接法(Neighbor-Joining)1000次泊松校验构建系统发育树[13]。
1.3.3盐析按0%~20%、20%~40%、40%~60%、60%~80%、80%~100%的硫酸铵饱和梯度进行分级沉淀。操作步骤:将粗酶液量好体积,倒入烧杯中,烧杯内放置磁性转子,将烧杯置于冰上,打开磁力搅拌器。按粗酶液的体积缓慢加入硫酸铵,使饱和度达到20%,然后在4 ℃下以13000 r/min的转速离心20 min,对沉淀以少量不含硫酸铵的20 mmol/L Tris-HCl (pH 7.4)重悬,测定蛋白含量和酶活;对上清继续添加硫酸铵,使饱和度达到40%,重复以上步骤,直至饱和度达到100%为止[14]。计算收集的各级沉淀的蛋白含量与酶活。取比酶活较高的分级沉淀蛋白,以少量20 mmol/L Tris-HCl (pH 7.4)重悬。
1.3.4最适反应温度测定将适量粗酶液加入至20 mmol/L Tris-HCl(pH 7.5)的测活体系中,将反应体系分别放置于40、50、60、70 ℃条件下,反应30 min后加入三氯乙酸终止,用Foline-Phenol试剂显色后测定OD680,实验设置3个重复[15]。
1.3.5最适反应pH测定将适量的粗酶液加入至不同pH值的缓冲液体系中,测定酪蛋白水解酶活力,缓冲液为:柠檬酸缓冲液(pH 4.0~6.0)、磷酸盐缓冲液(pH 6.0~8.0)、Tris-HCl缓冲液(pH 7.5~9.0)和甘氨酸-NaOH缓冲液(pH 9.0~12.0),各缓冲液浓度皆为20 mmol/L。酶活性测定反应在最适温度条件下进行,反应时间为30 min。重复3次,以最大酶活为对照(100%),比较粗酶液在不同pH条件下的相对酶活[16]。
1.4酶活测定
酶活测定参照文献[2, 7]中的方法进行。酶活力单位定义:每分钟水解酪蛋白产生1 μg酪氨酸所需的蛋白量(mg)为一个酶活力单位。
1.5蛋白含量测定
按Bradford方法进行,以牛血清蛋白作为标准蛋白,具体操作见参考文献[17]。
2结果与分析
2.1菌株筛选结果
从江西上高、铜鼓、南昌等地采集土样,稀释分离后涂布于以酪蛋白为唯一氮源的平板上,以酪蛋白水解圈为筛选标记进行筛选,选取水解圈/菌落直径较大的菌株划线纯化,得到3株菌株。菌落形态见图1。
挑取单菌落,革兰氏染色后镜检,菌体染色结果见图2。由图2可见:菌株G1和G2产芽孢,不膨大,芽孢中生,革兰氏染色阳性,菌体大小(0.8~1.0)μm×(2.0~3.0)μm;菌株V1不产芽孢,革兰氏染色阴性,菌体大小(0.2~0.5)μm×(0.8~1.0) μm。
将菌株G1、G2、V1分别接种于液体产酶培养基,在30 ℃下以180 r/min摇瓶培养48 h,以6000 r/min离心10 min;取上清液5 μL点样于酪蛋白平板,在30 ℃下反应30 min,观察粗酶液对酪蛋白的水解效果。从图2 d~图2f可以看出,3株菌的发酵液均对酪蛋白有较好的水解效果。
2.2菌株鉴定及系统发育树的构建
用高盐法提取各产酶菌株的总DNA,以DNA作模板,利用16S rRNA基因的通用引物27f和1492r进行PCR扩增,PCR产物经0.75%琼脂糖凝胶电泳后检测其纯度,结果如图3所示。

a:菌株G1菌落(正面); b:菌株G2菌落(正面); c:菌株V1菌落(正面)。

a:菌株G1革兰氏染色; b:菌株G2革兰氏染色; c:菌株V1革兰氏染色; d:菌株G1粗酶液在酪蛋白平板上的水解圈; e:菌株G2粗酶液在酪蛋白平板上的水解圈; f:菌株V1粗酶液在酪蛋白平板上的水解圈。
图23株产蛋白酶菌株的菌体形态及其粗酶液活性检测
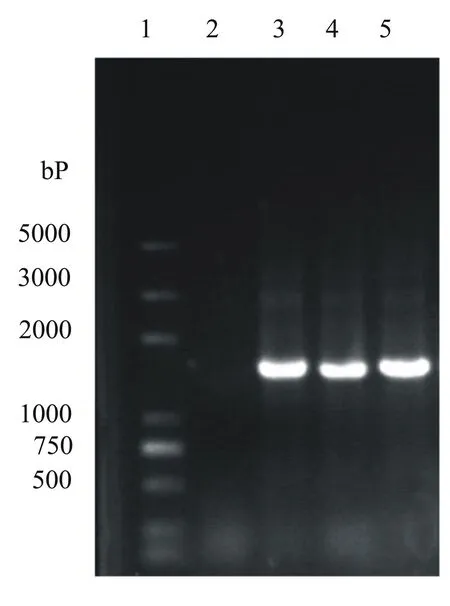
1:5000 Plus核酸Marker;2:阴性对照;3:菌株G1 16S rRNA;
对3株菌株构建16S rRNA系统发育树,结果(图4~图6)显示:菌株G1与巨大芽孢杆菌(Bacillusmegatherium)16S rRNA聚为一类(相似性97%);菌株G2与阿耶波多芽孢杆菌(BacillusaryabhattaiB8W22)16S rRNA聚为一类(相似性98%);菌株V1与寡养单胞菌(StenotrophomonaschelatiphagaLPM-5)16S rRNA聚为一类(相似性100%)。因此,分别将这3株菌命名为Bacillussp. G1、Bacillussp. G2和Stenotrophomonassp. V1。
2.3盐析结果
采用盐析法对粗酶液进行分级沉淀,在不同浓度的硫酸铵饱和溶液中,酶活力、蛋白含量和比酶活测定结果见图7。
由图7可知:菌株Bacillussp. G1的粗酶液在60%~80%硫酸铵饱和浓度的沉淀中有较高的比酶活(22.21 U/mg),其单位酶活为114.53 μg/(min·mg);菌株Bacillussp. G2的粗酶液在80%~100%硫酸铵饱和浓度的沉淀中有较高的比酶活(19.10 U/mg),其单位酶活为106.50 μg/(min·mg);菌株Stenotromonassp. V1的粗酶液在60%~80%硫酸铵饱和浓度的沉淀中有较高的比酶活(16.03 U/mg),其单位酶活为107.03 μg/(min·mg)。

图4 菌株Bacillus sp. G1 16S rRNA系统发育树

图5 菌株Bacillus sp. G2 16S rRNA系统发育树
2.4反应温度对酶活性的影响
分别取适量的盐析后的酶液在不同温度下进行反应,测定酶反应的最适温度。以各温度条件下的最高酶活为对照(100%),绘制温度-相对酶活曲线。由图8可知:菌株Bacillussp. G1和Bacillussp. G2所产蛋白酶的最适反应温度为40 ℃,在50 ℃时其相对酶活为78%,在30 ℃条件下相对酶活分别为60%和30%。当温度达60 ℃以上时失去活力,表明这两株菌所产的蛋白酶为中温蛋白酶;菌株Stenotromonassp. V1所产蛋白酶的最适反应温度为70 ℃,在40~60 ℃时其相对酶活为60%,但在30 ℃和80 ℃时均失去活力,表明该菌株所产的蛋白酶为高温蛋白酶。
2.5反应pH值对酶活性的影响
分别取适量的盐析后的酶液在不同pH条件下进行反应,酶反应温度选取各自的最适温度。以各pH条件下的最高酶活为对照(100%),绘制pH值-相对酶活曲线,见图9。

图6 菌株Stenotromonas sp. V1 16S rRNA系统发育树
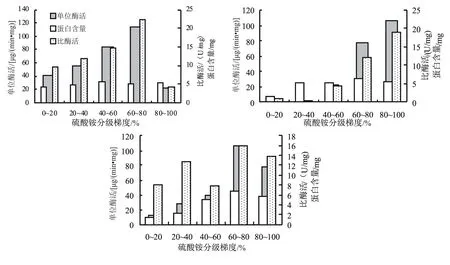
a: Bacillus sp. G1菌株粗酶液盐析结果; b: Bacillus sp. G2菌株粗酶液盐析结果; c:Stenotromonas sp. V1菌株粗酶液盐析结果。
从图9可见:菌株Bacillussp. G1所产蛋白酶的最适酶反应pH值为7.5,当pH在8.5以上和5.0以下时,其相对酶活丧失;在pH为6时,其相对酶活为35%, pH为8时相对酶活为98%, pH为7时相对酶活为80%(图9-a)。表明该菌株所产蛋白酶为中性蛋白酶。
菌株Bacillussp. G2所产蛋白酶的最适酶反应pH值为7.5,当pH在8.5以上和4.0以下时,其相对酶活丧失;在pH为6时,其相对酶活为34%, pH为8时相对酶活为8%, pH为7时相对酶活为80%(图9-b),表明该菌株所产蛋白酶为中性蛋白酶。
菌株Stenotrophomonassp. V1所产蛋白酶的最适酶反应pH值为8.0,当pH在10以上和5以下时,其相对酶活丧失;在pH为9时,其相对酶活为18%, pH为6时在柠檬酸缓冲液中很难测出酶活(4%), pH为7.5时相对酶活为80%(图9-c),表明该菌株所产蛋白酶为碱性蛋白酶。
3讨论
本研究获得的3株高产蛋白酶菌株中,Bacillussp. G1和Bacillussp. G2所产蛋白酶均为常温中性蛋白酶;对这两株菌株通过盐析初步分级纯化后,所得蛋白酶的比酶活分别为22.21和19.10 U/mg,与已报道的芽孢杆菌所产蛋白酶活力[18-19]相比不占优势,但通过培养条件优化后有望提高,具有一定的应用潜力。

图8 3株菌株所产蛋白酶的最适反应温度

a:菌株Bacillus sp. G1; b:菌株Bacillus sp. G2; c:菌株Stenotrophomonas sp. V1。
嗜麦芽窄食单胞菌(Stenotrophomonasmaltophilia),又称嗜麦芽黄单胞菌、嗜麦芽寡养单胞菌。由于该菌产生的蛋白酶多为高温碱性蛋白酶,因此具有较高的工业开发和应用价值,其相关研究近年来在国际上越来越受到重视。如来源于嗜麦芽窄食单胞菌PO-1的丝氨酸碱性蛋白酶,其分子量为36.0 kDa,最适pH 9.0,最适反应温度60 ℃[20];来源于Stenotrophomonasmaltophiliastrain S-1的碱性蛋白酶SmP-1,其分子量约为40.0 kDa,最适pH 12.0,最适反应温度50 ℃[21]。
菌株Stenotrophomonassp. V1所产的蛋白酶为高温碱性蛋白酶,通过盐析初步分级纯化后所得蛋白酶的比酶活为16.03 U/mg,最适反应温度为70 ℃,最适酶反应pH为7.5,与已报道的寡营养单胞菌所产碱性蛋白酶相比,其酶活力均高于从Stenotrophomonasmaltophiliastrain PO-1和Stenotrophomonasmaltophiliastrain S-1发酵液中所分离的碱性蛋白酶的,显示出很大的应用潜力。
参考文献:
[1] Senol M, Nadaroglu H, Dikbas N, et al. Purification of chitinase enzymes fromBacillussubtilisbacteria TV-125, investigation of kinetic properties and antifungal activity againstFusariumculmorum[J]. Annals of Clinical Microbiology and Antimicrobials, 2014, 13(1): 35.
[2] 邱秀宝,崔福绵.蛋白酶的研究和应用[J].微生物学通报,1982(5):13.
[3] Haki G, Rakshit S. Developments in industrially important thermostable enzymes: a review[J]. Bioresource Technology, 2003, 89(1): 17-34.
[4] Schallmey M, Singh A, Ward O P. Developments in the use ofBacillusspecies for industrial production[J]. Canadian Journal of Microbiology, 2004, 50(1): 1-17.
[5] Abidi F, Limam F, Marzouki M N. Purification and characterization of an alkaline protease prot 1 fromBotrytiscinerea[J]. Applied Biochemistry and Biotechnology, 2007, 141(2-3): 361-376.
[6] 方海红,胡好远.微生物碱性蛋白酶的研究进展[J].微生物学通报,2002,29(2):57-59.
[7] 翁海波,敬蔚然,杨丹丽,等.产高温碱性蛋白酶菌株筛选及酶学性质研究[J].郑州大学学报:工学版,2010(1):70-73.
[8] 唐雪明,王正祥,邵蔚蓝,等.碱性蛋白酶工程菌发酵条件及重组酶的纯化和性质的研究[J].生物工程学报,2002,18(6):729-734.
[9] 孙谧,洪义国.一株产低温碱性蛋白酶嗜冷海洋细菌YS—9412—130的分离和培养条件研究[J].海洋水产研究,2000,21(4):1-5.
[10] 林念炜,张锐,赵晶,等.南极产低温蛋白酶菌株Marinobactersp. R2的发酵条件及酶学性质研究[J].厦门大学学报:自然科学版,2004,43(6):865-869.
[11] Miller S, Dykes D, Polesky H. A simple salting out procedure for extracting DNA from human nucleated cells[J]. Nucleic Acids Research, 1988, 16(3): 12-15.
[12] Goodfellow M, Stackebrandt E. Nucleic acid techniques in bacterial systematics [M]. J Wiley, 1991.
[13] Dong W, Jiang S, Shi K, et al. Biodegradation of fenoxaprop-P-ethyl (FE) byAcinetobactersp. strain DL-2 and cloning of FE hydrolase geneafeH[J]. Bioresource Technology, 2015, 186: 114-121.
[14] Sha X M, Tu Z C, Liu W, et al. Effect of ammonium sulfate fractional precipitation on gel strength and characteristics of gelatin from bighead carp (Hypophthalmichthysnobilis) scale[J]. Food Hydrocolloids, 2014, 36: 173-180.
[15] Van Wormhoudt A, Le Chevalier P, Sellos D. Purification, biochemical characterization and N-terminal sequence of a serine-protease with chymotrypsic and collagenolytic activities in a tropical shrimp,Penaeusvannamei(Crustacea, Decapoda)[J]. Comparative Biochemistry and Physiology Part B: Comparative Biochemistry, 1992, 103(3): 675-680.
[16] Wang F, Hou Y, Zhou J, et al. Purification of an amide hydrolase DamH fromDelftiasp. T3-6 and its gene cloning, expression, and biochemical characterization[J]. Applied Microbiology and Biotechnology, 2014, 98(17): 7495-7499.
[17] Bradford M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1): 248-254.
[18] Aftab S, Ahmed S, Saeed S, et al. Screening, isolation and characterization of alkaline protease producing bacteria from soil[J]. Pak J Biol Sci, 2006(9): 2122-2126.
[19] Ray A K, Roy T, Mondal S, et al. Identification of gut-associated amylase, cellulase and protease-producing bacteria in three species of Indian major carps[J]. Aquaculture Research, 2010, 41(10): 1462-1469.
[20] De Toni C, Richter M, Chagas J, et al. Purification and characterization of an alkaline serine endopeptidase from a feather-degradingXanthomonasmaltophiliastrain[J]. Canadian Journal of Microbiology, 2002, 48(4): 342-348.
[21] Miyaji T, Otta Y, Shibata T, et al. Purification and characterization of extracellular alkaline serine protease fromStenotrophomonasmaltophiliastrain S-1[J]. Letters in Applied Microbiology, 2005, 41(3): 253-257.
(责任编辑:黄荣华)
Research on Screening, Identification and Enzymological Characteristics of Strains with High Yield of Protease
ZHAO Xiao-yan, ZENG Zhi-chi, MU Li-li, DENG Yue, WANG Fei*
(College of Bioscience and Bioengineering, Jiangxi Agricultural University, Nanchang 330045, China)
Abstract:Three bacteria strains producing protease were screened and isolated from soil by observation of clearing zones on casein agar plates, based on phenotypic and 16S rRNA gene phylogenetic analysis, G1, G2 and V1 were preliminary classed and named as Bacillus sp. G1, Bacillus sp. G2 and Stenotrophomonas sp. V1. Bacillus sp. G1, Bacillus sp. G2 and Stenotrophomonas sp. V1 were all aerobic strains. The proteases from Bacillus sp. G1, Bacillus sp. G2 and Stenotrophomonas sp. V1 were purified by the methods of ammonium sulfate precipitation, the specific activities of three proteases were up to 22.21 U/mg, 19.10 U/mg and 16.03 U/mg, respectively. The enzymes from Bacillus sp. G1 and G2 were optimally active at 40 ℃ and in 20 mmol/L phosphate buffered saline buffer(PBS, pH 7.0), and the alkaline protease from Stenotrophomonas sp. V1 was optimally active at 70 ℃ and in 20 mmol/L PBS buffer (pH 7.5).
Key words:Protease; Screening; Identification; Enzymological characteristics; Bacillus sp. G1; Bacillus sp. G2; Stenotrophomonas sp. V1
收稿日期:2015-10-07
基金项目:国家自然科学基金地区科学基金项目(31560031)。
作者简介:赵晓艳(1995─),女,从事生物与工程方面的研究。*通讯作者:王飞。
中图分类号:TQ925.2
文献标志码:A
文章编号:1001-8581(2016)04-0032-07
