花生根部耐盐相关miRNAs的鉴定与功能分析
2016-06-05张廷婷闫彩霞赵小波李春娟石程仁于久江单世华李荣贵
张廷婷,闫彩霞,赵小波,李春娟,石程仁,于久江,单世华*,李荣贵
(1.山东省花生研究所,山东 青岛 266100; 2.青岛大学生命科学学院,山东 青岛 266071;3.Food and Nutrition Biotechnologies,Germantown,Maryland 13314,USA)
花生根部耐盐相关miRNAs的鉴定与功能分析
张廷婷1,2,闫彩霞1,赵小波1,李春娟1,石程仁1,于久江3,单世华1*,李荣贵2*
(1.山东省花生研究所,山东 青岛 266100; 2.青岛大学生命科学学院,山东 青岛 266071;3.Food and Nutrition Biotechnologies,Germantown,Maryland 13314,USA)
miRNAs作为一种基因表达调控子在植物应答胁迫的过程中起着重要的作用,当花生受到盐胁迫时,也会有相应的miRNAs参与基因表达调控应答胁迫。本研究对200份花生品种进行萌发期耐盐性鉴定,获得了高耐盐花生品种3份,中耐盐花生品种5份,盐高敏感品种4份。并对不同耐盐性花生品种在盐胁迫处理条件下进行了抗氧化酶活性(SOD,POD,CAT)和MDA含量的测定。结果表明,耐盐性花生品种清除活性氧的能力大于盐敏感型。盐胁迫条件下,耐盐性花生植株MDA含量较少,受到的伤害相对较小,而盐敏感植株的受伤害程度最大,也证明了耐盐性花生品种在受到盐胁迫伤害时植物体内存在更强大的保护作用。通过小RNA测序及对靶基因序列进行功能分析和同源序列功能检索,获得了8条花生耐盐相关保守miRNAs序列,miR159-1,miR159-2,miR159-3,miR164-2,miR167-3,miR319-1,miR319-2,miR2111-1。荧光定量PCR测定结果表明,这些花生保守miRNAs受盐胁迫诱导,并调控其靶基因的应答反应。
花生;miRNA;靶基因;抗氧化酶活性;耐盐
花生是世界上重要的油料作物之一,也是我国主要的油料作物、经济作物和出口创汇作物。在油料作物中种植面积仅次于油菜,是国民经济发展的重要保障。中国是世界花生生产大国,种植面积仅次于印度,总产约占世界的40%,居首位,单产高出世界平均水平。即使如此也不能满足日益增加的需求,特别是入世以来,随着世界贸易的进一步扩大,人们生活水平的提高和膳食结构的变化,人们对花生及其制品需求量持续增加,提高花生产量势在必行。但随着人口的增加,耕地面积的不断减少,粮油争地矛盾日益突出,开发和利用盐渍化土地资源是提高花生产量的重要途径。植物在响应及适应盐胁迫的过程中,形成了一系列调控机制,如渗透调节、离子区域化、高盐诱导产生大分子蛋白等。其中,胁迫信号的感知和转导是引起植物产生理化响应及发生变化的关键步骤。植物耐盐能力的大小是一个多基因控制的数量性状,涉及生理生化多方面的因素,并由多种耐盐机制协调作用。全生育期耐盐鉴定发现花生本身耐盐能力有限[1],目前花生耐盐基因利用方面的研究较少,通过分子生物学手段研究花生耐盐机制具有重要的意义。
MicroRNAs(miRNAs)是一类长度为21nt左右、内源性、非编码单链小分子RNA,广泛存在于植物[2-3]、动物[4-6]和病毒[7-8]中,通过碱基互补对靶mRNA的翻译进行抑制或降解,参与细胞凋亡、生长控制、脂肪代谢、发育调控、信号转导、应对胁迫和抗病毒机制等[2,9-11]。miRNAs作为一种基因表达调控子在植物应答胁迫的过程中起着重要的作用。 Zhang等[12]发现了123种受各种胁迫诱导的包含miRNA序列的EST叠连群,这些环境胁迫包括干旱、盐碱、低温、高温、营养缺乏、氧化胁迫以及微生物侵染等。不同的植物,如拟南芥[13]、水稻[14-15]、玉米[16]、毛果杨[17]、大麦[18]以及普通烟草[19]在干旱和盐碱胁迫下会诱导不同miRNAs的差异表达,这些miRNAs包括miR156、miR159、miR165、miR167、miR168、miR169、miR319、miR393等,如过量表达miR169能增强番茄的抗旱能力。通过生物信息学和高通量测序的方法,许多保守和新的miRNAs在盐胁迫的大豆[20],花生[21-22],杨树[19,23],葡萄[24-25]等植物中被发现并进行了实验验证和功能分析。当花生受到盐胁迫时,也会有相应的miRNAs参与基因表达调控应答胁迫。
目前发掘植物miRNAs的方法主要有直接克隆法、高通量测序法和生物信息学法[26]。本研究利用高通量测序技术结合生物信息学分析及分子生物学实验等方法,发掘和鉴定花生苗期根部耐盐相关miRNAs及其靶基因,并研究miRNAs对盐胁迫的应答特性及对靶基因的调控作用模式,阐明miRNAs的功能。为通过增强或削弱内源性miRNAs的基因沉默效应来提高花生的耐盐能力奠定基础。
1 材料与方法
1.1 花生品种筛选与培养
以200份花生种质资源为筛选材料,进行萌发期耐盐性筛选鉴定试验。每个品种均采用直径13cm玻璃培养皿,覆双层灭菌滤纸,选取饱满一致的花生种子每皿10粒,处理与对照分别添加100mmol/L NaCl溶液和自来水各30mL,每组设3个重复,置于人工气候室(温度28℃,湿度60%,光照强度500μmol·m-2·s-1)。培养5d,统计萌发种子数,胚根长度、株高,计算萌发率和伤害度。
萌发率(%)=处理萌发数/对照萌发数×100
伤害度(%)=(对照平均根长-处理平均根长)/对照平均根长×100
根据种子萌发率、伤害度,筛选出不同耐盐类型(高耐盐、中耐盐、盐敏感)花生品种。本实验设定萌发率大于90%且伤害度低于20%的品种为耐盐品种,萌发率大于90%且伤害度低于50%的品种为中耐盐品种,萌发率小于70%且伤害度高于20%的品种为盐敏感品种。
以鉴选出的花生品种A025(耐盐)、山花10号(中耐盐)、D001(盐敏感)为鉴定材料,采用容积4L培养盒加自来水培养于人工气候室(温度28℃,湿度60%,光照强度500μmol·m-2·s-1),每盒30株,种子萌芽培养20d后,于200mmol/L NaCl溶液分别处理0、2、4、6、8h后取根,液氮冷冻后保存于-80℃冰箱备用。每组设3次重复。
1.2 测序与生物信息学分析
构建花生Small RNA文库,利用双末端测序(Paired-End)法,完成花生Small RNA(对照组和处理组2、4、6、8h混合根样品)测序,并进行生物信息分析。通过WebGO和InterPro分析,对靶基因序列进行功能分析和同源序列功能检索,获得了花生耐盐相关保守miRNAs序列,选择8条miRNA及其靶基因序列作为研究对象,以花生品种山花10号为材料,对盐胁迫下花生根部耐盐相关miRNAs及其靶基因的表达量进行测定。
1.3 RNA及miRNA的提取
花生样品RNA的提取参照宝生物GeneJET Plant RNA提取试剂盒说明书进行。miRNA的提取参照天根miRcute miRNA提取试剂盒说明书进行。提取样品采用1.2%琼脂糖凝胶电泳和Nanodrop2000分光光度计检测纯度和浓度。
1.4 抗氧化酶活性的测定
称取花生根样品(0.5g),按重量(g):体积(mL)=1∶9 (SOD的测定按照1∶4)的比例加入9倍体积的磷酸盐缓冲液(0.1mol/L pH7~7.4),冰水浴条件下制备成10%的组织匀浆,3500r/min离心10min后,取上清液。参照南京建成生物工程研究所的POD、SOD、CAT、MDA试剂盒方法进行测定并计算。
1.5 荧光定量PCR分析
Actin基因作为内参基因,引物5'-GAG GAG AAG CAG AAG CAA GTT G;5'-AGA CAG CAT ATC GGC ACT CAT C。miRNAs序列及引物设计如表1:
qRT-PCR 分析参照TaKaRa SYBR®PrimixEx TaqTMⅡ(Tli RNaseH Plus)试剂盒,采用ABI 7300/7500和StepOnePlusTM Real-Time PCR System进行。PCR反应液的组份配制如表2所示(反应液配制在冰上进行)。
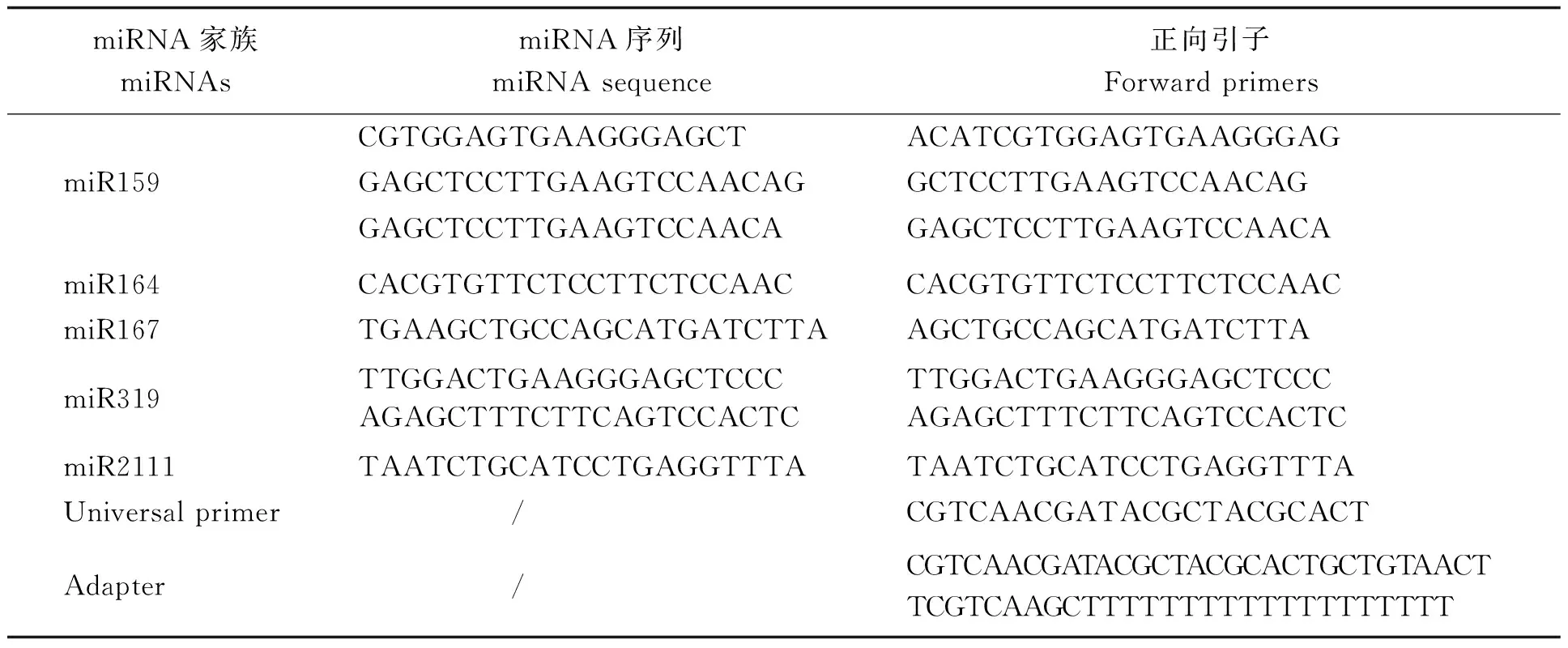
表1 miRNAs序列及引物设计Table 1 miRNA sequence and primer design
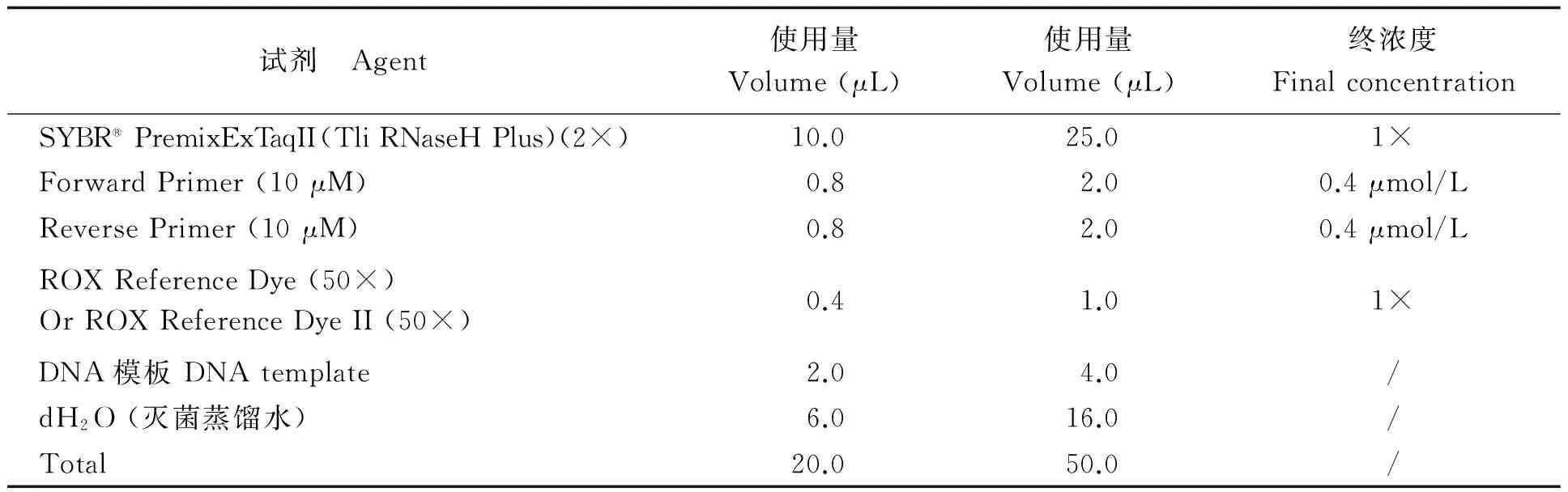
表2 PCR反应液的配制组份Table 2 Component of PCR reaction solution
反应条件:第一步预变性:95℃ 2min,第二步荧光收集:94℃ 15s,60℃ 15s,72℃ 20s共40个循环,第三步溶解曲线分析:65℃逐步升温到95℃间隔0.1℃收集一次荧光信号,第四步冷却:40℃冷却30s。每管重复3次。
2 结果与分析
2.1 耐盐花生品种筛选
以200份花生种质资源为筛选材料,进行萌发期耐盐性筛选鉴定试验,获得高耐盐花生品种3份,中耐盐花生品种5份,盐高敏感品种4份。表3为部分典型品种耐盐类型。
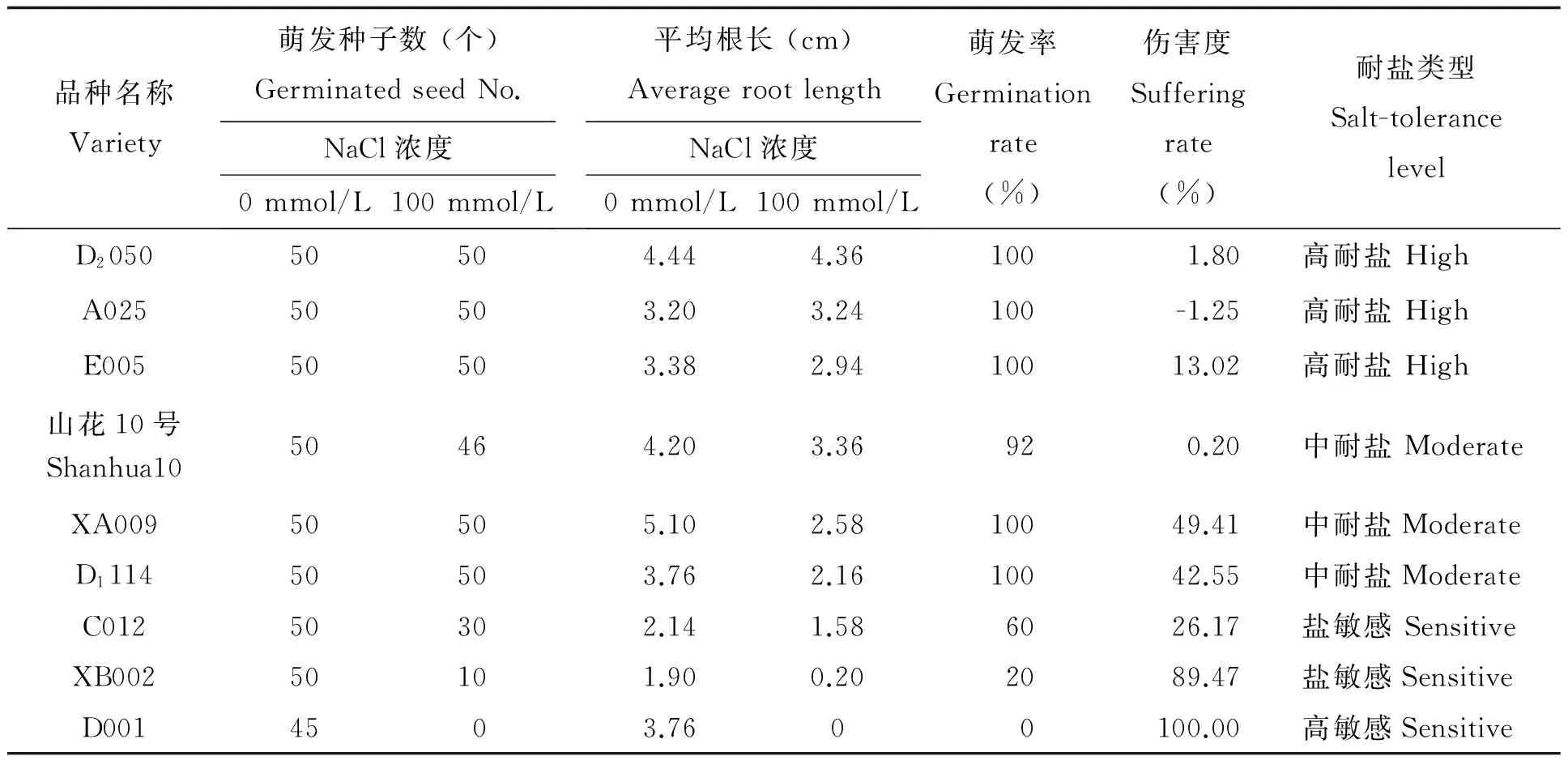
表3 耐盐筛选部分典型品种Table 3 Typical varieties of salt tolerance in screening
2.2 盐胁迫对不同花生品种根部抗氧化酶活性的影响
200mmol/L NaCl盐胁迫处理前与处理后,耐盐性高的花生品种抗氧化酶活性(SOD,POD,CAT)都明显高于盐敏感品种(图1)。盐胁迫处理不同时间,不同花生品种根的超氧化物歧化酶(SOD)活性都有不同的变化,耐盐性品种A025呈现先降低后升高的趋势,中耐盐品种山花10号和盐敏感品种D001呈现先升高后降低的趋势(图1A)。盐处理2h时,不同花生品种根的POD活性都出现明显的下降,但随着盐处理时间的延长至4h,6h,8h,POD活性都维持在一定水平变化不明显(图1B)。盐处理2h内,各品种的CAT活性没有明显变化,2h之后,A025开始升高,山花10号和D001开始下降,6h时都达到最高或最低并维持在一定水平不再变化(图1C)。表明耐盐花生品种植株的清除活性氧的能力大于盐敏感型,相同的盐处理时间,不同耐盐类型的抗氧化酶活性变化不一致,清除活性氧的能力受到不同程度的影响。
2.3 盐胁迫对不同花生品种根部丙二醛含量的影响
丙二醛(MDA)是膜脂过氧化的最终产物,它能抑制细胞保护酶的活性和降低抗氧化物的含量,从而加剧膜脂过氧化作用,其值大小可以反映根受到逆境伤害的程度。 在受到逆境伤害时,丙二醛的含量与细胞膜的伤害程度正相关。图2所示,在200mmol/L NaCl处理后,不同耐盐类型花生品种根的丙二醛含量都增加,然而盐敏感型品种D001丙二醛含量增加得多,耐盐品种A025增加较少,说明耐盐性花生根部细胞膜系统受到的损害相对较小,而盐敏感花生根部细胞膜系统受到的损害程度最大,也证明了耐盐性花生品种在受到盐胁迫伤害时植物体内存在更强大的保护作用。

图1 200mmol/L NaCl处理对不同耐盐性花生品种SOD,POD和CAT活性的影响Fig.1 The effects of 200mmol/L NaCl treatment on the activities of SOD,POD and CAT in roots of peanut plants注:图中数据为3个重复的平均值±S.E.。图2同此。Note:Values are means ± SD (n = 3) from one representative of three independent experiments.The same as in fig.2.
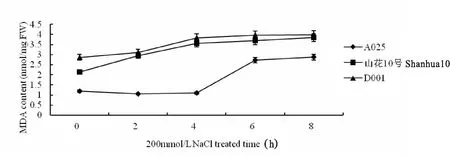
图2 200mmol/L NaCl处理对不同耐盐性花生品种丙二醛(MDA)含量的影响Fig.2 The effects of 200mmol/L NaCl treatment on the content of MDA in roots of peanut plants
2.4 盐胁迫对花生根部miRNA的影响
在200mmol/L NaCl胁迫条件下,随着胁迫时间的延长,miR159-1,miR159-2,miR159-3,miR164-2,miR167-3,miR319-1,miR319-2,miR2111-1及其靶基因的表达量都发生了不同的变化。如图3所示,在200mmol/L NaCl胁迫处理条件下,miR159-1,miR159-3,miR167-3,miR319-1,miR319-2表现为上调表达,其靶基因表达下调,miR159-2,miR164-2表现为表达下调,其靶基因表达上调。
在胁迫处理2h,6h,8h时,miRNA159-1表达量增加比较多,而在4h时,miRNA和靶基因检测表达无明显变化。miRNA159-2及其靶基因的表达受盐胁迫4h和8h时影响较大。在胁迫处理6h,8h时,miRNA159-3表达量增加比较多,耐盐作用更强。miR164-2靶基因1表达没有明显受到调控,靶基因2表达明显上调,但在6h,8h时,miR164-2表达下调较小,而靶基因2表达量却增加比较多,说明靶基因1 miR164-2调控影响不大,靶基因2明显受miR164-2的调控,但也会受到其他miRNA的调控,miRNA与靶基因的关系是一对多、多对多的调控关系。胁迫处理8h时,明显诱导了miR167-3的耐盐作用机制,表现出更强的调控作用。miR319-1的总体表达是上调的,其靶基因表达先下调后上调,二者也无明显规律,推测miR319-1与耐盐胁迫有一定相关性,miR319-1与其靶基因的关系也不是一对一的调控关系。miR319-2的表达受盐胁迫诱导,2h时最为敏感,在短暂的时间内就启动了耐盐调控机制。
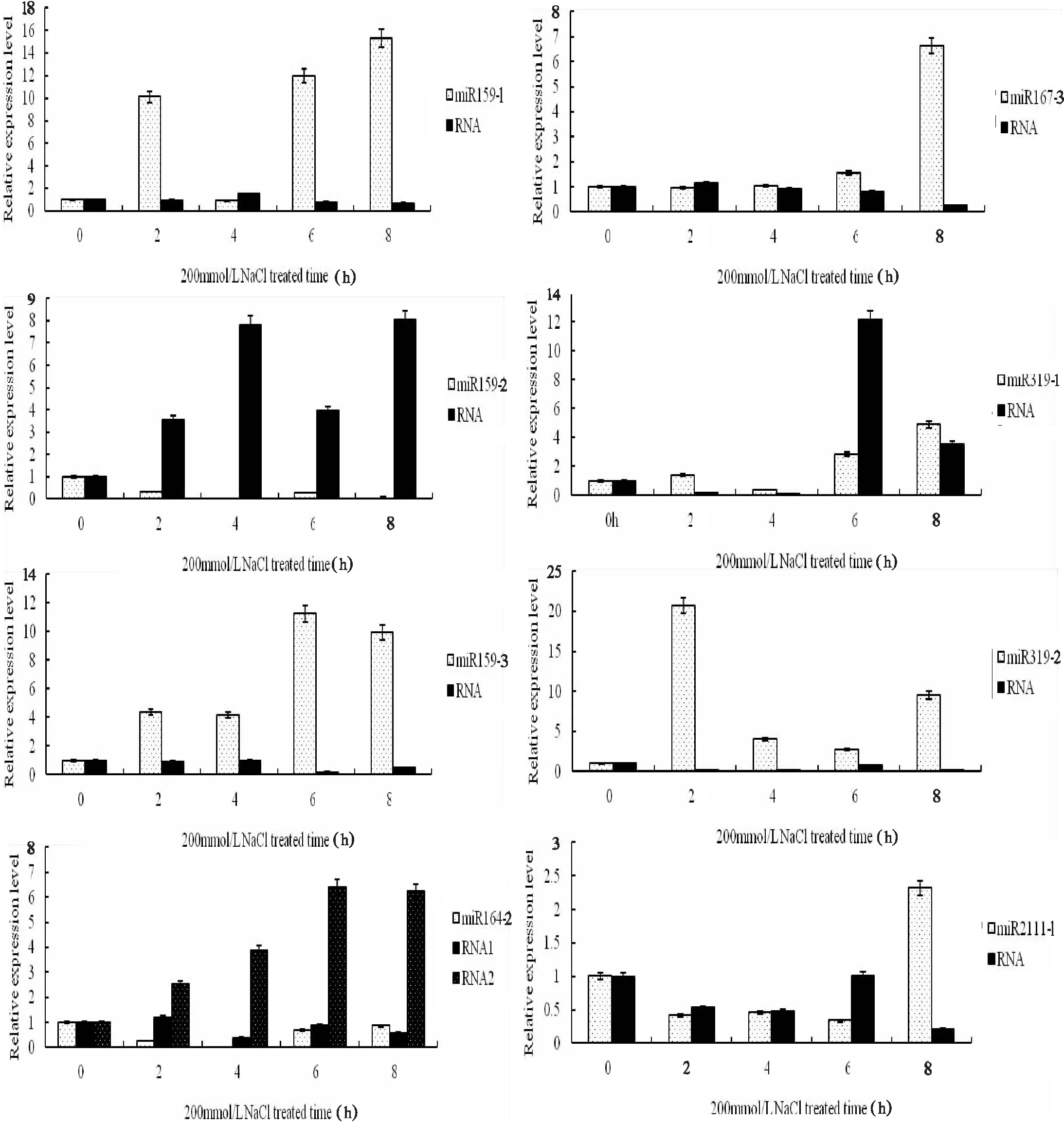
图3 山花10号在200mmol/L NaCl处理下,根部miR159-1,miR159-2,miR159-3,miR164-2,miR167-3,miR319-1,miR319-2,miR2111-1及其靶基因的相对表达量分析Fig.3 Relative expression levels of miR159-1,miR159-2,miR159-3,miR164-2,miR167-3,miR319-1,miR319-2,miR2111-1 and their target genes in peanut Shanhua10 under 200mmol/L NaCl treatment
miR2111-1表达先下调后上调,其靶基因表达也出现相似规律,可见靶基因受miRNA的调控不对应。盐胁迫处理6h以内,miR2111-1表达下调,而在8h时,表达明显增加,靶基因表达明显降低,可能是6h以内,miR2111-1及其靶基因的表达不只是受到盐胁迫诱导,而在8h以后,才明显受到盐胁迫诱导,启动了耐盐调控机制。也可能miR2111-1在花生受到盐胁迫时,并没有明显的关系,具体有待进一步研究确认。后续的研究中,需要研究胁迫8h以后的表达情况,完善数据。
3 讨论与结论
本研究对200份花生品种进行萌发期耐盐性鉴定,获得了高耐盐花生品种3份,中耐盐花生品种5份,盐高敏感品种4份。并对不同耐盐性花生品种在盐胁迫处理条件下进行了抗氧化酶活性(SOD,POD,CAT)和MDA含量的测定,结果表明耐盐花生品种植株的清除活性氧的能力大于盐敏感型。盐胁迫条件下,耐盐性花生根部MDA含量较少,细胞膜系统受到的损害相对较小,而盐敏感植株膜损害程度最大,也证明了耐盐性花生品种在受到盐胁迫伤害时植物体内存在更强大的保护机制。
通过对靶基因序列进行功能分析和同源序列功能检索,获得了miR159-1,miR159-2,miR159-3,miR164-2,miR167-3,miR319-1,miR319-2,miR2111-1 八条花生耐盐相关保守miRNAs。以花生品种山花10号为材料,在200mmol/L NaCl胁迫条件下,随胁迫时间延长,花生根部耐盐相关miRNAs及其靶基因的表达量都发生了不同的变化,也进一步证明了这些花生保守miRNAs在盐胁迫应答反应中的作用特点。与前人研究结果基本一致[13-19]。
钙依赖蛋白激酶(Calcium-dependent protein kinase,CDPKs)是miR167的靶基因之一[27-28]。植物感受到正常生长发育信号和应答外部的逆境胁迫信号后,导致植物细胞质内的Ca2+浓度发生改变,经过特殊的Ca2+信号转导途径,将钙信号逐级放大,并向下游继续传递,使植物表现出不同的生理生化反应。花生在盐碱地不能正常生长的较大影响因素就是Ca2+作用失调,深入研究miR167的作用机制将为提高花生耐盐种质创新提供重要的思路和途径。
[1] 朱统国,高华援,周玉萍,等.花生耐盐性鉴定研究进展[J].中国农学通报,2014,30(21):19-23.
[2] Bartel D P.MicroRNAs:genomics,biogenesis,mechanism,and function [J].Cell,2004,116:281-297.
[3] Palatnik J F,Allen E,Wu X L,et al.Control of leaf morphogenesis by microRNAs [J].Nature,2003,425:257-263.
[4] Lee R C,Feinbaum R L,Ambros V.TheC.elegansheterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14 [J].Cell,1993,75:843-854.
[5] Cheng A M,Byrom M W,Shelton J,et al.Antisense inhibition of human miRNAs and indications for an involvement of miRNA in cell growth and apoptosis [J].Nucleic Acids Research,2005,33:1290-1297.
[6] Reinhart B J,Slack F J,Basson M,et al.The 21-nucleotide let-7 RNA regulates developmental timing inCaenorhabditiselegans[J].Nature,2000,403:901-906.
[7] Pfeffer S,Zavolan M,Grasser F A,et al.Identification of virus-encoded microRNAs [J].Science,2004,304:734-736.
[8] Zhu J Y,Pfuhl T,Motsch N,et al.Identification of novel Epstein-Barr virus microRNA genes from nasopharyngeal carcinomas [J].Journal of Virology,2009,83:3333-3341.
[9] Jones-Rhoades M W,Bartel D P,Bartel B.MicroRNAs and their regulatory roles in plants [J].Annual Review Plant Biology,2006,57:19-53.
[10] Lang Q L,Jin C Z,Lai L Y,et al.Tobacco mircoRNAs prediction and their expression infected with Cucumber mosaic virus and Potato virus X [J].Molecular Biology Reports,2010,38(3):1523-1531.
[11] Khraiwesh B,Zhu J K,Zhu J H.Role of miRNAs and siRNAs in biotic and abiotic stress responses of plants [J].Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms,2012,1819:137-148.
[12] Zhang B H,Pan X P,Wang Q L,et al.Identification and characterization of new plant microRNAs using EST analysis [J].Cell Research,2005,15(5):336-360.
[13] Liu H H,Tian X,Li Y J,et al.Microarray-based analysis of stress-regulated microRNAs inArabidopsisthaliana[J].RNA,2008,14:836-843.
[14] Zhao B T,Liang R Q,Ge L F,et al.Identification of drought-induced microRNAs in rice [J].Biochemical and Biophysical Research Communications,2007,354:585-590.
[15] Zhou L G,Liu Y H,Liu Z C,et al.Genome-wide identification and analysis of drought-responsive microRNAsinOryzasativa[J].Journal of Experimental Botany,2010,61:4157-4168.
[16] Ding D,Zhang L F,Wang H,et al.Differential expression of miRNAs in response to salt stress in maize roots [J].Annuals of Botany,2009,103:29-38.
[17] Jia X Y,Wang W X,Ren L G,et al.Differential and dynamic regulation of miR398 in response to ABA and salt stress inPopulustremulaandArabidopsisthaliana[J].Plant Molecular Biology,2009,71:51-59.
[18] Kantar M,Unver T,Budak H.Regulation of barley miRNAs upon dehydration stress correlated with target gene expression [J].Funct Integr Genomics,2010,10(4):493-507.
[19] Frazier T P,Sun G,Burklew C E,et al.Salt and drought stresses induce the aberrant expression of microRNA genes in tobacco [J].Molecular Biotechnology,2011,49:159-165.
[20] Dong Z H,Shi L,Wang Y W,et al.Identification and dynamic regulation of microRNAs involved in salt stress responses in functional soybean nodules by high-throughput sequencing [J].International Journal of Molecular Sciences,2013,14:2717-2738.
[21] Zhao C Z,Xia H,Frazier T P,et al.Deep sequencing identifies novel and conserved microRNAs in peanuts [J].BMC Plant Biology,2010,10:3.
[22] Zhao C Z,Xia H,Cao T J,et al.Small RNA and degradome deep sequencing reveals peanut microRNA roles in response to pathogen infection [J].Plant Molecular Biology Reporter,2015,33(4):1013-1029.
[23] Li X Y,Bian H W,Song D F,et al.Flowering time control in ornamental gloxinia (Sinningiaspeciosa) by manipulation of miR159 expression [J].Annals of Botany,2013,111:791-799.
[24] Bottino M C,Rosario S,Grativol C,et al.High-throughput sequencing of small RNA transcriptome reveals salt stress regulated microRNAs in Sugarcane [J].PloS ONE,2013,8:e59423.
[25] Zhou M,Li D Y,Li Z G,et al.Constitutive expression of a miR319 gene alters plant development and enhances salt and drought tolerance in transgenic creeping bentgrass [J].Plant Physiology,2013,161:1375-1391.
[26] Xie F L,Zhang B H.Target-align:a tool for plant microRNA target identification [J].Bioinformatics,2010,26(23):3002-3003.
[27] Reddy A S N.Calcium:silver bullet in signaling [J].Plant Science,2001,160:381-404.
[28] Wang J J,Han SF,Li X J,et al.Molecular basis of plant signal transduction mediated by Calcium dependent protein kinase (CDPKs) [J].Acta Prataculturae Sinica.2009,18(3):241-250.
Identification and Functional Characterization of Salt Tolerance Related microRNAs in Roots of Peanut (ArachishypogaeaL.)
ZHANG Ting-ting1,2,YAN Cai-xia1,ZHAO Xiao-bo1,LI Chun-juan1, SHI Cheng-ren1,YU Jiu-jiang3,SHAN Shi-hua1*,LI Rong-gui2*
(1.ShandongPeanutResearchInstitute,Qingdao266100,China; 2.CollegeofLifeScience,QingdaoUniv.,Qingdao266071,China; 3.FoodandNutritionBiotechnologies,Germantown,Maryland13314,USA)
miRNAs as a gene expression regulator plays an important role in the process of plant response to stress.When peanut suffered from salt stress,there will be the corresponding miRNAs involved in the regulation of gene expression responding to stresses.3 salt-tolerant,5 moderate salt-tolerant and 4 high salt-sensitive peanut varieties were screened out from 200 peanut germplasm resources in germination and seedling stage.The results of antioxidase activities and malondialdehyde content indicated that the salt-tolerant plant had stronger ability to eliminate reactive oxygen species.And the salt-tolerant plants suffered less from salt treatment than the salt-sensitive ones,which also showed the self protective response.Through miRNA sequencing and Gene Ontology (GO) functional annotation of target gene sequences,eight salt tolerance related miRNA sequences (miR159-1,miR159-2,miR159-3,miR164-2,miR167-3,miR319-1,miR319-2,miR2111-1) were obtained.qRT-PCR analyses demonstrated that these peanut miRNAs are regulated by salt stress,resulting in appropriate biological responses of their target genes.
peanut; microRNA; target gene; antioxidase activities; salt-tolerance
10.14001/j.issn.1002-4093.2016.04.007
2016-10-27
国家国际科技合作专项项目(2015DFA31190);山东省农业科学院青年科研基金项目(2015YQN07);山东省花生产业技术体系项目(SDAIT-04-02);山东省良种工程项目;青岛市博士后基金项目(2015142)
张廷婷(1981-),女,山东泰安人,山东省花生研究所助理研究员,博士后,主要从事花生抗逆相关miRNAs功能分析研究。
*通讯作者:单世华(1971-),研究员,主要从事花生遗传育种与种质资源研究。E-mail:shhshan@sina.com
S565.2034; Q756
A
李荣贵(1969-),教授,主要从事松萎蔫病分子机制研究。E-mail:lrg@qdu.edu.cn
