一种嗜热菌Pif1解旋酶的表达纯化及活性分析
2016-06-03赵正阳刘娜女李海红奚绪光范三红
赵正阳,刘娜女,李海红,奚绪光,范三红
(西北农林科技大学 生命科学学院,陕西 杨凌 712100)
一种嗜热菌Pif1解旋酶的表达纯化及活性分析
赵正阳,刘娜女,李海红,奚绪光,范三红
(西北农林科技大学 生命科学学院,陕西 杨凌 712100)
[摘要]【目的】 利用大肠杆菌表达纯化嗜热脱铁去硫弧菌(Deferribacter desulfuricans)解旋酶DePif1,并对其结合与解旋DNA的活性进行分析,为Pif1家族解旋酶结构和功能的阐明奠定基础。【方法】 将促溶标签SUMO编码序列和人工合成的DePif1解旋酶编码序列依次连入pET15b载体,获得重组融合表达载体pET15b-SUMO-DePif1,然后将其导入E.coli BL21(DE3)菌株进行诱导表达;利用Ni-NTA亲和层析柱获得融合蛋白,SUMO蛋白酶酶切去除融合标签,再经Heparin和Ni-NTA柱分离获得无标签的纯化重组DePif1蛋白;采用荧光各向异性分析,研究pH和NaCl浓度对DePif1与DNA结合的影响及DePif1与不同底物(单链DNA、双链DNA和G4-DNA)的结合特性;使用基于荧光共振能量转移的stopped-flow技术,分析DePif1对不同底物(G4-DNA with 5′ 26 nt tail和dsDNA with 5′ 26 nt tail)的解旋活性。【结果】 每升菌液可获得9 mg纯度大于95%的DePif1解旋酶。DePif1结合不同DNA底物的强度依次为G4-DNA>单链DNA>双链DNA,其对G4-DNA的解旋活力大于双链DNA。【结论】 成功表达并纯化了嗜热脱铁去硫弧菌Pif1解旋酶,并证明其具有特异的G4-DNA结合和解旋能力。
[关键词]嗜热脱铁去硫弧菌;Pif1解旋酶;G4-DNA;表达纯化;解旋活性
解旋酶是生物体内一类参与几乎所有核酸代谢过程的分子马达,通常能结合并水解ATP,并利用水解释放的能量打开核酸双链间的氢键,为复制、转录、修复和重组等过程提供单链模板或反应中间物[1]。解旋酶编码基因突变常会导致严重遗传病的发生,如人类的Werner、Bloom、Fanconi 综合症等[2-3]。根据保守模体数量和序列差异,解旋酶可分为5个超家族[4]。Pif1家族解旋酶是一类依赖ATP的5′→3′解旋酶,属于解旋酶超家族Ⅰ(SFⅠ)的一个亚家族,广泛存在于原核、真核生物及病毒中。Pif1家族解旋酶均包含一个由300~500个氨基酸残基组成的Pif1结构域,该结构域中包含7个保守模体,在不同物种间高度保守[5-6]。
酿酒酵母的ScPif1是首个被发现的Pif1家族成员,是在研究影响线粒体DNA重组频率的基因时分离得到的[7]。之后的研究证明,ScPif1具有依赖单链DNA的ATP水解活性,并且在ATP和Mg2+存在时,能沿5′→3′方向解旋DNA-DNA或DNA-RNA底物[8-9];除此之外, ScPif1还能解旋G-四联体DNA (G-quadruplex,G4-DNA)结构,并且解旋速度高于其他双链DNA底物[10]。同时,ScPif1参与冈崎片段的加工[11],通过抑制端粒酶的活性来抑制端粒延伸[8,12],这些功能使其在维持染色体和线粒体DNA稳定性方面发挥着重要作用[13]。酿酒酵母中还存在另一个Pif1家族成员ScRrm3,是在筛选rDNA重组相关基因时发现的[14]。 DNA复制过程中,ScRrm3全程随复制叉移动,能帮助复制叉通过结合蛋白或特殊的DNA结构等障碍,并与DNA聚合酶催化亚基ε作用,是复制体的组成元件[15]。酵母双杂交和PULL-DOWN试验证实,Rrm3p可直接与增殖细胞核抗原(Proliferating Cell Nuclear Antigen,PCNA))相互作用[16]。另一个研究较深入的Pif1成员是裂殖酵母(Schizosaccharomycespombe)的Pfh1。截短的Pfh1具有依赖ATP和Mg2+的5′→3′DNA解旋酶活力[17]。当Pfh1缺失或突变时,线粒体DNA迅速丢失并能导致细胞死亡,因此其对维持酵母细胞核及线粒体基因组的完整是必需的[13,17]。
哺乳类基因组中只编码一个Pif1-Like蛋白,序列分析结果显示,人类与小鼠Pif1-Like蛋白(分别为hPif1和mPif1)具有84%的序列相似度[18]。免疫荧光分析显示,hPif1在细胞核和线粒体中均有存在[18]。hPif1在镁离子和单链DNA存在下,能有效地水解ATP,并且以ATP水解为动力,沿5′→3′方向解旋DNA-DNA双链、DNA-RNA杂合链、复制叉状结构和G4-DNA结构[19-21]。在人肿瘤细胞中,过量表达hPif1导致端粒缩短,抑制其过表达则端粒长度恢复。结合hPif1对DNA-RNA杂交链的解链试验、酵母双杂交以及免疫共沉淀试验,研究者认为hPif1通过解旋“端粒酶RNA/端粒DNA”杂合链来抑制端粒酶活性[18-19]。小鼠mPif1在胚胎和造血细胞中表达,虽然可与端粒酶相互作用,但并不抑制端粒酶活性[22]。
富含鸟嘌呤(G,guanine)的核酸序列可以通过4个鸟嘌呤的自组装产生四链DNA结构,即G4-DNA。全基因序列分析表明,潜在的G4-DNA序列不仅存在于真核生物端粒序列中,而且广泛散布在原核和真核基因组中[23-24]。G4-DNA的广泛存在暗示其具有特定的生物学功能。研究表明,基因启动子区的G4-DNA会影响基因的转录,而mRNA中的G4结构则影响蛋白质的翻译。G4-DNA结构的出现会成为复制与转录的障碍,因而需要G4-DNA结构特异的解旋酶。目前已发现的具有G4-DNA解旋活性的酶包括FANCJ、BLM、WRN和Pif1解旋酶。相对于其他DNA底物,Pif1解旋酶对G4-DNA具有更高的结合和解旋活力[10,21]。
研究表明,Pif1解旋酶在不同生物体中,甚至同一生物体的不同组织中具有不同的功能[6]。因此需要对不同来源的Pif1解旋酶进行研究,以更全面地了解Pif1家族的功能特点和作用机理。蛋白与靶DNA晶体的获得与解析对解旋酶功能的阐明具有决定性作用,而嗜热菌蛋白通常具有良好的热稳定性,有利于晶体的形成。已有多种嗜热菌解旋酶获得晶体结构,如DnaB、 RecG 和XPD等[25-27]。本研究以生活在深海热液喷发口的嗜热脱铁去硫弧菌(Deferribacterdesulfuricans)(最适生活温度60~65 ℃)的Pif1蛋白DePif1为对象,利用大肠杆菌系统实现DePif1的高效表达纯化,分析其对不同底物的结合和解旋能力,以期为研究Pif1家族解旋酶的功能及晶体结构奠定基础。
1材料与方法
1.1材料
1.1.1菌株及质粒E.coli2984、BL21(DE3)菌株, pET15b载体及PCR扩增获得的SUMO促溶标签编码序列,均为西北农林科技大学生命科学学院奚绪光教授实验室保存。
1.1.2工具酶及主要试剂NdeⅠ、EcoRⅠ-HF、XhoⅠ等限制性内切酶购自NEB公司,Prime STAR DNA Polymerase、T4 DNA ligase 购自Takara公司,所有试剂均为分析纯,所有缓冲液均由Millipore系统的超纯水配制,Ni-NTA、Heparin Sepharose Fast Flow等层析柱购自GE公司。
1.1.3DNA底物解旋试验中所用的DNA底物两条链分别标记荧光素(fluorescein,F)和六氯荧光素(hexachlorofluorescein,H),而DNA结合的荧光偏振试验中用到的DNA底物只有一条链标记荧光素。制备各种底物的单链DNA购自上海生工生物工程公司,均经过HPLC纯化,具体序列见表1。双链和G4-DNA底物通过单链DNA退火获得,方法是:将单链DNA置于退火缓冲液(20 mmol/L Tris-HCl,100 mmol/L NaCl,pH 8.0)中90 ℃变性3 min,然后自然降温退火。

表 1 结合和解旋试验中所用的DNA底物
注:F.荧光素;H.六氯荧光素。
Note:F.Fluorescein;H.Hexachlorofluorescein.
1.2方法
1.2.1DePif1表达载体的构建将SUMO促溶标签编码序列(上、下游引物中均含NdeⅠ/EcoRⅠ酶切位点)及由Biomatik公司合成的全长DePif1解旋酶编码序列(上、下游均含EcoRⅠ/XhoⅠ酶切位点),分别用NdeⅠ/EcoRⅠ和EcoRⅠ/XhoⅠ双酶切后,连入NdeⅠ/XhoⅠ双酶切并回收的pET15b表达载体,将连接反应产物导入E.coli2984菌株,扩增并鉴定,获得pET15b-SUMO-DePif1重组质粒。
1.2.2DePif1解旋酶在大肠杆菌中的表达纯化用重组质粒pET15b-SUMO-DePif1转化E.coliBL21(DE3)菌株,37 ℃、180 r/min培养至A600约为0.6,加入终浓度为0.3 mmol/L的IPTG,于28 ℃、160 r/min培养6 h,收集菌体,按照1(g)∶8(mL)的比例加入裂解缓冲液Buffer A(50 mmol/L Tris-HCl (pH 8.0),300 mmol/L NaCl,10 mmol/L imidazole,体积分数10% Glycerol)重悬,高压破碎,超声裂解打断DNA分子降低黏度。然后于4 ℃、13 000 r/min离心30 min,在上清中加入等体积4 mol/L硫酸铵溶液,4 ℃搅拌4 h,13 000 r/min离心20 min收集沉淀。用适当体积Buffer A溶解沉淀,透析平衡后载入Ni-NTA亲和层析柱,利用AKTA蛋白纯化仪梯度洗脱获得N端融合有His-tag和SUMO标签的融合蛋白。按照1∶100的质量比加入SUMO蛋白酶, 然后于4 ℃酶切16 h去除蛋白标签。酶切后的反应液载入Heparin Sepharose Fast Flow层析柱,再用AKTA蛋白纯化仪梯度洗脱纯化。收集包含目标蛋白的组分载入Ni-NTA亲和层析柱,则残留的标签蛋白与亲和柱结合,去标签的DePif1存在于穿出液中。使用10% SDS-PAGE检测样品纯度,用nano drop测定蛋白浓度,样品超滤浓缩后-80 ℃冻存待用。
1.2.3DePif1 的DNA结合能力检测采用荧光各向异性(FA)方法对DePif1的DNA 结合活性进行检测[28-29]。荧光标记的核酸底物不与蛋白结合时,高度自由旋转,表现出低的荧光各向异性;而当其与蛋白结合时,形成的复合物转动性降低,各向异性值增加,因此荧光标记的核酸底物的各向异性变化能反映蛋白与底物的结合情况。测定在Infinite F200 Pro型多功能酶标仪(TECAN)上完成,150 μL的反应体系中有20 mmol/L MES,5 nmol/L 荧光标记DNA底物(分别为16 nt-ssDNA、16 bp-dsDNA、G4-DNA),2 mmol/L MgCl2,2 mmol/L DTT,以及不同浓度的DePif1蛋白(0~120 nmol/L)。将反应体系pH分别设置为4.0,5.0,6.0,7.0,8.0和9.0,反应体系中的NaCl浓度分别为20,50,80,100,150和200 mmol/L,振荡混匀,37 ℃温育5 min,然后测定各样品的荧光各向异性值,分析pH和NaCl浓度对DePif1与G4-DNA结合的影响,并比较DePif1与单链DNA、双链DNA和G4-DNA的结合强度。
1.2.4DePif1的DNA解旋活性检测采用基于荧光共振能量转移(FRET)的stopped-flow 技术,对DePif1的DNA解旋活性进行测定[30]。在这种方法中,DNA底物两条链中一条链的3′端标记有荧光素(F,供体),另一条链的5′端标记有六氯荧光素 (HF,受体)。当两条链结合在一起时,荧光素发射的荧光被六氯荧光素淬灭。而当双链DNA被解旋酶分开时,荧光素和六氯荧光素间的能量传递被打破,从而导致荧光素的荧光发射增强。因而DNA链的打开与否可通过荧光信号的变化来监测。
为了选择DePif1和G4-DNA底物在解旋缓冲液中孵育的适宜温度及缓冲液中NaCl的适宜浓度,特进行了筛选试验。(1)孵育温度的筛选。将DePif1和G4-DNA底物(G4-DNA with 5′ 26 nt tail)分别置于40和25 ℃解旋缓冲液中孵育5 min,然后加入1 mmol/L ATP开启解链反应,通过监测激发荧光强度变化来分析解链过程,筛选适宜的孵育温度。
(2)NaCl浓度的筛选。在筛选出的适宜温度下,将缓冲液中NaCl浓度设为20和50 mmol/L,测定DePif1对G4-DNA底物的解旋能力,筛选适宜的NaCl浓度。测定使用Bio-logic SFM-400 混合器和Bio-Logic MOS450/AF-CD光学系统,反应池为1.5 mm×1.5 mm 微量比色皿,反应缓冲液包含20 mmol/L Tris-HCl (pH 7.5),20 mmol/L NaCl,2 mmol/L MgCl2,2 mmol/L DTT。DePif1解旋酶(100 nmol/L)与双链DNA底物(dsDNA with 5′ 26 nt tail,4 nmol/L)或G4-DNA底物(G4-DNA with 5′ 26 nt tail,4 nmol/L)在注射器3中预先于40 ℃ 孵育5 min,ATP(终浓度1 mmol/L)在注射器4中,将2个注射器中的样品快速混合后开始检测荧光强度变化,激发波长为429 nm,发射波长为525 nm。
2结果与分析
2.1DePif1表达载体的构建
将NdeⅠ/EcoRⅠ、EcoRⅠ/XhoⅠ双酶切后的SUMO标签基因(309 bp)和DePif1编码基因(1 533 bp)连入NdeⅠ/XhoⅠ双酶切的pET15b表达载体,获得pET15b-SUMO-DePif1重组质粒。在目标蛋白N-端分别融合有His-tag和SUMO标签,在SUMO标签和DePif1之间引入了SUMO蛋白酶切位点(图1-A)。重组质粒先通过PCR和酶切进行鉴定,最后通过测序确证无误后用于后续试验。图1-B为重组质粒pET15b-SUMO-DePif1的PCR鉴定结果,扩增获得了约1 800 bp片段,其包含了SUMO和DePif1编码区,与预期大小一致。图1-C为重组质粒pET15b-SUMO-DePif1的EcoRⅠ/XhoⅠ双酶切结果,出现的大小约1 500 bp的片段为DePif1编码序列。
2.2DePif1解旋酶的表达纯化
将构建好的重组质粒pET15b-SUMO-DePif1导入大肠杆菌BL21(DE3)进行诱导表达,然后依次通过Ni-NAT柱亲和纯化、SUMO蛋白酶切除标签、Heparin Sepharose Fast Flow及Ni-NTA柱层析去除SUMO蛋白酶和亲和标签等步骤,最终获得无标签的DePif1。纯化过程中各样品的SDS-PAGE分析结果如图2所示。由图2可见,菌体裂解上清液(泳道1)和Ni-NAT亲和纯化样品(泳道2)中均出现70 ku大小的预期条带(DePif1分子质量约为60 ku,SUMO标签约为10 ku),SUMO蛋白酶切后出现预期的60和10 ku 2条条带(泳道3)。再经过Heparin Sepharose Fast Flow和Ni-NTA柱,获得纯度大于95%的DePif1解旋酶,每升菌液可获得9 mg重组DePif1。
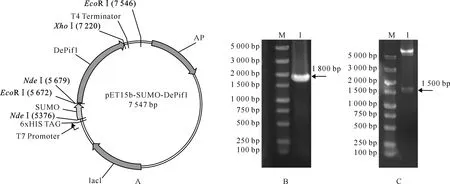
图 1表达载体pET15b-SUMO-DePif1的构建与鉴定
A.重组载体构建示意图;B.重组质粒PCR鉴定;C.重组质粒的双酶切鉴定;
M.DS TM 5000 DNA Marker;1.重组质粒pET15b-SUMO-DePif1
Fig.1Construction of expression vector pET15b-SUMO-DePif1
A.Schematic map of pET15b-SUMO-DePif1;B.Identification of the recombinant vector by PCR;
C.Double digestion of the pET15b-SUMO-DePif1 byEcoRⅠ andXhoⅠ;M.DS TM 5000 DNA Marker;1.pET15b-SUMO-DePif1

图 2DePif1蛋白的SDS-PAGE分析
M.Prestained protein ladder;1.菌体裂解液上清; 2.第1轮
Ni-NTA纯化;3.SUMO蛋白酶裂解; 4~5.Heparin柱纯化;
6.第2轮Ni-NTA纯化;7.浓缩后样品
Fig.2SDS-PAGE analysis of DePif1 protein
M.Prestained protein ladder;1.Supernatant of cell lysates;
2.Purified by first round Ni-NTA column;3.Cleaved by
SUMO protease;4-5.Purified by Heparin column;
6.Purified by second round Ni-NTA column;
7.Concentrated DePif1
2.3DePif1解旋酶与DNA底物的结合活性
首先运用荧光各向异性方法分析DePif1与G4-DNA结合的最适pH和NaCl浓度。由图3-A可见,pH为6.0时,不同浓度DePif1的各向异性值均明显高于其他pH,因而最适pH为6.0。由图3-B可见,当DePif1浓度为20,50,100 nmol/L时,DePif1与DNA的结合活性在NaCl浓度为20或50 mmol/L时差别不大,但显著高于其他NaCl浓度处理,但由于20 mmol/L时DePif1与单链底物结合不稳定,因此选择50 mmol/L NaCl进行试验。根据米氏方程得到DePif1对单链底物(16 nt-ssDNA)、双链底物(16 bp-ds DNA)、G4底物的Km值分别为3.02±0.65,18.82±1.70和2.71±0.57,表明DePif1对不同底物的结合强度依次为G4-DNA>单链DNA>双链DNA(图3-C)。
2.4DePif1的DNA解旋活性
从图4-A可以看出,DePif1在40 ℃时的解旋效率明显优于25 ℃。图4-B显示,在20 mmol/L NaCl作用下,DePif1的解旋速度和幅度均高于50 mmol/L NaCl。因此,将温度设为40 ℃,NaCl浓度设置为20 mmol/L,分析DePif1对双链DNA底物(dsDNA with 5′ 26 nt tail)和G4-DNA底物(G4-DNA with 5′ 26 nt tail)的解旋能力,结果(图4-C)显示,2种底物均能被DePif1解旋,但解旋效率存在较明显的差异。利用双指数拟合方法求得DePif1对双链DNA底物的解旋参数为:Afast=21.97%,kfast=0.724 6 s-1,Aslow=27.72%,kslow=0.216 5 s-1;对G4-DNA底物的解旋参数为:Afast=79.54%,kfast=0.543 8 s-1,Aslow=12.07%,kslow=0.099 6 s-1;A代表解旋幅度,fast、slow分别指解旋过程中的快反应和慢反应步骤,k指速率常数。因此,DePif1对G4-DNA的解旋活性高于双链DNA。

图 3DePif1与DNA底物结合的条件与强度
A.pH对DePif1与G4-DNA结合的影响;B.NaCl浓度对DePif1与G4-DNA结合的影响;C.DePif1与不同底物结合强度的比较
Fig.3Binding conditions and strengths of DePif1 with DNA substrates
A.Effects of pH on binding of DePif1 with G4-DNA;B.Effects of NaCl concentration on binding of DePif1 with G4-DNA;
C.Comparison of binding strengths of DePif1 with different substrates
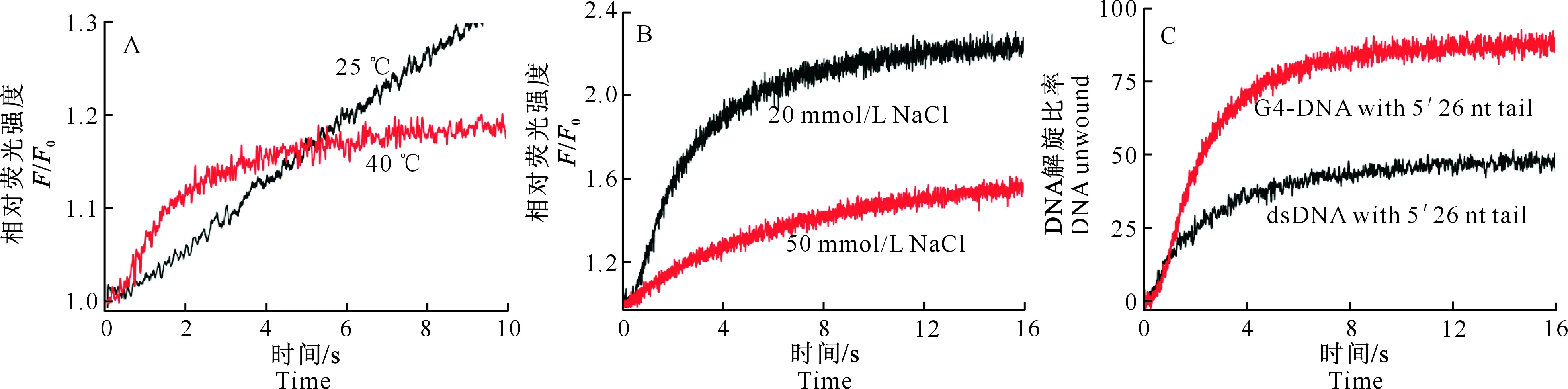
图 4DePif1 DNA解旋活性的FRET分析
A.温度对DePif1解旋活性的影响;B.NaCl浓度对DePif1解旋活性的影响;C.DePif1对 G4-DNA和
双链DNA底物的解旋活性比较;F0为底物自身荧光强度,F为加入DePif1后的实时荧光强度
Fig.4DNA unwinding activity of DePif1 by FRET
A.Effects of temperature on unwinding activity of DePif1;B.Effects of NaCl concentration on the unwinding activity of DePif1;
C.Comparison of unwinding activities of DePif1 to G4-DNA and duplex DNA;FandF0.The fluorescence
intensities of DNA substrates in the presence and absence of DePif1,respectively
3讨论
Pif1家族成员具有依赖ATP的5′→3′解旋活性,其可影响端粒、rDNA及线粒体DNA的复制,参与冈崎片段加工,在维持染色体和线粒体DNA稳定性方面发挥重要功能[6]。本研究建立了一种嗜热脱铁去硫弧菌(Deferribacterdesulfuricans)Pif1解旋酶(DePif1)的表达纯化流程,利用该流程每升菌液可获得9 mg纯度大于95%且具有生物活性的DePif1蛋白,这为后期DePif1解旋酶晶体的获得奠定了基础。为了使目标蛋白在大肠杆菌中更好地表达,本研究对DePif1的编码序列分析后,利用人工合成的方法,在不影响蛋白序列的前提下对DePif1的编码序列进行了修饰,优化了序列中的部分密码子,并通过序列优化降低了其转录产物的复杂二级结构对表达的不利影响。在表达过程中,目标蛋白N-端融合了His-tag和SUMO促溶标签,SUMO标签可以增加融合蛋白的可溶性,而His-tag便于融合蛋白的纯化,以及SUMO蛋白酶切后SUMO标签的去除。纯化过程中还引入了硫酸铵沉淀、复溶步骤,这有利于核酸、酯类等杂质的去除,避免对后续结合和解旋试验产生影响。
荧光各向异性(FA)是一种快速、灵敏的监测分子运动及其影响因素的方法,监测浓度可低至“nmol/L”级别[28-29]。本研究采用荧光各向异性法,确定了DePif1与G4-DNA底物结合的最适pH为6.0,最适NaCl浓度为20 mmol/L。但NaCl浓度为20 mmol/L时,DePif1与单链DNA结合不稳定。NaCl浓度为50 mmol/L时,DePif1对不同底物的结合强度顺序为G4-DNA>单链DNA>双链DNA。相较于传统的EMSA方法,基于荧光共振能量转移(FRET)的stopped-flow检测技术,能够在低浓度核酸条件下实时监测蛋白与核酸的反应过程。本研究采用此方法,对DePif1的解链活性进行了分析,结果表明在ATP和5′-单链存在的条件下,DePif1能有效解旋双链DNA和G4结构,且解旋G4结构的能力高于双链DNA。此外,DePif1在40 ℃时的解链活性高于25 ℃,这与其来自嗜热菌的实际相符。综上所述,DePif1结合和解旋G4-DNA的能力高于其他底物,而DePif1本身又是易结晶的嗜热菌蛋白,因而适用于解旋酶/G4-DNA共结晶研究。
[参考文献]
[1]Matson S W,Bean D W,George J W.DNA helicases:Enzymes with essential roles in all aspects of DNA metabolism [J].Bioessays,1994,16(1):13-22.
[2]Ellis N A,Groden J,Ye T Z,et al.The Bloom’s syndrome gene product is homologous to RecQ helicases [J].Cell,1995,83(4):655-666.
[3]Gray M D,Shen J C,Kamath-Loeb A S,et al.The Werner syndrome protein is a DNA helicase [J].Nature Genetics,1997,17(1):100-103.
[4]Caruthers J M,McKay D B.Helicase structure and mechanism [J].Current Opinion in Structural Biology,2002,12(1):123-133.
[5]Bessler J B,Zakian V A.The Pif1p subfamily of helicases:Region-specific DNA helicases [J].Trends in Cell Biology,2001,11(2):60-65.
[6]Bochman M L,Sabouri N,Zakian V A.Unwinding the functions of the Pif1 family helicases [J].DNA Repair,2010,9(3):237-249.
[7]Foury F,Kolodynski J.Pif mutation blocks recombination between mitochondrial rho+ and rho-genomes having tandemly arrayed repeat units inSaccharomycescerevisiae[J].Proceedings of the National Academy of Sciences,1983,80(17):5345-5349.
[8]Zhou J Q,Monson E,Teng S C,et al.Pif1p helicase,a catalytic inhibitor of telomerase in yeast [J].Science,2000,289(5480):771-774.
[9]Boulé J B,Zakian V A.The yeast Pif1p DNA helicase preferentially unwinds RNA-DNA substrates [J].Nucleic Acids Research,2007,35(17):5809-5818.
[10]Ribeyre C,Lopes J,Boulé J B,et al.The yeast Pif1 helicase prevents genomic instability caused by G-quadruplex-forming CEB1 sequencesinvivo[J].PLoS Genetics,2009,5(5):e1000475.
[11]Budd M E,Reis C C,Smith S,et al.Evidence suggesting that Pif1 helicase functions in DNA replication with the Dna2 helicase/nuclease and DNA polymerase δ [J].Molecular and Cellular Biology,2006,26(7):2490-2500.
[12]Schulz V P,Zakian V A.TheSaccharomycesPIF1 DNA helicase inhibits telomere elongation and de novo telomere formation [J].Cell,1994,76(1):145-155.
[13]Pinter S F,Aubert S D,Zakian V A.TheSchizosaccharomycespombePfh1p DNA helicase is essential for the maintenance of nuclear and mitochondrial DNA [J].Molecular and Cellular Biology,2008,28(21):6594-6608.
[14]Keil R L,McWilliams A D.A gene with specific and global effects on recombination of sequences from tandemly repeated genes inSaccharomycescerevisiae[J].Genetics,1993,135(3):711-718.
[15]Azvolinsky A,Dunaway S,Torres J Z,et al.TheS.cerevisiaeRrm3p DNA helicase moves with the replication fork and affects replication of all yeast chromosomes [J].Genes & Development,2006,20(22):3104-3116.
[16]Schmidt K H,Derry K L,Kolodner R D.SaccharomycescerevisiaeRRM3,a 5′ to 3′ DNA helicase,physically interacts with proliferating cell nuclear antigen [J].Journal of Biological Chemistry,2002,277(47):45331-45337.
[17]Zhou J Q,Qi H,Schulz V P,et al.Schizosaccharomycespom-bepfh1+Encodes an essential 5′ to 3′ DNA helicase that is a member of the PIF1 subfamily of DNA helicases [J].Molecular Biology of the Cell,2002,13(6):2180-2191.
[18]Mateyak M K,Zakian V A.Human PIF helicase is cell cycle regulated and associates with telomerase [J].Cell Cycle,2006,5(23):2796-2804.
[19]Zhang D H,Zhou B,Huang Y,et al.The human Pif1 helicase,a potentialEscherichiacoliRecD homologue,inhibits telomerase activity [J].Nucleic Acids Research,2006,34(5):1393-1404.
[20]George T,Wen Q,Griffiths R,et al.Human Pif1 helicase unwinds synthetic DNA structures resembling stalled DNA replication forks [J].Nucleic Acids Research,2009,37(19):6491-6502.
[21]Sanders C.Human Pif1 helicase is a G-quadruplex DNA-binding protein with G-quadruplex DNA-unwinding activity [J].Biochem J,2010,430:119-128.
[22]Snow B E,Mateyak M,Paderova J,et al.Murine Pif1 interacts with telomerase and is dispensable for telomere functioninvivo[J].Molecular and Cellular Biology,2007,27(3):1017-1026.
[23]Rawal P,Kummarasetti V B R,Ravindran J,et al.Genome-wide prediction of G4 DNA as regulatory motifs:Role inEscherichiacoliglobal regulation [J].Genome Research,2006,16(5):644-655.
[24]Johnson J E,Smith J S,Kozak M L,et al.Invivoveritas:Using yeast to probe the biological functions of G-quadruplexes [J].Biochimie,2008,90(8):1250-1263.
[25]Singleton M R,Scaife S,Raven N D,et al.Crystallization and preliminary X-ray analysis of RecG,a replication-fork reversal helicase fromThermotogamaritimacomplexed with a three-way DNA junction [J].Acta Crystallographica Section D:Biological Crystallography,2001,57(11):1695-1696.
[26]Bailey S,Eliason W K,Steitz T A.The crystal structure of theThermusaquaticusDnaB helicase monomer [J].Nucleic Acids Research,2007,35(14):4728-4736.
[27]Fan L,Fuss J O,Cheng Q J,et al.XPD helicase structures and activities:Insights into the cancer and aging phenotypes from XPD mutations [J].Cell,2008,133(5):789-800.
[28]Dou S X,Wang P Y,Xu H Q,et al.The DNA binding properties of theEscherichiacoliRecQ helicase [J].Journal of Biological Chemistry,2004,279(8):6354-6363.
[29]Xi X G,Deprez E.Monitoring helicase-catalyzed DNA unwinding by fluorescence anisotropy and fluorescence cross-correlation spectroscopy [J].Methods,2010,51(3):289-294.
[30]Zhang X D,Dou S X,Xie P,et al.RecQ helicase-catalyzed DNA unwinding detected by fluorescence resonance energy transfer [J].Acta Biochimica et Biophysica Sinica,2005,37(9):593-600.
Expression,purification and activity analysis of Pif1 helicase from thermophileDeferribacterdesulfuricans
ZHAO Zheng-yang,LIU Na-nü,LI Hai-hong,XI Xu-guang,FAN San-hong
(CollegeofLifeSciences,NorthwestA&FUniversity,Yangling,Shaanxi712100,China)
Abstract:【Objective】 This study expressed and purified the Pif1 helicase of thermophile Deferribacter desulfuricans in E.coli and analyzed its DNA binding and unwinding activity to lay foundation for the elucidation of the structure and function of Pif1 family helicases.【Method】 Based on the expression vector pET15b,a recombinant plasmid pET15b-SUMO-DePif1 was constructed by cloning the SUMO coding sequence and the synthetic DePif1 gene into the downstream of its His-tag.The recombinant plasmid was transformed into E.coli strain BL21(DE3) and the fusion DePif1 was induced by IPTG.Firstly,DePif1 with His6-SUMO tag from the supernatant of cell lysates was captured by affinity chromatography with a Ni-NTA column.Then,the fusion tag was cleaved by SUMO protease and tag-free DePif1 helicase was obtained by further purification with Heparin Sepharose Fast Flow and Ni-NTA chromatography.Finally,the effects of pH and NaCl concentrations on DNA binding,the characteristics of DePif1 binding with different substrates (single-stranded DNA,double-stranded DNA,and G4-DNA) and unwinding activities of DePif1 against different substrates (G4-DNA with 5′ 26 nt tail and ds DNA with 5′ 26 nt tail) were analyzed by fluorescence anisotropy and stopped-flow based FRET.【Result】 According to the above protocol,9 mg DePif1 with >95% purity was obtained for every liter of broth.DePif1 can bind with different DNA substrates with the binding activity in decreasing order of G4-DNA>ss-DNA>ds-DNA.Its unwinding activity to G4-DNA was higher than to ds-DNA. 【Conclusion】 DePif1 helicase was expressed and purified successfully.DePif1 can bind and unwind G4-DNA specially.
Key words:Deferribacter desulfuricans;Pif1 helicase;G4-DNA;expression and purification;activity analysis
[文章编号]1671-9387(2016)01-0169-08
[中图分类号]Q71
[文献标志码]A
[作者简介]赵正阳(1989-),男,河南洛阳人,在读硕士,主要从事生物大分子结构与功能研究。E-mail:yangokay@yeah.net[通信作者]范三红(1971-),男,陕西合阳人,副教授,博士,硕士生导师,主要从事生物化学与生物信息学研究。
[基金项目]国家自然科学基金项目(31370798,11304252)
[收稿日期]2014-04-25
DOI:网络出版时间:2015-12-0214:2510.13207/j.cnki.jnwafu.2016.01.025
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20151202.1425.050.html
E-mail:shfan@nwsuaf.edu.cn
