基于天然氨基酸的蛋白质修饰
2016-06-02侯颖钦北京大学化学与分子工程学院北京100871
侯颖钦 吕 华*(北京大学化学与分子工程学院,北京 100871)
基于天然氨基酸的蛋白质修饰
侯颖钦吕 华*
(北京大学化学与分子工程学院,北京 100871)
摘要:基于天然氨基酸的蛋白质修饰种类繁多,由于在不同的氨基酸上所进行的修饰会对蛋白质的结构和功能产生不同的影响,所以科学家们一直在探索基于天然氨基酸特异的蛋白质修饰策略。本文主要以半胱氨酸、酪氨酸和蛋白质氮端特异修饰为例,简要回顾目前基于天然氨基酸的蛋白质化学的相关工作。
关键词:蛋白质修饰;天然氨基酸;半胱氨酸;酪氨酸
1 背景介绍
对蛋白质进行化学修饰可以带来新的性质和功能,从而调控蛋白的生物活性。而事实上,蛋白质在生物体内的翻译后修饰是十分常见的现象,能极大地增加蛋白质的结构和功能的多样性。比如蛋白特定氨基酸的乙酰化、甲酰化、磷酸化、磺酸化、泛素化和糖基化等,可以影响细胞的信号传导、分化、迁移等活动[1]。因此,发展正交的化学反应对蛋白质中的天然氨基酸进行特异性修饰,具有重要的研究意义[2]。 由于蛋白质含有多种不同的氨基酸侧链官能团,为了保证修饰的特异性,这些反应必须具有较好的官能团选择性。此外,由于蛋白质多级结构的不稳定,对它们的修饰必须同时保证蛋白质的结构和功能不受破坏和影响。因此,在接近生理环境条件(≤37 ℃, pH 6–8,μmol·L–1浓度)下即可以进行的高效率、高特异性和高转化率的化学反应成为了此类反应的首选[3,4]。
本文主要以半胱氨酸、酪氨酸和蛋白质氮端氨基酸为例,总结近年来天然氨基酸特异性的蛋白质修饰,并对该研究领域的发展方向做出展望。
2 天然氨基酸特异性的蛋白质修饰
在蛋白质的氨基酸序列中,虽然半胱氨酸在数量上所占比例并不高,但是由于其具有显著的亲核性和氧化还原性,易于修饰,被广泛应用于蛋白的选择性修饰。
基于半胱氨酸的蛋白质修饰较为常用的方法包括二硫键的交换反应(图1a)[5]、烷基化(图1b)[5]以及与马来酰亚胺(图1c)[6]或乙烯基砜(图1d)[7,8]的麦克尔加成反应。Brik等利用二硫键的交换反应对α-球蛋白中的半胱氨酸进行修饰,在较为温和的条件(pH 7.0)下,1 h即可达到42%的产率,但是由于该反应可逆,所以转化产率难以提高,并且新生成的二硫键在还原性环境中较易被破坏,从而降低蛋白质修饰产物的稳定性。因此,Brik等又利用α-卤代羰基化合物(如碘乙酰胺等)与α-球蛋白中唯一的半胱氨酸反应,对其进行修饰以提高蛋白质修饰产物的稳定性。此外,马来酰亚胺和乙烯基砜与巯基的麦克尔加成反应是非常经典的修饰巯基的反应。目前,很多市售的巯基反应活性的荧光染料为马来酰亚胺修饰。所以,这也是对蛋白质中半胱氨酸修饰选择性较好的修饰方法。

图1 基于半胱氨酸的蛋白质修饰的经典反应
其他基于半胱氨酸的蛋白质修饰如图2、图3所示。根据所利用的化学反应机理不同分为极性反应和光催化反应。
Baker小组和Caddick小组研究了半胱氨酸与溴代马来酰亚胺的极性反应,分别通过半胱氨酸的可逆烷基化(图2a, 2b)和不可逆烷基化(图2c)对蛋白质进行修饰,不仅可以用于体外的蛋白质反应[9,10],还可以在活细胞中对蛋白质进行修饰[11]。而后,Caddick小组[12]通过两步反应利用2, 5-二溴代己二酰胺与半胱氨酸在较为温和的条件下(21–37 ℃)反应得到了较高的转化率(>95%)。半胱氨酸也可以与缺电子的烯丙基酰胺/酯或者烯丙基酮等反应生成乙烯基硫化物(图2d),这类反应速度快,生物正交性好。 Che等[13]利用带有铱配合物的烯丙基酰胺与半胱氨酸的反应对BSA(半胱氨酸个数多达70个,其中约一半左右的半胱氨酸互相形成二硫键)和HSA(含有72个半胱氨酸,其中32个互相形成二硫键)成功修饰,形成蛋白-一铱配合物杂化体,可以通过磷光实现生物成像。Wagner等[14]则研究了图2e中APN与半胱氨酸的反应,发现这类反应不仅反应速度快,并且生成的产物稳定性好,且在反应过程中对半胱氨酸具有良好的选择性。
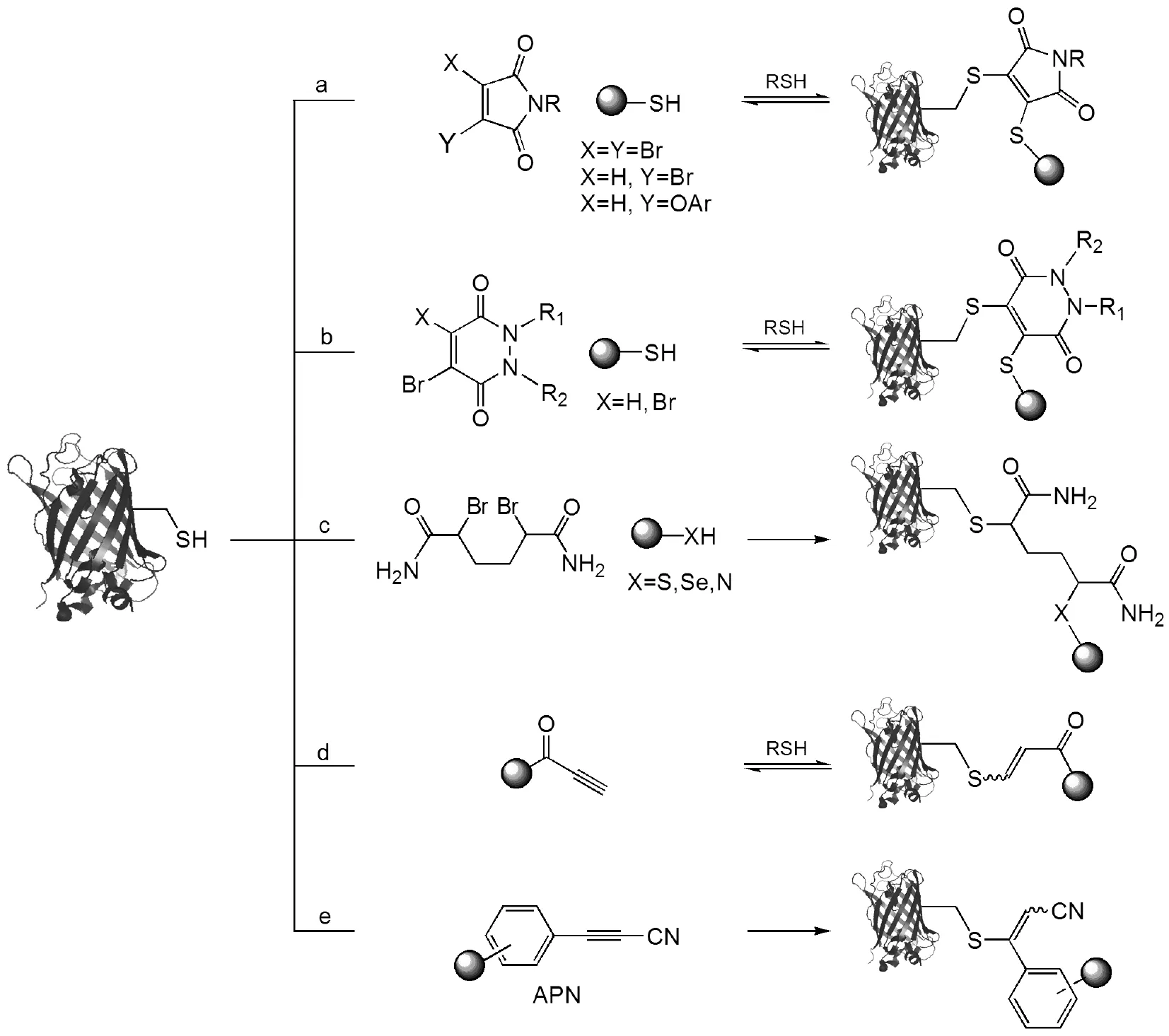
图2 基于半胱氨酸的蛋白质修饰的极性反应

图3 基于半胱氨酸的蛋白质修饰的光催化反应
如图3所示,在光催化反应中,包括3种反应,其中巯基-烯(thiol-ene)反应最常用到。利用thiolene[15–17]或巯基-炔(thiol-yne)[18]反应与蛋白质的半胱氨酸进行偶联时需要有自由基引发剂或者光激发反应。Liu等[16]在利用thiol-ene反应对蛋白进行乙酰化标记时通过蛋白质的溶解性研究表明,thiolene反应过程中自由基的产生并没有导致蛋白质变性,而Strieter等[17]在利用thiol-ene反应研究泛素的不同数目聚体的功能与结构时也发现,thiol-ene反应本身对泛素的结构与功能并没有太大影响,而是在不同位置和取向的偶联体对泛素在蛋白质水解过程中的作用会有较大影响。因此,可以推测自由基的产生对蛋白质本身的结构与性质并没有非常重要的影响。与thiol-ene蛋白质修饰方法相比,thiolyne反应可以进行两次修饰,因此可以分别引入两种不同的官能团,从而对蛋白质进行双官能团化修饰。
基于半胱氨酸的蛋白质修饰方法种类丰富,但是被大家广泛应用的是半胱氨酸与马来酰亚胺的反应和thiol-ene反应。由于绝大多数蛋白质序列中均含有半胱氨酸,所以该修饰方法适用范围广泛。但是,半胱氨酸在蛋白质中往往会形成二硫键,当在半胱氨酸上进行修饰时很可能会对蛋白质的结构或性质有一定的影响。所以科学家们也在积极探索在其他氨基酸上进行修饰。
2.2基于酪氨酸的蛋白质修饰
加州大学Berkeley分校的Francis小组和斯克里普斯研究所的Barbas小组在基于酪氨酸的特异性蛋白质标记方面做出了很多工作(图4)。Barbas和Francis等小组[19–21]利用带有吸电子基团的重氮化合物与酪氨酸酚羟基的邻位反应生成偶氮化合物,反应速度很快(<4 h),条件温和(pH 9.0, 4 ℃即可反应),并且对酪氨酸的选择性较好(图4a)。2004年,Francis小组[22]提出了图4b所示的Mannich类型的酪氨酸修饰方法。这种方法反应效率很高,并且条件温和,三组分反应使其能够引入多种官能团。2011年,Francis等[23]又利用硝酸铈铵作为催化剂实现了对酪氨酸的选择性修饰,反应速度更快(<1 h),且需要的底物浓度也比较低(图4c)。关于酪氨酸的选择性修饰还有酪氨酸酚羟基邻位和双键的反应等,反应效率均比较好[24]。

图4 基于酪氨酸的蛋白质修饰
基于酪氨酸的蛋白质修饰反应选择性较好,而且主要利用的是侧链酚环的富电子特性,所以从反应机理上来看基本一致,反应类型比较单一。
2.3基于天然氨基酸的蛋白质N-端特异性修饰
基于蛋白质N-端氨基酸的修饰主要分为两类,一类以氨基修饰为主(图5a, 5b)[25–27],这类反应通常对氨基酸种类的选择性不大,虽然N-端的氨基和赖氨酸中ε-氨基的pKa不同,通过控制反应体系的pH可以使其对氨基有一定的选择性,但是仍然不能做到完全选择性,例如图5a为Wong和Che等[26]的研究工作,利用烯酮化合物与氨基的反应对蛋白质的N-端进行修饰,基本上只要N-端有游离的氨基即可进行修饰。此外,可以通过串联反应或者利用反应本身对N-端氨基酸的选择性来实现N-端氨基的选择性修饰。如Francis等则利用磷酸吡哆醛将蛋白质N-端氧化成双羰基酮或醛,再与胺氧反应进行修饰,在pH 7.5的条件下,数小时(4 h)内就可以得到较高的转化效率[25],而后其又将磷酸吡哆醛换为具有同样作用的两个分子(称为Rapoport's盐),对谷氨酸有一定的选择性[27]。另一类则有一定的氨基酸选择性,例如图5c、5d所示, Neri等[28]利用醛与N-端半胱氨酸的成环反应将带有醛基的西马多丁与N-端带有半胱氨酸的单克隆抗体F8进行偶联以实现药物分子的靶向性输运。Francis等[29]则利用金属配合物作为催化剂,对N-端的脯氨酸进行修饰,如图5d所示,反应条件比较温和,选择性很好并且反应效率很高。
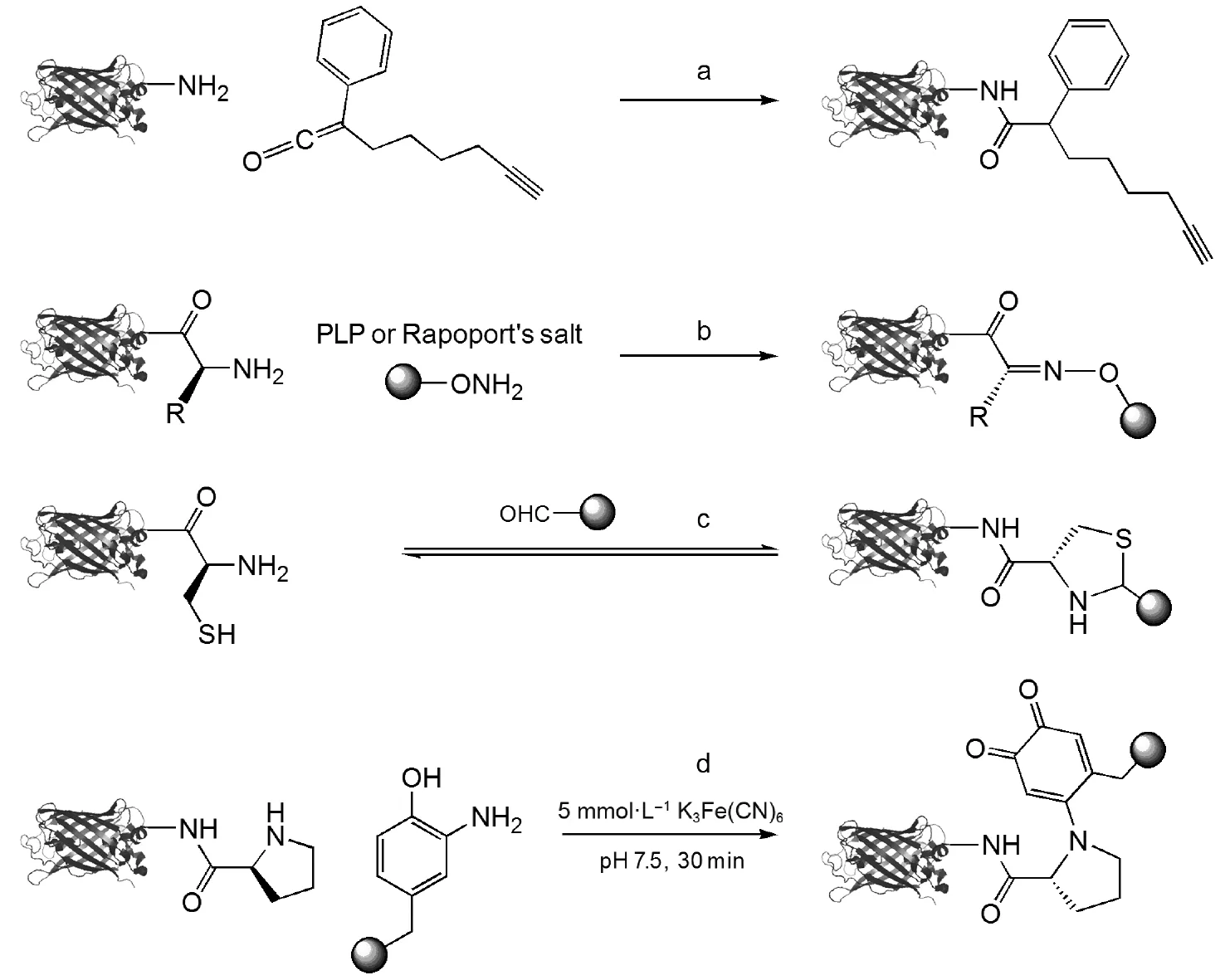
图5 基于天然氨基酸的蛋白质N-端修饰
2.4基于其他天然氨基酸的蛋白质修饰
赖氨酸是最传统的用于蛋白质修饰的氨基酸,然而由于蛋白质上的赖氨酸数目众多,这种方法通常无法很好地控制修饰的位点和数目,容易对蛋白质结构和功能带来影响。Barbas等[30]发展了基于催化抗体 (catalytic antibody)的赖氨酸修饰方法,但是并不具备普适性。基于其他天然氨基酸修饰的方法研究较少,目前有关于丝氨酸[31]、色氨酸[32]等的修饰方法,但是较为单一,并且应用较少。
3 总结与展望
基于天然氨基酸的蛋白质修饰在最小程度上避免了对蛋白质本身结构的改变,以期最大程度利用蛋白质本身的结构与性质,并且通过修饰基团对蛋白质的性质进行改善和提高。虽然这种利用蛋白质本身具有的氨基酸进行蛋白质修饰的方法简单快速,但却面临着选择性的问题。氨基酸选择性和修饰位点与数量的选择性一直是这类修饰方法所面临的挑战与问题,所以在蛋白质的天然氨基酸修饰方面科学家们将继续致力于选择性修饰的研究,并且近年来已经有很多利用非天然氨基酸进行蛋白质修饰的方法产生[4,33,34],这将为蛋白质修饰带来更多的选择策略。
参 考 文 献
[1]Walsh, C. T.; Garneau-Tsodikova, S.; Gatto, G. J., Jr. Angew. Chem. Int. Edit. 2005, 44 (45), 7342.
[2]Stephanopoulos, N.; Francis, M. B. Nat. Chem. Biol. 2011, 7 (12), 876.
[3]Sletten, E. M.; Bertozzi, C. R. Angew. Chem. Int. Edit. 2009, 48 (38), 6974.
[4]Boutureira, O.; Bernardes, G. J. Chem. Rev. 2015, 115 (5), 2174.
[5]Hemantha, H. P.; Bavikar, S. N.; Herman-Bachinsky, Y.; Haj-Yahya, N.; Bondalapati, S.; Ciechanover, A.; Brik, A. J. Am. Chem. Soc. 2014, 136 (6), 2665.
[6]Massa, S.; Xavier, C.; De Vos, J.; Caveliers, V.; Lahoutte, T.; Muyldermans, S.; Devoogdt, N. Bio. Chem. 2014, 25 (5), 979.
[7]Morales-Sanfrutos, J.; Lopez-Jaramillo, F. J.; Hernandez-Mateo, F.; Santoyo-Gonzalez, F. J. Org. Chem. 2010, 75 (12), 4039.
[8]Morales-Sanfrutos, J.; Lopez-Jaramillo, F. J.; Ortega-Munoz, M.; Megia-Fernandez, A.; Perez-Balderas, F.; Hernandez-Mateo, F.; Santoyo-Gonzalez, F. Org. & Biomol. Chem. 2010, 8 (3), 667.
[9]Nathani, R. I.; Chudasama, V.; Ryan, C. P.; Moody, P. R.; Morgan, R. E.; Fitzmaurice, R. J.; Smith, M. E.; Baker, J. R.; Caddick, S. Org. Biomol. Chem. 2013, 11 (15), 2408.
[10]Smith, M. E.; Schumacher, F. F.; Ryan, C. P.; Tedaldi, L. M.; Papaioannou, D.; Waksman, G.; Caddick, S.; Baker, J. R. J. Am. Chem. Soc. 2010, 132 (6), 1960.
[11]Moody, P.; Smith, M. E.; Ryan, C. P.; Chudasama, V.; Baker, J. R.; Molloy, J.; Caddick, S. ChemBioChem 2012, 13 (1), 39.
[12]Nathani, R.; Moody, P.; Smith, M. E.; Fitzmaurice, R. J.; Caddick, S. ChemBioChem 2012, 13, 1283.
[13]Shiu, H. Y.; Chong, H. C.; Leung, Y. C.; Zou, T.; Che, C. M. Chem. Commun. 2014, 50 (33), 4375.
[14]Koniev, O.; Leriche, G.; Nothisen, M.; Remy, J. S.; Strub, J. M.; Schaeffer-Reiss, C.; Van Dorsselaer, A.; Baati, R.; Wagner, A. Bioconjug. Chem. 2014, 25 (2), 202.
[15]Chalker, J. M.; Bernardes, G. J.; Davis, B. G. Acc. Chem. Res. 2011, 44 (9), 730.
[16]Li, F.; Allahverdi, A.; Yang, R.; Lua, G. B.; Zhang, X.; Cao, Y.; Korolev, N.; Nordenskiold, L.; Liu, C. F. Angew. Chem. Int. Edit. 2011, 50 (41), 9611.
[17]Valkevich, E. M.; Guenette, R. G.; Sanchez, N. A.; Chen, Y. C.; Ge, Y.; Strieter, E. R. J. Am. Chem. Soc. 2012, 134 (16), 6916.
[18]Lo Conte, M.; Grotto, D.; Chambery, A.; Dondoni, A.; Marra, A. Chem. Commun. 2011, 47 (4), 1240.
[19]Gavrilyuk, J.; Ban, H.; Nagano, M.; Hakamata, W.; Barbas, C. F., III. Bioconjug. Chem. 2012, 23 (12), 2321.
[20]Hooker, J. M.; Kovacs, E. W.; Francis, M. B. J. Am. Chem. Soc. 2004, 126 (12), 3718.
[21]Schlick, T. L.; Ding, Z.; Kovacs, E. W.; Francis, M. B. J. Am. Chem. Soc. 2005, 127 (11), 3718.
[22]Joshi, N. S.; Whitaker, L. R.; Francis, M. B. J. Am. Chem. Soc. 2004, 126 (49), 15942.
[23]Seim, K. L.; Obermeyer, A. C.; Francis, M. B. J. Am. Chem. Soc. 2011, 133 (42), 16970.
[24]Guo, H. M.; Minakawa, M.; Ueno, L.; Tanaka, F. Bioorg. Med. Chem. Lett. 2009, 19 (4), 1210.
[25]Gilmore, J. M.; Scheck, R. A.; Esser-Kahn, A. P.; Joshi, N. S.; Francis, M. B. Angew. Chem. Int. Edit. 2006, 45 (32), 5307.
[26]Chan, A. O.; Ho, C. M.; Chong, H. C.; Leung, Y. C.; Huang, J. S.; Wong, M. K.; Che, C. M. J. Am. Chem. Soc. 2012, 134 (5), 2589.
[27]Witus, L. S.; Netirojjanakul, C.; Palla, K. S.; Muehl, E. M.; Weng, C. H.; Iavarone, A. T.; Francis, M. B. J. Am. Chem. Soc. 2013, 135 (45), 17223.
[28]Casi, G.; Huguenin-Dezot, N.; Zuberbuhler, K.; Scheuermann, J.; Neri, D. J. Am. Chem. Soc. 2012, 134 (13), 5887.
[29]Obermeyer, A. C.; Jarman, J. B.; Francis, M. B. J. Am. Chem. Soc. 2014, 136 (27), 9572.
[30]Jürgen Wagner, R. A. L.; Barbas, C. F., III. Science 1995, 270, 1797.
[31]Shannon, D. A.; Weerapana, E. Curr. Opin. Chem. Biol. 2015, 24, 18.
[32]Antos, J. M.; Francis, M. B. J. Am. Chem. Soc. 2004, 126 (33), 10256.
[33]Dumas, A.; Spicer, C. D.; Gao, Z.; Takehana, T.; Lin, Y. A.; Yasukohchi, T.; Davis, B. G. Angew. Chem. Int. Edit. 2013, 52 (14), 3916.
[34]Li, J.; Lin, S.; Wang, J.; Jia, S.; Yang, M.; Hao, Z.; Zhang, X.; Chen, P. R. J. Am. Chem. Soc. 2013, 135 (19), 7330.
• 教学研究与改革•
Protein-Modification Based on Canonical Amino Acid
HOU Ying-QinLV Hua*
(College of Chemistry and Molecular Engineering, Peking University, Beijing 100871, P. R. China)
Abstract:Chemical modifications on certain amino acids can result in diverse effects on protein structures and functions. For this, numerous strategies have been developed for site-specific or residue-specific protein modifications. Here we summarize the latest progresses on proteinmodification based on canonical amino acids such as cysteine, tyrosine and N-terminus.
Key Words:Protein-modification; Canonical amino acid; Cysteine; Tyrosine
*通讯作者,Email: chemhualu@pku.edu.cn
doi:10.3866/pku.DXHX20160101www.dxhx.pku.edu.cn
中图分类号:O6;G64
