黑麦草EST—SSR分子标记开发及生物信息学分析
2016-05-30王秀华李家丽蒲艳艳丛晓翔林雪婷于鸿翔赵岩
王秀华 李家丽 蒲艳艳 丛晓翔 林雪婷 于鸿翔 赵岩


摘要:为加速分子标记在黑麦草中的应用,利用黑麦草EST数据库(GenBank/dbEST)开展了黑麦草EST-SSR信息分析、标记开发和EST功能分析研究。在25 752条黑麦草EST序列中,共发现346条SSR序列,占整个EST总数的1.344%。其中,三核苷酸基序最多(39.60%),次之为二核苷酸基序(31.79%)。三核苷酸基序以GGC/CCG出现频率最高,为8.38%,其次是CGC/GCG(7.51%)、GCC/CGG(4.05%)和ATG/TAC(2.02%),二核苷酸基序以CT/GA出现频率最高(14.45%),其次是GA/CT(10.98%)。根据这些含有SSR的EST序列,利用Primer Premier 5.0软件,对346条EST-SSR序列进行引物设计,共设计引物193对,其中分值在90分以上的有113对(32.66%)。利用GenBank的BlastN和BlastX程序对相应113条EST进行功能分析,72条EST序列与51种具有生物学功能的蛋白质同源。
关键词:黑麦草;EST-SSR;信息分析;引物设计;功能分析
中图分类号:S543+.601文献标识号:A文章编号:1001-4942(2016)10-0001-06
黑麦草属禾本科(Poaceae)黑麦草属(Lolium)植物。其中最具有商业价值的两个种为多年生黑麦草(Lolium perenne L.)和多花黑麦草(Lolium multiflroum L.),作为草坪草和优质牧草在我国均有大面积栽培和利用[1,2]。
目前,黑麦草中常用的分子标记技术主要有RAPD[3]、CAPS[4]、AFLP[4,5]、SSR[6]、EST-SSR[7]等。SSR(simple sequence repeat)标记在遗传图谱的构建[8]、遗传多样性和亲缘关系分析[9]、品种指纹图谱及纯度鉴定[10]、功能基因标记[11]等方面具有公认的优越性和广阔的应用前景。但传统的基因组SSR标记开发投入多、耗时长。近年来,随着GenBank中大量EST(expressed sequence tags)序列的公布,许多植物基于EST的SSR(EST-SSR)标记开发成为可能。与基因组SSR相比,EST-SSR具有开发成本低、物种间通用性高、可直接反映相关基因的多样性等突出优点。目前许多作物如小麦、大麦、高羊茅、蒺藜苜蓿等的EST-SSR已被开发并用于遗传作图、遗传多样性、基因发掘、比较作图等研究[12-15]。
本研究利用NCBI上的黑麦草EST序列查找SSR,对其进行信息分析,设计EST-SSR引物,开发黑麦草EST-SSR分子标记,并对相应EST进行功能分析。
1材料与方法
1.1EST序列来源
从http://www.ncbi.nlm.nih.gov/dbEST/下载黑麦草EST序列。
1.2EST-SSR的查找
登陆网站http://archive.gramene.org/db/markers/ssrtool,利用SSRIT(simple sequence repeat identification tool)软件在线查找二、三、四、五、六核苷酸5种类型的SSR。识别标准为:重复基序最小长度≥18 bp,即二、三、四、五、六核苷酸重复次数分别大于或等于10、7、5、4、3。
1.3EST-SSR引物设计
利用Primer Premier 5.0软件,根据SSR的侧翼区域设计引物。引物设计的主要参数:EST序列长度大于100 bp;SSR序列的开始和结束位置分别距5′和3′端不少于20 bp;引物长度18~22 bp;退火温度Tm值 50~65℃,而且上游和下游引物的Tm值相差不大于5℃;PCR扩增产物长度100~300 bp;得分90分以上,尽量避免引物二级结构的出现。
1.4EST功能分析
利用NCBI网站对筛选出的黑麦草EST序列进行Blast比对,包括BlastN和BlastX两种比对,得到与其他植物已知基因或蛋白相似的功能,获得该EST在草坪草中的预测功能。利用BlastN程序在核苷酸水平上进行同源性分析,认为序列比分值大于200,e-value ≤1×10-20的序列具有同源核苷酸片段,而比对的片段长度小于50 bp结果不予采纳;对符合上述要求的EST序列利用BlastX程序在翻译水平上对其进行分析,序列比分值大于80的结果认为有相似性显著的同源蛋白质。
2结果与分析
2.1源于黑麦草EST的SSR查找
从NCBI上共下载到黑麦草的25 752条EST序列(其中多年生黑麦草19 784条,多花黑麦草5 968条)。经SSR查找,共发现346条SSR序列,占整个EST数据库的1.344%;这些EST序列全长17 720 kb,平均每1 000 kb 出现19.53个SSR,即每51.21 kb出现1个SSR。含有二、三、四和五核苷酸重复基序分别占SSR总数的31.79%、39.60%、11.27%和17.34%,无六核苷酸重复基序,三核苷酸基序出现频率最高,次之为二核苷酸基序(表1)。
2.2黑麦草EST-SSR的特征分析
346个SSR中,二核苷酸基序以CT/GA出现频率最高,占SSR总数的14.45%,其次是GA/CT(10.98%)和AT/TA(1.73%)。三核苷酸基序以GGC/CCG出现的频率最高,为8.38%,其次是CGC/GCG(7.51%)、GCC/CGG(4.05%)和ATG/TAC(2.02%),其他类型出现频率较低。四核苷酸基序以GTTG/CAAC出现频率最高,为0.87%。五核苷酸以TGTCG/ACAGC(2.31%)和CTCAT/GAGTA(2.02%)出现频率较高,其余出现频率均较低(表2)。
2.3黑麦草EST-SSR引物的开发
利用Primer Premier 5.0软件,对346个EST-SSR序列进行引物设计,共设计引物193对(55.78%),分值在90分以上的有113对(32.66%),见表3。
2.4功能分析
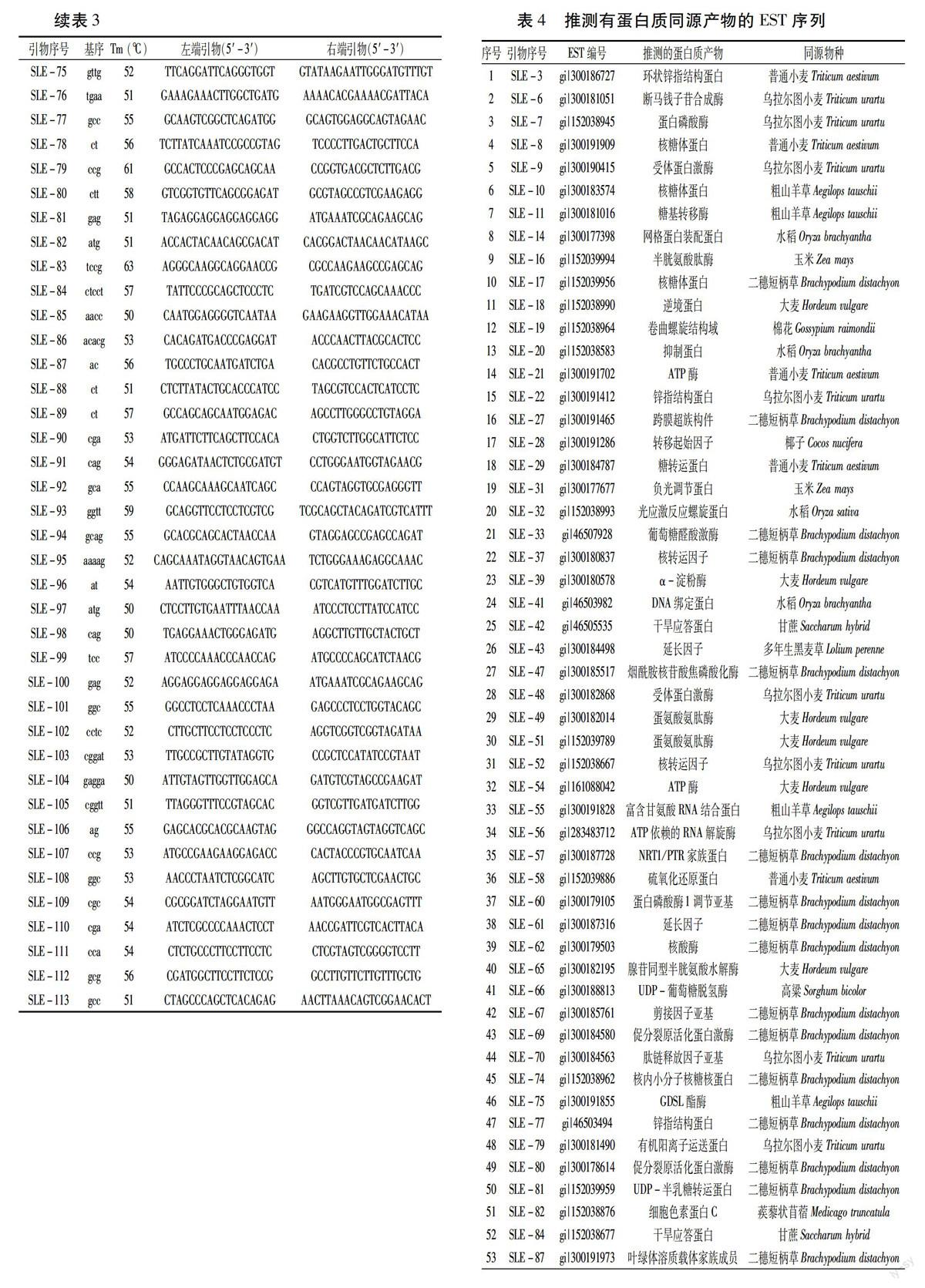
利用NCBI的BlastN和BlastX程序,对113条EST-SSR引物相应的EST序列进行比对分析。有78条EST序列与具有生物功能的核酸有同源性(未列出),有72条EST序列与具有生物功能的蛋白质有同源性(表4)。这72条蛋白序列分属于51种蛋白,大部分的同源产物来源于二穗短柄草(31.94%)或乌拉尔图小麦(15.28%)。
3讨论
目前,水稻[16]、小麦[17-19]、玉米[20]等主要粮食作物的EST-SSR标记已得到大量开发和应用。草类植物EST-SSR标记的开发和应用还较少。随着草坪草、牧草等草类植物EST和cDNA大规模测序的开展,相应EST数目也在急剧增加,其EST-SSR引物的开发与利用展现了广阔的前景[14,21-24]。截至2015年11月14日,在GenBank数据库(http://www.ncbi. nlm.nih. gov/dbEST)中找到的黑麦草的EST序列已达到25 752条。我们利用这25 752条EST序列,对346个EST-SSR序列进行引物设计,设计了113个分值90以上的引物对,为黑麦草增加了新的分子标记。
前人对EST-SSR的出现频率及重复基元出现频率进行了大量研究。关于EST中SSR出现频率,Chen等[17]对小麦EST-SSR进行开发时,每57.44 kb出现一个SSR;而李杰勤等[21]在高粱中研究结果为平均3.93 kb。这可能是由所研究的EST数据库不同及不同的EST-SSR搜索标准和统计标准所致。本研究在25 752条黑麦草EST序列中,共找到346条SSR序列,占整个EST数据库的1.344%,平均分布距离为51.21 kb。本研究结果和Chen等[17]研究结论相近。关于EST中不同核苷酸数目的重复基元出现频率,本研究中三核苷酸基序出现频率最高(39.60%),次之为二核苷酸基序(31.79%)。三核苷酸基序以GGC/CCG出现的频率最高,为8.38%,其次是CGC/GCG(7.51%),二核苷酸基序以CT/GA出现频率最高,占SSR总数的14.45%,其次是GA/CT(10.98%)。前人多数研究结果也为三核苷酸重复基元出现频率最高[25,26]。在水稻、玉米、大豆、高粱中二核苷酸重复基序出现频率最多的都是GA/CT[19],在水稻、玉米、大麦中,三核苷酸的CCG/GGC和AGG/TCC出现频率高[19,26,27]。本研究结果与在大多数植物基因组中三核苷酸重复出现频率较高的结果是一致的,二、三核苷酸不同基序出现频率也与多数结果一致。
利用生物信息学对EST序列进行功能分析,可推测该EST序列的功能,使随机测序而获得的EST序列与特定的生物功能相联系,有助于EST-SSR的进一步利用。而那些通过比对不能发现同源核苷酸或蛋白质的EST序列,可能是新的功能基因,EST-SSR引物就可作为这些新基因的分子标记,为基因克隆与功能验证奠定标记基础。高瑞娟等[28]在比对结球白菜EST时,94.8%(1102/1162)的EST可在蛋白质或核苷酸水平上找到同源类似物,大约77%的功能已知蛋白质来自拟南芥。本文利用NCBI的BlastN和BlastX程序对113条EST-SSR引物对应的EST序列进行了比对分析。结果表明,72条EST序列有生物学意义上的同源序列(63.72%),与高瑞娟等研究结果类似。我们比对的大部分的同源产物来源于二穗短柄草(31.94%)或乌拉尔图小麦(15.28%),得益于这两个物种特别是草类模式植物二穗短柄草近年来基因组研究的飞速发展。
4结论
在GenBank/dbEST中检索到黑麦草的EST序列25 752条,其中346个EST序列含有SSR(1.344%),共设计了分值90以上的EST-SSR引物113对。EST-SSR信息分析表明,三核苷酸基序最多,并以GGC/CCG出现的频率最高;次之为二核苷酸基序,并以CT/GA出现频率最高。对113条EST-SSR引物相应的EST序列进行Blast比对分析表明,有72条EST序列与具有生物功能的蛋白质有同源性,且分为51种蛋白质,大部分的同源产物来源于二穗短柄草(31.94%)或乌拉尔图小麦(15.28%)。
参考文献:
[1]
李杰勤, 王丽华, 詹秋文, 等. 20个黑麦草品系的SRAP遗传多样性分析[J]. 草业学报, 2013, 22(2): 158-164.
[2]刘春英, 孙学映, 朱体超, 等. 不同黑麦草品种生产性能比较与优势品种筛选[J]. 草业学报, 2014, 23(4): 39-48.
[3]董晓宁, 张晓佩, 李文杨. 18个黑麦草品种(系)的RAPD分析[J]. 福建农业学报, 2009, 24(3): 266-269.
[4]Miura Y, Ding C, Ozaki R, et al. Development of EST-derived CAPS and AFLP markers linked to a gene for resistance to ryegrass blast (Pyricularia sp.) in Italian ryegrass (Lolium multiflorum Lam.) [J]. Theoretical and Applied Genetics, 2005, 115(5): 811-818.
[5]Bert P F, Charmet G, Sourdille P, et al. A high-density molecular map for ryegrass (Lolium perenne) using AFLP markers [J]. Theoretical and Applied Genetics, 1999, 99(3): 445-452.
[6]Studer F, Klliker R, Muylle H, et al. EST-derived SSR markers used as anchor loci for the construction of a consensus linkage map in ryegrass (Lolium spp.) [J]. BMC Plant Biology, 2010, 10:177.
[7]Hirata M, Cai H, Inoue M, et al. Development of simple sequence repeat (SSR) markers and construction of an SSR-based linkage map in Italian ryegrass (Lolium multiflorum Lam.) [J]. Theoretical and Applied Genetics, 2006, 113(2):270-279.
[8]卢杰, 吕媛媛, 李杰勤, 等. 高丹草SSR引物设计及分子遗传框架图谱构建[J]. 中国草地学报, 2009, 31(2):28-33.
[9]Ipek A, Barut E, Gulen H, et al. SSR analysis demonstrates that olive production in the southern Marmara region in Turkey uses a single genotype [J]. Genetics and Molecular Research, 2009, 8(4): 1264-1272.
[10]Aitken K S, Jackson P A, McIntyre C L. A combination of AFLP and SSR markers provides extensive map coverage and identification of homo(eo)logous linkage groups in a sugarcane cultivar [J]. Theoretical and Applied Genetics, 2005, 110(5): 789-801.
[11]Li Y, Niu Y C, Chen X M. Mapping a stripe rust resistance gene YrC591 in wheat variety C591 with SSR and AFLP markers [J]. Theoretical and Applied Genetics, 2009, 118(2): 339-346.
[12]Holton T A, Christopher J T, McClure L, et al. Identification and mapping of polymorphic SSR markers from expressed gene sequences of barley and wheat[J]. Molecular Breeding, 2002, 9(2): 63-71.
[13]Saha M C, Rouf M A, Eujayl I, et al. Tall fescue EST-SSR markers with transferability across several grass species [J]. Theoretical and Applied Genetics, 2004, 109(4): 783-791.
[14]Barrett B, Griffiths A, Schreiber M, et al. A microsatellite map of white clover [J]. Theoretical and Applied Genetics, 2004, 109 (3): 596-608.
[15]Gupta S, Prasad M. Development and characterization of genic SSR markers in Medicago truncatula and their transferability in leguminous and non-leguminous species [J]. Genome, 2009, 52(9): 761-771.
[16]Yu J K, Rota M L, Kantety R V, et al. EST derived SSR markers for comparative mapping in wheat and rice[J]. Molecular Genetics and Genomics, 2004, 271(6): 742-751.
[17]Chen H M, Li L Z, Wei X Y, et al. Development, chromosome location and genetic mapping of EST-SSR markers in wheat [J]. Chin. Sci. Bull., 2005, 50: 2328-2336.
[18]Song W, Xie H, Liu Q, et al. Molecular identification of Pm12-carrying introgression lines in wheat using genomic and EST-SSR markers[J]. Euphytica, 2007, 158 (1): 95-102.
[19]Li L Z, Wang J J, Guo Y, et al. Development of SSR markers from ESTs of gramineous species and their chromosome location on wheat [J]. Proceedings of the Academy of Natural Science, 2008, 18: 1485-1490.
[20]Galvao K S, Ramos H C, Santos P H, et al. Functional molecular markers (EST-SSR) in the full-sib reciprocal recurrent selection program of maize (Zea mays L.) [J]. Genetics and Molecular Research, 2015, 14(3): 7344-7355.
[21]李杰勤, 王丽华, 詹秋文, 等. 高粱EST-SSR标记的建立及其在苏丹草中的应用初探[J]. 草业科学, 2010, 27(3): 112-117.
[22]陈永霞, 张新全, 谢文刚, 等. 利用EST-SSR标记分析西南扁穗牛鞭草种质的遗传多样性[J]. 草业学报, 2011, 20(6): 245-253.
[23]温莹, 逯晓萍, 任锐, 等. 高丹草EST-SSR标记的开发及其遗传多样性[J]. 遗传, 2013, 35(2): 225-232.
[24]Zeid M, Yu J K, Goldowitz I, et al. Cross-amplification of EST-derived markers among 16 grass species [J]. Field Crops Research, 2010, 118(1): 28-35.
[25]Gao L F, Tang J F, Li H W. Analysis of microsatellites in major crops assessed by computational and experimental approaches [J]. Molecular Breeding, 2003, 12: 245-261.
[26]Thiel T, Michalek W, Varshney R K, et al. Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L.) [J]. Theoretical and Applied Genetics, 2003, 106: 411-422.
[27]Cho Y G, Ishii T, Temnykh S, et al. Diversity of microsatellites derived from genomic libraries and GenBank sequences in rice (Oryza sativa L.) [J]. Theoretical and Applied Genetics, 2000, 100: 713-722.
[28]高瑞娟, 戴大鹏, 马荣才, 等. 结球白菜结球前期基因表达序列标签(EST)分析 [J]. 农业生物技术学报, 2004, 12(1): 24-29.
