中国常用橡胶树品种鉴定方法研究
2016-05-30王生瑞王师笈朱军黄家权
王生瑞 王师笈 朱军 黄家权


摘 要 橡胶树(Hevea brasiliensis)产生的胶乳是天然橡胶的最重要来源,不同品种产生胶乳的能力不同,但不同的橡胶树品种形态差异小,难以区分。利用简单的方法区分不同的橡胶树品种具有重要的意义。以天然橡胶生物合成相关的7个基因家族中的目的基因及启动子序列为模板,设计基因特异的PCR引物16对,分别扩增21个橡胶树栽培品种的基因组DNA,扩增条带的多态性可将19个品种区分开(除了云研77-4和南华1号外)。再利用橡胶小粒子编码蛋白基因(SRPP2)特异的引物,扩增云研77-4和南华1号的SRPP2基因,PCR反应产物直接测序,通过单核苷酸水平上的差异可准确的将二者区分开。本研究通过简单的PCR扩增及其扩增产物的直接测序,建立了一种稳定性好、简单快速、经济高效的鉴定橡胶树品种的方法。
关键词 橡胶树;克隆;引物设计;品种鉴定;单核苷酸差异
中图分类号 S794.1 文献标识码 A
Abstract The latex of the rubber tree(Hevea brasiliensis) is the most important source of natural rubber. The ability of latex production and adaptability of different rubber tree clones varied greatly, however, it is difficult to distinguish clones based on morphological traits, which is an important task in field work. In this study, sixteen pairs of gene specific primers were designed basing on gene and promoter sequences of genes related to natural rubber biosynthesis, these primer pairs were used respectively to amplify the genomic DNA of 21 rubber tree clones widely grown in China. The results showed that banding profile of these primer pairs could distinguish the 19 rubber tree clones, except Yunyan77-4 and Nanhua1. PCR amplification products using gene(SRPP2 encoding small rubber particle protein)specific primers were used for sequencing analysis, these two clones could be distinguished based on single nucleotide differences. In this study, a simple, stable and cost effective method using PCR band profile and PCR product sequencing to identify rubber tree clones was established.
Key words Rubber tree;Clone;Primer design;Clone identification;Single nucleotide difference
doi 10.3969/j.issn.1000-2561.2016.08.001
橡胶树(Hevea brasiliensis)原产于巴西亚马逊河流域赤道气候带,为多年生乔木,是典型的热带雨林树种。天然橡胶是橡胶树的次生代谢物,具有耐高温和高弹性等突出优点,是世界性工业原料和重要的战略物资,具有极高的经济价值及应用前景。在2 000多种产胶植物中,橡胶树是唯一大规模商业化种植的产胶植物。近年来,随着世界对天然橡胶需求量的不断增加,橡胶树栽培面积也迅速扩大。中国是天然橡胶的消费和生产大国,自1906年起就开始种植橡胶树,但受自然条件限制,橡胶树仅分布在海南、广东、云南等地。
目前世界上栽培的橡胶树品种均来自魏克汉种质,并通过引种、杂交等育种手段,选育和推广了多个高产优质的橡胶树品种。中国在生产中也使用了大量的橡胶树品种,仅中国栽培的橡胶树就超过了20种,并且早在20世纪70年代引进的RRIM600仍然在广泛种植。为了加快品种的更新换代,了解不同橡胶树品种的分布,以及在进行野外调查时确定调查对象的遗传背景,需要简单、快速、准确的橡胶树品种鉴定方法。
植物品种鉴定方法主要包括形态学鉴定法、物理化学鉴定法、生化鉴定法和DNA分子标记指纹图谱鉴定法等[1]。目前国内外品种鉴定仍以形态学鉴定为主,但形态标记的表现受环境影响较大、鉴定工作量大、周期长、受季节限制[2-3],并且橡胶树不同材料之间的差别较小,对普通的工作者而言,难以区分。近年来,分子标记方法(RFLP、AFLP、SSR、ISSR、SRAP、TRAP、SNP)的开发和利用在作物遗传多样性分析、遗传图谱构建、种质资源鉴定等方面的研究取得了一定的研究进展[4-5],这些技术可被用来有效的区分不同的品种,但这些技术的操作技术复杂,对DNA质量要求高,时间长,费用相对较高[6-7]。因此,亟需开发稳定性好、简单快速、经济高效的橡胶树品种鉴定方法。
本研究以天然橡胶生物合成相关的7个基因家族中的部分目的基因及启动子序列为模板,设计16对特异性扩增引物,对中国热带农业科学院橡胶研究所国家橡胶树种质圃保存的且在中国生产上广泛利用的21份魏克汉种质进行PCR扩增,结合PCR产物测序,以期建立一种简单快速、稳定且经济高效的橡胶树品种鉴定方法。
1 材料与方法
1.1 材料
所有的橡胶树材料均来自于中国热带农业科学院橡胶研究所国家橡胶树种质圃。采集橡胶树淡绿期的幼叶,铝箔包裹,作好记号后,放入液氮罐中带回实验室,置于-80 ℃的超低温冰箱中保存备用,共采集21个在中国栽培面积较大品种的叶片(表1)。
1.2 方法
1.2.1 橡胶树DNA提取 参照钟淦彬等[8]的方法,采用改良的CTAB法提取橡胶树幼嫩叶片的总DNA。
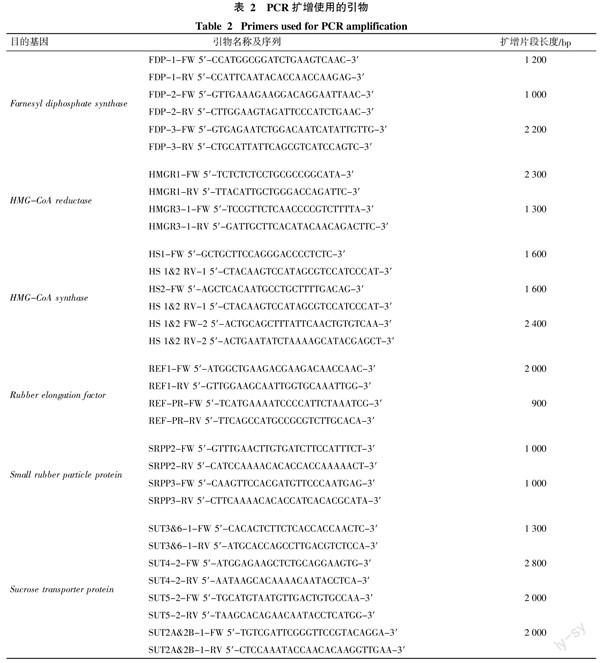
1.2.2 PCR反应及电泳检测 以巴西橡胶树天然橡胶生物合成(甲羟戊酸途径)相关的7个基因家族[9]中的目的基因及其启动子区序列为模板,设计16对引物(表2),由上海生物工程有限公司合成。反应体系:总体积为20 μL,其中正向和反向引物各1 μL(20 μmol/L),10xTrans Taq HiFi Buffer 2 μL,模板DNA 1 μL(50 ng/μL),2.5 mmol/L dNTPs 1.5 μL,HiFi DNA Polymerase 0.5 μL。PCR扩增条件为:94 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸2 min,共35个循环;72 ℃延伸10 min。所有材料和引物对进行PCR扩增的反应体系和条件均一致,反应产物在1.0%的琼脂糖凝胶上进行电泳,电泳结束后,在UVitec凝胶成像系统下观察拍照。
1.3 数据处理
1.3.1 数据统计与遗传相似性分析 对PCR反应扩增产物的琼脂糖凝胶电泳图谱进行条带统计分析,根据是否扩增出条带,转换为0、1数据,并利用NTSYS软件构建系统发育树。
1.3.2 单核苷酸多态性分析 利用SRPP2基因特异引物对云研77-4和南华1号进行PCR反应,将获得的产物直接进行DNA测序(上海生物工程有限公司),每个样品至少进行4次PCR产物测序,以保证基因测序结果的准确性,然后用DNAMAN进行序列的拼接,并与GenBank上登录的SRPP2的cDNA序列(AY237009.1,来自于橡胶树A775)做多重序列比对,以探测单核苷酸序列的多态性。
2 结果与分析
2.1 特异性PCR产物的多态性分析
本研究共使用25对引物,其中16对引物可在不同样本中扩增出清晰的条带(表2和表3)。引物REF1扩增产物经琼脂糖凝胶的分离结果显示条带清晰,无杂质,大小一致,除6、8、9、16号样本未扩增出条带,剩余样本均可扩增出清晰可见的条带(图1)。引物SRPP3扩增产物经琼脂糖凝胶的分离结果显示条带清晰,无杂质,大小一致,除9号样本未扩增出条带,剩余样本均可扩出清晰可见的条带(图2)。结果表明,16对特异性引物在21份供试材料中共扩增出条带183条,大小约为1 000~2 000 bp,用同一对引物在不同品种橡胶树中扩增结果不同,同一个品种用全部引物扩增出的条带数目也不同,具有一定的扩增多态性,可进一步的统计分析。
2.2 品种间的遗传相似性分析
数据统计分析结果显示,利用16对基因特异性引物扩增不同DNA样本,不同遗传背景的样本间具有明显的差异,同一对引物在不同品种橡胶材料中扩增出的条带数目变化很大,而条带大小基本一致,如引物SRPP3能在除RRIC 52外的其余20个品种中扩增出清晰可见的条带,引物HS1/HS2和SUT5-2只能在PB86中扩增出清晰可见的条带,在其他品种中均未能扩增出条带。不同品种样本利用全部特异性引物扩增出的条带数目也不同,如PB86用全部引物可扩增出14条条带,RRIC52用全部引物只能扩增出1条条带(表3)。利用统计分析出的数据构建系统发育树,结果表明,大部分品种(除云研77-4和南华1号外)存在PCR扩增产物带型的差异,可直接通过多个PCR反应,根据扩增条带的有无加以区分(图3),但云研77-4和南华1号使用这些引物扩增的结果没有差异,不能被区分开。
2.3 单核苷酸多态性(SNP)分析
为了准确区分云研77-4和南华1号,利用基因特异性引物SRPP2对云研77-4和南华1号进行PCR反应,获得的产物直接进行DNA测序,然后利用DNAMAN进行序列的拼接,并与引物SRPP2在GenBank中登陆的cDNA序列(AY237009.1)进行多重序列比对。共考察了SRPP2的3个区域,包括2个内含子区和1个外显子区,分别定义为Intron1、Exon、Intron2,Intron1(505 bp)中共有33个位点发生碱基突变,Exon(513 bp)中也有6个位点发生碱基突变,3个位点发生碱基缺失,Intron2 (335 bp)中有10个处位点发生碱基突变。总体而言,内含子区域发生的碱基突变位点显著多于外显子区域(表4)。根据云研77-4和南华1号在等位基因SRPP2上的单核苷酸多态性,可将二者区分开。
3 讨论与结论
目前,中国和东南亚各植胶国的橡胶树栽培品种主要是由魏克汉从原产地亚马逊河流域引种的22株野生种质驯化繁衍而成的,遗传基础比较狭窄。而中国的自育品种绝大多数又都可追溯到从东南亚引进的少数几个无性系,如 PR107、RRIM600等,因此中国橡胶树品种的遗传基础更加狭窄[10]。在本研究中21个无性系的相似系数变异范围仅为0.60~1.00,也证明了这一点。虽然有经验的研究人员可通过形态学的差异区分不同橡胶树品种,但不同品种之间的形态学的差异较小,并且受环境因素的影响。从分子水平上对橡胶树品种进行快速准确的鉴定,能为橡胶树遗传育种前期的品种鉴定工作提供帮助,对橡胶树种质资源的保护、橡胶树野外品种调查等工作均具有现实的指导意义。
一种理想的作物品种分子鉴定方法应具有以下特点:多态性高;重复性和稳定性好;带型清晰,容易统计;在染色体上均匀分布;共显性;简单快速,易自动化;标准化;开发和使用成本低廉[11-12]。目前报道的同一种作物的品种鉴定多利用PCR技术。因此,适宜的PCR反应体系及其优化利用是作物品种分子鉴定技术的研究内容之一[13-14]。虽然许多传统的分子标记技术方法均已被用来鉴定不同作物的品种,但在实际操作中,由于DNA用量大,耗费成本高,操作繁琐,不能简单快速的区分不同的品种[15-16]。本研究中所采用的鉴定橡胶品种的方法,只需获得少量的基因组DNA,设计基因特异性引物,进行PCR扩增,利用琼脂糖电泳检测及PCR产物测序,最多1周时间就可完成橡胶树品种的快速鉴定,在本过程中,标准化的PCR反应体系保证了结果的重复性和稳定性,方法操作非常简单且费用较低,条带清晰明显,可靠性高,在大部分的实验室就能完成。由于本研究选用的是基因特异的PCR扩增,不能从基因组的水平来区分不同品种,同时,少部分品种利用本研究设计的基因特异引物也不能区分,在今后的工作中,可通过增加目标基因的扩增来实现,但同时也可通过对单个基因的序列进行分析,获得差异化的snp位点,从而达到区分不同品种的目的。
参考文献
[1] 匡 猛, 杨伟华, 许红霞,等. 分子标记技术在棉花品种鉴定上的研究进展[J]. 棉花学报, 2009, 21(4): 330-334.
[2] Wang H, Sun H, Kwon WS, et al. Molecular identification of the Korean ginseng cultivar “Chunpoong” using the mitochondrial nad7 intron 4 region[J]. Mitochondrial DNA,2009, 20(2-3): 41-45.
[3] Noli E, Teriaca M, Sanguineti M, et al. Utilization of SSR and AFLP markers for the assessment of distinctness in durum wheat[J]. Molecular Breeding, 2008, 22(2): 301-313.
[4] De Riek J, Everaert I, Esselink D, et al. Assignment tests for variety identification compared to genetic similarity-based methods using experimental datasets from different marker systems in sugar beet[J]. Crop Science, 2007, 47(5): 1 964-1 974.
[5] Welsh J, Honeycutt R J, Mcclelland M, et al. Parentage determination in maize hybrids using the arbitrarily primed polymerase chain reaction(AP-PCR)[J]. Theoretical & Applied Genetics, 1991, 82(82): 473-476.
[6] Joshi D, Shrotria P, Singh R, et al. Assessment of RAPD and ISSR marker systems for establishing distinctiveness of forage Sorghum(Sorghum bicolor L. Moench) varieties as additional descriptors for plant variety protection[J]. Indian Journal of Genetics and Plant Breeding, 2011, 71(1): 25-36.
[7] Sarao N K, Vikal Y, Singh K, et al. SSR marker-based DNA fingerprinting and cultivar identification of rice(Oryza sativa L.)in Punjab state of India[J]. Plant Genetic Resources,2010, 8(1): 42-44.
[8] 钟淦彬, 李维国, 郑学项, 等. 一种快速高效适于橡胶树叶片AFLP分析的DNA提取方法[J]. 热带农业科学, 2010, 30(5):1-5.
[9] 邹 智, 杨礼富, 王真辉,等. 橡胶树中橡胶的生物合成与调控[J]. 植物生理学通讯, 2009, 59(12): 1 231-1 238.
[10] 王印肖, 徐秀琴,韩宏伟. 分子标记在品种鉴定中的应用及前景[J]. 河北林业科技, 2006, 34(9): 46-49.
[11] 焦仁海, 孙发明, 刘兴二,等. 玉米DNA分子标记及其研究进展[J]. 中国农学通报, 2006, 22(4): 48-51.
[12] 李菊芬, 许 玲,马国歲. 利用分子标记技术鉴定西瓜杂交种纯度[J]. 上海农业学报, 2009, 25(1): 72-75.
[13] 安泽伟, 赵彦宏, 程 汉,等. 橡胶树EST-SSR标记的开发与应用[J]. 遗传, 2009, 31(3): 311-319.
[14] 吴则东, 江 伟,马龙彪. 分子标记技术在农作物品种鉴定上的研究进展及未来展望[J]. 中国农学通报, 2015, 31(33):172-176.
[15] 朱岩芳. 农作物品种分子标记鉴定及指纹图谱构建研究[D]. 杭州: 浙江大学, 2013.
[16] 梁 勇, 李焕秀,何 艳. 几种DNA分子标记在种子纯度鉴定上的应用进展[J]. 广西农业科学, 2008, 39(1): 17-20.
