可可泛素结合酶基因家族的生物信息学初步分析
2016-05-30赵秋芳董晨决登伟陈宏良
赵秋芳 董晨 决登伟 陈宏良
摘 要 泛素结合酶(E2s)促进底物泛素化或者与E3s链接,是靶蛋白泛素化的关键酶,在泛素-蛋白酶体途径中起重要作用。利用可可(Theobroma cacao L.)全基因组测序数据,共鉴定出45个E2s基因家族成员,包括39个UBC和6个UEV基因。通过生物信息学方法,对可可E2s家族的基本理化性质、基因结构、二级结构预测、亚细胞定位、进化关系等方面进行初步分析。结果表明:可可E2s基因CDS长度在441(TcUEV3)~3 651 bp(TcUBC7)之间,对应的编码蛋白氨基酸数目在146(TcUEV3)~1 216 aa(TcUBC7)之间,编码蛋白分子量在16.39~134.71 ku之间。E2s基因外显子在1~11之间,多数基因外显子数目在5~7之间。E2s基因在10条染色体上均有分布,1号染色体为7个,数量最多;6号染色体2个基因,数量最少。可可E2s蛋白大多为不稳定蛋白,且均为亲水性蛋白,其二级结构以α-螺旋和无规则卷曲为主要构成元件。大多数可可E2s蛋白定位在细胞核,少数定位在内质网或者细胞质。进化树分析表明:可可E2s蛋白被分为20个亚家族,包括 16个UBC亚家族和4个UEV亚家族,第Ⅵ亚家族E2s数目最多为9个;E2s家族蛋白在物种进化过程中具有高度保守性。
关键词 可可;泛素结合酶(E2s);基因家族;生物信息学
中图分类号 S571.3 文献标识码 A
细胞内蛋白质的产生和降解必须保持平衡,才能维持细胞的稳态和正常功能。泛素-蛋白酶体途径(Ubiqutin-proteasome pathway, UPP)是细胞内蛋白质选择性降解的重要途径,广泛参与植物生长发育相关过程,尤其在维持细胞功能、细胞衰老、胚胎发育、光形态建成、组织分化、昼夜节律控制、花器官发育、激素信号响应、抵御生物和非生物胁迫等方面发挥着重要作用[1-9]。泛素化过程主要由泛素活化酶(ubiquitin-activating enzymes, E1s)、泛素结合酶(ubiquitin-conjugating enzymes, E2s)和泛素连接酶(ubiquitin-protein ligases, E3s)3种主要的酶来完成[10]。其中E2s是蛋白泛素化的中间环节,在泛素化系统中,负责将E1s激活的泛素分子转移至底物或者E3s,调节目标蛋白聚泛素链的形成,并与E3s共同确定底物的特异性[11-13]。
E2s蛋白在真核生物中广泛存在,所有E2s蛋白均包含由150左右的氨基酸组成保守催化结构域,称为UBC domain,内含有1个高度保守的半胱氨酸活性位点[14-15]。另外还存在一类UEV(ubiqutin E2 variants)蛋白[16],该蛋白家族在序列和结构上与E2s相似,但是缺少半胱氨酸催化位点,其功能也与典型的E2s蛋白有所不同[17-18]。随着全基因组测序技术的发展,基于E2s蛋白所具有的高度保守的UBC结构域,E2s家族已在多个真核生物中被鉴定出来,如酵母[15]、线虫[19]、人类[20]、拟南芥[21]、水稻[13]、番茄[22]、香蕉[23]、玉米[24]等。但目前对E2s蛋白家族的功能研究仍然偏少,仅在拟南芥上有较为深入的研究。如研究表明拟南AtUBC1和AtUBC2参与叶片发育和植物成花抑制基因的激活,atubc1-1 atubc2-1的双突变体表现出拟南芥莲座叶减少和花期明显提前的突变表型[5];AtUBC13在DNA复制后修复以及N-末端序列的蛋白降解方面发挥着重要作用[25];另外COP10作为拟南芥的UEV蛋白,其在植物的光形态建成中起着重要作用[26]。
可可(Theobroma cacao L.)与咖啡、茶一同被称为世界三大饮料作物,原产于亚马逊河上游热带雨林,主要分布在南北纬10°以内地带。可可的营养丰富,滋味醇香,具有兴奋与滋补作用,主要被用来制作饮料、巧克力、糕点等高档食品,具有较高的经济价值[27-28]。目前,世界上有超过50个国家进行规模种植[29]。随着可可全基因组的测序成功,更多的可可基因资源可供挖掘利用,也为鉴定可可E2s基因提供数据支持。本研究从可可全基因组数据库中分析筛选E2s家族基因,并利用生物信息学的方法,对筛选到的E2s家族进行理化性质、基因结构、染色体定位、二级结构、亚细胞定位预测、系统进化等方面进行初步分析,以期为后续开展可可E2s家族基因相关研究提供参考。
1 材料与方法
1.1 材料
以热带特色作物可可(Theobroma cacao L.)为研究对象,基因数据来源于Cacao Genome Database数据库(http://www.cacaogenomedb.org/)及Phytozome基因组数据库(http://phytozome.jgi.doe.gov/pz/portal.html)。
1.2 方法
1.2.1 拟南芥、水稻和可可E2s家族基因序列的获取
根据文献报道,在TAIR数据库(http://www.arabidopsis.org/)和水稻RAP 数据库(http://rapdb.dna.affrc.go.jp/)中分别提取48个拟南芥[19]和48个水稻[20]E2s基因的CDS序列和蛋白序列,以FASTA格式保存。用拟南芥的E2s蛋白序列在Phytozome基因组数据库(http://phytozome.jgi.doe.gov/pz/portal.html)中,通过基因查找和序列比对,查找可可基因组中所有E2s基因的CDS和蛋白序列,并利用SMART(http://smart.embl-heidelberg.de/)在线分析软件对候选基因的氨基酸序列结构域进行鉴定,凡是含有UBC保守结构域的蛋白即为E2s家族成员。
1.2.2 可可E2s基因结构分析 利用Gene Structure Display Server GSDS在线软件(http://gsds.cbi.pku.edu.cn/)对可可的E2s基因结构进行作图,可可编码区序列(CDS)与基因组DNA序列来自Phytozome(http://phytozome.jgi.doe.gov/pz/portal.html)基因组数据库。
1.2.3 可可E2s蛋白的氨基酸序列属性分析 将获得的可可E2s蛋白的氨基酸序列投入Ex-PAsy (http://www.expasy.org/)站点,利用其中的Prot-Param软件在线分析E2s蛋白的分子量、等电点、不稳定系数、脂肪指数和疏水性等物理属性。二级结构分析采用在线SOPMA程序(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html),所有参数均为默认值。亚细胞定位采用Plant-mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/#)进行分析。
1.2.4 可可和拟南芥、水稻E2s家族蛋白系统进化分析 利用Clustal W对可可、拟南芥和水稻中所有E2s蛋白序列进行序列比对,结合MEGA6.06软件构建可可、拟南芥及水稻E2s蛋白家族的无根进化树,进化树生成采用邻接法(neighbor joining,NJ),参数设置:使用neighbor-joining法则的P-距离(P-distance)模型构建,选择了成对删除(pairwise deletion)空位(gap)的选项,Bootstrap method取值1 000。
2 结果与分析
2.1 可可E2s家族基因鉴定和基因相关信息分析
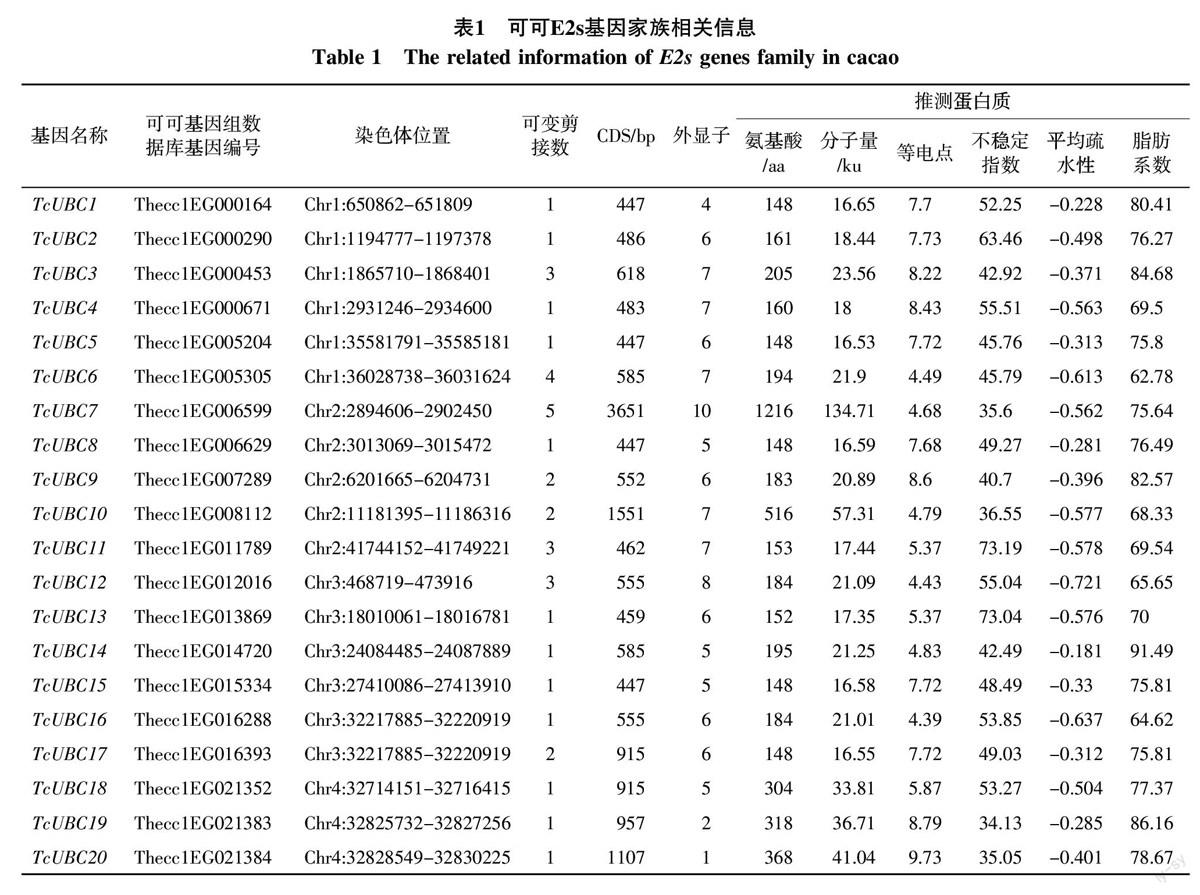
E2s蛋白均包含高度保守的催化结构域(UBC domain),根据其UBC结构域,通过基因查找和序列比对,最终确定45个可可E2s基因,其中包含39个UBC和6个UEV基因,为了描述方便,将筛选到的E2s基因根据其在染色体上的位置,分别命名为TcUBC1-39和TcUEV1-6(见表1)。由表1可以看出,可可E2s家族基因序列在转录后会产生1~5个可变剪接,其中TcUBC7和TcUBC23的可变剪接数为5个,数量最多。可变剪接被认为是导致蛋白质功能多样性的重要原因之一,使一个基因可编码多个不同转录产物和蛋白产物,已有研究表明,可变剪接在产生受体多样性、调节生长发育等方面起决定性作用[30]。鉴于较多的可可E2s基因存在可变剪接,在分析基因结构特征时,仅选择最主要的可变剪接体进行分析(详见Phytozome基因组数据库的注释)。已鉴定到的可可E2s基因CDS长度在441(TcUEV3)~3 651 bp(TcUBC7)之间,跨度较大,对应的编码蛋白氨基酸数目在146(TcUEV3)~1 216 aa(TcUBC7)之间,编码蛋白分子量在16.39~134.71 ku之间。蛋白质等电点分析结果表明可可E2s蛋白包含酸性、中性、碱性3种蛋白,其中等电点小于6.5的蛋白有19个,显酸性;6.5~7.5之间的蛋白有4个,显中性;大于7.5的蛋白个数有23个,显碱性。蛋白不稳定指数分析发现可可E2s蛋白大多数属于不稳定蛋白,不稳定指数>40;TcUBC7/10/19/20/21/24/30和TcUEV2/3/4/5共10个E2s蛋白为稳定蛋白。疏水性分析显示平均疏水性(GRAVY)在-0.894(TcUBC33)~-0.07(TcUEV5)之间,表明所有可可E2s蛋白均为亲水性蛋白。可可E2s蛋白脂肪系数(AI)在62.78(TcUBC6)~91.49(TcUBC14)之间(表1)。
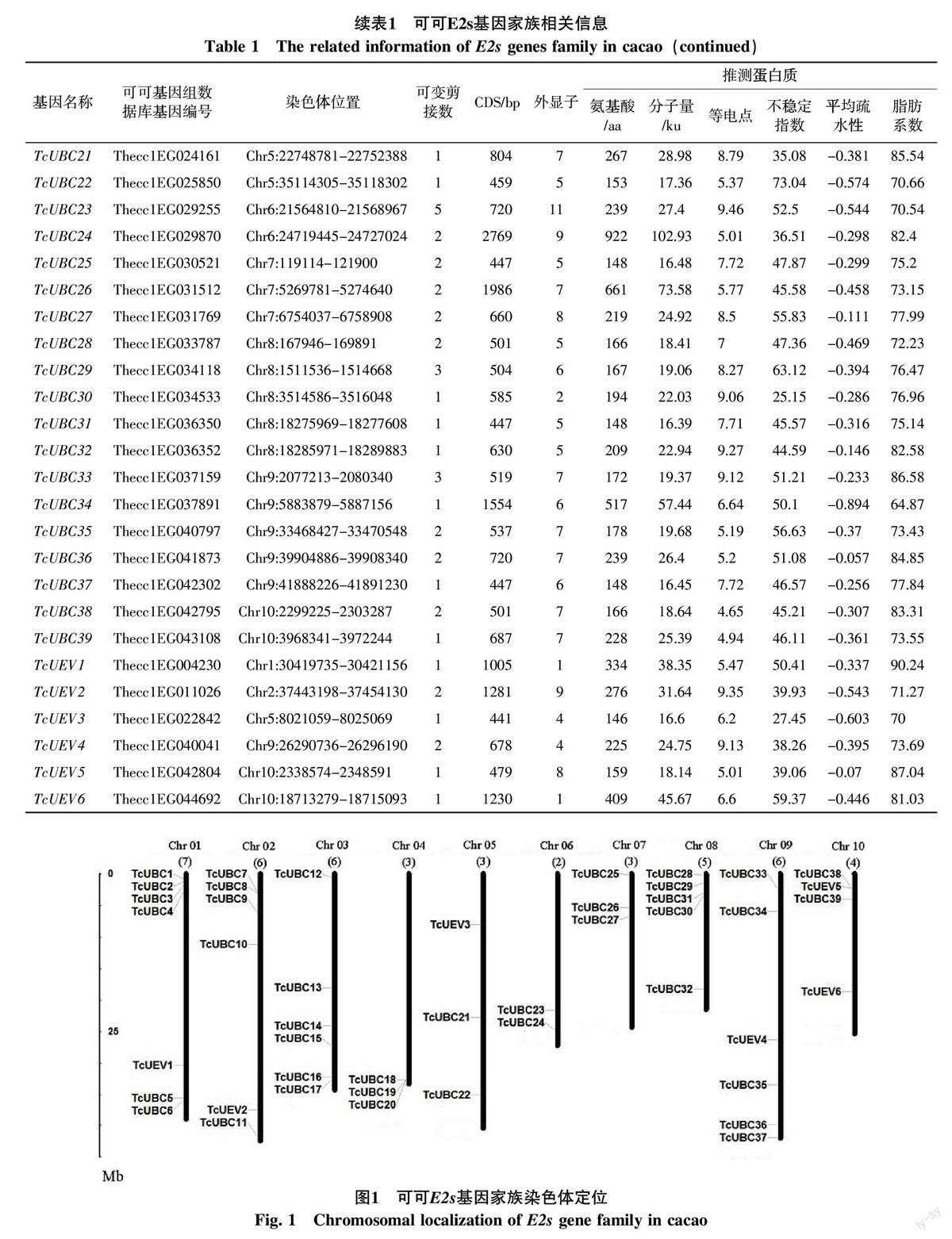
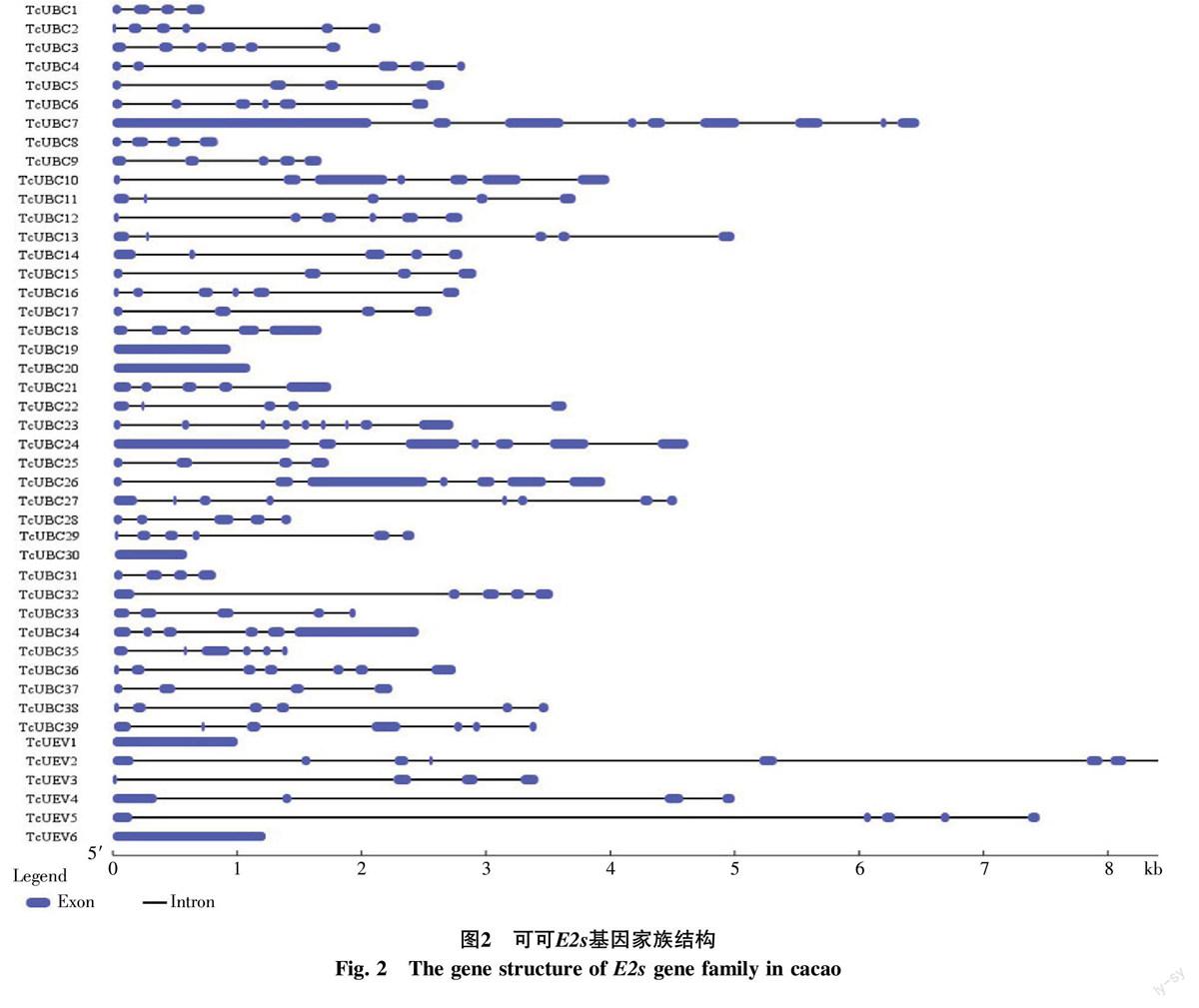
可可E2s家族基因在10个染色体上均有分布,但分布并不均匀(图1)。1号染色体数量最多为7个;2号、3号和9号染色体上有6个基因;8号染色体有5个基因;10号染色体有4个;4、5和7号染色体均分布有3个E2s基因;6号染色体分布有2个基因,数量最少。可可E2s基因结构分析结果显示:E2s基因含1个(TcUBC20、TcUEV6)至11(TcUBC23)个外显子,而多数基因的外显子数目在5~7之间(表1和图2)。
2.2 可可E2s蛋白的二级螺旋结构、亚细胞定位预测及UBC结构域分析
可可E2s蛋白的二级结构进行预测结果如表2所示。可可E2s蛋白均由α-螺旋、扩展链结构、β-转角和无规则卷曲4种形式组成,以无规则卷曲为主的蛋白有24个,所占百分比在33.3%~47.66%之间,以α-螺旋为主要构成元件蛋白有19个,所占百分比在33.72%~53.09%之间,另外TcUBC15和TcUBC17α-螺旋和无规则卷曲所占百分比一致,均为40.54%。可可E2s蛋白扩展链结构和β-转角所占百分比例较小,说明可可E2s蛋白的二级结构以α-螺旋和无规则卷曲为主。
蛋白质亚细胞定位分析发现可可E2s蛋白大多定位于细胞核中,少数蛋白被定位在内质网或者细胞质中,如TcUBC11、TcUBC13、TcUBC22和TcUBC27定位在细胞质和细胞核中,TcUBC36定位在细胞核和内质网中,而TcUBC18和TcUBC38仅定位在内质网中。
尽管E2s蛋白均含有一个由150个氨基酸组成的高度保守的UBC结构域,但是N端和C端的大小和结构上仍然存在很大差别,而这些侧翼序列参与底物的选择,二聚反应和其他相关过程,往往导致E2s蛋白间的功能差异[31-32]。根据是否具有N端和C端延长链,将E2s蛋白分为4大亚类,ClassⅠ仅含有UBC/UEV结构域,ClassⅡ包含UBC/UEV结构域和C端延长的蛋白序列,ClassⅢ包含UBC/UEV结构域和N端延长的蛋白序列,ClassⅣ包含UBC/UEV结构域、N端和C端延长的蛋白序列[33]。通过SMART程序分析可可E2s蛋白的UBC结构域(图3)。可可E2s蛋白UBC结构存在4大亚类,其中属于ClassⅣ亚类的E2s蛋白为16个,数量最多,其氨基酸数目在159~1 216 aa之间(表1);其次为ClassⅠ亚类,为14个,氨基酸数目在148~166 aa之间(表1),ClassⅡ和ClassⅢ亚类分别包含9个和6个E2s蛋白,氨基酸数目分别在161~276 aa和172~517 aa之间(表1)。ClassⅠ亚类仅含有UBC结构,因此其氨基酸数目相对较少;ClassⅢ亚类仅包含C端延长和UBC结构域,而TcUBC34(517 aa)长于ClassⅣ亚类中除了TcUBC7(1 216 aa)和TcUBC26(661 aa)之外的所有蛋白,E2s蛋白结构域之间的差异也导致其功能间的差异。
2.3 可可E2s蛋白的系统进化树分析
为了分析可可E2s家族的系统发育关系,利用可可45个E2s蛋白,模式植物拟南芥48个E2s蛋白[21]和水稻48个E2s蛋白[13](均包括其UEV蛋白)构建系统发育树(图4),并参照拟南芥E2s蛋白亚家族分类[21]。进化分析将45个可可E2s蛋白分为20个亚家族,包括16个UBC亚家族和4个UEV亚家族。各亚家族所含E2s蛋白数量差异较大,其中Ⅵ亚家族包含可可E2s蛋白为9个,数量最多,分别是TcUBC1/5/8/15/17/25/31/32/37;其次为Ⅺ家族,包含6个E2s蛋白,分别为TcUBC7/10/19/20/24/26。而Ⅸ、Ⅹ、Ⅻ、ⅩⅢ、ⅩⅤ、ⅩⅥ共6个亚家族中,仅包含一个E2s蛋白,分别为TcUBC33、TcUBC21、TcUBC14、TcUBC30、TcUBC27、TcUBC34。另外,Ⅰ、Ⅱ、Ⅴ、Ⅷ、ⅩⅣ共5个亚家族均含有2个E2s蛋白。TcUBC4和TcUBC28属于Ⅰ亚家族,TcUBC3和TcUBC9属于Ⅱ亚家族,TcUBC36和TcUBC38属于Ⅴ亚家族,TcUBC35和TcUBC39属于Ⅷ亚家族,TcUBC18和TcUBC23属于ⅩⅣ亚家族。TcUBC11/12/13属于Ⅲ亚家族。6个UEV蛋白被分为ⅩⅦ、ⅩⅧ、ⅩⅨ和ⅩⅩ四个亚家族,其中TcUEV1和TcUEV6属于ⅩⅦ亚家族;TcUEV2和TcUEV3属于ⅩⅧ亚家族;TcUEV4和TcUEV5分别属于ⅩⅨ和ⅩⅩ亚家族。构建系统发育树可以用来识别不同物种间直系同源基因和物种内旁系同源基因,通常直系同源基因的发生会促使新物种的形成,而旁系同源基因产生则是新功能的来源[34]。进化树分析鉴定出6对旁系同源蛋白,分别为TcUBC2和TcUBC29、TcUBC3和TcUBC9、TcUBC4和TcUBC28、TcUBC35和TcUBC39、TcUEV2和TcUEV3、TcUEV1和TcUEV6。可可E2s蛋白与拟南芥和水稻均存在直系同源蛋白,校验参数(bootshap)>90%的分别有10对和8对,其中TcUBC14、TcUBC18、TcUBC23、TcUBC33、TcUEV4在拟南芥和水稻中均存在其直系同源蛋白。
3 讨论与结论
近年来,随着基因组测序技术的不断提高,越来越多的植物完成全基因组的测序工作,这也促使相关基因家族研究快速发展。E2s家族在植物生长发育方面起着非常重要的作用,而目前仅在拟南芥[21]、水稻[13]、番茄[22]、玉米[24]等模式植物中有部分研究,许多作物仍然亟待开展E2s基因家族的相关研究。本研究通过对可可全基因组全面查找、各种软件和在线工具分析,共鉴定出45个E2s基因,其中包括39个UBC基因和6个UEV基因,其E2s基因数目与水稻(48)和拟南芥(48)相似。E2s基因家族在生物演变过程中不断扩大,低级真核生物的E2s基因数目少于高级真核生物[20]。已有报道低级真核生物酵母中发现13个[15],线虫中20个[19],绿藻中19个(数据未发表);而在高级植物和动物中E2s基因数目较多,比如在人类中发现37个[20],番茄中52个[22],香蕉中74个[23],玉米中75个,其中69个UBC基因和6个UEV基因[24]。
为了揭示可可E2s家族的进化关系,将拟南芥、水稻和可可共141个E2s蛋白构建系统发育树。系统进化分析将可可E2s蛋白分为20个亚家族,各亚家族所含蛋白数目差别很大,Ⅵ亚家族包含9个E2s蛋白,Ⅺ亚家族为6个。拟南芥和水稻的Ⅵ和Ⅺ亚家族也包含较多的E2s蛋白,但在数目上有差别,如二者Ⅵ亚家族均包含8个E2s蛋白,而Ⅺ亚家族中包含4个和9个E2s蛋白。可可在亚家族Ⅸ、Ⅹ、Ⅻ、ⅩⅢ、ⅩⅤ、ⅩⅥ仅有1个E2s蛋白,拟南芥[21]和水稻[35]这些亚家族也包含较少E2s蛋白。本研究发现大多数的亚家族包含拟南芥、水稻和可可3个物种,而少数亚家族仅包括1个或2个物种。如ⅩⅢ和ⅩⅦ亚家族包括拟南芥和可可2个物种;而ⅩⅩ亚家族仅包含可可E2s蛋白(TcUEV5),这表明该蛋白可能是生物进化过程中可可所特有的E2s蛋白。以往研究发现水稻和拟南芥E2s蛋白亚家族非常相似,揭示在单子叶和双子叶植物分化之前E2s家族已经存在。本研究发现可可与拟南芥和水稻间均存在直系同源蛋白,也进一步证明植物E2s蛋白具有共同的起源,进化过程高度保守。另外,同一亚家族E2s在基因结构和UBC结构域上往往具有较高的相似性[24]。在可可Ⅵ亚家族中TcUBC8/15/25/31/32均含有5个外显子,基因结构相似;且TcUBC1/5/8/15/17/25/31/37UBC结构域也相似,均属于ClassⅠ亚类。同一亚家族的E2s蛋白在功能上往往也较为相似,进化分析表明TcUEV4和AtCOP10之间校验参数(bootshap)达98%,因此根据前人研究可以推测TcUEV4可能对可可的光形态建成起着重要作用,但其具体功能仍需后续验证。
可可属于典型的热带作物,适宜在高温、高湿且较为荫蔽的环境下种植,但我国热区面积有限,适宜可可种植区域较少,且在生产上容易遭受低温和干旱等逆境胁迫,影响可可产业发展。泛素结合酶作为泛素化途径中的关键酶,在调控植物生长、发育和响应逆境胁迫中发挥重要作用。而目前,可可E2s家族基因相关研究仍未见报道。本研究对可可E2s基因家族进行了初步分析,为将来深入研究该基因家族的表达调控、结构和功能等提供参考,也为进一步揭示该基因家族参与可可生长、发育的调控以及响应逆境胁迫的机制提供理论依据。
参考文献
[1] Vierstra R D. Proteolysis in plants: mechanisms and functions[J]. Plant Molecular Biology, 1996, 32(1-2): 275-302.
[2] Welchman R L, Gordon C, Mayer R J. Ubiquitin and ubiquitin-like proteins as multifunctional signals[J]. Nature Reviews Molecular Cell Biology, 2005, 6(8): 599-609.
[3] Sadanandom A, Bailey M, Ewan R, et al. The ubiquitin-proteasome system: central modifier of plant signaling[J]. New Phytologist, 2012, 196(1): 13-28.
[4] Dreher K, Callis J. Ubiquitin, hormones and biotic stress in plants[J]. Annals of Botany, 2007, 99(5): 787-822.
[5] Xu L, Ménard R, Berr A, et al. The E2 ubiquitin-conjugating enzymes, AtUBC1 and AtUBC2, play redundant roles and are involved in activation of FLC expression and repression of flowering in Arabidopsis thaliana[J]. The Plant Journal, 2009, 57: 279-288.
[6] Cui F, Liu L, Zhao Q, et al. Arabidopsis ubiquitin conjugase UBC32 is an ERAD component that functions in brassinosteroid-mediated salt stress tolerance[J]. The Plant Cell, 2012(24): 233-244.
[7] Smalle J, Vierstra R D. The ubiquitin 26S proteasome proteolytic pathway[J]. Plant Biology, 2004, 55: 555-590.
[8] Santner A, Estelle M. The ubiquitin-proteasome system regulates plant hormone signaling[J]. The Plant Journal, 2010, 61: 1 029-1 040.
[9] Park C, Chen S, Shirsekar G, et al. The Magnaporthe oryzae effector AvrPiz-t targets the RING E3 Ubiquitin Ligase APIP6 to suppress pathogen-associated molecular pattern-triggered immunity in rice[J]. The Plant Cell, 2012 (24): 4 748-4 762.
[10] Glickman M H, Ciechanover A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction[J]. Physiological Reviews, 2002, 82(2): 373-428.
[11] Bae H, Kim W T. The N-terminal tetra-peptide(IPDE)short extension of the U-box motif in rice SPL11 E3 is essential for the interaction with E2 and ubiquitin-ligase activity[J]. Biochemical and Biophysical Research Communications, 2013, 433(2): 266-271.
[12] Ye Y, Rape M. Building ubiquitin chains: E2 enzymes at work[J]. Nature Reviews Molecular Cell Biology, 2009, 10: 755-764.
[13] Bae H, Kim W T. Classification and interaction modes of 40 rice E2 ubiquitin-conjugating enzymes with 17 rice ARM-U-box E3 ubiquitin ligases[J]. Biochemical and Biophysical Research Communication, 2014, 444(4): 575-580.
[14] Criqui M C, de Almeida Engler J, Camasses A, et al. Molecular characterization of plant ubiquitin-conjugating enzymes belonging to the UbcP4/E2- C/UBCx/UbcH10 gene family[J]. Plant Physiology, 2002,130: 1 230-1 240.
[15] Michelle C, Vourc'h P, Mignon L, et al. What was the set of ubiquitin and ubiquitin-like conjugating enzymes in the eukaryote common ancestor?[J]. Journal of Molecular Evolution, 2009, 68(6): 616-628.
[16] Thomson T M, Lozano J J, Loukili N, et al. Fusion of the human gene for the polyubiquitination coeffector UEV1 with Kua, a newly identified gene[J]. Genome Research, 2000, 10(11): 1 743-1 756.
[17] Kerscher O, Felberbaum R, Hochstrasser M. Modification of proteins by ubiquitin and ubiquitin-like proteins[J]. Cell Development Biology, 2006, 22: 159-180.
[18] Hochstrasser M. All in the ubiquitin family[J]. Science, 2000, 289(5479): 563-564.
[19] Jones D, Crowe E, Stevens T A, et al. Functional and phylogenetic analysis of the ubiquitylation system in Caenorhabditis elegans: ubiquitin-conjugating enzymes, ubiquitin-activating enzymes, and ubiquitin-like proteins[J]. Genome Biology, 2001, 3(1): 0002.1-0002.15.
[20] van Wijk S J L, Timmers H T M. The family of ubiquitin-conjugating enzymes(E2s): deciding between life and death of proteins[J]. The Faseb Journal, 2010, 24(4): 981-993.
[21] Kraft E, Stone S L, Ma L, et al. Genome analysis and functional characterization of the E2 and RING-type E3 ligase ubiquitination enzymes of Arabidopsis[J]. Plant Physiology, 2005, 139(4): 1 597-1 611.
[22] Wang Y, Wang W, Cai J, et al. Tomato nuclear proteome reveals the involvement of specific E2 ubiquitin-conjugating enzymes in fruit ripening[J]. Genome Biology, 2014, 15(12): 548.
[23] Dong C, Hu H, Jue D, et al. The banana E2 gene family: genomic identification, characterization, expression profiling analysis[J]. Plant Science, 2016, 245: 11-24.
[24] Jue D, Sang X, Lu S, et al. Genome-wide identification, phylogenetic and expression analyses of the ubiquitin-conjugating enzyme gene family in maize[J]. PloS One, 2015, 10(11): 21-35.
[25] Broomfield S, Hryciw T, Xiao W. DNA postreplication repair and mutagenesis in Saccharomyces cerevisiae[J]. Mutation Research/DNA Repair Reports, 2001, 486(3): 167-184.
[26] Lau O S, Deng X W. Effect of Arabidopsis COP10 ubiquitin E2 enhancement activity across E2 families and functional conservation among its canonical homologues[J]. Biochemical Journal, 2009, 418(3): 683-690.
[27] Buamah R, Dzogbefia V P, Oldham J H. Pure yeast culture fermentation of cocoa(Theobroma cacao L): effect on yield of sweatings and cocoa bean quality[J]. World Journal of Microbiology and Biotechnology, 1997, 13(4): 457-462.
[28] Beavan M, Kligerman A, Droniuk R, et al. Production of microbial cocoa butter equivalents[M]. Industrial Applications of Single Cell Oils. AOCS Press: Champaign, 1992: 156-184.
[29] 李付鹏, 秦晓威, 伍宝朵, 等. 可可蔗糖磷酸合成酶基因家族进化及组织表达分析[J]. 热带作物学报, 2015, 36(9): 1 608-1 613.
[30] Tack D C, Pitchers W R, Adams K L. Transcriptome analysis indicates considerable divergence in alternative splicing between duplicated genes in Arabidopsis thaliana[J]. Genetics, 2014, 198(4): 1 473-1 481.
[31] Arrigoni A, Grillo B, Vitriolo A, et al. C-terminal acidic domain of ubiquitin-conjugating enzymes: A multi-functional conserved intrinsically disordered domain in family 3 of E2 enzymes[J]. Journal of Structural Biology, 2012, 178(3): 245-259.
[32] Schumacher F R, Wilson G, Day C L. The N-terminal extension of UBE2E ubiquitin-conjugating enzymes limits chain assembly[J]. Journal of Molecular Biology, 2013, 425(22): 4 099-4 111.
[33] Bachmair A, Novatchkova M, Potuschak T, et al. Ubiquitylation in plants: a post-genomic look at a post-translational modification[J]. Trends in Plant Science, 2001, 6(10): 463-470.
[34] Thornton J W, De Salle. Gene family evolution and homology: Genomics meets phylogenetics[J]. Annual Review of Genomics and Human Genetics, 2000, 1(1): 41-73.
[35] Zhiguo E, Zhang Y, Li T, et al. Characterization of the ubiquitin-conjugating enzyme gene family in rice and evaluation of expression profiles under abiotic stresses and hormone treatments[J]. PloS One, 2015, 10(4): 1-24.
