生物信息学在蛋白质(酶)改造及设计中应用的新进展
2016-05-13孙伟峰钟文娟伍圆明林泉秀陈祥贵
孙伟峰,钟文娟,孙 彬,伍圆明,苟 赟,林泉秀,陈祥贵,刘 义
(西华大学食品与生物工程学院,食品生物技术四川省高校重点实验室,四川 成都 610039)
生物信息学在蛋白质(酶)改造及设计中应用的新进展
孙伟峰,钟文娟,孙彬,伍圆明,苟赟,林泉秀,陈祥贵,刘义
(西华大学食品与生物工程学院,食品生物技术四川省高校重点实验室,四川 成都 610039)
摘要:蛋白质(酶)的改造和设计是人类面对纷繁复杂自然界的一个重大挑战,是自然进化的突发事件,是人类不断掌握自然规律的必然结果,对人类更深入地了解蛋白质结构与功能,促进生物蛋白工业生产具有重大的意义。本文介绍了新型蛋白质(酶)改造和设计的新思路和实施过程,以及目前生物酶改造和设计的一些前沿研究团队和研究成果,以期为生物酶或蛋白质的改造与设计提供借鉴。
关键词:生物信息学; 蛋白质改造;酶设计
从1965年首次获得溶菌酶(PDB:HEWL)晶体结构开始[1],人类就一直试图寻找和掌握蛋白质(酶)折叠成特定构象的奥秘。从DNA水平、蛋白质的一级序列、二级结构到高级结构,人们一直在不断地探索以求掌握生命的秘密。由于早期研究条件所限,仅能达到蛋白二级结构肽的设计水平,尽管如此,蛋白质(酶)改造和设计还是取得了丰硕的成果:研究者从7个氨基酸序列形成的α-helix出发,并在此基础上不断改进,成功设计了(helix-loop-helix- loop-helix)和α/β 折叠等新型蛋白[1],这些经典设计方法一直沿用至今;最近Koga等[2]在《Nature》杂志上报道了关于设计二级结构α-helix和β-sheet的一般规律。
随着计算机技术和各种算法的迅速发展,许多优秀的软件(如Metal Search和Dezyme)被开发出来用于改造和设计新型蛋白质(包括改造和设计新型金属蛋白)[1],而分子力学和量子化学的迅猛发展,为开发更加优秀的软件用于改造和设计更为复杂的蛋白进一步创造了条件[3-9]。近年来,由美国华盛顿大学Baker实验室组织的每年一次的国际蛋白质结构预测技术评估CASP大赛,被称之为蛋白质设计方向的“奥林匹克”,鼓励和吸引了全世界蛋白质结构研究科研人员的积极参与,新的算法和新型蛋白质设计方法得以不断开发[10]。在2010年的第九届全球蛋白质结构预测比赛中,我国中科院生物物理所蒋太交研究组发展的Jiang_Assembly蛋白质结构预测服务器获得佳绩,进入了前20名,这标志着我国在蛋白质三维结构预测领域进入了世界上游水平[11]。
尽管如此,在涉及新型蛋白质(酶)改造和设计的诸多领域还有待加强。当前合成生物学和代谢工程已经允许我们设计比较精细的细胞工厂用于生产人类所需的高价值的化学物质;但若想真正实现生物催化的工业化,高效生物催化剂——酶,特别是人类还未探知的(也可能是自然界本不存在的)酶成为了不可或缺的关键因素,因而无论是从工业价值还是理论价值来讲,新型酶设计都显得尤为重要。
1传统蛋白质工程在改造和设计新型蛋白质(酶)过程中的不足
目前蛋白质(酶)改造和设计大多指的是传统概念上的酶设计,如通过点突变、片段优化替换等各种传统蛋白质工程手段,或者涉及常规生物信息学的分子改造等而获得新型酶[12];但是有些酶由于蛋白质骨架性质不够优良,很难通过这些传统手段“点穴式”小修小补而获得性能改变,或使某些性能得到巨大提升。如多数D型氨基酸的生产,由于自然界缺乏直接催化的相应酶,并且通过传统蛋白质工程手段很难获得,只好采用其他酶多步催化“绕路走”的办法,有时需要用几个酶联合催化才能实现,再加上工序复杂导致产品得率损失等多种因素,给工业生产带来巨额成本。如采用海因酶-氨基酸酰胺水解酶2种酶催化法生产D型氨基酸,由于氨甲酰水解酶的活性、稳定性较海因酶低,在生产过程中氨甲酰水解酶成为了限制性因素,导致D型氨基酸产率较低[13]。此外,某些行业中关键限制性酶的缺乏,限制了其产物的应用。如在医药行业中,默克制药公司开发的西塔列汀(sitagliptin)是用于治疗糖尿病的特效药,而作为西塔列汀中的活性结构模块的β-芳香丙氨酸,如果用有机法合成,产物往往是对消旋体,这大大制约了这种特效药的应用。尽管如此,目前β-芳香丙氨酸也只能通过此法生产,因为生物酶法也缺少可以直接催化生成β-芳香丙氨酸的高活性新型酶种[14]。
虽然目前一些传统的蛋白质(酶)技术和相关的辅助技术(例如酶工程手段和分子模拟等)已经取得了长足的进步,但若想实现新型的蛋白质(酶),特别是自然界不存在的蛋白质(酶)的改造和设计,其难度仍然非常大。这主要是由于包括蛋白质结构与功能理论、进化理论,蛋白质工程手段(点突变技术)、有机化学理论、量子化学理论和计算机科学等在内的各学科的理论和技术没有系统、有机地融合。例如研究酶催化机制的文献虽然比较多,但实际上大量的研究成果多停留在理论水平。这主要是由于机制研究对科学性严谨性的高要求,酶催化机制研究往往比较理想化和理论化,与实际脱节,从而使得研究过程付出昂贵的造价和冗长的时间周期[15],最后研究所得的机制成果却很难得以实际应用;因此,多学科有效融合是克服传统蛋白质(酶)改造和设计缺陷的有效途径之一。
2新型蛋白质(酶)的改造和设计思路
改造和设计新型蛋白质(酶),特别是自然界不存在的全新酶种,需要突破传统理论的诸多束缚,更要强化多学科理论和技术的有效融合。近年来,生物酶学理论得到了迅速的发展,在新型酶的改造和设计中,出现了把酶蛋白的折叠fold模式与酶活性中心分离分别加以研究的设计思路。该思路把酶的进化理论与折叠模式理论有机地结合起来,指出如果酶活性中心氨基酸与折叠模式(如TIM桶)在进化上不存在必然的联系,即折叠模式中活性中心氨基酸占有率越低,则这种折叠骨架即为可进化的折叠,反之则为不可进化的折叠(如二氢叶酸还原酶折叠DHFR)(图1)。
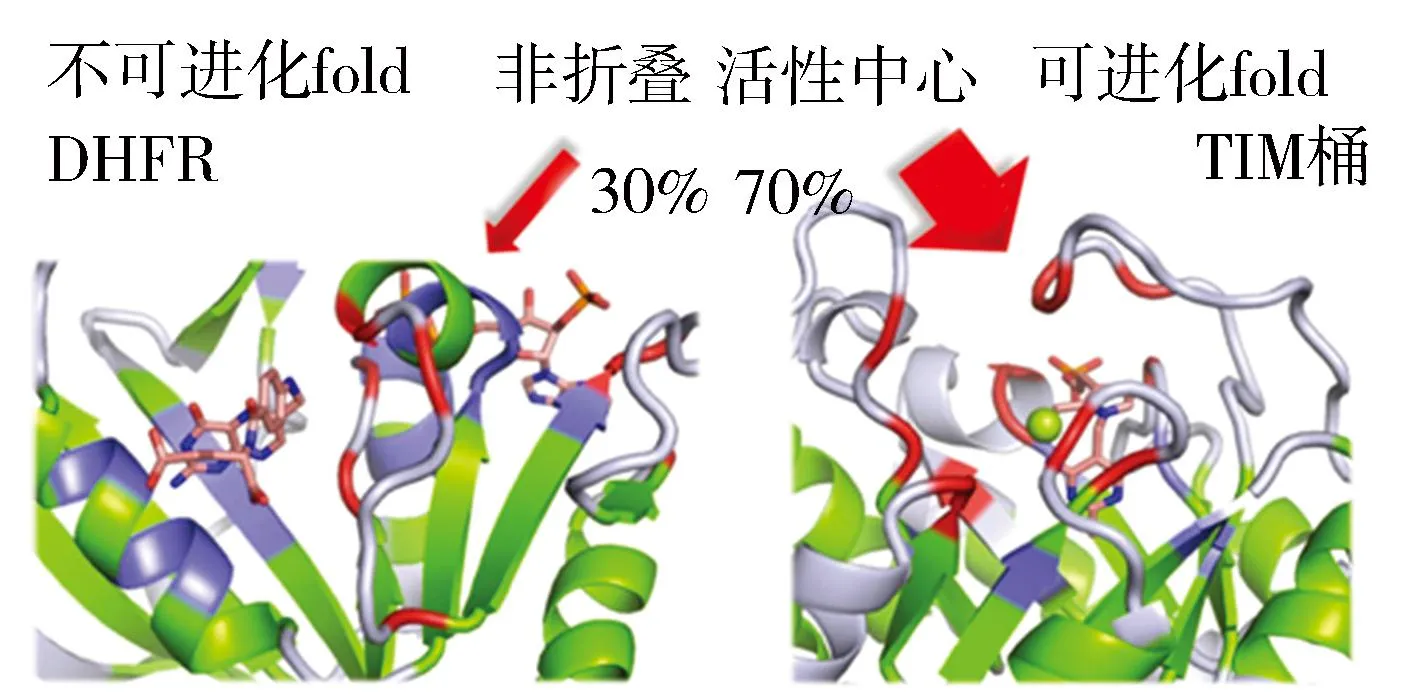
图1 可进化与不可进化折叠模式
生命体如何选择与特定进化功能(活性中心)匹配的蛋白质骨架则与其生理功能相关:如果在生理上蛋白质的某种功能要求持续存在,则支持这种生理功能长期发挥稳定性作用的蛋白质骨架会被选择[16-17]。自然界进化这一原则表明,可将某一生理(如酶催化)功能关键结构(酶活性中心)安装在性能达到工业生产要求的蛋白质骨架上,实现可工业应用的新型酶的改造和设计。在该理论的指导下,可先提取酶的活性中心theoz -yme(即完成催化反应所需的必需氨基酸),然后将其与决定酶优良特性(高稳定性、可溶性、高表达量、耐酸碱等)的蛋白骨架进行匹配,最后选择匹配较好、酶学属性更强的新型酶进行实际生产,从而获得更具工业应用价值的新型酶种(图2)。
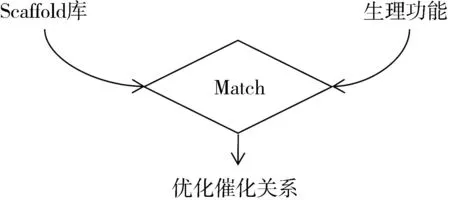
图2 新型酶的改造和设计思路
3新型酶的改造和设计工具及流程
3.1酶改造和设计的常用软件
随着各种优秀算法、软件和更廉价的高配置计算机服务器的出现,生物信息学预测的精度大大提高,为理论和实践开辟了另一片广阔的天空。与酶改造和设计相关的模拟软件有Namd, Discovery Studio、Amber、Autodock、Threader、Pymol、Vmd、Ligandscout、Rosetta、Foldit、Chem3d、Chemdraw、Mopac、Foldx、Triton、Mole和Meme Suite等[18-20]。这些软件各有侧重,掌握这些软件的原理和使用方法,不但有助于深刻理解酶学的各种理论,更有助于减少不必要的实验,提高实验的成功率。
3.2新型酶改造和设计的Rosetta和Foldit软件
目前国际上盛行的蛋白质(酶)设计软件是Rosetta和Foldit[21]。这2种软件涵盖了蛋白质折叠研究、新酶设计和抗体识别设计等,同时也代表了2种不同的蛋白质改造和设计思想。Rosetta是一种全计算机支持的蛋白质改造和设计软件,而Foldit是一种基于人脑直觉的软件。有时人脑的一个简单的直觉,是很难靠计算机完成的。Rosetta的成功经典案例很多,如:2008年第1个由Rosetta成功设计出可应用于生产的Kemp elimination的酶[22];同年Retro-Aldol (醛缩)酶设计成功;2010年Diels-Alder Reaction(双烯合成)酶也设计成功[23];2012年Khare等用Rosetta第一个设计了用于水解有机磷的含Zn金属酶[24]。虽然Rosetta的成功案例逐年增多,但基于人脑直觉的蛋白质Foldit游戏设计方法发展也很迅速。就如同玩游戏般的改造和设计蛋白质,不但吸引了全世界蛋白质科研爱好者的广泛参与,也频有高水平的研究成果出现。如最近Eiben等利用Foldit游戏玩家的灵感,完成了增强双烯合成酶活性的研究,相关成果[25]发表在Natrue Biotechnology杂志上。
由于Foldit是根据人脑直觉进行设计的,所以没有具体的改造和设计流程。Rosetta的新酶改造和设计有一套比较固定的操作流程:首先通过分析提取已有或是从头设计某一催化功能活性中心的结构三维模型,利用生物信息学计算获得该三维模型完成催化所必需的各种约束条件;再将该模型装配到(通过构象搜索到的可匹配的)性能优良的蛋白骨架中(如TIM桶),经分子模拟优化获得一系列氨基酸序列;最终通过微生物高效异源表达进行实验验证。与传统的从序列到结构的改造和设计方法相比,Rosetta这种新型改造和设计方法是从设计底物反应到设计结构模型到最终序列的逆过程,由于有大量生物信息学计算参与,因而更具灵活性和创造性(图3)。
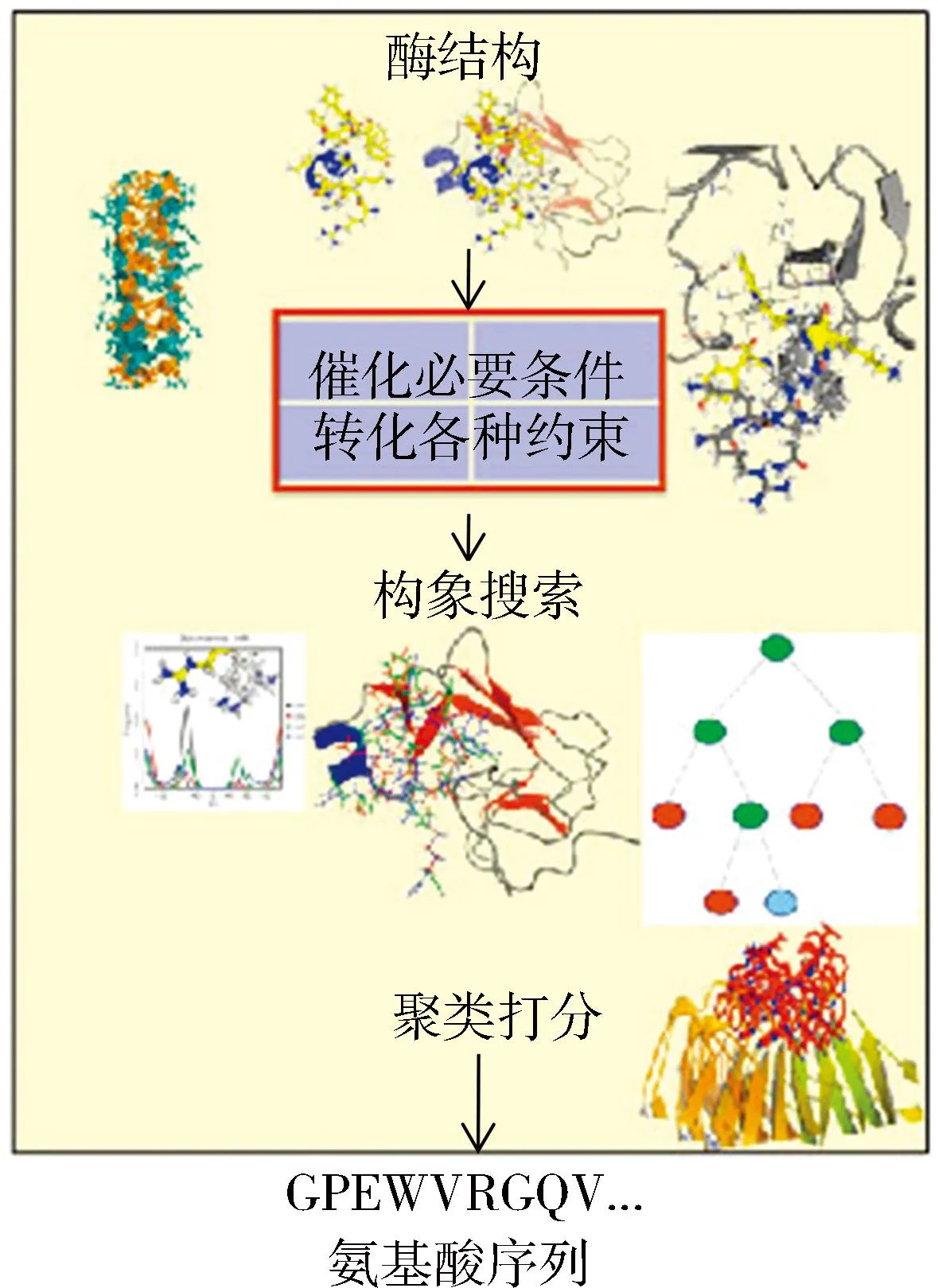
图3 Rosetta新型蛋白质(酶)设计
4国际上蛋白质(酶)改造和设计的前沿团队
巨大的工业需要促使国际知名团队对蛋白质(酶)改造和设计展开了深入的研究。1991年Hellinga等[26]利用自己开发的Dezymer蛋白设计软件成功设计了一个类超氧化物歧化酶。目前在此领域较前沿的团队有美国华盛顿大学Baker团队、美国普林斯顿大学Floudas团队、以色列魏兹曼研究所Tawfik团队和加利福尼亚大学戴维斯分校Florian Richter团队等。David Baker团队主要围绕蛋白质结构预测、新酶设计和抗体设计等蛋白质方向进行研究,目前已在分子模拟、量子化学计算、Rosetta软件设计和蛋白结晶方向取得丰硕的成果,特别是改造和设计自然界不存在的酶,如Kemp elimination 酶[22]、Diels-Alder Reaction酶[23]等;Dan S. Tawfik团队是以色列著名的酶学研究团队之一,该团队提出的根据蛋白质序列进化关系系统设计点突变的理论和方法,经常能够得到酶活与稳定性成百倍提升的正向结果,并在此基础上揭示了蛋白质进化的理论[27];Jens Erik Nielsen团队在耐(嗜)酸碱性改造中发展出了一套成功的理论,他们所开发的pKaTool和pKD软件等,能随意设计酶分子中任一位置氨基酸的pKa值,通过计算可预测出该酶的pH谱,这对有些需要进行酸碱改造的酶工程提供了一种有效的理论和方法[28]。
5展望
酶的酶学属性与工业属性有时并不完全相同,而酶学的理论研究更是难以准确实现酶工业属性的改造,因此利用蛋白质分子改造和设计方法来设计、改变或加强酶的某一工业属性(如耐酸碱性、稳定性和酶活性分子改造等)显得尤为重要。了解国际上最前沿的蛋白质分子改造和设计的相关理论,是我国蛋白质(酶)学科快速发展的必要途径。目前从大分子酶来设计小分子(如抑制剂)的理论与成功的例子较多,但是对于跨学科逆向设计和基于小分子设计新型大分子生物酶,由于其理论和技术都很复杂,国外也还是刚刚开始起步,且大多集中在医药领域,我国在该领域方面起步也比较晚;因此更应加大投入,争取在该领域迎头赶上。
参考文献
[1]Nanda V, Koder R L. Designing Artificial Enzymes by Intuition and Computation[J]. Nature Chemistry, 2009, 2(1):15.
[2]Koga N, Tatsumi-Koga R, Liu G, et al. Principles for Designing Ideal Protein Structures[J].Nature,2012,491(7423):222.
[3]Siegel J B, Smith A L, Poust S, et al. Computational Protein Design Enables a Novel One-carbon Assimilation Pathway[J]. Proceedings of the National Academy of Sciences, 2015, 112(12):3704.
[4]Gonen S, DiMaio F, Gonen T, et al. Design of Ordered Two-dimensional Arrays Mediated by Noncovalent Protein-protein Interfaces[J]. Science, 2015, 348(6241):1365.
[5]De Santis E, Ryadnov M G. Peptide Self-assembly for Nanomaterials: the Old New Kid on the Block[J]. Chemical Society Reviews, 2015,44(22):33.
[6]Christopher J A, Aves S J, Bennett K A, et al. Fragment and Structure-Based Drug Discovery for a Class C GPCR: Discovery of the mGlu5 negative allosteric modulator HTL14242 (3-chloro-5-[6-(5-fluoropyridin-2-yl) pyrimidin-4-yl] benzonitrile) [J]. Journal of Medicinal Chemistry, 2015, 58(16):6653.
[7]Burgess N C, Sharp T H, Thomas F, et al. Modular Design of Self-Assembling Peptide-Based Nanotubes[J]. Journal of the American Chemical Society, 2015, 137(33):10554.
[8]Loquet A, Sgourakis N G, Gupta R, et al. Atomic Model of the Type III Secretion System Needle[J].Nature,2012, 486(7402):276.
[9]Gordon S R, Stanley E J, Wolf S, et al. Computational Design of an α-Gliadin Peptidase[J]. Journal of the American Chemical Society, 2012, 134(50):20513.
[10]Moult J. A decade of CASP: Progress, Bottlenecks and Prognosis in Protein Structure Prediction[J]. Current Opinion in Structural Biology, 2005, 15(3):285.
[11].England L, Jiang T.Reliability of Cu Wire Bonding to Al Metallization[C]//In: Electronic Components and Technology Conference. Las Vegas: IEEE, 2007: 1604.
[12]Borgo B, Havranek JJ. Motif‐directed Redesign of Enzyme Specificity[J]. Protein Science, 2014, 23(3):312.
[13]胡晓佳, 李强, 丛进阳. 重组大肠杆菌共表达D-海因酶和N-氨甲酰基-D-氨基酸酰胺水解酶[J]. 过程工程学报, 2006, 6(6):948.
[14]Huisman G W, Collier S J. On the Development of New Biocatalytic Processes for Practical Pharmaceutical Synthesis[J]. Current Opinion in Chemical Biology, 2013, 17(2):284.
[15]Boone C D, Pinard M, McKenna R, et al. Catalytic Mechanism of α-class Carbonic Anhydrases: CO2Hydration and Proton Transfer. In: Carbonic Anhydrase: Mechanism, Regulation, Links to Disease, and Industrial Applications[M]. Gainesville:Springer, 2014: 31.
[16]Dellus-Gur E, Toth-Petroczy A, Elias M, et al. What Makes a Protein Fold Amenable to Functional Innovation Fold Polarity and Stability Trade-offs[J]. Journal of Molecular Biology, 2013, 425(14):2609.
[17]Tokuriki N, Tawfik D S. Stability Effects of Mutations and Protein Evolvability[J]. Current Opinion in Structural Biology, 2009, 19(5):596.
[18]Phillips J C, Braun R, Wang W, et al. Scalable Molecular Dynamics with NAMD[J]. Journal of Computational Chemistry, 2005, 26(16):1781.
[19]Kumar V A, Mohan K. In Silico Analysis of Indoles Against 1KE8 Inhibitors Using Autodock[J]. British Journal of Pharmaceutical Research, 2013, 3(3):446.
[20]Khoury G A, Liwo A, Khatib F, et al. a Coopetition for Protein Structure Prediction[J]. Proteins: Structure, Function, and Bioinformatics, 2014, 82(9):1850.
[21]Khatib F, Cooper S, Tyka M D, et al. Algorithm Discovery by Protein Folding Game Players[J]. Proceedings of the National Academy of Sciences, 2011, 108(47):18949.
[22]Röthlisberger D, Khersonsky O, Wollacott AM, et al. Kemp Elimination Catalysts by Computational Enzyme Design[J]. Nature, 2008, 453(7192):190.
[23]Siegel J B, Zanghellini A, Lovick H M, et al. Computational Design of an Enzyme Catalyst for a Stereoselective Bimolecular Diels-Alder Reaction[J]. Science, 2010, 329(5989):309.
[24]Khare S D, Kipnis Y, Takeuchi R, et al. Computational Redesign of a Mononuclear Zinc Metalloenzyme for Organophosphate Hydrolysis[J]. Nature Chemical Biology, 2012, 8(3):294.
[25]Eiben C B, Siegel J B, Bale J B, et al. Increased Diels-Alderase Activity Through Backbone Remodeling Guided by Foldit Players[J]. Nature Biotechnology, 2012, 30(2):190.
[26]Hellinga H W, Richards F M. Construction of New Ligand Binding Sites in Proteins of Known Structure: I. Computer-aided Modeling of Sites with Pre-defined Geometry[J]. Journal of Molecular Biology, 1991, 222(3):763.
[27]Goldsmith M, Tawfik D S. Enzyme Engineering by Targeted Libraries[J]. Methods in Enzymology, 2013, 523:257.
[28]Ludwiczek M L, D’Angelo I, Yalloway G N, et al. Strategies for Modulating the pH-dependent Activity of a Family 11 Glycoside Hydrolase[J]. Biochemistry, 2013, 52(18):3138.
(编校:叶超)
Research Progress of Molecular Modification and Design of Novel Proteins (Enzymes) Based on Bioinformatics
SUN Weifeng, ZHONG Wenjuan, SUN Bin, WU Yuanming, GOU Yun, LIN Quanxiu, CHEN Xianggui, LIU Yi
(SchoolofFoodandBioengineering,FoodBiotechnologyLaboratoryofSichuanUniversities,XihuaUniversity,Chengdu610039China)
Abstract:The molecular modification and design of proteins (enzymes) are major challenges for human beings in the face of complex nature. They happened in accident incidents and adhered to inevitable law in the natural evolution of proteins, which is meaningful for the protein industry. In this paper, new strategies and the implementation process for the molecular modification and design of novel biological enzymes are introduced, as well as some advanced research teams and their achievements, in order to provide reference for proteins molecular modification and design.
Keywords:bioinformatics; proteins molecular modification; enzymes design
doi:10.3969/j.issn.1673-159X.2016.02.013
中图分类号:Q816
文献标志码:A
文章编号:1673-159X(2016)02-0067-5
基金项目:国家自然科学基金(21506172);四川省教育厅项目(15205445);西华大学校重点项目(z1420524);西华大学校重点项目(z1420525);西华大学食品生物技术重点实验室项目(szjj2015-013,szjj2015-014);大学生创新创业训练计划(201510623003,201510623053)。
收稿日期:2015-11-12
第一作者:张伟峰(1982—),女,讲师,博士,主要研究方向为食品生物技术。
·生物工程·
