浓香型白酒窖池窖泥中原核微生物菌群的研究
2016-04-14张宿义敖宗华王松涛税梁扬李德林泸州龙泉窖酒业有限公司四川泸州66606泸州老窖股份有限公司四川泸州66000国家固态酿造工程技术研究中心四川泸州66000泸州科源生物科技有限公司四川泸州66606
罗 杰,张宿义,,敖宗华,王松涛,税梁扬,杨 艳,李德林,张 兵(.泸州龙泉窖酒业有限公司,四川泸州66606;.泸州老窖股份有限公司,四川泸州66000;3.国家固态酿造工程技术研究中心,四川泸州66000;.泸州科源生物科技有限公司,四川泸州66606)
浓香型白酒窖池窖泥中原核微生物菌群的研究
罗杰1,张宿义1,2,3,敖宗华2,3,王松涛2,税梁扬4,杨艳4,李德林4,张兵4
(1.泸州龙泉窖酒业有限公司,四川泸州646606;2.泸州老窖股份有限公司,四川泸州646000;3.国家固态酿造工程技术研究中心,四川泸州646000;4.泸州科源生物科技有限公司,四川泸州646606)
摘要:提取浓香型窖池窖壁泥和窖底泥样品总DNA,构建窖泥细菌16S rRNA文库,经过16S rRNA扩增片段的限制性酶切分析(ARDRA)、DNA序列测定和系统发育关系分析,对比研究了窖壁泥和窖底泥细菌群落组成。数据分析结果表明,窖池窖壁泥和窖底泥中细菌种类及数量的差异性较大。窖壁泥中优势菌属为Clostridiumsp、Caloramatorsp和Desulfotoma- culum carboxydivorans,所占比例分别为17%、8%和6%。窖底泥优势菌属为Clostridium acidurici、Ruminococcus bromii、Proteiniphilumsp和Parabacteroides merdae,所占比例分别为18%、8%、7% 和6%。初步揭示了窖池窖壁泥和窖底泥中细菌系统发育多样性及相互之间存在的差异。
关键词:窖泥;16S rRNA序列;克隆文库;白酒
窖泥在浓香型白酒发酵过程中具有重要的作用,它是窖泥功能菌生长繁殖的载体,也是浓香型白酒呈香呈味物质生成的主要场所。因此研究浓香型白酒窖泥微生物的菌群组成和动态变化,对于进一步分离和开发应用窖泥微生物资源、了解窖泥微生物与白酒发酵之间的关系具有重要的意义。目前,大多数研究主要还是采用传统的微生物培养结合分子生物学手段,从可培养的角度针对不同窖龄、不同地域的窖泥中可培养微生物的数量、群落组成及动态变化进行了较为系统的研究[1-2],并且分离、鉴定出很多以前从未报道的窖泥微生物[3-6]。
然而,由于窖泥中可分离培养的微生物仅占窖泥微生物的极少数,因此采用传统分离培养方法难以全面的反映窖泥生态系统微生物群落组成及演替规律。近年来,随着微生物生态学的发展,分子生物学技术越来越多的应用于窖泥微生物群落结构和动态变化规律的研究,利用16S rRNA技术对窖泥中未培养微生物的多样性及系统发育的研究得以飞速发展[7-14]。
窖池不同空间位置窖泥所处的微生态不尽相同,窖泥中微生物群落存在典型的空间异质性,不同层面窖泥的生态因子也存在一定的差异。这种差异影响着窖泥中微生物的繁殖与代谢,对中国浓香型白酒风味的形成起着重要作用。鉴于此,本研究以窖池窖壁泥和窖底泥为对象,利用16S rRNA技术构建窖泥原核微生物克隆文库,探索不同层面窖泥中细菌群落构成,并结合相关微生物的特性对窖池生香原理进行讨论,为探索浓香型白酒风味物质形成机理提供了一定科学依据。
1 材料与方法
1.1样品采集
窖泥样品:采集自某浓香型名酒企业酿酒车间。窖壁泥的取样方法是从该窖池四周窖壁接近中心一点各取5 g窖泥,将窖壁泥混合均匀,分装后密封冷冻保存。窖底泥的取样方法是从窖底四个角落和接近中心点各取5 g窖泥,将窖底泥混合均匀,分装后密封冷冻(-80℃)保存。
1.2试剂与仪器
DNA提取试剂盒,dNTP,Taq DNA聚合酶,HinfI和Csp6I购自上海生工生物公司;X-gal(50mg/mL),IPTG (0.5 mM),氨苄青霉素(50mg/mL),Tripton(4%);PCR扩增引物由上海生工生物公司合成。
高速冷冻离心机(SIGMA),DyNA Quant 200浓度测定仪(美国,PHARMACIABIOTECH),核酸电泳仪(BIORAD),GDS凝胶成像系统(英国,UVP),台式离心机(上海安亭仪器厂),PCR仪(美国PE公司,MJ),人工气候培养箱(哈尔滨东明医疗仪器厂),自动灭菌锅(日本,SANYO),超低温冰箱(-80℃)(美国,FORMA SCIENTIFIC),电子天平(美国,SARTORIUS)。
1.3窖泥样品的预处理和总DNA提取
取1~2 g窖泥样品,用5 mL 0.1 M的磷酸缓冲液(pH7.0)悬浮,加入玻璃珠,漩涡振荡器充分振荡5 min。200×g离心5 min,收集上清液(含菌体),弃去沉淀,洗涤3次,200×g离心5 min,收集上清液(含菌体),弃去沉淀。9000×g高速离心5 min,弃上清液,收集沉淀(菌体)。收集的菌体用5 mL 0.1 M的磷酸缓冲液(pH7.0)悬浮,9000×g离心5 min,洗涤3次。洗净的菌体悬浮在5 mL磷酸缓冲液(pH7.0)中,用枪吹打后漩涡振荡器振荡均匀。然后按试剂盒上的操作步骤提取窖泥DNA。
1.4细菌16S rRNA基因克隆文库的构建与测序与比对
细菌16S rRNA全长的PCR扩增所用的引物采用Di CelloF等所用的引物,可以扩增16S rRNA约1.5 kb的片段。序列如下;P0:5'-GAGAGTTTGATCCTGGCTCAG-3';P6:5'-CTACGGCTACCTTGTTACGA-3'。PCR扩增条件为:94℃变性4 min,接下来30个循环,分别是94℃变性45 s,56℃退火45 s,72℃延伸1 min 45 s,最后一个循环72℃延伸10 min。PCR产物用1%琼脂糖凝胶电泳分离检验,电压约11 V/cm,电泳时间30 min。通过胶回收试剂盒回收纯化,纯化后的PCR产物连接到pGEM-T easy vector载体上。再将其转化到大肠杆菌DH10B化学感受态细胞内,涂布于加有100µL 0.5 Mm IPTG和25µL 50mg/mL的X-Gal的Amp选择性平板上涂板。37℃培养16 h后,用灭菌的牙签随机挑取192个窖壁泥细菌阳性克隆子编号50B(1-192)和192个窖底泥细菌阳性克隆子编号50D(1-192),加入2 mL EP管中(含有Amp 的LB液体培养基),37℃过夜培养,长出单菌落后放4℃备用。再通过菌落PCR确定阳性克隆子,用移液枪吸取10µL菌液悬于20µL 0.5% Tripton-100中,室温静置10 min,离心机6000×g离心1 s,使用时取3µL上清液作为模板。引物对T7(5'-GGCCGCGGGAATTC GATT-3')/SP6(5'-GCGAATTCACTA GTGATT-3'),扩增程序同上。用限制性内切酶HinfI(MBI)和Csp6I(promega)消化提取的重组质粒50B(1-192号)和50D(1-192号)。酶切反应均在37℃下水浴消化3 h,酶切产物用1.5%琼脂糖凝胶进行分离,电压11 V/cm,时间45 min,EB染色。根据酶切图谱对克隆进行分型,综合比较2种内切酶酶切结果,对窖壁泥和窖底泥分别选取84个和80个阳性克隆子,委托上海捷瑞生物技术有限公司进行序列测定。
得到的序列通过Genebank(http://www.ncbi.nlm.nih.gov/BLAST/)进行比较鉴定,寻找与目的序列同源性较高且通过聚类分析能聚到一类的已知分类地位的菌种的16S rRNA序列。通过BioEdit和Clustal X程序多重比对后用Mega5.05软件Neighbor-Joining法进行1000次步长计算构建系统发育树图[16-17]。
2 结果与分析
将测得的细菌16S rRNA序列与数据库进行比对。每一条16S rRNA基因文库内的序列均与数据库中相似性较高的序列进行聚类分析,然后选择聚类的自展支持率较高的序列。结果表明,克隆子与数据库内参比序列间相似度较高,达到80%~100%,它们大多数与土壤、堆肥、厌氧生物反应器等环境中的不可培养微生物或可培养微生物相关。由于分析的序列较多,针对窖壁泥和窖底泥细菌16S rRNA基因文库内的厚壁菌门克隆子分别构建了系统发育树(见图1、图2)。
两个克隆文库中都检测到厚壁菌门、拟杆菌门和放线菌门的微生物,而且厚壁菌门所占比例是最高的。变形菌门仅在窖壁泥中鉴定出,Synergistetes仅存在于窖底泥中。研究表明窖壁泥和窖底泥菌群结构存在明显差异(各门所占比例详见图3)。
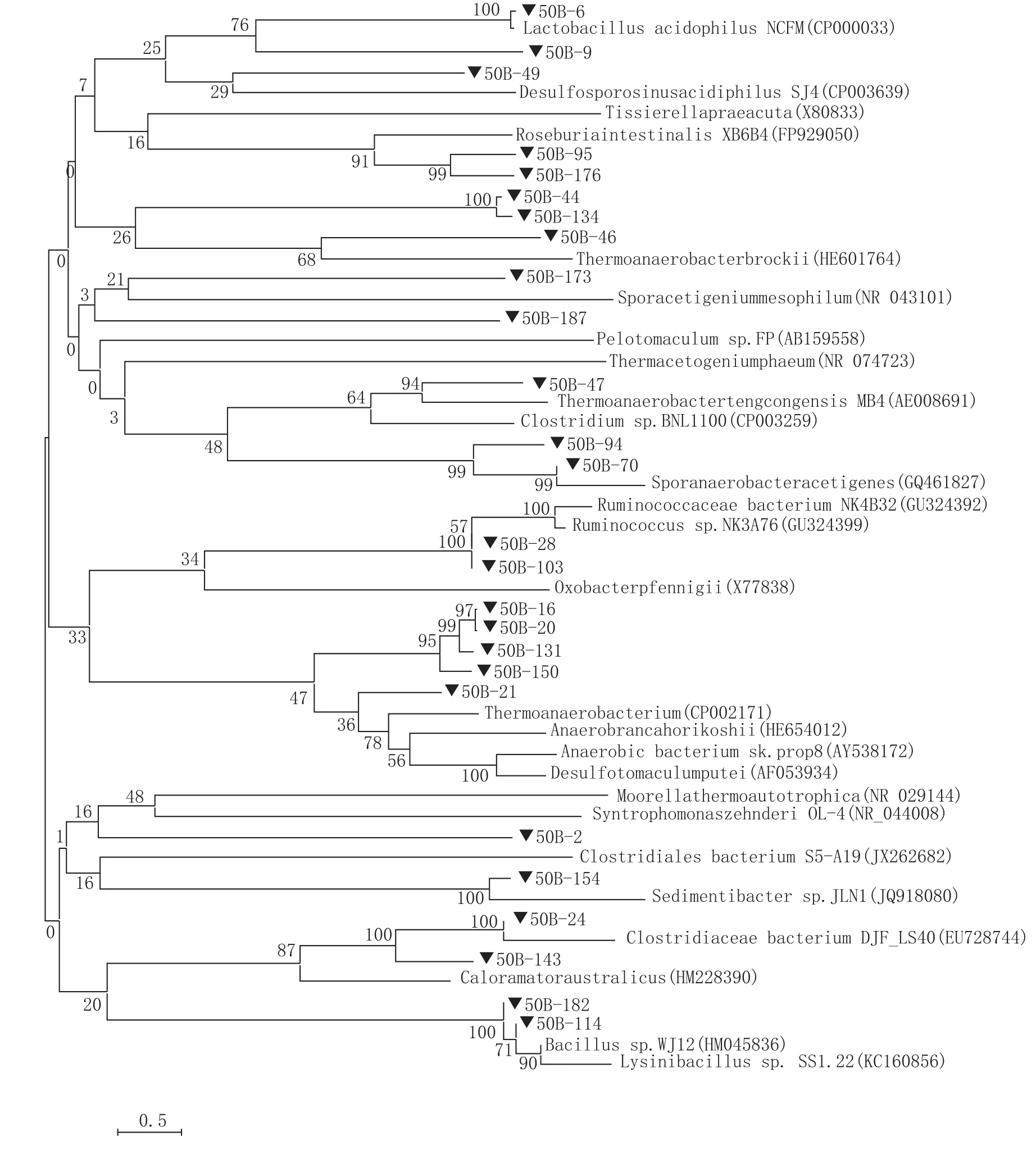
图1 基于厚壁菌门克隆子序列构建的窖壁细菌16S rRNA基因系统发育树
窖壁文库得到37个OTUS,主要分布于16科和7个科级分类未知的梭菌纲和拟杆菌纲类群内。窖底文库得到35个OTUS,主要分布于14个科和9个科级分类未知的梭菌纲类群内。窖壁泥中的优势菌属为Clostridium sp.、Caloramatorsp和Desulfotomaculum carboxydivorans,窖底泥中的优势菌属为Clostridium acidurici、Ruminococcus bromii、Proteiniphilumsp和Parabacteroides merdae(详见表1)。
窖壁和窖底克隆文库中克隆子16S rRNA序列的比对结果见表2。
从表2可知,窖泥中微生物种类较多,其中大部分微生物在窖壁泥和窖底泥中均有分布。对存在于窖壁泥中微生物主要功能特性进行分析发现:肠杆菌属(Enterobacter)兼性厌氧,发酵葡萄糖,产酸产气(通常CO2∶H2=2 ∶1);醋杆菌属(Acetobacter)能够在有空气的条件下将乙醇转化为乙酸;假黄色单胞菌属(Pseudoxa-nthomonas)嗜热厌氧具有降解亚硝酸盐的能力;嗜酸栖热菌属(Acidothermus)最适生长温度55℃,能分泌糖苷水解酶和碳水化合物酯酶,这些水解酶能够水解植物细胞壁和其他的一些碳水化活物;纤维菌属(Cellulomonas)化能异养菌,可呼吸代谢也可发酵代谢,在好氧和厌氧条件下都能利用葡萄糖和其他碳水化合物产酸;Moorella嗜热微生物,能产乙酸;Unclassified Ruminococcaceae能降解纤维素;乳酸菌(Lactobacillus)发酵分解糖代谢,终产物中50%以上是乳酸,最适生长温度30~40℃。

图2 基于厚壁菌门克隆子序列构建的窖底细菌16S rRNA基因系统发育树
针对存在于窖底泥中微生物主要功能特性进行分析发现:Aminobacterim与甲烷菌存在共生关系[18-21];Unclassified Peptostreptococcaceae营养需求复杂,可发酵氨基酸或碳水化合物产生低级脂肪酸、二氧化碳和氢,有时可产生琥珀酸或乙醇,通过异型发酵形成乳酸,但一般不是主要产物;Alkalitalea可发酵甘油、果糖和乳酸。
3 讨论
通过对窖壁泥和窖底泥克隆文库的分析,厚壁菌门(Firmicute)和拟杆菌门(Bacteroidetes)均为窖壁泥和窖底泥中两大优势菌群。其中窖壁泥中有少量的变形菌门克隆子,窖底泥中存在一定的Synergistetes门克隆子。窖壁泥和窖底泥中优势菌属存在一定的差异:窖壁泥中的优势菌属为Clostridiumsp、Caloramatorsp和Desulfo-tomaculum carboxydivorans,所占比例分别为17%、8% 和6%。窖底泥中的优势菌属为Clostridium acidurici、Ruminococcus bromii、Proteiniphilumsp和Parabacteroides merdae,所占比例分别为18%、8%、7%和6%。

图3 16S rRNA克隆文库中操作分类单元和克隆子在不同门的分布

表1 窖壁泥和窖底泥样品中优势菌属在细菌域的序列比对
长期生产实践表明:由于窖底糟醅与窖泥的接触面积最大,故窖池底部被认为是整窖中生产优质调味酒的主要区域,而中层糟醅及上层糟醅的酒质相对差一些。这或许与窖壁泥、窖底泥微生物差异性有关。
运用16S rRNA技术分析窖壁泥和窖底泥的微生物群落结构,研究传统食品发酵功能微生物的生态特征与规律,将其与传统的菌种分离鉴定和白酒固态发酵检测等手段相结合,对判断和鉴定白酒生产中与特征风味物质相关的关键微生物、指导生产工艺改进,具有重要的理论和实践意义。

表2 浓香型白酒50年窖泥16S rDNA克隆文库中窖壁和窖底克隆子在不同属的分布
参考文献:
[1]汪江波,万朕,李莉,等.稻花香窖泥微生物群落变化研究[J],酿酒科技,2010(11):36-39.
[2]易彬,任道群,唐玉明,等.不同窖龄窖泥微生态变化研究[J].酿酒科技,2011(6):32-34.
[3]王岩,吴衍庸.浓香型酒窖中生丝微菌的分离与特性[J].微生物学通报,1991,18(2):75-78.
[4]刘莉萌,张斌,东秀珠.浓香型白酒窖池中片球菌的分离与鉴定[J].酿酒科技,2007(2):22-25.
[5]王俪鲆,张良,刘来雁,等.泸州古酿酒窖池中两株产甲烷杆菌比较研究[J].应用与环境生物学报,2010,16(6):840-844.
[6]张霞,武志芳,张胜潮,等.贵州浓香型白酒窖池可培养细菌系统发育分析[J].酿酒科技,2010(12):23-27.
[7]赵辉,敞颜,王葳,等.浓香型白酒窖泥中高产己酸兼性厌氧细菌的分离鉴定[J].食品科学,2012,33(5):177-182.
[8]刘基银.人工窖泥培养方法[J].酿酒科技,2006(8):129-130.
[9]胡承,应鸿,许德富,等.窖泥微生物群落的研究及其应用[J].酿酒科技,2005(3):34-38.
[10]游玲,王松,冯瑞章,等.宜宾产区浓香型白酒窖房空气、窖泥和糟醅可培养细菌的相关性[J].食品科学,2012,33(9):188-192.
[11]王涛,田时平,赵东,等.浓香型白酒窖泥、出窖糟醅细菌区系的相似性分析[J].食品科学,2012,33(7):193-197.
[12]张文学,向文良,乔宗伟,等.浓香型白酒窖池糟醅原核微生物区系的分类研究[J].酿酒科技,2005(7):22-26.
[13]向文良,张文学,胡承,等.中国浓香型白酒窖池中原核微生物的特性及系统发育分析[J].四川大学学报:工程科学版,2005,37(1):39-42.
[14]唐云容,钟方达,张文学.浓香型酒窖泥微生物菌群多样性及系统发育分析[J].酿酒科技,2011(12):22-26.
[15]王海英,张文学,施思,等.水井坊窖底泥微生物群落结构的DGGE分析[J].中国酿造,2012,31(2):38-41.
[16]Thompson J D,Gibson T J,Plewniak F,et al.The Clustal X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Res,1997,25(24):4876-4882.
[17]Kumar S,Tamura K,Nei M.MEGA3:integrated software for molecular evolutionary genetics analysis and sequence alignment[J].Brief Bioinform,2004,5(2):150-163.
[18]Sekiguchi Y,Imachi H,Susilorukmi A,et al.Tepidanaerobacter syntrophicus gen.nov.,sp.nov.,an anaerobic,moderately thermophilic,syntrophic alcohol and lactat-edgrading bacterium isolated from thermophilic digested sludges[J].International Journal of Systematic and Evolutionary Microbiology,2006,56:1621-1629.
[19]Han C,Mwirichia R,Chertkov O,et al.Complete genome sequence of Syntrophobotulus glycolicus type strain (FlGlyR)[J].Standards in Genomic Sciences,2011,4(3):371-80.
[20]Qiu Y L,Sekiguchi Y,Hanada S,et al.Pelotomaculum terephthalicum sp.nov.and Pelotomaculum isophthalicum sp.nov.:two anaerobic bacteria that degrade phthalate isomers in syntrophic association with hydrogenotrophic methanogens[J].Archives of microbiology,2006,185(3):172-182.
[21]Baena S,Fardeau M L,Labat M,et al.Aminobacterium mobile sp.nov.,a new anaerobic amino-acid-degrading bacterium[J].International Journal of Systematic and Evolutionary Microbiology,2000,50 (Pt1):259-64.
Prokaryotic Bacteria Communities in Pit Mud of Nongxiang Baijiu
LUO Jie1,ZHANG Suyi1,2,3,AO Zonghua2,3,WANG Songtao2,SHUI Liangyang4,YANG Yan4,LI Delin4and ZHANG Bing4
(1.Luzhou Longquanjiao Distillery Co.Ltd.,Luzhou,Sichuan 646606;2.Luzhou Laojiao Co.Ltd.,Luzhou,Sichuan 646000;3.National Engineering Technology Research Center of Solid-state Brewing,Luzhou,Sichuan 646000;4.Luzhou Keyuan Biological Technology Co.Ltd.,Luzhou,Sichuan 646606,China)
Abstract:Total DNA were extracted from pit wall mud and pit bottom mud for Nongxiang Baijiu production for the construction of clone libraries of 16S rRNA.The bacteria composition and diversity in pit mud between pit wall and pit bottom were contrasted by amplified 16S rRNA restriction analysis (ARDRA),DNA sequencing and phylogenetic analysis.The results showed that there was significant difference in the varieties and the quantity of bacteria from pit wall mud and pit bottom mud,the dominant bacteria species in pit wall mud included Clostridiumsp,Caloramatorsp and Desulfotomaculum carboxydivorans (17%,8% and 6% of total bacteria quantity),the dominant bacteria species in pit bottom mud included Clostridium acidurici,Ruminococcus bromii,Proteiniphilumsp and Parabacteroides merdae (18%,8%,7% and 6% of total bacteria quantity).This research preliminarily revealed the difference in bacteria diversity between pit wall mud and pit bottom mud.
Key words:pit mud;16S rRNAsequence;clone library;Baijiu
通讯作者:敖宗华(1971-),男,博士,硕士生导师,高级工程师,发表学术论文数十篇,E-mail:aozh@lzlj.com.cn。
作者简介:罗杰(1989-),男,四川荣县人,硕士,主要从事酿酒生物技术与应用。
收稿日期:2015-10-16
中图分类号:TS262.3;TS261.4;TS261.1
文献标识码:A
文章编号:1001-9286(2016)02-0055-06
DOI:10.13746/j.njkj.2015404
优先数字出版时间:2015-12-30;地址:http://www.cnki.net/kcms/detail/52.1051.TS.20151230.0923.005.html。
