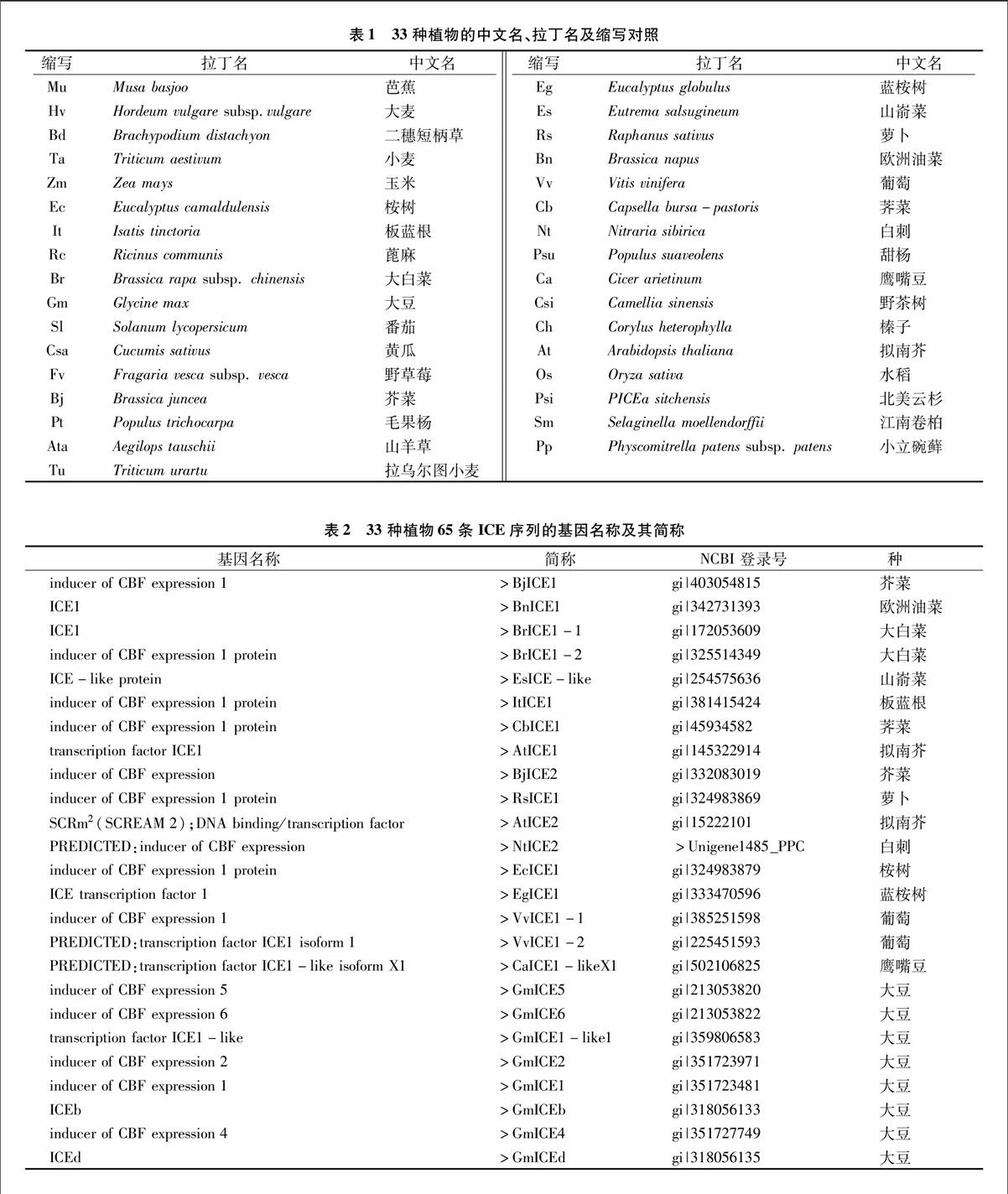
植物ICE蛋白基因家族的系统进化分析
2016-04-11陈露杨立明罗玉明
陈露 杨立明 罗玉明


摘要:ICE是植物体内的一类bHLH转录因子,在植物低温胁迫中发挥重要作用。本研究利用来自不同物种包括苔藓、蕨类、裸子、被子植物的ICE蛋白序列,用Clustal X程序对其氨基酸序列进行比对,并用邻近法构建系统进化树,进一步了解ICE家族在植物界的分子进化情况。结果表明,ICE蛋白家族基本上按苔藓、蕨类、裸子、单子叶和双子叶植物而分别聚类,尽管不同来源的ICE序列有一定差异,但HLH结构域在各植物物种中具有很好的保守性,这说明ICE蛋白在进化上具有保守性;被子植物的ICE形成不同的分支,单子叶植物和双子叶植物原始的ICE蛋白有可能不同。
关键词:植物;ICE;序列比对;系统进化分析
中图分类号: Q943.2文献标志码: A
文章编号:1002-1302(2016)02-0042-06
收稿日期:2015-02-27
基金项目:国家高技术研究发展计划(编号:2013AA102705);国家自然科学基金(编号:30900871);江苏省自然科学基金(编号:(BK2011409)。
作者简介:陈露(1989—),女,江苏靖江人,硕士研究生,从事植物生物技术研究。E-mail:chenlu1201@163.com。
通信作者:罗玉明(1963—),男,江苏涟水人,教授,研究生导师,主要从事植物生物技术研究。Tel:(0517)83525128;E-mail:yumingluo@163.com。
ICE(inducer of CBF expression)是CBF[C-repeat-binding factors,又称DREBs(dehybration responsive element factors)]冷响应通道的上游调控因子,目前主要在拟南芥中发现[WTBX][STBX]ICE1和ICE2[WTBZ][STBZ]这2种ICE基因。ICE一般为组成型表达,常温下没有活性,低温诱导可使[WTBX][STBX]ICE1[WTBZ][STBZ]成活性状态,编码一个类似MYC的螺旋-环-螺旋型(bHLH)转录激活因子,诱导CBF基因的表达,从而提高植物的抗寒性,在转基因植株生长发育过程中没有异常表现[1]。研究表明,ICE2可以直接影响[WTBX][STBX]CBF1[WTBZ][STBZ]基因的表达,将[WTBX][STBX]ICE2[WTBZ][STBZ]基因转入拟南芥发现,拟南芥体内[WTBX][STBX]CBF1[WTBZ][STBZ]基因表达量明显上升,转基因拟南芥可在-20 ℃环境下正常生长[2-3]。由此可见,ICE在植物低温抗性中有着重要的作用。有研究证明,ICE1-CBF转录级联反应在植物响应低温胁迫过程中发挥着至关重要的作用[4]。
目前,依赖CBF的信号转导通路被视为植物低温胁迫应答的主要途径[4-7]。在ICE1-CBF的调控路径中,ICE1在低温诱导下与CBF3启动子顺式作用元件相互结合,并激活CBF3转录,调控CBF及下游抗冷相关基因的转录,也可以与R2R3型MYB转录因子AtMYB15相互作用,负调控CBF基因的表达[2,8]。ICE1蛋白的磷酸化可能涉及到低温胁迫过程中ICE1的活性调控。另外,HOS1编码一种E3连接酶,参与ICE1蛋白的泛素化降解,在低温胁迫过程中,HOS1与ICE1相互作用并降解ICE1,通过ICE1调节CBF的转录。有研究表明,ICE1蛋白在冷胁迫时明显降解,而在HOS1突变体中,ICE1蛋白受冷胁迫降解的程度明显降低[9-10]。
鉴于ICE在植物低温抗性中的重要作用,本研究通过对来源于植物的ICE蛋白进行同源比对,分析系统发生关系,拟找出ICE基因在植物界的系统进化关系,为研究该基因的功能进化提供参考。
1材料与方法
1.1序列信息的获取与筛选
为获得ICE家族的各成员,分别以拟南芥ICE基因的蛋白质序列AtICE1、AtICE2,使用Blastp程序搜索绿色植物数据(http://blast.be-md.ncbi.nlm.nih.gov/Blast.cgi),database选择为non-redundant protein sequences (nr),organism选择为plants(taxid:3193),max target sequences设置为250,其余均为默认参数。对获得的搜索结果用Pfam(http://pfam.xfam.org/)进行分析,把具有与拟南芥ICE基本结构特征的序列保留下来做后续分析。
1.2多序列比对及系统进化树的构建
以氨基酸全序列比对结果为基础,用Mega 5.10软件构建系统进化树[11]。采用Neighbor-joining法运行相应参数[12]:Poisson model模式,缺口设置为Pairwise deletion;校验参数为bootstrap=1 000。系统发生树中,ICE家族各亚族的分组是在相似理论和分类学基础上完成,并用Clustal X软件[13]进行检验。
1.3模体识别
使用MEME模体搜索工具以识别ICE家族相关蛋白质所共有的模体,并对相关参数进行修改,将可找到的模体最大值调整为25,每个模体的最大宽度调整为100,其他均为默认值;MEME在线软件识别的模体通过Smart和Pfam模体识别工具[14-15]进行进一步检验。
2结果与分析
2.1植物ICE基因的鉴定
利用模式植物拟南芥中已测序的2个ICE蛋白基因序列,执行Blastp搜索、关键词搜索、结构域搜索等,以在植物中获得更多的ICE基因;将得到的初始目标序列经过整理、合并、筛选,通过Mega 5.10软件构建系统进化树,并进行逐一比对分析,去除不符合条件的序列,最终确定33个植物(表1)基因组中的65个ICE蛋白质基因(表2)用于分析,其中包括拟南芥2个模板ICE蛋白基因。按照从低等植物到高等植物的顺序依次为:2条来自苔藓植物,即藓纲的小立碗藓;2条来自蕨类植物,即石松纲的江南卷柏;1条来自裸子植物,即松柏纲的北美云杉;21条来自单子叶植物的禾本科和芭蕉科,包括山羊草、小麦、乌拉尔图小麦、水稻、二穗短柄草、大麦、玉米、芭蕉共8种植物;39条来自双子叶植物,包括芥菜、欧洲油菜、蓖麻、大白菜、山嵛菜、板蓝根、荠菜、拟南芥、萝卜、白刺、桉树、蓝桉树、葡萄、鹰嘴豆、大豆、野茶树、番茄、黄瓜、野草莓、榛子、甜杨、毛果杨共22种植物。
2.2序列特异性分析
由图1可见,65个ICE蛋白质基因中,有63、63、61、62、63个基因分别包含模体1、模体2、模体3、模体5和模体6;基因GmICE5、GmICE1-like2缺失模体1,基因GmICE5、GmICE6缺失模体2,基因GmICE5、GmICE6、HvICE2-3、PsiICE1缺失模体3,基因GmICE5、MuICE1-3、MuICE1-1缺失模体5,PpICE1、PpICE2缺失模体6。利用NCBI中Blastp对65个植物的ICE氨基酸序列进行结构域搜索,结合MEME做出蛋白序列25个模体(表3)中的模体2、模体3序列进行分析,并用Smart和Pfam进行检验,结果表明,不同来源的ICE氨基酸序列有一定的差异,但仍有一个保守结构域HLH存在,HLH是ICE氨基酸序列的功能区域,包含3个功能位点,分别为DNA结合区域(核酸结合位点)、E-box (5-CANNTG-3)/N-box (5-CACGC/AG-3)的特异性位点、二聚接口(多肽结合位点)。
2.3植物ICE基因家族的系统发生关系
由图2可见,ICE蛋白家族基本按苔藓、蕨类、裸子、单子叶和双子叶植物而分别聚类,说明该基因在进化上具有保守性;大部分允许ICE蛋白亚族分离的节点,自展值都比较小;自上往下,将ICE家族分为4个亚组,分别被称为Ⅰ、Ⅱ、Ⅲ、
Ⅳ,其中第Ⅰ大亚组包含所有双子叶植物,第Ⅱ大亚组全部为单子叶植物,第Ⅲ大亚组可分为2个小亚组A、B,其中,小亚组A全部为单子叶植物,小亚组B为裸子植物,第Ⅳ大亚组可分为2个小亚组C、D,其中,小亚组C为蕨类植物,小亚组D为苔藓类植物;Ⅲ和Ⅳ位于系统发生树的基部,其在进化地位上是最原始的类型。
3结论
对植物ICE蛋白基因家族进行分析发现,尽管不同来源的ICE序列表现出一定的差异,但HLH结构域在各植物物种中都有很好的保守性,这说明该基因在进化上具有保守性。对双子叶植物来说,大多数同源基因聚类到一起,如 BrICE1-1 和BrICE1-2,VvICE1-1和VvICE1-2,GmICE5、GmICE6、GmICE1-like1、GmICE2、GmICE1、GmICEb、GmICE4和GmICEd,GmICE1-like2和GmICE3,CsiICE1和CsiICE2,PsuICE1和 PsuICE1;对单子叶植物、蕨类植物、苔藓植物来说,也发生同样的现象。这说明植物ICE蛋白基因家族的进化模式, 应归因于物种特定的扩张,也可以说,植物进化历程
中,细胞扩增过程中的基因复制及细胞分化是ICE蛋白基因家族的主要进化模式。因此,进化树分支末端的基因可能是代表最近复制过程中突变的基因[16],这为物种特定扩张学说提供了有力支持。同时,研究发现,被子植物中的ICE基因形成不同的分支,这表明单子叶植物和双子叶植物可能有不同的原始ICE蛋白基因。
[HS2][HT8.5H]参考文献:[HT8.SS]
[1][ZK(#]郑银英,崔百明,常明进,等. 转拟南芥ICE1基因增强烟草抗寒性的研究[J]. 西北植物学报,2009,29(1):75-79.
[2]Chinnusamy V,Ohta M,Kanrar S,et al. ICE1:a regulator of cold-induced transcriptome and freezing tolerance in Arabidopsis[J]. Genes & Development,2003,17(8):1043-1054.
[3]王翠花,刘沙,张瑞富,等. 植物抗寒分子生物学研究概况及展望[J]. 辽宁农业科学,2014(1):45-48.
[4]Chinnusamy V,Zhu J H,Zhu J K. Cold stress regulation of gene expression in plants[J]. Trends in Plant Science,2007,12(10):444-451.
[5]Zhu J H,Dong Ch H,Zhu J K. Interplay between cold-responsive gene regulation,metabolism and RNA processing during plant cold acclimation[J]. Current Opinion in Plant Biology,2007,10(3):290-295.
[6]Chinnusamy V,Zhu J K,Sunkar R. Gene regulation during cold stress acclimation in plants[J]. Methods in Molecular Biology,2010,639:39-55.
[7]Zhou M Q,Shen C,Wu L H,et al. CBF-dependent signaling pathway:a key responder to low temperature stress in plants[J]. Critical Reviews in Biotechnology,2011,31(2):186-192.
[8]Agarwal M,Hao Y J,Kapoor A,et al. A R2R3 type MYB transcription factor is involved in the cold regulation of CBF genes and in acquired freezing tolerance[J]. Journal of Biological Chemistry,2006,281(49):37636-37645.
[9]Dong C H,Hu X Y,Tang W P,et al. A putative Arabidopsis nucleoporin,AtNUP160,is critical for RNA export and required for plant tolerance to cold stress[J]. Molecular and Cellular Biology,2006,26(24):9533-9543.
[10][ZK(#]Dong C H,Agarwal M,Zhang Y Y,et al. The negative regulator of plant cold responses,HOS1,is a RING E3 ligase that mediates the ubiquitination and degradation of ICE1[J]. Proceedings of the National Academy of Sciences of the United States of America,2006,103(21):8281-8286.
[11]蒋瑶,陈其兵. 植物CBF1转录因子的生物信息学分析[J]. 林业科学,2010,46(6):43-50.
[12]王波,孙君社,翟玉盼,等. 植物Ⅲ型聚酮合酶基因家族的分子进化分析[J]. 生物技术通报,2011(1):83-89,94.
[13]金谷雷,汪旭升,朱军. 水稻14-3-3蛋白家族的生物信息学分析[J]. 遗传学报,2005,32(7):726-732.[HJ]
[FK(W46][TPCL222.tif;S+2mm][FK)]
[14][ZK(#]Schultz J C R,Bork P. Smart:a web-based tool for the study of genetically mobile domains[J]. Nucleic Acids Research,2000,28(1):231-234.
[15]Sonnhammer E R,Durbin R. Pfam:a comprehensive database of protein domain families based on seed alignments[J]. Proteins,1997,28(3):405-420.
[16]Xiong Y Q,Liu T Y,Tian C G,et al. Transcription factors in rICE:a genome-wide comparative analysis between monocots and eudicots[J]. Plant Molecular Biology,2005,59(1):191-203.
