苹果遗传图谱的构建与QTL定位研究进展
2016-04-11黄金凤王冬梅闫忠业吕天星
黄金凤++王冬梅++闫忠业++吕天星++王颖达++刘志
摘要:遗传图谱构建及QTL定位对苹果育种具有十分重要的意义,高密度饱和的遗传图谱是有效QTL定位的基础,QTL定位是基因精细定位、克隆及后续指导育种实践的基础。综述了国内外苹果遗传图谱构建和QTL定位的研究进展,其中对图谱构建综述包括分子标记类型及数目、作图群体及群体大小的选择、图谱距离、连锁群数目等,QTL定位综述内容包括国内外抗病、品质和农艺性状相关QTL的研究进展,同时对图谱构建及QTL定位中存在问题进行分析并对发展前景进行了展望。
关键词:苹果;遗传图谱;QTL定位
中图分类号: S661.103文献标志码: A
文章编号:1002-1302(2016)02-0004-05
收稿日期:2015-01-13
基金项目:现代农业产业技术体系建设专项(编号:CARS-28);国家科技支撑计划(编号:2013BAD02B01-4)。
作者简介:黄金凤(1985—),女,辽宁阜新人,研究实习员,主要研究方向为分子标记辅助育种。 E-mail:huangfeng1002@163.com。
通信作者:刘志,博士,研究员,研究方向为分子标记辅助育种。E-mail:lnliuzhi@163.com。[HJ]
苹果是世界上除柑橘和香蕉外的第三大水果,也是我国第一大水果。我国是世界第一大苹果生产国,苹果栽培面积、产量均占世界苹果总面积、总产量的40%以上[1],因此,加快优良苹果品种选育对我国苹果产业发展具有十分重要的意义。果树常规育种手段主要是杂交育种、芽变选种,常规育种方法周期长,育种目标不够明确,带有一定的盲目性,而且效率低,这些特点严重制约苹果育种产业的发展。分子标记技术能够显著缩短育种年限,未来分子标记育种将成为辅助常规育种的有效手段。利用分子标记技术构建饱和的遗传图谱,随后基于图谱对苹果的数量性状位点(quantitative trait locus,QTL)进行定位,是分子标记育种的重要组成部分。QTL定位可以使作物复杂数量性状尤其是经济性状的遗传改良和分子操纵成为可能,具有巨大的应用前景[2]。
1苹果遗传图谱的构建
1.1用于连锁图谱构建的分子标记
利用遗传重组率作为基因间的距离而得到的图谱称为遗传连锁图谱,将分子标记技术应用于遗传连锁图谱的构建便产生了分子连锁图谱[3]。最早被用于构建苹果遗传图谱的标记是同功酶标记和RFLP(restriction fragment length polymorphism,限制性片段长度多态性)、RAPD(random amplified polymorphismic DNA,随机扩增多态性DNA)标记[4],随后AFLP(amplified fragment length polymorphis,扩增片段长度多态性)、SCAR(sequence characterized amplified region,序列特异扩增区域)、SSR(simple sequence repeats,简单重复序列)、EST(expressed sequence tag,表达序列标签)、ISSR(inter-[JP3]simple sequence repeats,简单重复序列间区)、SRAP(sequence-related amplified polymorphism,相关序列扩增多态性标记)等标记陆续被应用到遗传图谱构建上,伴随着测序技术的发展,SNP(single nucleotide polymorphisms,单核苷酸多态性)也越来越多地被应用在遗传图谱构建上。相较之前的标记,SNP不仅具有多态性好、广泛均匀分布于全基因组的特点,而且随着高通量测序及生物信息学数据分析技术的发展,可以实现大规模自动化检测[5]。尽管构建图谱的标记方法很多,但每种方法都有其优缺点,因此,可以将几种标记方法相结合应用于遗传图谱构建,以发挥每种标记的优势,最大程度覆盖苹果基因组,更好地为性状定位甚至后续基因克隆、功能验证等服务。
1.2苹果作图群体
果树为多年生木本,具有生长周期长、基因组杂合度高等特点,很难通过杂交获得纯系,很难得到F2群体,一度导致构建图谱工作难以开展。“双假测交”(double pseudo test cross)理论的提出,使得利用分子标记技术构建苹果的分子遗传图谱成为可能[6]。其原理是高度杂合的果树品种亲本都相当于纯合亲本的杂种一代,对亲本中所拥有的杂合位点而言,杂交后代相当于“F1”自交产生的“F2”群体[7]。因此,苹果杂交群体F1代即可用于遗传作图,大大简化并推动了苹果遗传图谱的构建工作[8]。
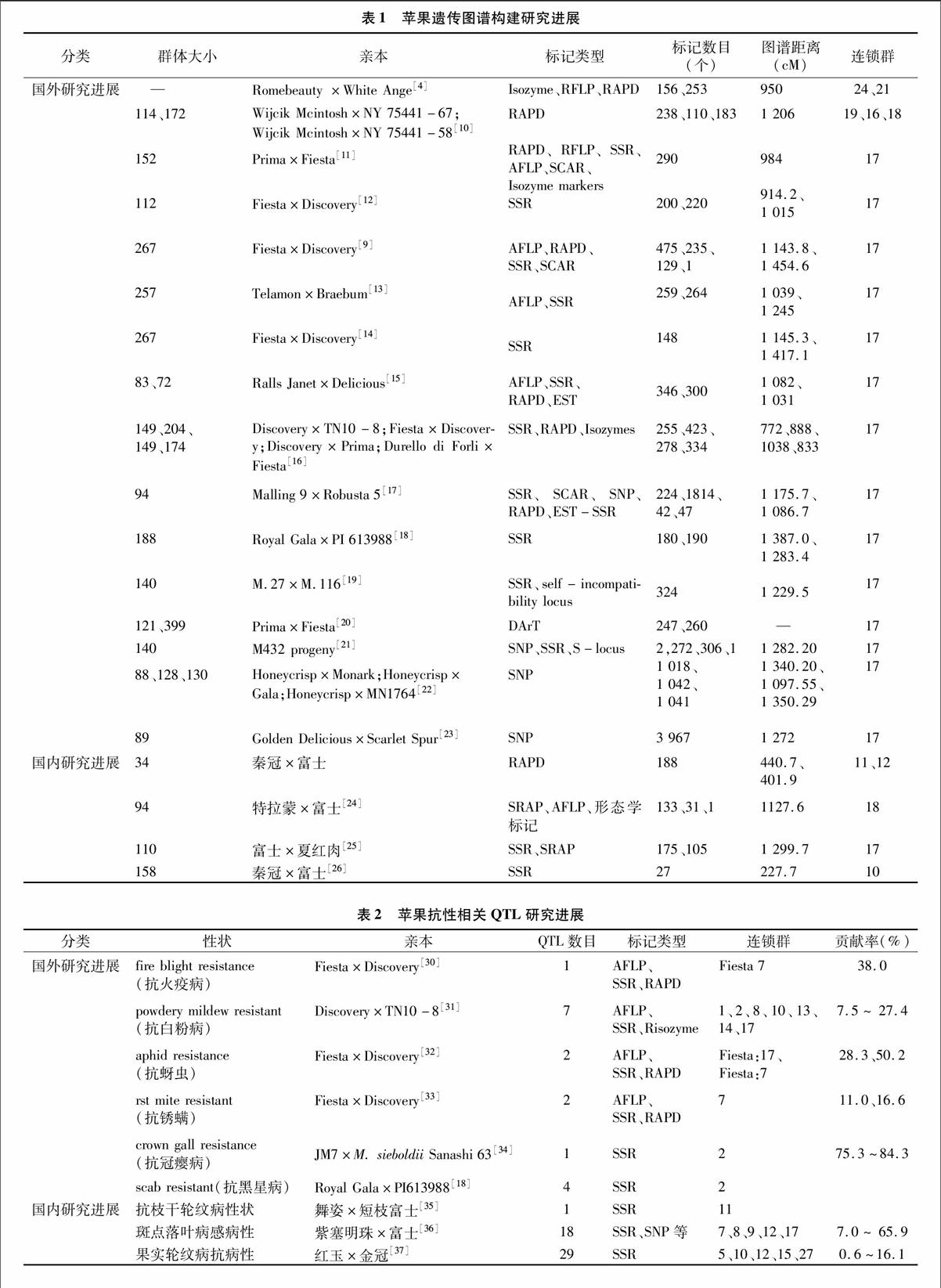
[BT2-*5]1.3分子连锁图谱的构建研究进展
自1994年Hemmat等构建了第1张苹果遗传图谱以来,世界上至少有几十张遗传图谱被发表。以Fiesta×Discovery杂交F1群体的267个后代单株构建了目前认为是最完整、饱和度最大的苹果遗传连锁图谱,该图谱包含840个标记(475个AFLP、235个RAPD、129个SSR、1个SCAR分子标记)[9]。部分图谱的基本情况见表1。从表1可以看出,国外对苹果遗传图谱研究中作图群体大小均在85以上;亲本包含品种、品系、砧木等;标记类型从最初的同工酶标记到大规模测序开发SNP标记,不断向前发展;标记数目总体不断增加,由最初的几百个上升到几千个;随着标记数目的增加,图谱距离也在不断扩大,目前已发表的最大图谱距离为1 454.6 cM;连锁群最终稳定为17个,与染色体组相对应。国内对苹果遗传图谱构建的研究开始较晚,群体、标记数目相对较少,有待进一步加强。
2苹果QTL定位研究进展
苹果许多农艺性状如果实大小、产量、成熟期等都是数量性状。数量性状受多基因控制,因此遗传基础比较复杂,而且表型受环境因素影响较大,变异呈现出一定的连续性。数量性状定位标记的实质是分析QTL与分子标记之间的关系。目前苹果的许多QTL已被定位,如抗病性、品质、产量等。
2.1抗性相关QTL
目前,关于苹果抗性相关QTL研究较多(表2)。抗性相关QTL包括抗病性、抗逆性等,抗病性研究包括抗黑星病、白粉病、轮纹病、斑点落叶病(真菌病害)、火疫病、冠瘿病(细菌病害),其中研究较多的是抗黑星病方面:利用RAPD标记寻找与抗黑星病基因连锁的标记,OPAM192200和OPAL07580与Vf遗传距离为(0.9±0.9)cM[27]。抗黑星病基因[WTBX][STBX]Vd3[WTBZ][STBZ]被定位到LG1上,连锁的SSR标记CH-Vf1,遗传距离为1 cM[28]。研究表明,抗黑星病基因[WTBX][STBX]Va1[WTBZ][STBZ]被定位在LG1上,与其连锁的分子标记为SSR CH-Vf1遗传距离为1 cM,[WTBX][STBX]Va2[WTBZ][STBZ]也在LG1上与CH-Vf1相距15 cM[29]。
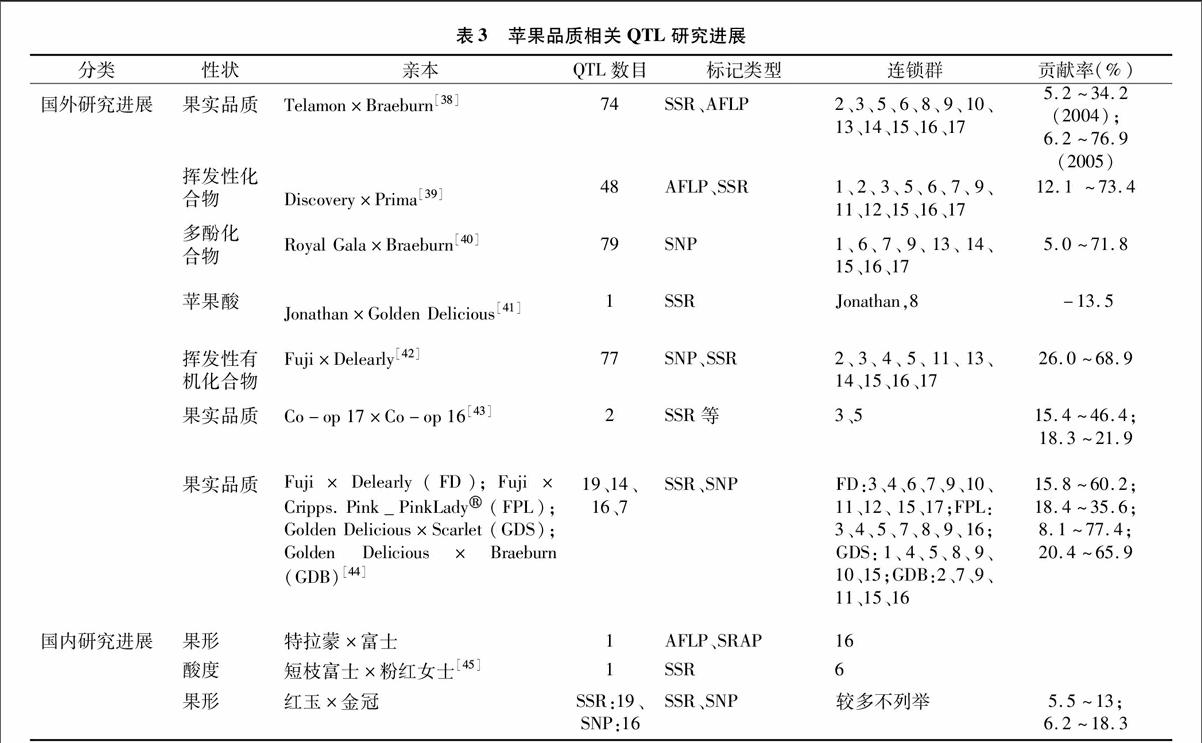
2.2品质相关QTL
目前许多果实品质相关性状已被定位,包括果皮颜色、果肉硬度、单果质量、可溶性固形物、果形、酸度、挥发性化合物、多酚化合物等一系列性状,部分品质相关QTL定位情况见表3。
2.3农艺性状相关QTL
利用Wijcik Mcintosh×NY 75441-58杂交群体F1代172个个体,构建遗传图谱,高度增量、节间数、节间长、基直径增加、基底直径、分枝数等性状被定位,可解释6.6%~41.6%表型变异[46]。3个茎粗和2个叶片大小相关的QTL被定位,5个开花习性相关的QTL被定位在第7、10、17连锁群上[47]。利用SSR、AFLP标记构建Telamon×Braeburn后代群体的图谱,将控制柱型性状的Co基因定位在Telamon的第15连锁群上,与其最近的连锁标记为COL和ECAAMCGA385,距离分别为23.62、4.77 cM[48]。A、B 2个群体中初始营养芽分化时期相关的QTL均在第9连锁群上,最多可分别解释40.1%、44.6%的表型变异[49]。国内对农艺性状相关QTL研究相对较少。刘遵春等利用红富士×红肉苹果杂交群体定位叶片相关性状的QTL共20个分布于1、2、3、4、5、7、8、10、11、12、16连锁群上,可解释11.63%~16.36%的表型变异[50]。
3问题与展望
3.1遗传图谱构建存在问题
目前苹果遗传图谱标记少,除SNP标记外,一般定位到遗传图谱上的标记多为几百个,基本没有上千,而且开发标记成本也比较高,图谱的密度以及均匀度不够。由于遗传图谱构建作者各自选用不同的作图群体、标记类型,使得很难将多个图谱整合加以利用。
3.2QTL定位存在问题
QTL定位是分子遗传图谱构建的重要应用之一,只有把相应的目标基因定位于图谱上,才能发挥图谱的遗传育种作用[51]。目前苹果QTL数目少,一些重要性状有待于进一步定位,标记与性状间距离较大,后续基因定位,功能验证不足。当前分子标记辅助的定位只能是初级定位,仅有少部分进行了精细定位或者克隆并应用到了育种实践,大多分子标记定位的位置、效应随材料、时间、地点而异,而且成本较高,所以很少真正与高产育种实践结合起来[52]。
3.3发展前景的展望
目前,苹果全基因组序列测序工作已经完成,将大大加快
苹果功能基因研究步伐,随着分子生物学技术的发展和生物信息学的应用,高密度遗传图谱的构建,影响苹果产量、品质、抗性的QTL数目、位置和效应等数量性状的遗传规律将进一步被人们认识并利用。将会有越来越多的优良性状QTL被精细定位、克隆并验证其功能,以QTL定位为基础的分子标记辅助选择育种乃至分子设计育种将在苹果育种中发挥巨大作用。
[HS2][HT8.5H]参考文献:[HT8.SS]
[1][ZK(#]王金政,薛晓敏,路超. 我国苹果生产现状与发展对策[J]. 山东农业科学,2010(6):117-119.
[2]田翠,张涛,蒋开锋,等. 水稻QTL定位研究进展[J]. 基因组学与应用生物学,2009,28(3):557-562.
[3]刘杰. 马铃薯分子连锁图谱及QTL研究进展[C]. 中国作物学会马铃薯专业委员会年会暨学术研讨会论文集,2006:239-245.
[4]Hemmat M,Weeden N F,Manganaris A G,et al. Molecular marker linkage map for apple[J]. The Journal of Heredity,1994,85(1):4-11.
[5]Helyar S J,Hemmer-Hansen J,Bekkevold D,et al. Application of SNPs for population genetics of nonmodel organisms:new opportunities and challenges[J]. Molecular Ecology Resources,2011,11(Suppl 1):123-136.
[6]Weeden N F,Hemmatt M,Lawson D M,et al. Development and application of molecular marker linkage maps in woody fruit crops[J]. Euphytica,1994,77(1/2):71-75.
[7]李英慧,韩振海,许雪峰.分子标记技术在苹果育种中的应用[J]. 生物技术通报,2002(6):11-13,10.
[8]常源升. 苹果果形性状遗传分析与QTL定位[D]. 北京:中国农业大学,2014.
[9]Liebhard R,Koller B,Gianfranceschi L,et al. Creating a saturated reference map for the apple (Malus ×domestica Borkh.) genome[J]. Theoretical and Applied Genetics,2003,106(8):1497-1508.
[10][ZK(#] Conner P J,Brown S K,Weeden N F. Randomly amplified polymorphic DNA-based genetic linkage maps of three apple cultivars[J]. Journal of the American Society for Horticultural Science,1997,122:350-359.
[11]Maliepaard C,Alston F H,van Arkel G,et al. Aligning male and female linkage maps of apple (Malus pumila Mill.) using multi-allelic markers[J]. Theoretical and Applied Genetics,1998,97(1/2):60-73.
[12] Liebhard R,Gianfranceschi L,Koller B,et al. Development and characterisation of 140 new microsatellites in apple (Malus×domestica Borkh.)[J]. Molecular Breeding,2002,10,217-241.
[13] Kenis K,Keulemans J. Genetic linkage maps of two apple cultivars(Malus×domestica Borkh.)based on AFLP and microsatellite markers[J]. Molecular Breeding,2005,15:205-219.
[14] Silfverberg-Dilworth E,Matasci C L,van de Weg W E,et al. Microsatellite markers spanning the apple (Malus×domestica Borkh.)genome[J]. Tree Genetics & Genomes,2006,2(4):202-224.
[15]Igarashi M,Abe Y,Hatsuyama Y,et al. Linkage maps of the apple (Malus×domestica Borkh.) cvs ‘Ralls Janet and ‘Delicious include newly developed EST markers[J]. Molecular breeding,2008,22:95-118.
[16]Ndiaye A,van de Weg W E,Kodde L P,et al. Construction of an integrated consensus map of the apple genome based on four mapping populations[J]. Tree Genetics & Genomes,2008,4(4):727-743.
[17]Celton J M,Tustin D S,Chagné D,et al. Construction of a dense genetic linkage map for apple rootstocks using SSRs developed from Malus ESTs and Pyrus genomic sequences[J]. Tree Genetics & Genomes,2009,5(1):93-107.
[18] Wang A,Aldwinckle H,Forsline P,et al. EST contig-based SSR linkage maps for Malus×domestica cv Royal Gala and an apple scab resistant accession of M. sieversii[J]. The Progenitor Species of Domestic Apple,2012,29:379-397.
[19]Fernández-Fernández F,Antanaviciute L,van Dyk M M,et al. A genetic linkage map of an apple rootstock progeny anchored to the Malus genome sequence[J]. Tree Genetics & Genomes,2012,8(5):991-1002.
[20]Schouten H J,van de Weg W E,Carling J,et al. Diversity arrays technology (DArT) markers in apple for genetic linkage maps[J]. Molecular Breeding:New Strategies in Plant Improvement,2012,29(3):645-660.
[21]Antanaviciute L,Fernández-Fernández F,Jansen J,et al. Development of a dense SNP-based linkage map of an apple rootstock progeny using the Malus infinium whole genome genotyping array[J]. BMC Genomics,2012,13:203.
[22]Clark M D,Schmitz C A,Rosyara U R,et al. A consensus‘Honeycrispapple (Malus×domestica) genetic linkage map from three full-sib progeny populations[J]. Tree Genetics & Genomes,2014,10:627-639.
[23]Gardner K M,Brown P,Cooke T F,et al. Fast and cost-effective genetic mapping in apple using next-generation sequencing[J]. G3 (Bethesda),2014,4(9):1681-1687.
[24]司鹏. 苹果分子遗传图谱构建及其部分性状SRAP分析[D]. 北京:中国农业科学院,2010.
[25]刘遵春. 新疆野苹果(Malus sieversi)核心种质与遗传图谱构建的研究[D]. 泰安:山东农业大学,2010.
[26]李爽. 苹果SSR标记体系的优化及遗传连锁图谱构建初探[D]. 杨凌:西北农林科技大学,2011.
[27]Tartarini S. RAPD markers linked to the Vf gene for scab resistance in apple[J]. Theoretical and Applied Genetics,1996,92(7):803-810.
[28]Soriano J M,Joshi S G,van Kaauwen M,et al. Identification and mapping of the novel apple scab resistance gene [WTBX][STBX]Vd3[WTBZ][STBZ][J]. Tree Genetics & Genomes,2009,5(3):475-482.
[29]Dunemann F,Egerer J. A major resistance gene from Russian apple ‘Antonovka conferring field immunity against apple scab is closely linked to the Vf locus[J]. Tree Genetics & Genomes,2010,6(5):627-633.
[30]Khan M A,Duffy B,Gessler C,et al. QTL mapping of fire blight resistance in apple[J]. Molecular Breeding,2006,17(4):299-306.
[31]Calenge F,Durel C E. Both stable and unstable QTLs for resistance to powdery mildew are detected in apple after four years of field assessments[J]. Molecular Breeding,2006,17(4):329-339.
[32]Stoeckli S,Mody K,Gessler C,et al. QTL analysis for aphid resistance and growth traits in apple[J]. Tree Genetics & Genomes,2008,4(4):833-847.
[33]Stoeckli S,Mody K,Patocchi A,et al. Rust mite resistance in apple assessed by quantitative trait loci analysis[J]. Tree Genetics & Genomes,2009,5(1):257-267.
[34]Moriya S,Iwanami H,Takahashi S,et al. Genetic mapping of the crown gall resistance gene of the wild apple Malus sieboldii[J]. Tree Genetics & Genomes,2010,6(2):195-203.
[35]殷豪. 苹果枝干轮纹病抗病性状分子标记及几丁质酶基因克隆研究[D]. 青岛:青岛农业大学,2011.
[36]敬小康,吕松,刘欣颖,等. Malus asiatica×M.domestica种间杂种苹果斑点落叶病感病性的QTL定位[J]. 中国农业大学学报,2014,19(6):140-147.
[37]崔镁沙,庄艳,申飞,等. 苹果果实轮纹病抗病性QTL定位及相关基因的初步预测[J]. 果树学报,2014,31(5):793-800.
[38]Kenis K,Keulemans J,Davey M W. Identification and stability of QTLs for fruit quality traits in apple[J]. Tree Genetics & Genomes,2008,4(4):647-661.
[39]Dunemann F,Ulrich D,Boudichevskaia A,et al. QTL mapping of aroma compounds analysed by headspace solid-phase microextraction gas chromatography in the apple progeny ‘DiscoveryבPrima[J]. Mol Breeding,2009,23:501-521.
[40]Chagné D,Krieger C,Rassam M,et al. QTL and candidate gene mapping for polyphenolic composition in apple fruit[J]. BMC Plant Biology,2012,12:12.
[41]Zhang Q,Ma B,Li H,et al. Identification,characterization,and utilization of genome-wide simple sequence repeats to identify a QTL for acidity in apple[J]. BMC Genomics,2012,13:537.
[42]Cappellin L,Farneti B,Di Guardo M,et al. QTL analysis coupled with PTR-ToF-MS and candidate gene-based association mapping validate the role of Md-AAT1 as a major gene in the control of flavor in apple fruit[J]. Plant Molecular Biology Reporter,2015,33(2):239-252.
[43]Potts S M,Khan M A,Han Y,et al. Identification of quantitative trait loci (QTLs) for fruit qality taits in apple[J]. Plant Mol Biol Rep,2014,32:109-116.
[44]Costa F. MetaQTL analysis provides a compendium of genomic loci controlling fruit quality traits in apple[J]. Tree Genetics & Genomes,2014,11(1):819.
[45]王雷存,樊红科,高华,等. 苹果酸度基因(Ma)SSR标记及遗传分析[J]. 园艺学报,2012,39(10):1885-1892.
[46]Conner P J,Brown S K,Weeden N F. Molecular-marker analysis of quantitative traits for growth and development in juvenile apple trees[J]. Theoretical and Applied Genetics,1998,96(8):1027-1035.
[47]Liebhard R,Kellerhals M,Pfammatter W,et al. Mapping quantitative physiological traits in apple (Malus×domestica Borkh.)[J]. Plant Molecular Biology,2003,52(3):511-526.
[48]Kenis K,Keulemans J. Study of tree architecture of apple (Malus×domestica Borkh.) by QTL analysis of growth traits[J]. Molecular Breeding,2007,19(3):193-208.
[49]Dyk M M,Soeker M K,Labuschagne I F,et al. Identification of a major QTL for time of initial vegetative budbreak in apple (Malus×domestica Borkh.)[J]. Tree Genetics & Genomes,2010,6:489-502.
[50]刘遵春,苗卫东,刘大亮,等. 苹果叶片相关性状的QTL定位及其遗传效应分析[J]. 西北植物学报,2014,34(3):481-487.
[51]贾小丽. 化感水稻的遗传图谱构建及其QTL定位[D]. 福州:福建农林大学,2006.
[52]梅德圣,李云昌,王汉中. 作物产量性状QTL定位的研究现状及应用前景[J]. 中国农学通报,2003,19(5):83-88.
