粘虫CYP6AE88和CYP332A1基因的鉴定和生物信息学分析
2016-04-09李敏李生才王青张虎芳
李敏,李生才,王青,张虎芳*
(1.太原师范学院 生物系,山西 太原 030031; 2.山西农业大学 农学院,山西 太谷 030801)
粘虫CYP6AE88和CYP332A1基因的鉴定和生物信息学分析
李敏1, 2,李生才2,王青1,张虎芳2*
(1.太原师范学院 生物系,山西 太原 030031; 2.山西农业大学 农学院,山西 太谷 030801)
摘要:鉴定粘虫Mythimna separata CYP450家族基因,分析其序列特征并推测可能的功能,为研究粘虫杀虫剂抗性分子机制提供理论依据。以棉铃虫Helicoverpa armigera CYP6AE20v2和CYP332A1作为询问序列,通过双向Blast方法分别检索粘虫转录组中CYP6AE和CYP332A cDNA序列,通过生物信息学的方法预测基因所编码的蛋白质序列的理化性质、疏水性、跨膜区、结构域、3D结构等以及可能的功能,并采用最大似然法(Maximum Likelihood,ML)分析夜蛾科代表物种的CYP6AE和CYP332A亚家族序列的系统发育关系。从粘虫转录组测序数据中鉴定出CYP6AE和CYP332A两个亚家族的基因,分别命名为CYP6AE88(GenBank登录号:KU145393)和CYP332A1(GenBank登录号:KU145394)。cDNA序列分析显示,CYP6AE88和CYP332A1的全长分别为1 713 bp和1 815 bp,分别编码509和503个氨基酸。蛋白质序列基本性质分析显示:CYP6AE88蛋白理论相对分子质量为58.9 kD,等电点是7.95;CYP332A1蛋白理论相对分子质量为58.4 kD,等电点是8.03。CYP6AE88和CYP332A1分别在5-22位和5-23位氨基酸之间有一个典型的疏水性区域;分别在2~21位和2~24位氨基酸之间形成一个典型的跨膜区。两个蛋白均不存在信号肽,亚细胞定位均为细胞质。蛋白质结构域分析分析显示:两个蛋白均含P450特有的5个特征序列,并且含有多个酶结合位点。3D结构分析显示,CYP6AE88含有α 螺旋的数目是18条和β 折叠数目是9股,CYP332A1含有α螺旋的数目是19条和β折叠10股。CYP6AE的系统发育分析显示:粘虫CYP6AE88与(草地贪夜蛾CYP6AE43 + 海灰翅夜蛾CYP6AE49)聚为一支(BP=72%),然后再与棉铃虫CYP6AE20v2/50/19形成单系(BP=100%);CYP332A的聚类分析显示:粘虫CYP332A1与棉铃虫CYP332A1聚为一支,bootstrap值为100%。研究结果为进一步研究粘虫CYP6AE88和CYP332A1基因杀虫剂抗性分子机制奠定了基础。
关键词:粘虫;CYP450;CYP6AE88;CYP332A1;生物信息学分析
粘虫Mythimnaseparata(Walker)属于鳞翅目,夜蛾科,是一种远距离迁飞害虫,也是粮食作物的重大害虫。其发生危害具有发生范围广,危害世代多;危害的作物种类和组织多;暴发性明显;发生危害历史长等特点[1]。在中国,1950-2013年的64年中有20年全国粘虫发生面积在666.7万hm2(1亿亩)以上,2012年总损失达657.8万t[2]。在亚洲和澳洲其它国家以及太平洋一些岛屿上也常发生粘虫危害[3,4]。喷洒化学杀虫剂是现阶段防治粘虫的主要方法,然而随着杀虫剂的应用不断增加,粘虫对杀虫剂产生了一定程度的抗药性[5,6]。
细胞色素P450(CYP)、羧酸酯酶(CXE)和谷胱甘肽S-转移酶(GST)是昆虫体内三种重要的代谢解毒酶。其中细胞色素P450 是细胞色素P450酶系的末端氧化酶,在杀虫剂的氧化、还原反应中起到重要作用。系统发育分析表明昆虫的P450蛋白可分为4个大的分支:CYP2族,CYP3族,CYP4族和Mitochondrial CYP族[7],其中CYP3族包括CYP6、CYP9、CYP28、CYP300、CYP400、CYP408及CYP413等家族成员,与代谢外源性物质及植物天然化合物有关[8,9]。如溴氰菊酯抗性的棉铃虫中CYP4L5,CYP4L11,CYP6AE11,CYP332A1 和CYP9A14基因产生了过量表达[10];CYP6G1与黑腹果蝇(DrosophilamelanogasterMeigen)对DDT抗性有关[11];CYP6M2能够引起冈比亚按蚊(Anophelesgambiae)对恶虫威、拟除虫菊酯和DDT三种杀虫剂的抗性[12];CYP6BQ23的过量表达是欧洲油菜露尾甲(Meligethesaeneus)对拟除虫菊酯杀虫剂产生抗性主要机制[13]。因此,鉴定粘虫抗药性相关基因,研究其功能,对于阐释粘虫抗药性分子机理乃至粘虫病虫害的有效控制具有指导意义。
本研究基于粘虫转录组测序数据,采用双向Blast方法搜索得到粘虫的CYP6AE88和CYP332A1 cDNA序列,并通过生物信息学方法分析其序列特征,对编码的蛋白质的结构和功能进行预测,并运用最大似然法(maximum likelihood,ML)构建鳞翅目夜蛾科代表性物种的系统发育关系,为深入研究粘虫CYP6AE88和CYP332A1基因的功能以及参与杀虫剂抗性的分子机制奠定了基础。
1材料与方法
1.1数据来源
粘虫(M.separate,Ms)饲养于人工气候箱中,饲养条件为:RH≥75%,温度保持在≤25 ℃,L∶D 为14∶10,未接触任何药物。提取卵,1~6龄幼虫,蛹,成虫的总RNA后,等量混合建库。对原始测序数据进行过滤后,得到高质量序列进行从头拼接,获得53 159 618 高质量的 clean reads,21 051(69.05%)unigenes被注释到Nr, Swiss-Prot, KEGG, COG, Go 和 Nt databases等数据库中。转录组数据提交至NCBI(http://www.ncbi.nlm.nih.gov/),可通过以下3种方式获得:BioProject: PRJNA301662;BioSample: SAMN03105778和Short Read Archive (SRA): SRP065982。其它昆虫的CYP450同源序列直接下载自NCBI的GenBank数据库。
1.2序列鉴定
在粘虫转录组功能注释结果中,将“P450”设置为关键词进行搜索,将搜索到的预测序列在NCBI中利用BlastX程序进行比对。如果与已知P450蛋白序列一致性高于40%(E值 ≤ e-7),则认为其可能是粘虫的P450基因[14]。然后分别以棉铃虫的CYP6AE20v2(GenBank登录号:KM016742)和CYP332A1(GenBank登录号:KM016708)作为问询序列在NCBI中进行BlastX搜索,找出粘虫的CYP6AE和CYP332A亚家族基因[15]。
1.3序列分析
利用软件对粘虫CYP6AE88和CYP332A1的cDNA的序列特征、编码蛋白质的结构和功能进行预测分析,选用的软件见表1。

表1 分析内容及所用软件的名称和网址
1.4CYP6AE和CYP332A同源性与系统发育分析
从BlastX结果中分别统计粘虫CYP6AE和CYP332A与其它昆虫统同源氨基酸的一致率(identity)和相似率(positive)。以粘虫的CYP6AE88和CYP332A1为问询序列,用NCBI的BlastX检索代表性鳞翅目昆虫以及外群的CYP6和CYP332的同源性序列。将黑腹果蝇的CYP6A14序列设为外群,采用最大似然法,用MEGA 6.0软件分析粘虫、海灰翅夜蛾,草地贪夜蛾,棉铃虫,烟草天蛾,家蚕,稻纵卷叶螟,黑腹果蝇的CYP6AE序列之间的系统发育关系,1 000次重复计算bootstrap(BP)值。采用最大似然法,用MEGA 6.0软件对粘虫、棉铃虫、家蚕、烟草天蛾的CYP332A序列进行聚类分析。
2结果
2.1cDNA序列分析
通过比对得出粘虫CYP6AE和CYP332A亚家族的两条基因序列,分别命名为CYP6AE88(GenBank登录号:KU145393)和CYP332A1(GenBank登录号:KU145394)。这两个基因的cDNA均为该基因的全长序列,其中前者cDNA全长为 1 713 bp,包括1 509 bp的编码区,编码502个氨基酸。后者cDNA全长为1 815 bp,包括1 512 bp的编码区,编码526个氨基酸。蛋白质结构域分析表明这两个基因均为CYP超家族基因,cDNA编码的氨基酸序列包含昆虫细胞色素P450的5个保守结构域(图1a, b),分别为WxxxR,A/GGxE/DTT/S,ExxR,PxxFxPE/DRF和FxxGxxxCxG(亦称血红素结合区)[16]。
2.2CYP6AE88和CYP332A1所编码的蛋白质特性分析及功能预测
2.2.1蛋白质一级结构特性分析
软件ProtParam预测结果表明,CYP6AE88的编码蛋白的分子式为C2803H4303N715O779S25,理论相对分子质量为58.9 kD,等电点是7.95,不稳定指数为34.64,亲水性平均数为-0.238;CYP332A1 的编码蛋白的分子式为C2690H4104N666O742S25,理论相对分子质量为58.4 kD,等电点是8.03,不稳定指数40.16,亲水性平均数-0.205。软件ProtScale预测结果表明CYP6AE88在N端5-22位氨基酸之间有一个典型的疏水性区域;

图1a 粘虫CYP6AE88 cDNA序列及其翻译的氨基酸序列Fig.1a cDNA and its translated amino acid sequence of CYP6AE88 from M. separata 注:起始密码子和终止密码子在图中被红色线框标出,左侧编号为核苷酸和氨基酸对应的编号,P450保守结构域:WxxxR,A/GGxE/DTT/S,ExxR,PxxFxPE/DRF和FxxGxxxCxG 用红色下划线标出。图1b同。Note:The start and stop codon are marked with red boxes,the numbers on the left are the positions of nucleotides and amino acids on the sequences,and the insect P450 conserved motifs including WxxxR,A/GGxE/DTT/S,ExxR,PxxFxPE/DRF and FxxGxxxCxG are underlined in bold red lines.The same for Figure 1b.

图1b 粘虫CYP332A1 cDNA序列及其翻译的氨基酸序列Fig.1b cDNA and its translated amino acid sequence of CYP332A1 from M. separata
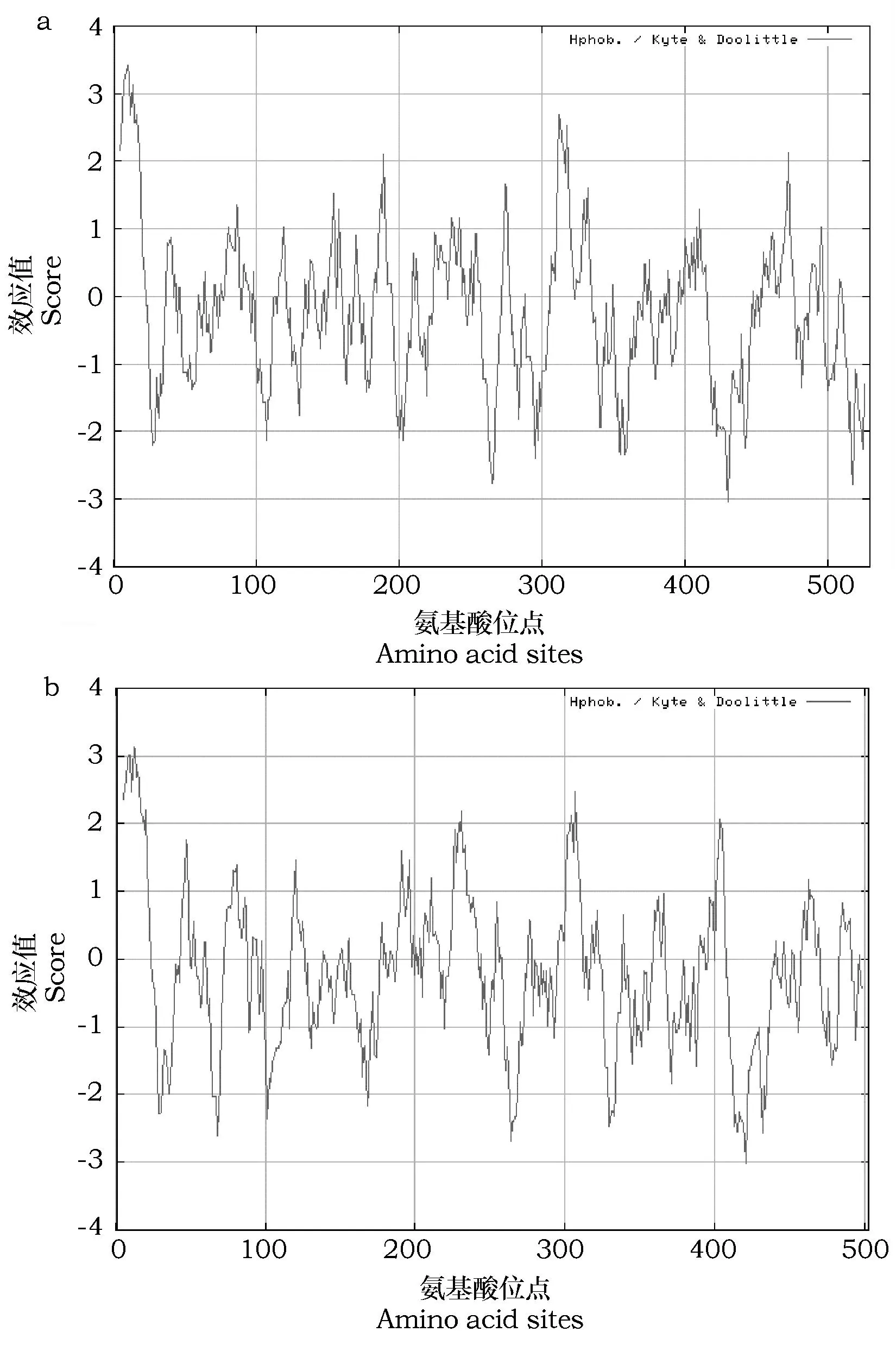
图2 粘虫CYP6AE88(a)和CYP332A1(b)蛋白序列的疏水区预测Fig.2 Hydrophobic region predication of the amino acid sequence of M. separata CYP6AE88 (a) and CYP332A1 (b) proteins. 注:正值代表疏水区,负值代表亲水区。Note: Positive values represent the hydrophobic regions and negative values represent the hydrophilic regions.
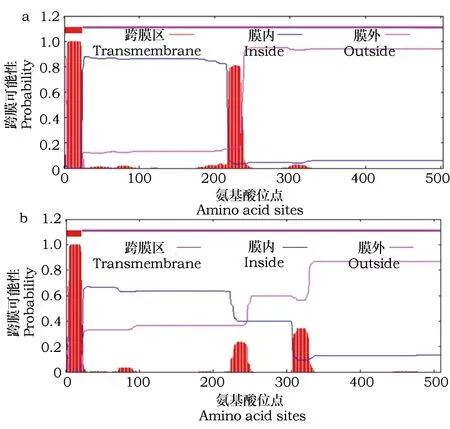
图3 粘虫CYP6AE88(a)和CYP332A1(b)蛋白跨膜区域Fig.3 The deduced transmembrane domain topology model of M. separata CYP6AE88 (a) and CYP332A1 (b) proteins
CYP332A1在N端5-23位氨基酸之间有一个典型的疏水性区域(图2a,2b)。利用TMHMM Server v.2.0软件对粘虫两个蛋白是否存在跨膜区进行预测,发现CYP6AE88编码的蛋白质跨膜片段约在2~21位氨基酸之间(图3a),预测膜上的一些物质与蛋白质有结合部位;CYP332A1编码的蛋白质在约2~24位氨基酸之间具有跨膜片段(图3b),同样预测膜上的一些物质与蛋白质有结合部位。SignalP 4.1预测2个基因的编码蛋白均不存在信号肽,亚细胞定位均为细胞质。
2.2.2蛋白功能位点分析
利用ScanProsite软件对粘虫CYP6AE88和CYP332AE1进行功能位点分析,预测CYP6AE88含有8个蛋白激酶C磷酸化位点、3个酪氨酸激酶磷酸化位点、6个N端酰基化位点、5个酪蛋白激酶Ⅱ磷酸化位点、1个N端糖基化位点和1个细胞色素P450-半胱氨酸血红素配体信号区(表2a)。CYP332AE1含有2个N端糖基化位点、2个酪氨酸激酶磷酸化位点、5个酪蛋白激酶Ⅱ磷酸化位点、6个蛋白激酶C磷酸化位点、4个N端酰基化位点、1个酰胺化位点和1个细胞色素P450-半胱氨酸血红素配体信号区(表2b)。
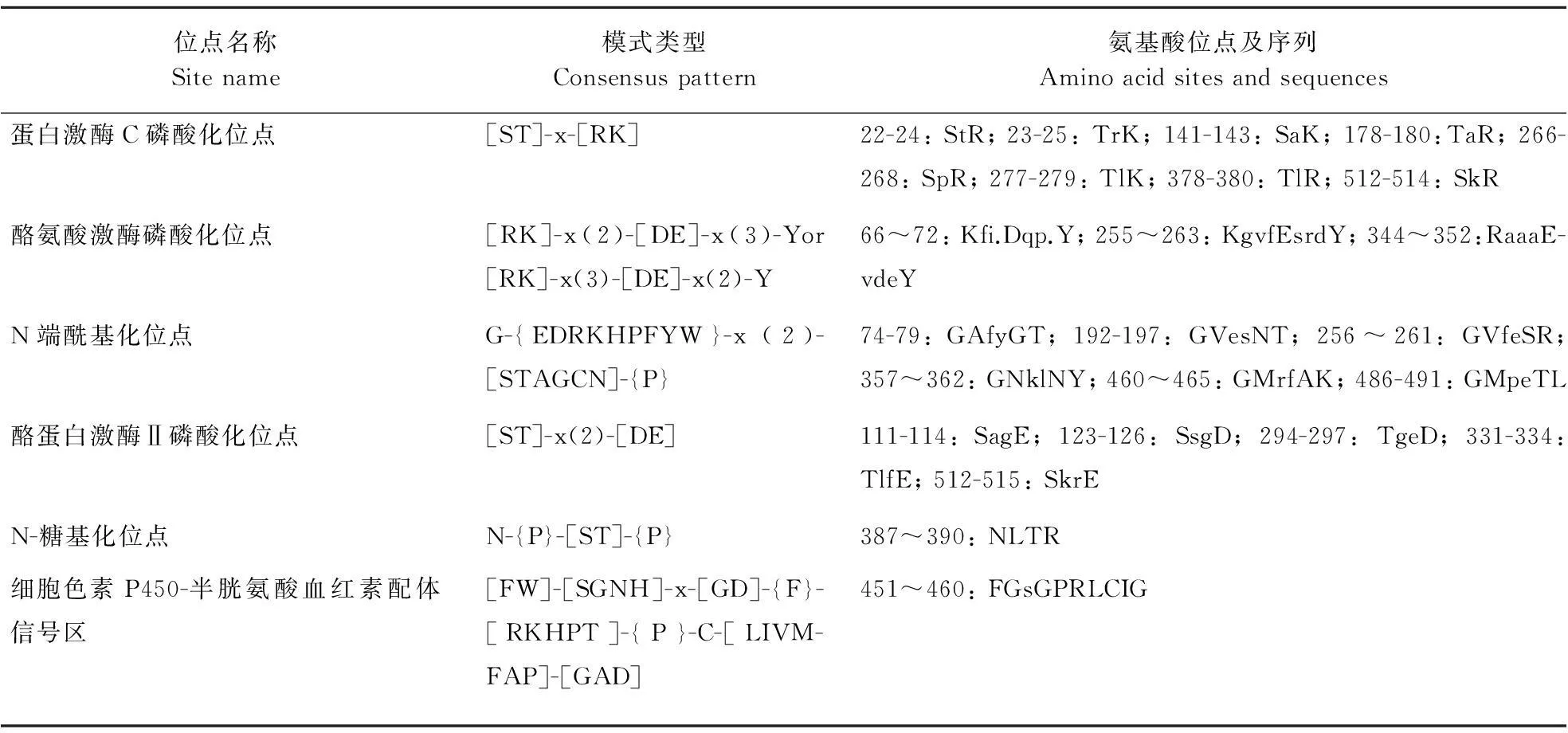
表2a ScanProsite预测的粘虫CYP6AE88蛋白功能位点
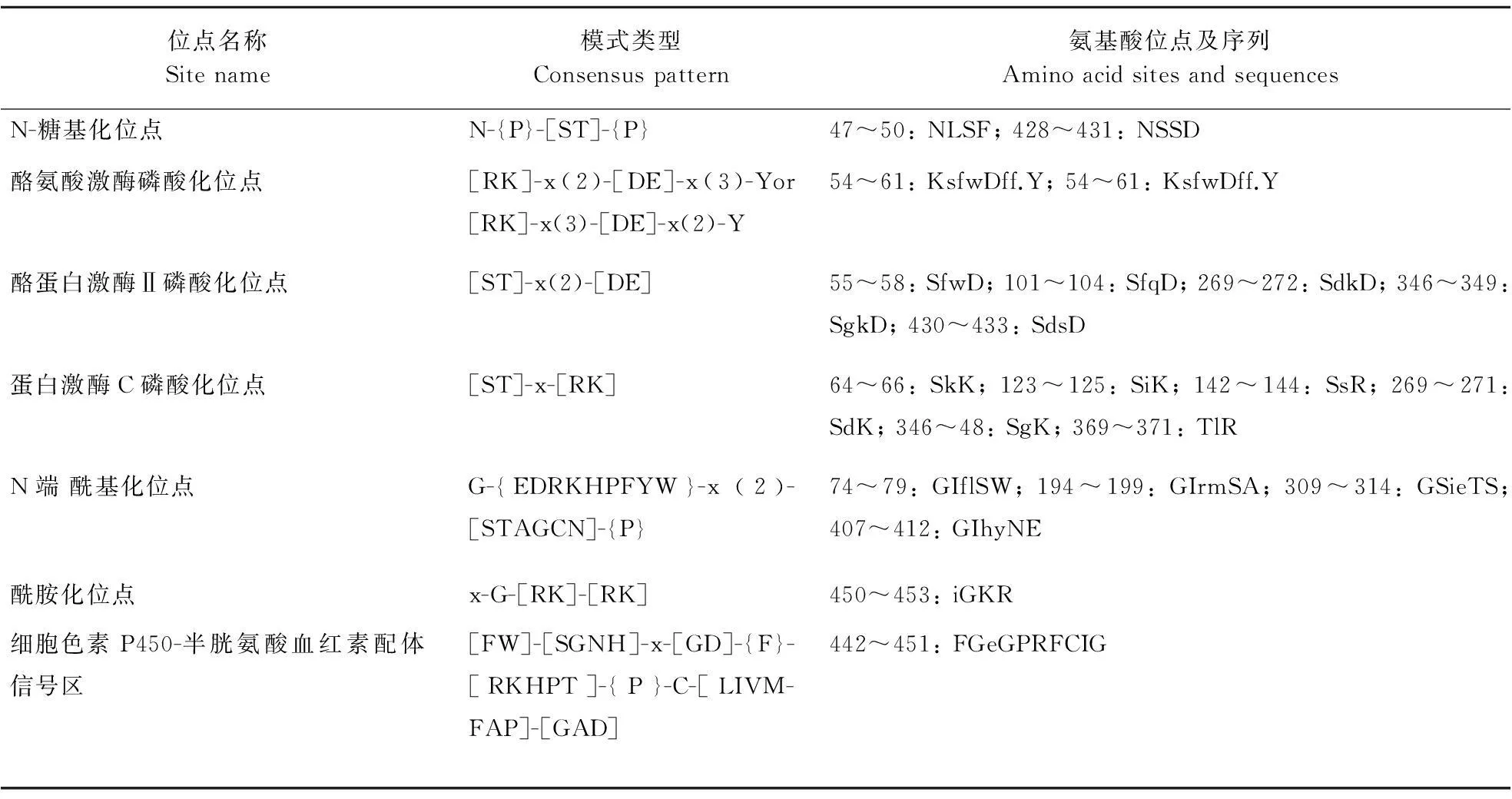
表2b ScanProsite预测的粘虫CYP332AE1蛋白功能位点
2.2.3蛋白质结构域分析
蛋白质结构域分析表明,CYP6AE88的5个保守结构与昆虫P450的保守结构存在正常的差异:昆虫P450的Meander区(PxxFxPE/DRF)结构域中的第4个氨基酸为苯丙氨酸,而CYP6AE88的(PxxYxPE/DRF)中的第4个氨基酸为酪氨酸,两者均为芳香族氨基酸,仅有一个基团——苯环上羟基的差别。CYP332A1的5个保守结构与昆虫P450的保守结构也存在一定差异:昆虫P450的Meander 区(PxxFxPE/DRF)中的苯丙氨酸被CYP332A1的结构域(PxxWxPE/DRF) 中色氨酸代替,两者均为芳香族氨基酸,也仅有一个基团的差别,一个为苯基,一个为吲哚基。以上两蛋白的5个结构保守区在图4a和图4b中用红色线框标出。
2.2.4蛋白质的空间结构预测
应用GORIV方法在线分析粘虫CYP6AE88基因和CYP332A1基因编码蛋白质的二级结构,分析显示: CYP6AE88蛋白质中无规卷曲占49.31%,所占比例最大,α-螺旋占30.06%,延伸链占20.63%(图5a);在CYP332A1蛋白质中无规卷曲占44.14%,所占例最大,α-螺旋占34.99%,延伸链占20.87%(图5b)。由以上数据可以推测,两个蛋白质二级结构中无规卷曲是所占比例最大,α-螺旋和延伸链则可能分散于整个蛋白质中。
通过 PSI-BLAST的搜索CYP6AE88和CYP332AE1与人CYP3A4 氨基酸序列一致性最高,所得相似性参数为32.17%和26.77%,选择人CYP3A4 氨基酸序列作为同源建模的模板进行建模。利用SWISSMODEL建模如图6a,b所示,得到CYP6AE88和CYP332A1的蛋白质三级结构。使用Swiss-PdbViewer4.1.0分析建模所得三级结构进行,软件运行结果显示: CYP6AE88有α螺旋18条和β折叠9股, CYP332A1有α螺旋19条和β折叠10股。
2.3粘虫CYP6AE和CYP332A亚家族相似性和系统发育分析
将粘虫CYP6AE88蛋白序列与CYP6AE家族氨基酸序列进行比对,结果表明粘虫CYP6AE88与海灰翅夜蛾CYP6AE49和草地贪夜蛾CYP6AE43氨基酸序列相似性最高,一致率分别为75%和71%,相似率分别为相似率为87%和84%,其次是棉铃虫,一致率和相似率分别为69%和85%;将粘虫CYP332A1蛋白序列与CYP332A家族氨基酸序列进行比对,结果表明粘虫CYP332A1与棉铃虫CYP332A1氨基酸序列相似性最高(一致率为78%,相似率为91%),其次是家蚕和烟草天蛾,一致率均为63%(见表3)。

图4a 粘虫CYP6AE88基因与昆虫CYP6AE亚家族基因氨基酸序列比较Fig.4a Amino acid sequence alignment of CYP6AE88 gene from M. separata with CYP6AE genes from other Noctuidae species 注:Ms:粘虫;Sl:海灰翅夜蛾;Sf:草地贪夜蛾;Ha:棉铃虫 Helicoverpa armigera; Mse:烟草天蛾 ;Bm:家蚕;Cm:稻纵卷叶螟。图中用红色线框标出了P450的5个保守结构域,黑色、灰色和白色阴影分别代表氨基酸序列保守性为100%,80%和80%以下。图4b同。Note:Ms:Mythimna separata;Sl: Spodoptera_littoralis;Sf:Spodoptera frugiperda;Mse:Manduca sexta;Bm: Bombyx mori;Cm:Cnaphalocrocis medinalis。Five conserved domains are line-boxed out in the figure. Black, grey and white shade denote amino acids with 100%, 80% and below 80% identity, respectively.The same for Figure 4b.
选择粘虫CYP6AE88和其它6种夜蛾科昆虫的同源氨基酸序列,以黑腹果蝇CYP6G1氨基酸序列为外群构建系统发育树,结果表明草地贪夜蛾CYP6AE43 和海灰翅夜蛾CYP6AE49首先聚为一支(BP=100%),然后与粘虫CYP6AE88构成单系(BP=72%),这三者再与棉铃虫CYP6AE20v2/50/19形成一支(BP=97%),系统发育树所有节点的bootstrap值均大于65%(图7a)。选择粘虫CYP332A1和其他3种夜蛾科昆虫的同源氨基酸序列进行聚类分析,结果表明粘虫CYP332A1与棉铃虫CYP332A1聚为一支,bootstrap值为100%,聚类树所有节点的bootstrap值均大于95%(图7b)。

图4b 粘虫CYP332A1基因与昆虫CYP332A亚家族基因氨基酸序列比较Fig.4b Amino acid sequence alignment of CYP332A1 gene from M. separata with CYP332A genes from other Noctuidae species Ms:粘虫 Mythimna separata; Ha:棉铃虫 Helicoverpa armigera; Bm:家蚕 Bombyx mori; Mse: 烟草天蛾 Manduca sexta.
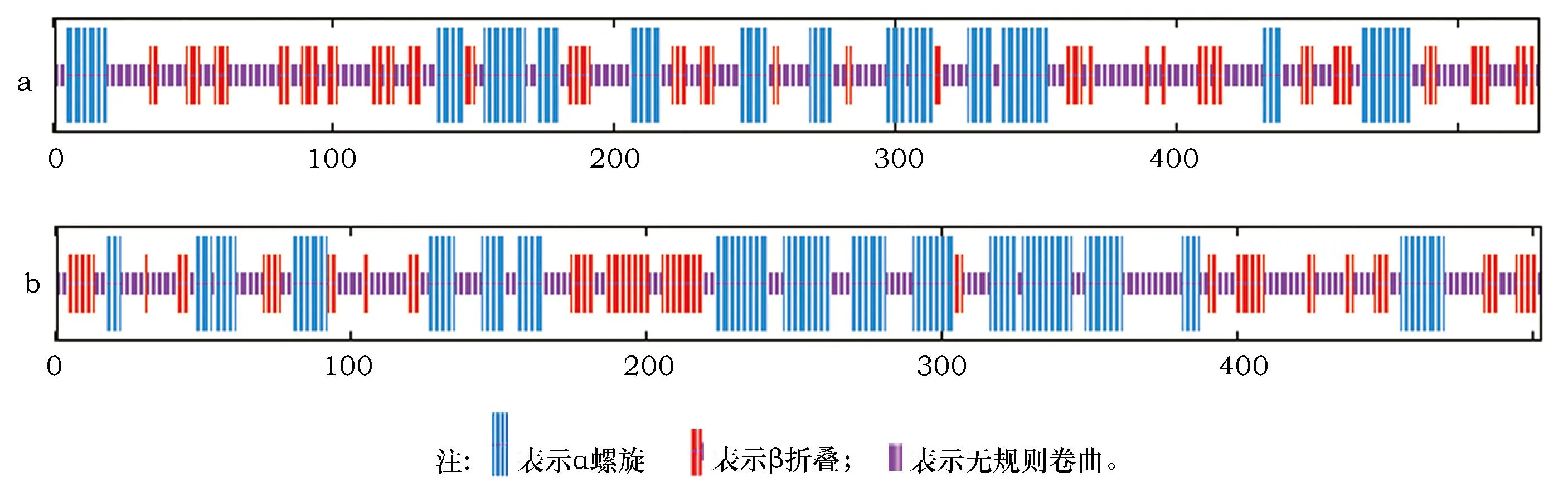
图5 粘虫CYP6AE88(a)和CYP332A1(b)蛋白二级结构预测Fig.5 Predicted secondary structure of M. separata CYP6AE88 (a) and CYP332A1 (b) proteins
表3a粘虫CYP6AE88与7种其它昆虫的GenBank登录号、相似率和一致率
Table 3aThe GenBank accession number, positive and identity in homologous CYP6AE sequences betweenM.separataand 7 other insect species

物种SpeciesGenBank登录号Accessionnumber相似率/%Positive一致率/%Identity海灰翅夜蛾AFP20593.18775草地贪夜蛾AID55427.18471棉铃虫AID54896.18569烟草天蛾ADE05581.17862家蚕BAM73891.17660稻纵卷叶螟CAX94849.17455黑腹果蝇NP_610389.35634
3讨论
CYP6AE亚家族基因在鳞翅目的草地贪夜蛾、稻纵卷叶螟、海灰翅夜蛾、家蚕、棉铃虫和烟草天蛾均有报道,分别有2、4、4、16、16和2个该亚家族基因,本研究发现了粘虫CYP6AE亚家族具有1个基因。CYP332A亚家族基因位于15号染色体上,截止2015年12月,仅在鳞翅目部分昆虫中有报道:草地贪夜蛾、稻纵卷叶螟和家蚕各1个,在棉铃虫和烟草天蛾中各2个,很可能为在其邻近转座子的作用下进行了一次复制[10,17~20],其它昆虫中暂未发现,或许与鳞翅目的某些特殊功能有关,需要进一步研究。也正由于在其它目昆虫中尚未发现CYP332A亚家族基因,所以在后续建树时未选择外群,对鳞翅目的代表种类进行聚类分析。
表3b粘虫CYP332A1与3种其它昆虫的GenBank登录号、相似率和一致率
Table 3bThe GenBank accession number, positive and identity in homologous CYP332A sequences betweenM.separataand 3 other Noctuidae species

物种SpeciesGenBank登录号Accessionnumber相似率/%Positive一致率/%Identity棉铃虫AID54860.19178家蚕NP_001108340.18263烟草天蛾ADE05588.17863ADE05587.18062
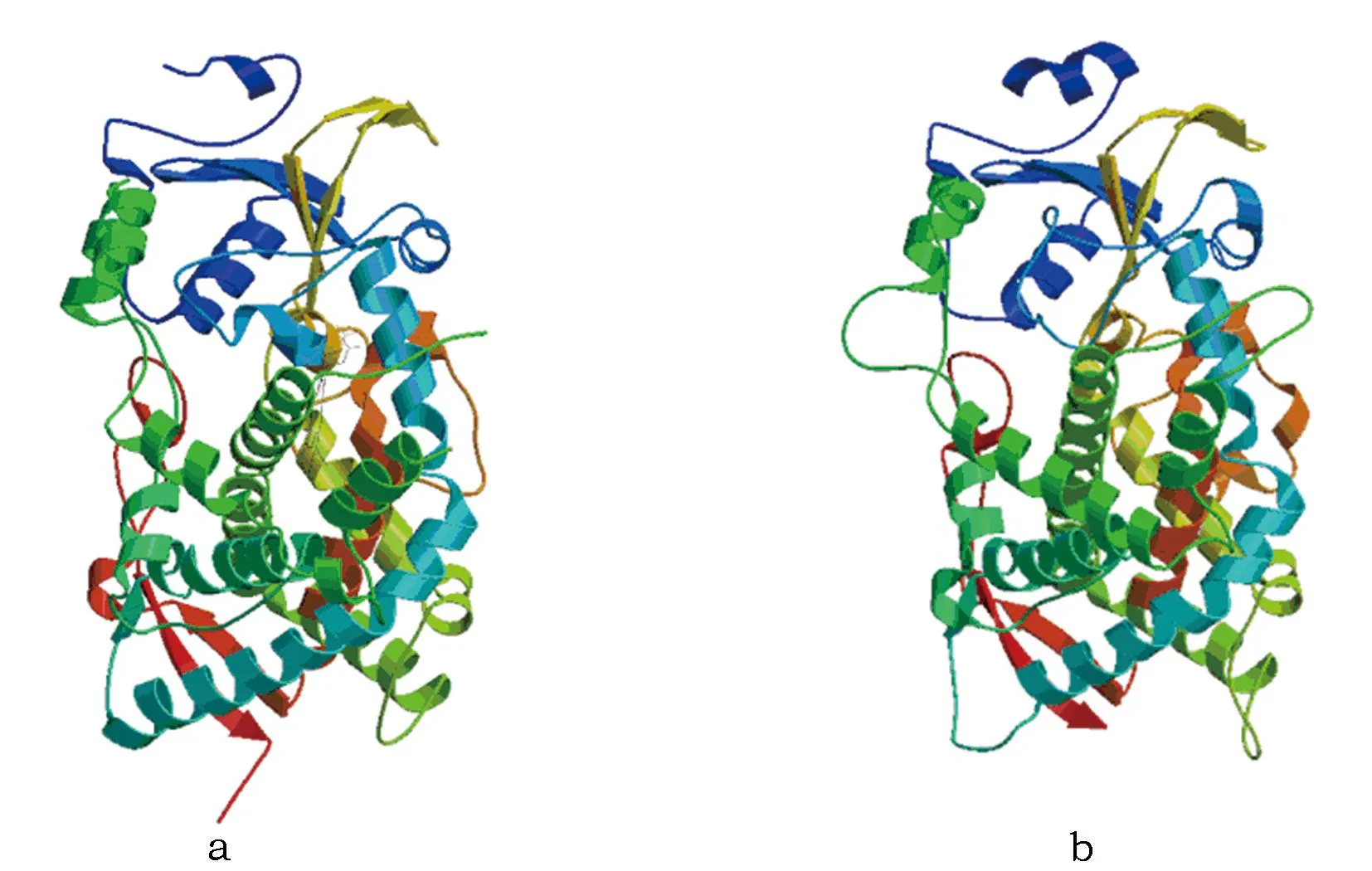
图6 粘虫CYP6AE88(a)和CYP332A1(b)的三维结构预测Fig.6 The 3D structure of M. separata CYP6AE88 (a) and CYP332A1 (b) proteins

图7a 基于最大似然法构建的7种夜蛾科昆虫CYP6AE亚家族的系统发育树Fig.7a The phylogenetic tree of homologous CYP6AE amino acid sequences of 7 Noctuidae species 注:各序列名称后为序列的GenBank 登录号,>50%的bootstrap值标记在树的分支节点上。各种中文名及其CYP6AE亚家族蛋白序列的GenBank登录号见表3。图7b同。Note: The GenBank accession numbers are listed after the sequence names. The bootstrap values greater than 50% are indicated on each branch.Species name and GenBank accession number are seen in Table 3a, b. The same for Figure 7b.
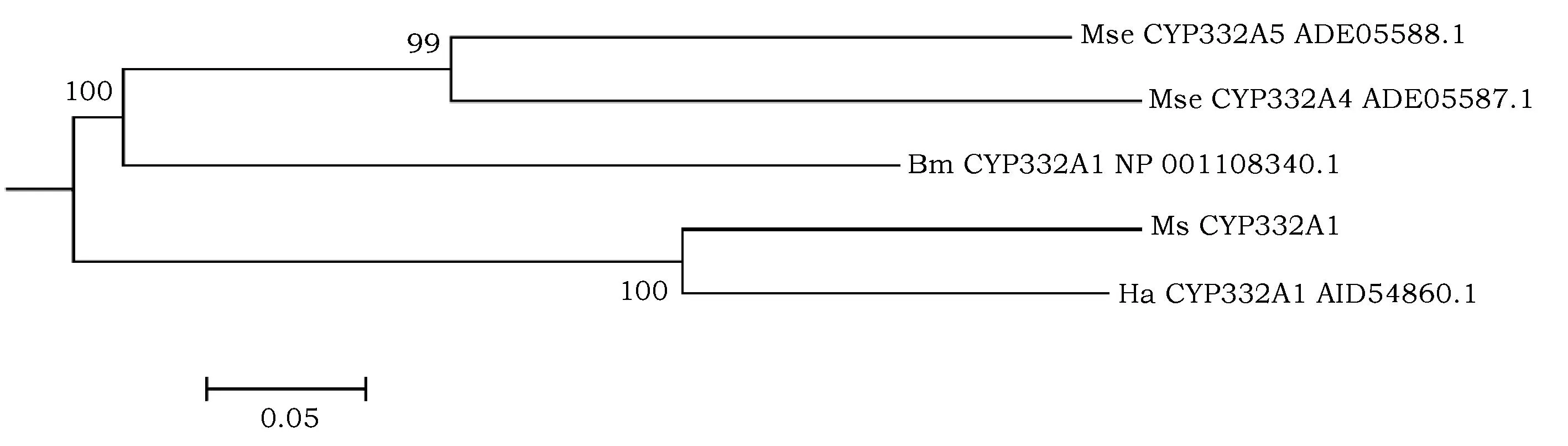
图7b 基于最大似然法构建的4种夜蛾科昆虫CYP332A亚家族的系统发育树Fig.7b The phylogenetic tree of homologous CYP332A amino acid sequences of 4 Noctuidae species
本研究基于粘虫转录组数据获得了其CYP6AE88和CYP332A1的cDNA全长序列,以其编码的氨基酸序列与夜蛾科其他昆虫同源序列对比,发现P450家族的5个保守序列在两个基因均存在,证明上述的两条序列均为细胞色素 P450家族的成员。不同的是粘虫的这两个基因的Meander区都存在一个氨基酸的替换:昆虫P450的Meander区(PxxFxPE/DRF)结构域中的第4个氨基酸为苯丙氨酸,在粘虫CYP6AE88 和CYP332A1的相同位置分别被替换为酪氨酸和色氨酸,三者均为芳香族氨基酸,这可能与不同的昆虫物种有关。
真核生物的CYP蛋白质的分子量大多在55~60 kD这一范围内[21,22],在本研究中,粘虫CYP6AE88的理论分子量为58.9 kD,CYP332A1理论分子量为58.4 kD,位于上述范围内。软件ProtParam tool和TMHMM Server v. 2.0推测以上两个蛋白为亲水性的膜结合蛋白,关于其功能的研究有待进一步推进。
通过相似性(表3a,b)和系统发育或聚类树(图7a,b)的比较发现,相似性高的序列在系统发育树或聚类树上聚为一支。在图7a中,粘虫CYP6AE88与草地贪夜蛾CYP6AE43首先聚为一支,然后再与棉铃虫CYP6AE50成为单系。粘虫CYP6AE88与草地贪夜蛾CYP6AE43的一致率为71%,相似率为84%,与棉铃虫CYP6AE50的一致率为69%,相似率为85%,可见序列的一致率的大小与树形拓扑结构更吻合。
本研究基于粘虫转录组测序信息鉴定出CYP6AE88和CYP332A1两个亚家族基因,丰富了粘虫乃至鳞翅目CYP450超家族基因的数据;并利用多种生物信息学软件,全面分析了其序列特征,预测了蛋白功能,构建了系统发育树,为粘虫的杀虫剂抗性分子机制研究提供了一定的理论基础。
参考文献
[1]江幸福, 张蕾, 程云霞,等. 我国粘虫研究现状及发展趋势[J]. 应用昆虫学报, 2014, 51(4): 881-889.
[2]姜玉英, 李春广, 曾娟,等. 我国粘虫发生概况: 60年回顾[J]. 应用昆虫学报, 2014,51(4): 890-898.
[3]Sharma H C, Davies J C. The Oriental Armyworm, Mythimna separata (Wlk.) Distribution, biology and control: a literature review. Miscellaneous Report No 59 [M]. London:Overseas Development Administration, Wrights Lane, 1983:18-29.
[4]Singh BB, Singh R. Major rice insect pests in northeastern UP [J]. Inernational Journal of Life Sciences Biotechnology and Pharma Research, 2014, 21:124-143.
[5]杨春龙, 龚国玑, 谭福杰等. 粘虫抗药性监测及其机制的初步研究[J]. 植物保护, 1995, 21(3):124-143.
[6]董杰, 刘小侠, 岳瑾等. 北京地区粘虫对5种杀虫剂的抗药性[J]. 农药学学报, 2014, 16(6): 687-692.
[7]Feyereisen R. Evolution of insect P450[J]. Biochemical Society Transactions, 2006, 34(Pt 6): 1252-1255.
[8] 冷欣夫,邱星辉. 细胞色素P450酶系的结构、功能与应用前景[M]. 北京: 科学出版社, 2001: 140-171.
[9] Feyereisen R. Insect CYP genes and P450 enzymes [M]. In: Gilbert LI ed. Insect Molecular Biology and Biochemistry. Oxford: Elsevier Press, 2012:236-316.
[10]Brun-Barale A, Héma O, Martin T, et al. Multiple P450 genes overexpressed in deltamethrin-resistant strains of Helicoverpa armigera[J]. Pest Management Science, 2010, 66(8): 900-909.
[11]Jones RT, Bakker SE, Stone D, et al. Homology modelling of Drosophila cytochrome P450 enzymes associated with insecticide resistance [J]. Pest Management Science, 2010, 66(10): 1106-1115.
[12]Edi CV, Luc D, Jenkins AM, et al. Cyp6 p450 enzymes and ace-1 duplication produce extreme and multiple insecticide resistance in the malaria mosquito Anopheles gambiae[J]. Plos Genetics, 2014, 10: e1004236.
[13]Zimmer CT, Bass C, Williamson MS, et al. Molecular and functional characterization of CYP6B23, a cytochrome p450 conferring resistance to pyrethroids in European populations of pollen beetle, Mligethes aeneus[J]. Insect Biochemistry and Molecular Biology, 2014, 45(1): 18-29.
[14]艾均文, 王根洪, 李艳红,等. 家蚕P450基因CYP18A1的克隆、序列分析及转录活性[J]. 昆虫学报, 2008, 51(3): 237-245.
[15]唐尧, 乔梁, 张玉娟,等.中华按蚊CYP6Y亚家族基因的鉴定和生物信息学分析[J]. 昆虫学报, 2014:6(6): 663-672.
[16]Feyereisen R. Insect cytochrome P450 [M].In:Gilbert LI,Iatrou K,Gill SS ed.Comprehensive Molecular Insect Science, Oxford: 2005:1-77.
[17]Jensen NB, Zagrobelny M, HjernK, et al. Convergent evolution in biosynthesis of cyanogenic defence compounds in plants and insects[J]. Nature Communications, 2011, 2: 273. doi:10.1038/ncomms 1271.
[18]Sezutsu H, Le Goff G, Feyereisen R. Origins of P450 diversity [J]. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 2013, 368(1612): 20120428.
[19]Jouraku A, Yamamoto K, Kuwazaki S, et al. KONAGAbase: a genomic and transcriptomic database for the diamondback moth, Plutella xylostella [J]. BMC Genomics, 2013, 14: 464.
[20]Pauchet Y, Wilkinson P, Vogel H, et al. Pyrosequencing the Manduca sexta larval midgut transcriptome: messages for digestion, detoxification anddefence [J]. Insect Molecular Biology, 2010, 19(1): 61-75.
[21]Nikou D, Ranson H, Hemingway J. An adult-specific CYP6 P450 gene is overexpressed in a pyrethroid-resistant strain of the malaria vector, Anopheles gambiae [J]. Gene, 2003: 318(5):91-102.
[22]车燕飞, 张玉娟, 唐尧,等. 中华按蚊CYP6P5基因的生物信息学鉴定及特征分析[J]. 中国媒介生物学及控制杂志, 2014, 25(3): 205-210.
(编辑:张贵森)
Identification and bioinformatics analyses of genesofCYP6AE88 andCYP332A1 inMythimnaseparata
Li Min1, 2, Li Shengcai2, Wang Qing1, Zhang Hufang2*
(1.DepartmentofBiology,TaiyuanNormalUniversity,Taiyuan030031,China; 2.CollegeofAgriculture,ShanxiAgriculturalUniversity,Taigu030801,China)
Abstract:To identify CYP450 genes of Mythimna separate, analyze their bioinformatic characteristics and infer the possible functions for further research on their roles in insecticide resistance. CYP6AE88 and CYP332A1 of M.separata were retrieved from its transcriptome annotation data and identified with bidirectional Blast using Helicoverpa armigera CYP6AE20v2 and CYP332A1 as query, respectively. The structure, characteristics and possible functions of CYP6AE88 and CYP332A1 genes were analyzed using bioinformation methods. The phylogenetic relationships of CYP6AE88 and CYP332A1 sequences of M. separate and representative noctuid species was constructed using maximum likelihood methods, respectively. Two CYP450 genes were identified from M.separata transcriptome sequenced data and named as CYP6AE88 (GenBank accession number: KU145393) and CYP332A1 (GenBank accession number: KU145394), respectively. They were 1590 bp and 1542 bp in length,encoding 509 and 503 amino acids, respectively. Basic properties of the protein CYP6AE88 and CYP332A1 showed that the molecular weight was 58.9kD and 58.4 kD, the isoelectric point was 7.95 and 8.03, the predicted hydrophobic region was 5-22 and 5-23, the transmembrane region was 2-21 and 2-24, respectively. Both of the two deduced amino acid sequences had no signal peptide sequence and were localized in cytoplasm. Protein domain prediction showed that the encoded proteins of these two genes contained five P450 characteristic sequences and had multiple enzyme active sites. The 3D structural prediction showed that CYP6AE88 had 18 α-helix and 9 β-strands, while CYP332A1 had 19 α-helix and 10 β-strands. Phylogenetic analysis of CYP6AE showed that M.separate CYP6AE88 were grouped with (Spodoptera frugiperda CYP6AE43 + Spodoptera littoralis CYP6AE49) (BP=72%), and then formed monophyletic relationship with Helicoverpa armigera CYP6AE20v2/50/19 (BP=100%). Clustering analysis of CYP332A showed that M.separate CYP332A1 and H.armigera CYP332A1 were the first cluster (BP=100%). This study laid a foundation for further study on the roles of CYP6AE88 and CYP332A1 in M.separate insecticide resistance.
Key words:Mythimna separata; CYP450; CYP6AE88; CYP332A1; Bioinformatics analysis
中图分类号:S433.4
文献标识码:A
文章编号:1671-8151(2016)03-0191-10
基金项目:中国博士后研究经费(134845);国家自然科学基金项目(31501840);山西省科技攻关项目(NO.20150311010-7);太原师范学院大学生创新创业项目(CXCY1610)
作者简介:李敏(1983-),女(汉),山东莱芜人,博士研究生,研究方向:昆虫分子生物学*通讯作者:张虎芳,教授,硕士生导师。Tel:0354-6288225,E-mail: zh_hufang@sohu.com
收稿日期:2015-12-05修回日期:2016-01-06
