沉香DNA提取方法及种质资源聚类分析研究*
2016-03-20钟志敏吴文如赖小平张桂芳
钟志敏,吴文如**,赖小平**,张桂芳,黄 松
(1. 广州中医药大学 中药学院 广州 510006;2. 东莞广州中医药大学中医药数理工程研究院 东莞 523808)
Zhong Zhimin1, Wu Wenru1, Lai Xiaoping1, Zhang Guifang1, Huang Song2
(1.School of Chinese Materia Medica, Guangzhou University of Chinese Medicine, Guangzhou 510006, China;
2. Dongguan Mathematical Engineering Academy of Chinese Medicine, Guangzhou University of Chinese Medicine, Dongguan 523808, China)
沉香DNA提取方法及种质资源聚类分析研究*
钟志敏1,吴文如1**,赖小平1**,张桂芳1,黄 松2
(1. 广州中医药大学 中药学院 广州 510006;2. 东莞广州中医药大学中医药数理工程研究院 东莞 523808)
目的:分别建立适用于提取不同类型沉香样品(叶片、干燥枝条、药材)DNA的方法,通过ITS2序列对沉香药材进行种质资源聚类分析。方法:根据沉香叶片、干燥枝条、药材的组织差异,分别采用试剂盒法和优化的CTAB法提取DNA;利用紫外分光光度仪、琼脂糖凝胶电泳、PCR扩增及ITS2序列测定来检测DNA的纯度、浓度及可用性,以比较不同DNA提取方法的效率;将样品测序结果与从GenBank下载的沉香属(Aquilaria spp.)、拟沉香属(Gyrinops spp.),及沉香药材常见混伪品的ITS2序列,进行聚类分析。结果:优化的CTAB法提取得到的DNA纯度比试剂盒法稍低,但浓度更高。两种方法所提沉香总DNA 进行ITS2序列的PCR扩增成功率和测序成功率均为100%。ITS2序列能准确鉴别出4个沉香混伪品和2个拟沉香物种,且15个沉香样品ITS2序列与Aquilaria subintegra(泰国)、Aquilaria sinensis(中国)、Aquilaria crassna(柬埔寨、泰国)三个沉香物种均100%相似。结论:本研究中的两种DNA提取方法均可用于沉香DNA条形码研究;优化CTAB法比试剂盒法更适用于药材DNA 的提取,该方法为有关中药材DNA的提取提供了参考;ITS2序列可为沉香种质资源的鉴定和系统发育分析提供必要的实验依据。
CTAB法 试剂盒法 DNA条形码 ITS2 聚类分析
据2015版《中国药典》记载,沉香(Aquilariae Lignum Resinatum)是瑞香科植物白木香Aquilaria sinensis(Lour.)Gilg含有树脂的木材,其味辛、苦,性微温,具有行气止痛、温中止呕、纳气平喘等功效。沉香是中国、日本、印度及其他东南亚国家的传统名贵药材和天然香料,其在药用、精油、香料等方面的市场需求量巨大,原材料远销中东、日韩、欧美等国家和地区。沉香物种来源丰富,从南亚次大陆的东北部到印度尼西亚群岛、巴布年亚新几内亚群岛,分布着至少20余种的沉香属植物,均可产沉香[1-3]。中国沉香药材可分为国产和进口两类,国产沉香唯一法定基源为瑞香科沉香属植物白木香[4];进口沉香来源较多,其中以奇楠沉香最为贵重,但其品种来源不明[5]。由于不同品种、等级、产区的沉香价格差异极大,市场上以劣充好或以假乱真的现象十分严重,按传统的鉴别检验标准,难以判断沉香药材的基源植物和真伪优劣,故沉香药材的质量评价难度较大。
为更好地保护和利用沉香属植物资源及其物种的多样性,国内外研究学者在沉香形态学表型多样性[6,7]、细胞水平染色体多样性[7-9]、生化水平等位酶多样性[10]等方面开展了大量研究。近年来,DNA条形码技术被逐渐应用到沉香的物种鉴定及遗传进化分析中[11-14],并取得了良好效果。高晓霞[13]、刘少烽[14]、邵敏[15]等利用改良CTAB法或PTB法从不同来源的沉香中成功提取到DNA。然而,沉香药材种类繁多,一些树脂含量高、经过高温干燥处理或者年代久远的沉香药材DNA的提取效果仍然很不理想。
本研究根据沉香叶片、干燥枝条、商品药材组织形式及所含成分的种类和含量的不同,分别采用试剂盒法和优化CTAB法提取三类沉香样品(叶片、干燥枝条、药材),以期建立适于提取不同类型沉香样品DNA的最佳方法,以满足其DNA条形码研究的需要;并将样品测序结果与GenBank中已有沉香属物种、拟沉香属物种、及沉香药材常见混伪品的ITS2序列进行比对分析和种质资源聚类分析,鉴定本实验沉香样品的种质来源,并尝试从分子水平上鉴定沉香药材的真伪优劣。
1 材料与方法
1.1 实验材料
沉香叶片和干燥枝条采自广州中医药大学药圃时珍山和药王山,经中药鉴定学教研室吴文如副教授鉴定其原植物为瑞香科白木香Aquilaria sinensis(Lour.)Glig,用70%乙醇消毒和无菌水冲洗晾干后,-20℃保存;国产和进口沉香药材分别从国内外药材市场购买,硅胶干燥器中保存(表1)。

表1 供试沉香材料
1.2 主要仪器
BSA224S电子分析天平(德国ViBRA 公司),Water Bath SY-1210恒温水浴锅(美国Crystal 公司),IKA®VORTEX GENIUS 3涡旋混匀机(美国Barnstead公司),BCD-260WDBD超低温冰箱(丹麦Arctiko 公司),5424DQ261173台式高速离心机(德国Eppendorf 公司),Centrifuge 5424R台式冷冻高速离心机(德国Eppendorf 公司),Power PacTMBasic水平电泳仪(美国BioRad 公司),Thermo Cycler Block 5020PCR 扩增仪(美国BioRad 公司),NanoDrop 2000超微量紫外分光光度计(美国Thermo 公司),Tanon 2500凝胶成像仪(上海天能科技有限公司)。
1.3 主要试剂
Goldview 电泳染料(日本TaKaRa 公司,批号:D1210),PrimeSTAR®HS(Premix)(日本TaKaRa公司,批号:#A4101A),DNeasy Plant Mini Kit(德国Qiagen 公司,批号NO.145017297)、Plant Genomic DNA Kit(北京天根生化科技有限司,批号:#03825)、CTAB(美国Amresco 公司);其余均为国产分析纯试剂;引物由华大生物科技有限公司合成。
1.4 操作方法
1.4.1 采用优化CTAB-A法提取沉香叶片基因组DNA
(1)称取沉香新鲜叶片100 mg,加入0.01 g PVP-40,液氮研磨至粉末状,转入2 mL离心管;(2)加入1 000 μL DNA洗涤液[100 mmol·L-1Tris-HCl(pH=8.0),0.14 mol·L-1NaCl,20 mmol·L-1EDTA]和1%的β-巯基乙醇,涡旋1 min,4℃,12 000 rpm·min-1,离心10 min,弃上清;(3)按步骤(2)重复操作两次;(4)加入700 μL 65℃预热的2%CTAB提取液[100 mmol·L-1Tris-HCl(pH=8.0),1.4 mol·L-1NaCl,20 mmol·L-1EDTA,3%CTAB(m/ v)],涡旋1 min,65℃水浴30 min;(5)加入700 μL酚:氯仿:异戊醇(体积比为=25∶24∶1),涡旋30 s,室温,12 000 rpm·min-1,离心10 min;(6)取上清,按步骤(5)重复一次;(7)取上清,加等体积氯仿:异戊醇(体积比为=24∶1),涡旋30 s,室温,12 000 rpm·min-1,离心10 min;(8)取上清,加等体积预冷异丙醇,轻轻混匀,挑出絮状物,用70%乙醇洗涤两次,再用无水乙醇洗涤一次,室温风干,50 μL TE溶解,-20℃保存。
1.4.2 采用优化CTAB-B法提取沉香干燥枝条基因组DNA
(1)取干燥枝条50 mg,液氮研磨至粉末状,转入2 mL离心管;(2)加入700 μL 65℃预热的2%CTAB提取液[100 mmol·L-1Tris-HCl(pH=8.0),1.4 mol·L-1NaCl,20 mmol·L-1EDTA,3%CTAB(m/v)],10 μL 1%PVP-40,2 μL 1%的β-巯基乙醇,涡旋1 min,65℃水浴60 min;(3)室温,12 000 rpm·min-1,离心10 min,取上清,加入等体积的Tris-饱和酚,涡旋30 s,12 000 rpm·min-1,离心10 min;(4)取上清,加入等体积酚∶氯仿∶异戊醇(体积比为=25∶24∶1),涡旋30 s,室温,12 000 rpm·min-1,离心10 min;(5)取上清,加等体积氯仿∶异戊醇(体积比为=24∶1),涡旋30 s,室温,12 000 rpm·min-1,离心10 min;(6)按步骤(5)重复操作一次;(7)加入1/10体积3 mol·L-1醋酸钠和2.5倍体积无水乙醇,-20℃沉淀2 h;(8)室温,12 000 rpm·min-1,离心10 min,用70%乙醇洗涤两次,再用无水乙醇洗涤一次,室温风干,50 μL TE溶解,-20℃保存。
1.4.3 采用优化CTAB-C法提取沉香药材基因组DNA
(1)取沉香药材粉末50 mg,液氮研磨,加入0.01 g PVP-40,2 μL蛋白酶K(20 mg·mL-1),35 μL 20% SDS,2 μL 1M DTT和500 μL DNA洗涤液[100 mmol·L-1Tris-HCl(pH=8.0),0.14 mol·L-1NaCl,20 mmol·L-1EDTA],继续在液氮条件下研磨2 min,转入2 mL离心管,用1 mL DNA洗涤液清洗研钵并将清洗液倒入离心管,室温,12 000 rpm·min-1,离心10 min;(2)弃上清,加入1 000 μL DNA洗涤液和2 μL 1M DTT,涡旋1 min,4℃,12 000 rpm·min-1,离心10 min,弃上清;(3)同1.4.1步骤(3)-(7);(4)取上清,加等体积预冷异丙醇,轻轻混匀,-20℃沉淀2 h;(5)室温,12 000 rpm·min-1,离心10 min,用70%乙醇洗涤两次,再用无水乙醇洗涤一次,室温风干,20 μL TE溶解,-20℃保存。
1.4.4 试剂盒法
(1)参照Plant Genomic DNA Kit试剂盒说明书提取沉香叶片、干燥枝条、药材基因组DNA;(2)参照DNeasy Plant Mini Kit试剂盒说明书提取沉香药材基因组DNA。
1.5 ITS2序列扩增体系和程序
扩增体系和程序参照Prime STAR®HS(Premix)说明书并进行优化。叶片和干燥枝条样品扩增体系为50 μL:Prime STAR®HS(Premix) 25 μL; 正向引物ITS2 S2F[16](5’-ATGCGATACTTGGTGTGAAT- 3’)、反向引物ITS2 S3R[16](5‘- GACGCTTCTCCAGACTACAAT- 3’)各 0.7 μL(20 μmol·L-1);模板 DNA 2 μL;无菌去离子水定容到 50 μL。药材样品的扩增体系为50 μL:Prime STAR®HS(Premix) 25 μL; 正反向引物(20 μmol·L-1)各 0.8 μL;模板 DNA 6 μL;无菌去离子水定容到 50 μL。叶片和干燥枝条样品扩增程序:94℃预变性 2 min;98℃变性10 s,55℃退火 10 s,72℃延伸30 s,40 次循环;72℃延伸 5 min。药材样品的退火温度为58℃,其余程序同叶片和干燥枝条。
1.6 DNA模板和PCR产物的检测
用超微量紫外分光光度计测定DNA A260 nm/A 280 nm的值及浓度。将DNA模板及扩增产物在1.5%的琼脂糖凝胶上电泳分离,电压100 V,电泳22 min。凝胶成像仪中观察成像并拍照保存。
1.7 ITS2序列测定及聚类分析
将电泳条带单一、清晰、明亮的PCR 扩增产物送至广州华大生物科技有限公司进行双向测序。所有序列用The ITS2 Database 在线工具(http∶//its2. bioapps.biozentrum.uni-wuerzburg.de/)及SnapGene®Viewer(Version2.6.2) 软件剪切并校正,除去序列两端质量差的碱基,用BioEdit(Version 7.01)进行多重比对,在GenBank 数据库中进行Blast,最后用MEGA(Version 6.0)软件,选择邻接法(Neighbor-Joining,NJ)构建系统进化树,并以Kimura.2-parameter model做1 000次可信度分析。
2 结果与分析
2.1 不同DNA提取方法提取效率比较分析
由于沉香不同组织所含化学成分差异较大,叶片中主要含有多糖、脂质类成分,因此需先用DNA洗涤液洗涤数次;干燥枝条中尤其是树皮中含有大量纤维[17]、蛋白质及酚类成分,需要加入Tris饱和酚除去;而含树脂药材中主要含有色酮类、甾体类、萜类、挥发油类成分[18-20],需用蛋白酶K、SDS、DTT等除杂,并在加入DNA洗涤液时充分研磨。本实验优化CTAB法提取的沉香总DNA A260/A280的值绝大多数分布在1.7-2.0之间,浓度达到几十到几百ng·μL-1(表2),叶片、干燥枝条总DNA电泳条带单一、清晰,但由于不同产地、不同结香方法、不同树种来源、不同质量等级、不同加工处理方法的市售沉香药材化学成分种类及含量差异较大[18-22],样品S1、S2总DNA的纯度较低,浓度差异也较大(图1)。试剂盒法提取的沉香总DNA A260/A280的值绝大多数分布在1.8-2.0之间,但浓度只有几到几十ng·μL-1,且去盐程度较低,因此其A260/A230出现负值(表2)。叶片和干燥枝条总DNA电泳条带单一、清晰,而药材总DNA未见明显电泳条带(图1)。

表2 试剂盒法和优化CTAB法提取15个样品总DNA的纯度和浓度
2.2 沉香ITS2-PCR扩增结果分析

图1 试剂盒法和优化CTAB法提取沉香DNA电泳图
沉香叶片和干燥枝条DNA的PCR产物电泳条带均单一、清晰且明亮(图2 -G、H、J、K)。但由于沉香药材样品质量差异,其相应的PCR产物电泳条带亮度也参差不齐;此外,药材DNA降解较严重,其PCR产物总体亮度也较叶片和干燥枝条低且有弥散;由于优化CTAB法所得药材DNA的纯度较低,其PCR扩增产物电泳主条带下方有非特异性条带,且在点样孔有杂质污染(图2)。
2.3 ITS2聚类分析
本研究15个沉香样品(GenBank接收号为KX351409-KX351421和KX369227-KX369228)及GenBank中下载的拟沉香属、沉香属及其常见混伪品(泡桐木、马尾松、檀香、降香)的ITS2序列比对、分析结果见表3、图3。ITS2序列能准确鉴别出4个沉香混伪品和2个拟沉香物种。15个沉香样品间ITS2序列与A. subintegra(泰国)、A. sinensis(中国)、A. crassna(柬埔寨、泰国)3个沉香物种来源的ITS2序列[23]及陈士林[16]等提供的GenBank中沉香序列均100%相似,且K2P距离均为0,因此可判断本研究所选取的序列可靠,并确认本实验所用15种沉香为其中一种。
3 讨论
目前,新鲜植物叶片DNA的提取已有较多可行的方法,但由于不同物种叶片中主要化学成分的种类和含量差异,目前还没有一种通用的DNA提取方法。木材DNA提取的方法仍处于试验和摸索阶段。1988年,De Filippis[24]利用改良的CTAB法成功提取刺槐Robinia pseudoacacia木材DNA,后继学者们也采用改良CTAB或商业试剂盒的方法先后从松属Pinus spp.[25],栎属Quercus spp.[26,27],龙脑香科Dipterocarpaceae[28]等多种木材中提取出可用于PCR扩增的DNA。而中药材样品来源复杂,所含成分的种类和含量千差万别,针对不同药材、不同药用部位进行DNA提取方法的摸索对于促进中药材分子生物学研究是十分必要的。为此,罗焜[29]等按照中药材药用部位及成分的差异建立了改良CTAB法,该方法提取DNA的PCR扩增成功率达90%,为中药材DNA的提取及后续的DNA条形码研究奠定了重要基础。
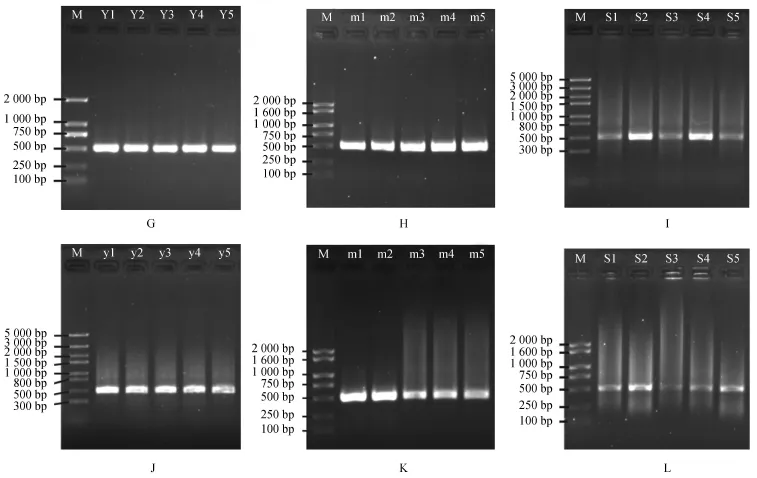
图2 试剂盒法和优化CTAB法提取15个样品PCR产物电泳图

与高晓霞[13]、刘少烽[14]、邵敏[15]等仅针对白木香叶片或者药材的DNA提取方法相比,本研究系统全面的建立了适用于不同类型沉香样品(叶片、干燥枝条、药材)DNA提取的方法及优化的PCR体系和程序,为后续DNA条形码研究及对沉香药材进行种质资源聚类分析奠定基础,具有一定创新性。本研究结果表明,试剂盒法所提取的沉香叶片、干燥枝条、药材总DNA的ITS2-PCR扩增成功率和测序成功率均为100%,但其成本较高,且得到的样品浓度较低。本实验建立的优化CTAB法提取的DNA浓度均较试剂盒法高,ITS2-PCR扩增成功率亦为100%,符合送样测序标准,且成本较低。此外,沉香不同组织需用不同的DNA提取方法和PCR反应条件;即使组织类型相同,在 DNA含量较低,杂质含量较多,储存年代久远的市售沉香药材之间,DNA提取效果及PCR扩增结果仍存在差异。此结果与罗焜[29]等的研究结果一致,即应当根据药材加工方法、贮藏时间、炮制工艺的不同,甚至是中成药等实际情况对提取方法及PCR反应条件进行调整。

图3 样品与拟沉香、沉香及其常见混伪品ITS2序列NJ树
从本实验基因组DNA紫外分光光度仪纯度的检测结果与DNA模板及PCR产物电泳检测结果来看,尽管总DNA A260/A280的值不在1.8-2.0的纯度范围内,甚至有些沉香药材样品的DNA模板电泳分离未见主条带,但由于所用的优化CTAB法提取的DNA浓度较高,试剂盒法提取的DNA纯度较高,且使用了高保真的PCR预混酶,其PCR扩增成功率仍能达到100%。A260/A230产生负值主要是由于在浓度很低的DNA溶液中存在一些杂质干扰,但这并不影响所得DNA的可用性,因为尽管DNA有一些污染,其仍然能进行PCR反应,且PCR产物经纯化后仍可用于ITS2序列的测定。可见,紫外检测和电泳检测仍存在偏差,在总DNA含量较少又含有杂质的情况下,应结合PCR扩增结果综合分析,必要的情况下,还可以结合测序结果的测序成功率来评估所提取DNA的可用性。
近年来,DNA条形码作为一项新兴技术被大量用于物种鉴别和亲缘关系分析,然而ITS2作为应用最广的植物DNA条形码,并不能100%的鉴别亲缘关系较近的物种。ITS2能100%的鉴别沉香常见混伪品及两个拟沉香物种,但A. subintegra(泰国)、A. sinensis(中国)、A. crassna(柬埔寨、泰国)三个沉香物种的ITS2序列完全一致,不存在差异。综上,ITS2序列能适用于较高分类等级的系统学研究,能够简便的鉴别沉香的真伪,但无法100%鉴别沉香属内亲缘关系较近的物种来源,实际工作中仍需结合传统的分类鉴定方法来综合判断。此外,随着第三代测序技术的发展和测序成本的降低,一些更具前景,能更高效精确的对亲缘关系较近的物种进行鉴别的植物DNA条形码片段被广泛地发掘应用。其中,叶绿体全基因组序列在贝母属[30]、石斛属[31]等药用植物的鉴别中得到广泛的应用并展现出良好的鉴别效率。因此,用沉香全叶绿体基因组作为“超级条形码”进行沉香鉴定及系统进化研究,或从沉香叶绿体全基因组序列中筛选出能鉴别亲缘关系较近的沉香物种的特异性DNA条形码,将为日后沉香分子鉴定及系统进化分析的研究提供参考。
1 Santisuk T. Taxonomy, geography and ecology of Aquilaria Lamk. (Thymelaeaceae: Aquilarioideae) in contimental Asia. Second International Agarwood Conference, 2007.
2 Kiet L C, Keasler PJA, Eurlings M, et al. A new species of Aquilaria (Thymelaeaceae) from Vietnam. Blumea J Plant Taxon Plant Geogr, 2005, 50(1): 135-141.
3 Oyen LPA, Nguyen X D, et al. Plant resources of south-east Asia No.19. Essential oil Plants. Bogor: Prosea Foundation Publisher, 1999.
4 国家药典委员会, 中华人民共和国药典(一部). 北京: 北京化学工业出版社, 2015: 172.
5 Eurlings MCM, Gravendeel B, et al. TrnL-trnF sequence data imply paraphyly of Aquilaria and Gyrinaps (Thymelaeaceae) and provide new perspectives for agarwood identification. Plant Syst Evol, 2005, 254:1.
6 赵翾. 白木香[Aquilaria sinensis (Lour.) Spreng]的遗传多样性研究.广州:华南理工大学博士士学位论文 , 2006.
7 赵翾,赵树进. 白木香群体的表型多样性分析. 华南理工大学学报:自然科学版, 2007,35(4):117-122.
8 申彦晶,赵树进. 白木香核型与Giemsa C-带带型研究. 中药材, 2007, 30(7): 762-765.
9 申彦晶,焦旭雯,赵树进. 不同居群白木香的染色体研究. 广西植物,2009,29(2):192-197.
10 黄崇才. 白木香Aquilaria sinensis (Lour.)Gilg种质形态学与细胞学研究. 广州: 广州中医药大学硕士学位论文, 2009.
11 申彦晶,谭雪梅,赵翾,等. 我国不同地区白木香核糖体DNA内转录间隔区碱基序列分析. 中华中医药杂志, 2009, 24(4): 539-541.
12 牛宪立,姬可平,吕国庆. 白木香rDNA ITS序列测序鉴定的初步研究. 广东农业科学,2010, 2: 167-169.
13 高晓霞,刘少烽,陈晓东,等. 基于ITS2条形码序列鉴定商品沉香基源植物. 时针国医国药, 2015,26(8):1932-1935.
14 刘少烽,陈晓东,朱爽,等. 沉香DNA的提取及其ITS2-PCR体系的优化. 中国实验方剂学杂志, 2015,21(2):7-11.
15 邵敏,周鹤峰,唐厉波,等. 沉香随机扩增多态性DNA反应体系的建立. 时珍国医国药,2009,20(1):74-76.
16 陈士林. 中国药典中药材DNA条形码标准序列. 北京:科学出版社, 2015:25.
17 杨春芳. 沉香叶化学成分及其生物学功效研究. 广州: 华南师范大学硕士学位论文 , 2012.
18 李嶶,梅文莉,左文键,等. 国产人工打洞沉香的化学成分研究.热带亚热带植物学报, 2016, 24(3): 342-347.
19 王红妮,董文化,梅文莉,等. 柯拉斯那沉香化学成分分析. 热带生物学报,2016,7(1):111-116.
20 杨林,乔立瑞,谢丹,等. 国产沉香中的倍半萜类和单萜类化学成分. 中国中药杂志, 2012,37(13):1973-1976.
21 谷田,彭海刚. 奇楠沉香挥发油化学成分分析. 广东化工, 2012,39(228): 257-259.
22 陈晓颖,高英,李卫民. 不同结香方法与国产沉香挥发性化学成分的相关性研究. 中国药房,2012,23(11): 1017-1020.
23 Lee S Y, Ng W L, Mahat M N, et al. DNA Barcoding of the Endangered Aquilaria (Thymelaeaceae) and Its Application in Species Authentication of Agarwood Products Traded in the Market. PLoS ONE, 2016, 11(4):E0154631.
24 Filippis L D, Magel E. Differences in genomic DNA extracted from bark and from wood of different zones in Robinia trees using RAPD-PCR. Trees, 1998, 12(6):377-384.
25 Reynolds M M, Williams C G. Extracting DNA from submerged pine wood. Genome, 2004. 47: 994-997.
26 Deguilloux M F, Pemonge M H, Petit R J. Novel perspectives in wood certification and forensics: dry wood as a source of DNA. Proc Biol Sci, 2002, 269:1039-1046.
27 Dumolin-Lapègue S, Pemonge M H, Gielly L, et al. Amplification of oak DNA from ancient and modern wood. Mol Ecol, 1999, 8(12): 2137-2140.
28 Rachma Yanti Y, Leinemann L, Gailing O, et al. Extraction, amplification and characterization of wood DNA from Dipterocarpaceae. Plant Mol Biol Report, 2006, 24: 45-55.
29 罗焜,马培,姚辉,等. 中药DNA条形码鉴定中的DNA提取方法研究. 世界科学技术-中医药现代, 2012,14(2):1433-1439.
30 李滢,姚辉,宋经元,等. 基于吋绿体全基因组的贝母属特异性D NA条形码的筛选. 世界科学技术-中医药现代化, 2016,18(1):24-28.
31 周红,马双姣,杨培,等.基于DNA序列的石斛属药用植物鉴定研究进展. 世界科学技术-中医药现代化, 2016,17(5):950-957.
A Study on the DNA Extraction Methods of Aquilaria and Cluster Analysis of Its Germplasm Resources
This study aimed at establishing DNA extraction methods suitable for Aquilaria samples of different types, such as leaves, dry branches and medicinal herbs for its authentication by DNA barcode, with cluster analysis of its germplasm resources with ITS2 sequences. As the differences between leaves, dry branches and medicinal materials of Aquilaria., kit method and optimized CTAB method were adopted to extract DNAs. Ultraviolet spectrophotometry, agarose gel electrophoresis, PCR and ITS2 sequences were applied to detect the concentration, purity, availability of DNA, respectively. Then the extraction efficiency of the two methods were compared. The ITS2 sequences of Gyrinops spp., Aquilaria spp., and common adulterants of Aquilaria downloaded from GenBank and the sequenced results were clustered and analyzed. As a result, it was found that the purity of DNA extracted by the optimized CTAB method was lower than that by the kit method, while its concentration using the optimized CTAB method was much higher than that using the kit method. The total DNAs extracted by the two methods were amplified with PCR. Both the amplification success rate and the sequencing success rate for ITS2 were 100%. Four adulterants of Aquilaria and two species of Gyrinops were accurately identifyied using ITS2 sequences. The similarity between the fifteen samples and the three species of Aquilaria, including Aquilaria subintegra (Thailand), Aquilaria sinensis (China), and Aquilaria crassna (Cambodia, Thailand), was 100%. In conclusion, both the kit method and the optimized CTAB method can be applied to the studies of DNA barcode. However, the optimized CTAB method was more suitable for the DNA extraction of Chinese materia medica, which provided a reference for the DNA extraction of Aquilaria and other herbal medicines. The ITS2 sequence also provided scientific basis of germplasm identification and phylogenetic analysis.
CTAB method, kit method, DNA barcode, ITS2, cluster analysis
Zhong Zhimin1, Wu Wenru1, Lai Xiaoping1, Zhang Guifang1, Huang Song2
(1.School of Chinese Materia Medica, Guangzhou University of Chinese Medicine, Guangzhou 510006, China;
2. Dongguan Mathematical Engineering Academy of Chinese Medicine, Guangzhou University of Chinese Medicine, Dongguan 523808, China)
10.11842/wst.2016.10.003
R282.2
A
(责任编辑:马雅静,责任译审:朱黎婷)
2016-07-01
修回日期:2016-07-21
* 港澳台科技合作专项项目(2014DFH30010):粤港澳岭南中药综合开发研究,负责人:赖小平;澳门科技大学中药质量研究国家重点实验室开放课题(MUST-SKL-2016-07):基于LAMP技术进行铁皮石斛的特异性分子鉴定研究,负责人:吴文如;广州中医药大学留学人员科技活动资助计划——薪火计划(A1-AFD015142Z09):基于环介导等温扩增技术建立人参与西洋参的快速分子鉴别新方法,负责人:吴文如。
** 通讯作者:吴文如,博士,副教授,主要研究方向:中药分子鉴定研究;通信作者:赖小平,本刊编委,教授,博士生导师,主要研究方向:岭南中药。
