基于重组酶聚合酶扩增技术(RPA)的葡萄卷叶伴随病毒3号检测方法
2016-03-17乾义柯胡白石赛铁尔汗
张 娜, 乾义柯, 魏 霜, 胡白石,陆 平, 赛铁尔汗,
刘中勇3, 张永江5, 张祥林6
(1.伊犁师范学院, 新疆伊宁 835000;2.伊犁出入境检验检疫局, 新疆伊宁 835000;3.汕头出入境检验检疫局, 广东汕头 515041;
4 .南京农业大学, 南京 290000;5.中国检验检疫科学研究院植物检疫研究所, 北京 100029;
6.新疆出入境检验检疫局, 乌鲁木齐 830052)
基于重组酶聚合酶扩增技术(RPA)的葡萄卷叶伴随病毒3号检测方法
张 娜1, 乾义柯2, 魏 霜3, 胡白石4,陆 平2, 赛铁尔汗2,
刘中勇3, 张永江5, 张祥林6
(1.伊犁师范学院, 新疆伊宁835000;2.伊犁出入境检验检疫局, 新疆伊宁835000;3.汕头出入境检验检疫局, 广东汕头515041;
4 .南京农业大学, 南京290000;5.中国检验检疫科学研究院植物检疫研究所, 北京100029;
6.新疆出入境检验检疫局, 乌鲁木齐830052)
摘要:【目的】建立一种快速、灵敏的重组酶聚合酶扩增技术(RPA)检测葡萄卷叶伴随病毒3(Grapevine leafroll-associated virus 3,GLRaV-3)方法。【方法】根据GLRaV-3已测序和已报道的HSP70基因(Heat shock protein 70)保守序列,设计用于RPA检测的特异性引物,建立GLRaV-3的RPA检测方法,并对其特异性和灵敏度进行验证。【结果】建立RPA检测方法能够从GLRaV-3带毒株中检测到约380 bp的特异性条带,仅需在37℃下恒温反应40 min,无需特殊的仪器设备,经特异性评价特异性好,且该方法与普通PCR检测灵敏度基本一致。【结论】建立的RPA检测方法特异性强、灵敏度高,无需特殊的仪器设备,适合GLRaV-3的快速检测方法。
关键词:葡萄卷叶伴随病毒3;RPA;检测方法
0 引 言
【研究意义】葡萄卷叶病毒病(Grapevineleafroll-associatedvirus,GLRaV)是仅次于葡萄扇叶病毒病的一种世界性葡萄病毒病,在国内分布较为普遍,田间感病率较高,对葡萄产量和品质影响很大,且引起该病的病毒是由多种病毒单独或复合侵染造成[1-3]。现已报道的葡萄卷叶伴随病毒有11种,分别为GLRaV- 1~9,GLRaV-Dr,GLRaV-De[4, 5],我国已鉴定明确的葡萄卷叶伴随病毒种类共有6种,即GLRaV- 1~5和7[6, 7]。GLRaV已被证明属于典型的Clostero病毒,仅分布于韧皮部和叶片,其传播不是靠机械,通常是靠感染的母株或接穗等繁殖材料[8]。其中葡萄卷叶伴随病毒3(Grapevineleafroll-associatedvirus3,GLRaV-3)是最重要的一种,为葡萄卷叶病毒属(Ampleovirus)模式Hya种,基因组全长约18 kb,包含13个开放阅读框架,分子量为35 kDa的CP由ORF6编码[9]。它分布最广泛造成的经济损失最大,而且也是这几种病毒中唯一具有自然介体的病毒[10-12]。GLRaV具有半潜隐性,导致葡萄植株生长衰弱,果实成熟延迟,果粒大小不均,着色不良,含糖量减少,产量下降,对葡萄的生长发育有显著影响。因此,研究葡萄卷叶病毒检测方法对该类病毒检验检疫和葡萄无毒苗木生产具有重要意义。【前人研究进展】目前,检测葡萄卷叶病毒的方法主要有指示植物法、酶联免疫吸附法(ELISA),反转录聚合酶链式反应(RT-PCR)和实时荧光PCR法。指示植物检测法比较简单,但其检测速度很慢,而且无法确定侵染病毒的种类,灵敏度也较低。葡萄卷叶伴随病毒ELISA检测方法[13]灵敏度高,操作简单,但所需检测时间也在1~2,而且抗体制备周期长,费用较高,灵敏度不及RT-PCR。利用分子生物学技术建立的RT-PCR[14]、免疫捕捉RT-PCR[15]、实时荧光RT-PCR[16]、探针杂交[17, 18]以及LAMP方法[19]检测结果更为快速、灵敏,但所需要的检测设备和探针试剂成本较高。RPA(Recombinase polymerase amplifcation)是一项新型的等温扩增技术[20],其主要原理为利用重组酶和引物形成微丝在模板DNA上搜索到与之完全互补配对的序列时,在单链DNA结合蛋白的帮助下使模板DNA解链,引物于模板DNA开始配对,并在DNA聚合酶的作用下复制延伸。该方法主要具备以下优点:(1)仅需1对引物即可完成扩增,不需要太复杂的引物设计过程;(2)只需要37 ℃下恒温反应即可,不需要特殊的热循环设备;(3)反应时间仅需40 min;(4)结果易于判断,不像LAMP产物的弥散带,RPA扩增产物根据引物设计位点具有特定大小的条带。【本研究切入点】目前国内未见报道有关RPA技术在葡萄病毒检测方面的应用,研究依据GLRaV-3的HSP70基因(Heat shock protein 70)保守序列,设计用于RPA检测的特异性引物,建立GLRaV-3的RPA检测方法。【拟解决的关键问题】利用RPA技术,建立一种简便高效、特异性强、灵敏度高的方法来快速检测GLRaV-3,以期为葡萄病毒病害的田间诊断、预测预报和葡萄无毒苗木的生产等提供更好的技术支持。
1材料与方法
1.1 材 料
含葡萄卷叶伴随病毒1 (GLRaV-1)、葡萄卷叶伴随病毒2(GLRaV-2)、葡萄卷叶伴随病毒3(GLRaV-3)、葡萄卷叶伴随病毒4(GLRaV-4)、葡萄卷叶伴随病毒5(GLRaV-5)及葡萄病毒A(GrapevinevirusA,GVA)的葡萄叶片采自新疆伊宁,阴性对照为脱毒葡萄苗,样品经PCR检测和序列测定确认后-80℃冻干保存。
1.2 方 法
1.2.1 总RNA提取及cDNA的合成
参照试剂盒操作(植物总RNA提取试剂盒,天根生物科技有限公司),提取感病葡萄叶片的总RNA。并参照反转录试剂盒(PrimeScript RT-PCR Kit,TaKaRa)的操作,将提取的RNA制备成cDNA,-20℃保存备用。
1.2.2引物设计
参考RPA引物设计的要求[21],根据GLRaV-3已测序和已报道的HSP70基因的保守序列,设计检测葡萄卷叶伴随病毒3号的RPA检测引物,扩增条带380 bp。上游引物GLRaV-3-F:5′-ATTGAGGAACAATTTGGGGGACATCCGGCAA
TACT-3′;下游引物GLRaV-3-R:5′-GAACCGACGTATCAATATCTAACGGCTGCCGACTA-3′。引物由上海生工生物工程有限公司合成。
1.2.3RPA检测
1.2.1中制备的cDNA为模板,采用GLRaV-3-F和GLRaV-3-R引物进行RPA扩增,同时设置阴性对照(模板为脱毒葡萄苗叶片cDNA)。RPA扩增体系的配制方法如下:向含有冻干酶粉的0.2 mL TwistAmp 反应管(TwistAmp Basic kits,Twist)中加入再水化缓冲液(Rehydration Buffer)29.5 μL,去离子水12.5 μL,上、下游引物各2 μL(终浓度为0.4 μmol/L),模板cDNA 1 μL,最后再加入醋酸镁溶液2.5 μL(280 mmol/L)。将RPA扩增体系充分混匀,置于37℃的金属浴上反应40 min。RPA反应结束后,向RPA扩增产物中加入50 μL苯酚/氯仿溶液,充分混匀后12 000 r/min离心2 min,取上清液5 μL于1.5 %琼脂糖凝胶电泳,在凝胶成像系统上观察结果。
1.2.4RPA检测方法特异性评价
按照1.2.4 RPA检测反应体系,对供试样品GLRaV-3以及GLRaV-1、GLRaV-2、GLRaV-4、GLRaV-5、GVA共6种病毒的cDNA进行检测,同时用脱毒葡萄苗的cDNA做阴性对照,对所建立的RPA检测方法特异性进行评价。
1.2.5RPA检测方法灵敏度评价
将GLRaV-3带毒株的总RNA制备的cDNA进行10倍梯度稀释,共6个稀释度,以梯度稀释的cDNA为模板,按照1.2.4所示的RPA检测反应体系进行RPA灵敏度实验,并参照已报道的GLRaV-3检测引物P3U/P3D[22]进行PCR灵敏度实验,扩增产物长度为653 bp,比较两种方法检测灵敏度。
2结果与分析
2.1 GLRaV-3的RPA检测方法的建立
以含GLRaV-3的葡萄叶片的cDNA为模板,并以脱毒葡萄苗的cDNA为阴性对照,建立了RPA检测方法检测GLRaV-3。GLRaV-3扩增条带与预期目的条带大小一致约380 bp,阴性对照脱毒葡萄苗没有条带度,建立的RPA检测体系能够准确检测到GLRaV-3。图1
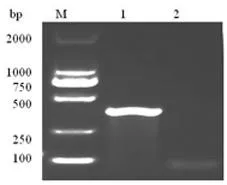
M:Marker DL 2000;1:GLRaV-3;2:阴性对照
M:Marker DL 2000;1:GLRaV-3;2:Negative control
图1 RPA技术检测GLRaV-3
Fig. 1 Detection GLRaV-3 by RPA
2.2 RPA检测方法的特异性评价
GLRaV-3的RPA特异性评价结果显示,GLRaV-3样品能够检测到380 bp的特异性条带,其它5种样品GLRaV-1、GLRaV-2、GLRaV-4、GLRaV-5及GVA均未检测到条带,证明该方法具有较强的特异性。图2
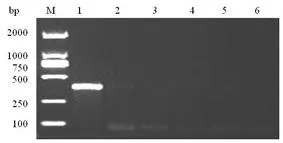
M:Marker DL 2000;1:GLRaV-3;2:GLRaV-1;3:GLRaV-2;4:GLRaV-4;5:GLRaV-5;6:葡萄A病毒
M:Marker DL 2000;1:GLRaV-3;2:GLRaV-1;3:GLRaV-2;4:GLRaV-4;5:GLRaV-5;6:GVA
图2 GLRaV-3的RPA特异性检测结果
Fig. 2 The specificity results of GLRaV-3 by RPA
2.3 RPA检测方法与普通PCR方法的灵敏度对比
GLRaV-3的RPA检测方法与普通PCR方法灵敏度比较检测结果显示,普通PCR反应设置在30个循环时,当稀释度为10-3时,仍可以检测到约653 bp的目的条带;RPA检测方法反应时间设置在40 min,当稀释度为10-3时,也可以检测约380 bp的目的条带,当稀释度为10-4时,两种方法均未能扩增到目的条带。说明两种方法灵敏度相当。图3,图4
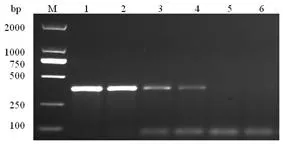
M:Marker DL 2000;1-6:cDNA稀释度依次为100、10-1、10-2、10-3、10-4、10-5
M:Marker DL 2000;1-6:The dilution of cDNA were 100、10-1、10-2、10-3、10-4、10-5
图3 GLRaV-3的RPA检测灵敏度
Fig. 3The detection sensitivity of GLRaV-3 by RPA
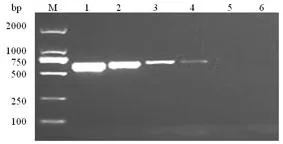
M:Marker DL 2000;1-6:cDNA稀释倍数依次为100、10-1、10-2、10-3、10-4、10-5
M:Marker DL 2000;1-6:The dilution of cDNA were 100、10-1、10-2、10-3、10-4、10-5
图4 GLRaV-3的PCR检测灵敏度
Fig. 4 The detection sensitivity of GLRaV-3 by PCR
3 讨 论
RPA技术是近几年兴起的恒温扩增技术,目前主要应用于医学病原菌、转基因的检测中[23-27]。研究以葡萄卷叶伴随病毒模式种GLRaV-3为研究对象,建立了GLRaV-3的RPA检测方法,并对其特异性和灵敏度进行了评价,为植物病毒的检测提供一种新的简便、快捷的检测方法。
以RPA技术建立的恒温扩增体系不需要 PCR 仪等热循环设备,37℃下就能对样品进行检测,而LAMP等温扩增技术需要在60~65℃下进行反应,因此RPA技术反应温度更容易达到。基于以上优点,Crannell等[28]建立一种不依赖任何仪器设备,仅仅利用人体体温来完成扩增反应的RPA检测方法。这大大增强了该技术的适用范围,尤其适合于现场检测。
LAMP等温扩增技术需要针对靶基因上6个区段设计4条引物才能完成扩增,引物设计复杂,需要进行大量的优化。而RPA 技术仅需一对引物就可完成整个扩增,引物设计相对简单。另外,LAMP的扩增条带为弥散带,而RPA技术的扩增条带根据引物设计位点,为特定大小的单一条带,RPA的扩增产物还可以进一步分析。因此,可以类似于普通多重PCR原理根据不同产物片段大小来判断结果。基于以上优点,Kersting等建立了一套含扩增内标的四重RPA技术用于检测三种病原菌[29]。
已报道的LAMP检测技术相对灵敏度在0.1%~0.01%(质量分数),与已报道的RPA检测技术灵敏度相当[30-31]。研究对RPA检测技术与RT-PCR检测技术的灵敏度进行了比较,发现两种技术灵敏度相当,这与已报道的文献结果一致。而利用添加了探针的RPA实时荧光检测技术,能够进一步提高检测的准确性和灵敏度[25-27]。当然RPA作为一种新兴起的检测技术,还有许多不足等待完善。首先,RPA体系对于蛋白活性的要求高于Taq酶,体系中任意一种酶失活都将导致 RPA 体系停止工作;其次,RPA扩增体系中存在大量蛋白酶,使得 RPA 扩增产物必须除去蛋白后才能电泳或进行后续试验。再次,RAA 体系与 PCR 同样采用正反引物与模版杂交,容易出现非特异性扩增。目前, RAA 法还处于初期阶段, 但是对于一个刚出现的新技术而言, 能够在体外较低的温度下最适温度(37℃)快速扩增出目的片段,已经是很好的开始。研究所做工作为 RPA 技术运用于植物病毒检测提供了积累对 RPA 技术的进一步发展具有积极意义。相信随着反应体系和反应条件的不断优化,RPA 法能够利用它的优势,在医药、农业等领域,特别是在检测(医学检测、动植物检疫和野外微生物检测)等方面广泛发挥作用。
4 结 论
以葡萄卷叶伴随病毒属模式种GLRaV-3为研究对象,建立了其RPA检测方法。该方法只需一对引物即可在37℃恒温条件下完成检测,并能准确区分其它几种参试葡萄病毒株,其扩增条带为一条380 bp的特异条带,扩增产物纯化后即可用于下游测序、克隆、酶切等分子生物学实验。研究所建立的RPA检测方法在40 min反应条件下的灵敏度与普通PCR在30个扩增循环下的检测灵敏度相当,反应时间缩短,不需要特殊的热循环仪,适合基层单位和现场检测使用,为葡萄病毒病害的田间诊断、预测预报和葡萄脱毒苗的生产等提供了更为简便高效的技术方法,在植物病毒快速检测方面具有一定的实践意义。
参考文献(References)
[1] Dovas, C. I., & Katis, N. I. (2003). A spot multiplex nested rt-pcr for the simultaneous and generic detection of viruses involved in the aetiology of grapevine leafroll and rugose wood of grapevine.JournalofVirologicalMethods, 109(2):217-226.
[2] 王升吉, 尚佑芬, 赵玖华, 等. 葡萄卷叶病毒主要检测技术比较[J]. 山东农业科学, 2008, 35(3): 95-98.
WANG Sheng-ji, SHANG You-fen, ZHAO Jiu-hua, et al. (2008). Comparison of main detecting technologies of grapevine leafroll associated virus [J].ShandongAgriculturalSciences, 35(3): 95-98. (in Chinese)
[3] 侯义龙. 葡萄卷叶病毒RT-PCR检测技术[J]. 植物保护学报, 2003, 30(3): 305-308.
HOU Yi-long. (2003). Detection of GLRaV by RT-PCR [J].Actaphytophylacicasinica, 30(3): 305-308. (in Chinese)
[4] Maliogka, V. I., Dovas, C. I., & Katis, N. I. (2008). Evolutionary relationships of virus species belonging to a distinct lineage within the ampelovirus genus.VirusResearch, 135(1):125-135.
[5] Ghanem-Sabanadzovic, N. A., Sabanadzovic, S., Uyemoto, J. K., Golino, D., & Rowhani, A. (2010). A putative new ampelovirus associated with grapevine leafroll disease.ArchivesofVirology, 155(11):1,871-1,876.
[6] 裴光前, 董雅凤, 张尊平, 等. 4种葡萄卷叶伴随病毒多重RT-PCR检测[J]. 植物病理学报, 2010, 40(1): 21-26.
PEI Quang-qian, DONG Ya-feng, ZHANG Zun-ping, et al. (2010). Detection of four grapevine leafroll-associated viruses by multiplex RT-PCR [J].ActaPhytopathologicaSinica, 40(1): 21-26. (in Chinese)
[7] 范旭东, 董雅凤, 张尊平,等. 葡萄卷叶伴随病毒4和5简并引物和多重PCR检测[J]. 植物保护, 2012, 38(6): 95-97.
FAN Xu-dong, DONG Ya-feng, ZHANG Zun-ping, et al. (2012). Detection of GLRaV-4 and 5 using degenerate primer and mutiplex PCR methods [J].PlantProtection, 38(6): 95-97. (in Chinese)
[8] 刘三军, 郭卫东, 张秋叶, 等. 葡萄卷叶病毒3 RT-PCR检测技术研究[J]. 果树学报, 2002, 19(1): 15-18.
LIU San-jun, GUO Wei-dong, ZHANG Qiu-ye, et al. (2002). Deteetion of grapevine leafroll associated virus 3 by Reverse Transcription-Polymerase Chain Reaction [J].JournalofFruitScience, 19(1): 15-18. (in Chinese)
[9] Kai-Shu, L., Hai-Ying, Z., & Dennis, G. (2004). Complete nucleotide sequence and genome organization of grapevine leafroll-associated virus 3, type member of the genus ampelovirus..JournalofGeneralVirology,85(Pt 7):2,099-2,102.
[10] Habili, N. (1995). Natural spread and molecular analysis of grapevine leafroll-associated virus 3 in australia.Phytopathology, 85(11):1,418-1,422.
[11] Cabaleiro, C., & Segura, A. (2006). Temporal analysis of grapevine leafroll associated virus 3 epidemics.EuropeanJournalofPlantPathology, 114(4):441-446.
[12] C. L. PETERSEN , & Charles, J. G. (1997). Transmission of grapevine leafroll-associated closteroviruses by pseudococcus longispinus and p. calceolariae.PlantPathology, 46(4):509-515.
[13] 陈建军, 刘崇怀, 古勤生, 等. DAS-ELISA、RT-PCR和IC-RT-PCR检测葡萄卷叶病毒3的比较研究[J]. 果树学报, 2003, 20(3): 173-177.
CHEN Jian-jun, LIU Chong-huai, GU Qin-sheng, et al. (2003). Comparative Studies on DAS-ELISA, RT-PCR and IC-RT-PCR for Detection Grapevine leaforll-associated virus 3 [J].JournalofFruitScience, 20(3): 173-177. (in Chinese)
[14] Thompson, J. R., Fuchs, M., Fischer, K. F., & Perry, K. L. (2012). Macroarray detection of grapevine leafroll-associated viruses.JournalofVirologicalMethods, 183(2):161-169.
[15] Engel, E., Girardi, C. P., Arredondo, V., Dominguez, C., Perez-Acle, T., & Valenzuela, P. (2008). Genome analysis and detection of a chilean isolate of grapevine leafroll associated virus-3.VirusGenes, 37(1):110-118.
[16] Osman, F., & Rowhani, A. (2006). Application of a spotting sample preparation technique for the detection of pathogens in woody plants by rt-pcr and real-time pcr (taqman).JournalofVirologicalMethods, 133(2):130-136.
[17] Fatima, O., Christian, L., Deborah, G., & Adib, R. (2008). Comparison of low-density arrays, rt-pcr and real-time taqman ; rt-pcr in detection of grapevine viruses.JournalofVirologicalMethods, 149(2):292-299.
[18] Saldarelli, P., Minafra, A., Martelli, G. P., & Walter, B. (1994). Detection of grapevine leafroll-associated closterovirus iii by molecular hybridization.PlantPathology, 43(1):91-96.
[19] 范旭东, 董雅凤, 张尊平, 等. 沙地葡萄茎痘相关病毒的 RT-LAMP 检测方法[J]. 植物病理学报, 2013, 43(3): 286-293.
FAN Xu-dong, DONG Ya-feng, ZHANG Zun-ping, et al. (2013). RT-LAMP assay for detection of Grapevine rupestris stem pitting-associated virus [J].ActaPhytopathologicaSinica, 43(3): 286-293. (in chinese)
[20] Lutz, S., Weber, P., Focke, M., Faltin, B., Hoffmann, J., & Müller, C., et al. (2010). Microfluidic lab-on-a-foil for nucleic acid analysis based on isothermal recombinase polymerase amplification (rpa).labchip.LabonAChip, 10(7):887-893.
[21] Daher, R. K., Stewart, G., Boissinot, M., Boudreau, D. K., & Bergeron, M. G. (2015). Influence of sequence mismatches on the specificity of recombinase polymerase amplification technology.Molecular&CellularProbes, 29(2):116-121.
[22] Turturo, C., Saldarelli, P., Yafeng, D., Digiaro, M., Minafra, A., & Savino, V., et al. (2005). Genetic variability and population structure of grapevine leafroll-associated virus 3 isolates.JournalofGeneralVirology, 86(Pt 1):217-224.
[23] Xu, C., Li, L., Jin, W., & Wan, Y. (2014). Recombinase polymerase amplification (rpa) of camv-35s promoter and nos terminator for rapid detection of genetically modified crops.InternationalJournalofMolecularSciences, 15(10):18,197-18,205.
[24] 邓婷婷, 黄文胜, 程奇, 等. 重组酶聚合酶扩增技术检测转基因水稻中的Cry1Ab/c 基因[J]. 中国食品学报, 2015, 15(3): 187-193.
DENG Ting-ting, HUANG Wen-sheng, CHENG Qi, et al. (2015) . Detection of Cry1Ab/c gene in genetically modified rice by recombinase polymerase amplification [J].JournalofChineseInstituteofFoodScienceandTechnology, 15(3): 187-193. (in Chinese)
[25] Euler, M., Wang, Y., Nentwich, O., Piepenburg, O., Hufert, F. T., & Weidmann, M. (2012). Recombinase polymerase amplification assay for rapid detection of rift valley fever virus.JournalofClinicalVirologytheOfficialPublicationofthePanAmericanSocietyforClinicalVirology,54(4):308-312.
[26] Milena, E., Yongjie, W., Peter, O., Herbert, T., Raquel, E., & Pedro, A., et al. (2012). Recombinase polymerase amplification assay for rapid detection of francisella tularensis.JournalofClinicalMicrobiology, 50(7):2,234-2,238
[27] Boyle, D. S., Lehman, D. A., Lorraine, L., Dylan, P., Mitra, S., & Niall, A., et al. (2013). Rapid detection of hiv-1 proviral dna for early infant diagnosis using recombinase polymerase amplification.Mbio, 4(2):49-52.
[28] Zachary Austin, C., Brittany, R., & Rebecca, R. K. (2014). Equipment-free incubation of recombinase polymerase amplification reactions using body heat.PlosOne, 9(11):112,146.
[29] Kersting, S., Rausch, V., Bier, F. F., & Nickisch-Rosenegk, M. V. (2014). Multiplex isothermal solid-phase recombinase polymerase amplification for the specific and fast dna-based detection of three bacterial pathogens.MicrochimicaActa, 181(13-14):1,715-1,723.
[30] Kiddle, G., Hardinge, P., Buttigieg, N., Gandelman, O., Pereira, C., & Mcelgunn, C. J., et al. (2012). Gmo detection using a bioluminescent real time reporter (bart) of loop mediated isothermal amplification (lamp) suitable for field use.BmcBiotechnology, 12(1):15.
[31] Rostamkhani, N., Haghnazari, A., Tohidfar, M., & Moradi, A. (2011). Rapid identification of transgenic cotton (gossypium hirsutum l.) plants by loop-mediated isothermal amplification.CzechJournalofGenetics&PlantBreeding, 47(4):140-148.
Fund project:Supported by Science and Technology Xinjiang Supporting Program (2013911090)
Based on Recombinase Polymerase Amplification , the Method of
Detection ofGrapevineleafroll-associatedvirus3
ZHANG Na1, QIAN Yi-ke2, WEI Shuang3, HU Bai-shi4, LU Ping2, Saitiieerhan2,
LIU Zhong-yong3, ZHANG Yong-jiang5, ZHANG Xiang-lin6
( 1.YiliNormalCollege,YiningXinjiang835000,China; 2.YiliEntry-ExitInspectionandQuarantineBureau,YiningXinjiang835000,China;3.ShantouEntry-ExitInspectionandQuarantineBureau,ShantouGuangdong515041,China; 4.NanjingAgriculturalUniversity,Nanjing290000,China; 5.InstituteofPlantQuarantine,ChineseAcademyofInspectionandQuarantine,Beijing100029,China; 6.XinjiangEntry-ExitInspectionandQuarantineBureau,Urumqi830052,China)
Abstract:【Objective】 The objective of this study is to develop a recombinase polymerase amplification (RPA) assay for the detection of Grapevine leafroll-associated virus 3 (GLRaV-3).【Method】Specific RPA primers were designed based on HSP 70 (heat shock protein 70) gene of GLRaV-3. A RPA assay was developed for the detection of GLRaV-3 after optimization reaction condition. The specificity and sensitivity of this assay were tested. 【Result】The RPA assay proved to be specific. Only a 380 bp fragment were detected in GLRaV-3 by RPA assay. The sensitivity of this RPA assay was consistent with conventional PCR. What was needed was just to keep the temperature at 37 ℃ about 40 min in RPA reaction without using other special equipments. 【Conclusion】This RPA assay proved to be rapid, sensitive and specific for the detection of GLRaV-3.
Key words:Grapevine leafroll-associated virus 3;RPA;method of detection
通讯作者:张祥林(1964-),男,新疆乌鲁木齐人,研究员,硕士生导师,研究方向为植物检疫,(E-mail)XL6479@163.com
作者简介:张娜(1983-),女,山西翼城人,实验师,研究方向为植物保护,(E-mail)yucixue@163.com
基金项目:新疆维吾尔自治区科技支疆项目(2013911090)
收稿日期:2015-07-21
中图分类号:S41;S182
文献标识码:A
文章编号:1001-4330(2016)02-0302-07
doi:10.6048/j.issn.1001-4330.2016.02.016
