红菇属分子生物学的研究进展*
2016-03-14王碧涵李宗菊袁晓芹马绍宾
王碧涵,李宗菊,左 奎,袁晓芹,马绍宾
(云南大学生命科学学院,云南 昆明 650091)
红菇属分子生物学的研究进展*
王碧涵,李宗菊,左 奎,袁晓芹,马绍宾**
(云南大学生命科学学院,云南 昆明 650091)
从种类鉴定、系统发育关系两个方面概述了近年来国内外对红菇属(Russula)的研究进展。其中,重点归纳了4种常见分子生物学技术(ITS、LSU、RPB2及RAPD序列分析)在红菇属研究中的应用,并对红菇属的分子系统学研究作了展望。
红菇属;新种;亲缘关系;DNA分子标记
红菇属(Russula) 隶属于担子菌门(Basidiomycotina)伞菌纲(Agaricomycetes)红菇目(Russulales)红菇科(Russulaceae)。该属子实体各个部位(特别是菌褶)很脆,菌柄短而粗,菌盖常有鲜艳的颜色,菌褶通常有囊状体,子实体通常肉质,菌肉由较典型菌丝缠绕着成群球状胞组成,菌丝无锁状联合,孢子具淀粉质纹饰[1]。迄今为止,全世界已有记载的种类大约有750种(包括变种)[2]。红菇属在世界范围内分布广泛,而在中国发现的新种约占已知红菇属种类的8%,表明了中国红菇属资源的独特性[3]。
红菇常发生于针叶林、阔叶林和混交林地上,和一些树种形成外生菌根,与植物群落有着密切的对应关系[4]。许多种类具有很高的食用价值和药用价值,其味道鲜美,营养丰富,部分红菇还具有改善糖代谢紊乱、调节体内脂质代谢[5]、抗癌[6]等作用。但红菇属亦有很多种类是有毒的,食用后可能引起肠胃炎型中毒症状[7]。传统的形态学鉴定主要通过观察新鲜样本的菌盖和菌柄的颜色、形状、大小,以及菌褶的颜色、着生状态,孢子印颜色、孢子纹饰、糊状反应等[8]。但传统形态学分类也有许多不足,例如由于地域分隔,红菇属种内的形态多样性偏高,亲缘种较难鉴定。因此近年来引入的分子生物学方法,如ITS、LSU、RPB2序列分析和RAPD等方法,可以为红菇属的鉴定提供依据和补充[9]。
1 红菇属分子生物学研究中的常用技术
1.1 ITS(internal transcribed space)
内转录间隔区,即3个高度保守的编码核糖体的基因(18S rDNA、5.8S r DNA、28S rDNA)之间的间隔区(图1)[10]。由于其进化相对迅速并且具有多态性,因而适合于等级水平较低的系统学研究[11],是目前真菌界亦是红菇属鉴定新种最常用的分子标记方法之一。然而ITS序列分析也存在很多不足之处,如片段较小,受到基因库完善程度、高度同源性序列,物种ITS区可变程度等一些因素的影响,并不能鉴定出所有真菌[12]。必要时需结合传统的形态学和其他分子标记方法,才能使结果更加准确。

图1 ITS结构Fig.1 The structure of ITS(internal transcribed space)
1.2 LSU(large subunit)
核糖体大亚基,即核糖体中较大的亚基,是28S rDNA基因上保守区和可变区中片段较长的基因[13]。由于其不同的结构域在进化上差别较大,而在真菌系统发育研究中一般选择某一变异较大的结构域来研究,因此可用于属及属以上的研究[14],而在对红菇属进行种类鉴定时,LSU序列分析方法可与ITS序列分析方法同时使用,使鉴定结果更加准确。
1.3 RAPD(randomly amplified polymorphic DNA)
随机扩增多态性DNA分析(randomly amplified polymorphic DNA),即运用随机引物扩增寻找多态性DNA片段,作为分子标记。RAPD是由Williams等[15]于1990年发展起来的一种技术。其基本原理是,利用人工合成的较短的随机排列碱基顺序的核酸单链(通常为10个核苷酸)为引物,在一种热稳定DNA多聚酶的作用下,对所研究的未知序列的基因组DNA进行PCR扩增,产生一组不连续的分子量为200 bp~2 000 bp的DNA片段。RAPD技术是近些年由PCR技术衍生的一种常用的技术,由于其无须事先了解目的基因序列、无须使用同位素、所需样品量较少、引物具有普遍适应性、比较适用于自动化操作和分析[16],而逐渐成为研究真菌亲缘关系以及遗传多样性的常用方法之一,尤其适用于种下水平的分类学研究,如鉴定子实体和分离菌株的亲缘关系,但在红菇属中的应用较少。
1.4 RPB2(the second largest RNA polymerase subunit)
RPB2,即编码RNA聚合酶Ⅱ第二大亚基的基因,与BTF3(general transcription factor) 结合可以形成复杂的转录活性[17],主要负责蛋白质编码基因的转录。由于其单拷贝和进化速率慢,较其他基因相对保守,因而可与ITS、LSU等分子标记方法结合,应用于红菇属的系统发育研究中。
2 红菇属的分子生物学研究
2.1 种类鉴定
2.1.1 鉴定未知红菇样本
ITS经常与形态鉴定结合被用以鉴定未知样本,常用于各属种大型野生真菌的鉴定,在红菇属中也被广泛的应用。田慧敏等[18]用形态学和ITS序列鉴定了内蒙古自治区赛罕乌拉国家级自然保护区的4个红菇样本,分别为铜绿红菇(Russula aeruginea)、细弱红菇(R.gracillima)、斑点红菇(R.maculata)和触黄红菇(R.luteotacta)。菌物学家发现新种后,也需要用分子标记的方法进行证实。Li等[19]在中国东北部长白山发现新种吉林红菇(R.jilinensis),并用ITS序列鉴定(图2),最大简约法构建的系统发育树可看出R.jilinensis与R.curtipes是近缘种,其形态学上也较为相似,但可用菌盖颜色及栖息地区分。Li等[20]在中国西南发现2个新种:R.atroaeruginea和R.sichuanensis,对这两个新种进行了形态和ITS序列分析的描述,并利用ITS系统发育树得以证实。Steven等[21]对发现于圭亚那的卡帕赖马高原的3个红菇新种R.myrmecobroma、R.paxilliformis和R. gelatinivelata进行了形态学和生态学描述及分类讨论,并用分子数据证实了分类群的亲缘关系。
2.1.2 鉴定组织分离株的真伪
红菇属有较多种类属于高经济价值的蘑菇,比如大红菇(Russula alutacea)、正红菇(R.vinosa),在我国东南沿海地区以及东南亚国家广受欢迎;青头菌(R.virescens)在云南等西南省份都有食用的传统。由于红菇科真菌全部为外生型共生菌,目前没有纯人工栽培的报道,国内仅见乳菇属的松乳菇(Lactarius deliciosus)、红汁乳菇(L.hatsudake) 半人工栽培的例子。因此分离筛选红菇纯培养菌株,保存其种质资源,对于红菇后续驯化开发具有重要意义。用ITS序列测定除了可以鉴定红菇新种外,亦可用于真菌组织分离株与子实体之间的对比。如颜军等[22]采用组织分离法从市售牛肝菌子实体分离得到两株性状稳定的分离株,用ITS序列分析将其分别鉴定为牛肝菌科(Boletaceae)成员及广义美味牛肝菌(Boletus edulis)。用ITS序列分析在红菇属中也有相似的案例。王桂文等[23]比对了组织分离株和红菇子实体的ITS序列,发现分离株的ITS序列全长小于子实体的ITS序列全长,并且组织分离株与红菇属真菌的遗传距离大,由此推测组织分离株可能不是红菇的分离株而是子实体的寄生菌或污染菌。
在鉴定组织分离株方面,随机扩增多态性DNA(RAPD)亦可提供可靠依据。孙文波等[24]用RAPD分析了红菇(Russulasp.) 子实体和分离菌株的DNA多样性,结果表明分离菌株和子实体之间的DNA相似性非常高。吴学谦等[25]对采自野外的正红菇(R.vinosa)子实体及其组织分离菌株进行RAPD和ITS分析,结果显示分离到的菌株并非来自其子实体,子实体内存在多种伴生菌。上述这些结果表明RAPD既可以单独使用,也可与其他分子标记方法,如与ITS结合,可高效准确地鉴定红菇属组织分离菌株的真伪,使食用纯培养红菇的安全性得到保障。
2.1.3 鉴定商品红菇
通过ITS序列比对,可以清晰分辨形态相似的毒菌和食用菌。红菇属中的亚稀褶黑菇和稀褶黑菇就是一个重要的例子(表1)。二者在形态上较为相似,肉眼难以分辨,因此导致的中毒事件在民间时有发生。有报道称,鉴定两者可以使用ITS序列分析方法,在ITS区域中,不同采集地的亚稀褶黑菇(Russula subnigricans) 与稀褶黑菇 (R.nigricans)的5.8S rDNA为同源性100%,而两侧ITS之间却表现出的种内和种间的多态性。其中种内差异均不超过5%,而种间差异高达10%左右。因此可以依据此数据来分辨剧毒的亚稀褶黑菇与可食用的稀褶黑菇[26]。陈新华[27]结合ITS及LSU序列分析,首次对广东梅州商品红菇进行形态学和分子学鉴定,由此进一步分析了该商品红菇营养成分及功能成分。
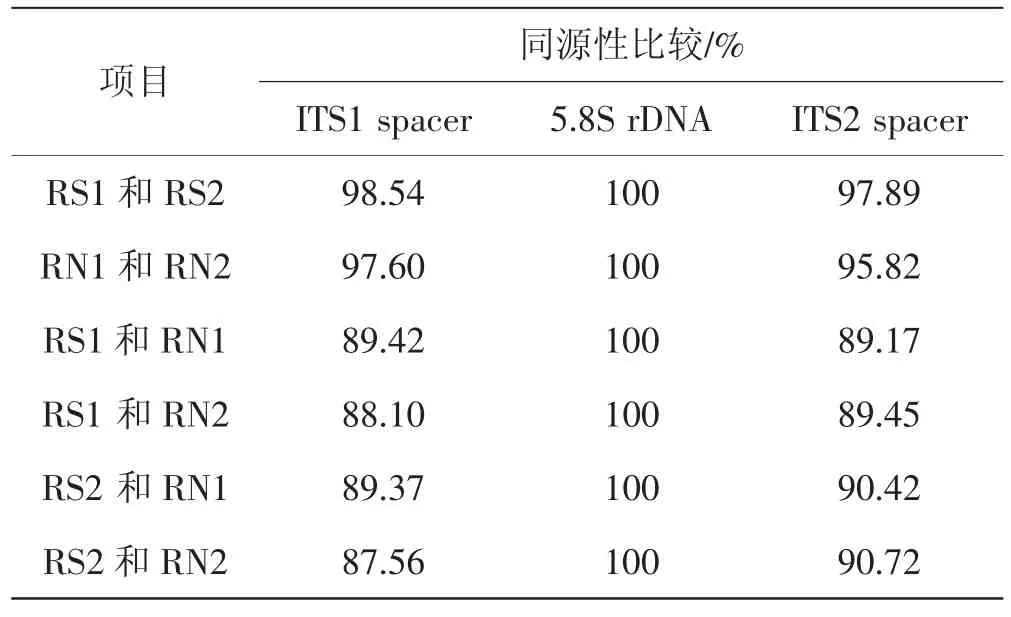
表1 亚稀褶黑菇和稀褶黑菇ITS分布长度比较Tab.1 Comparison of the ITS and rDNAs length variation between Russula subnigricans(RS)and R.nigricans(RN)
2.1.4 确定分类地位
在基因库完善程度较好的情况下,利用得到的ITS序列可以验证或重新确定物种的分类地位。Somanjana等[28]首次对点柄黄红菇(Russula senecis)完整的ITS序列进行测序,并通过系统发育分析证实了其分类地位在Ingratula亚属的Ingratae列下的Foetentinae亚组内。不仅如此,不同种类的红菇,由于生存环境相似,在长期的进化中可能出现一些相同或相似的形态特征,这就可能导致不同种类的红菇被划分成同一种类;然而有些属于同一种类的,由于环境不同,在长期进化过程中,其形态特征也可能发生变化;加之有些用于分类的形态特征可能是多系起源,具有同一形态特征的种类在系统发育树上却相距较远[29]。这3种情况都比较容易导致分类错误的产生。Razaq等[30]利用ITS序列分析鉴定了Russula foetentoides,将来自巴基斯坦的样本与来自喜马拉雅森林的样本鉴定为不同的种。Looney等[31]在分类学的框架下分析了25个由W.A.Murrill描述的北美红菇种的历史样本,结果显示,R.albimarginata和R.emeticiformis是一种美国东部常见的物种R.vinacea的同种异名;4个未知分类群(R. albimarginata、R.pinophila、R.subcremeiceps和R. subrubescens)已分化到亚组水平;对R.westii提出了另一种分类方式,对比了源自加拿大的样本和R. levyana,二者ITS1序列相似性高达99%,但形态差异却很大,说明单独使用ITS1序列可能不足以划分红菇属内物种。
2.2 系统发育分析
2.2.1 亲缘关系
ITS分子标记是构建系统发育树,研究种间亲缘关系远近的常用方法之一。曹政等[32]根据ITS序列分析结果得出,采自中国吉林省的标本与Russula firmula聚在同一分支上,而与其它近缘种存在明显的差异。在乳菇属中,也有利用ITS序列鉴定亲缘关系的例子。陈玉华等[33]基于ITS序列计算出江西的松乳菇 (Lactarius deliciosus)与意大利、西班牙、法国与西班牙的序列间遗传距离最大为0.012,而云南与法国的序列间遗传距离为0,是同源序列;系统发育树显示松乳菇和红汁乳菇 (Lactarius hatsudake)、橙色乳菇 (L.akahatsu)亲缘关系较近。不仅如此,在样本采自同一地区,同属于红菇科,但不同属的情况下,通过ITS序列分析,也能分析比较出少量样本的亲缘关系。付立忠等[34]对采自浙江丽水山区的4个红菇科样本进行ITS区段克隆测序和序列特征比较分析,并在核酸序列数据库GenBank中对其ITS序列进行了同源性检索比对,系统发育分析表明,红菇属的赭盖红菇 (R.mustelina)、绿菇 (R. virescens)和青灰红菇(R.parazurea)三者间序列差异较小,更具有亲缘关系。如果将样本的来源范围扩大,鉴定亲缘关系则同时需要ITS标记和LSU标记,才能获得较准确的结果。Myung等[35]利用ITS和LSU标记鉴定了红菇亚属Compacta的稀褶黑菇(R. nigricans) 和亚稀褶黑菇(R.subnigricans) 的系统发育关系,发现与日本的亚稀褶黑菇相比,韩国的亚稀褶黑菇与北美的R.eccentrica更具有亲缘关系,说明韩国的亚稀褶黑菇被误分类,应修正为R.eccentrica。但也有研究表明,在对特定的亚属内种进行亲缘关系分析时,LSU序列标记并不是最佳方法,而是可将RPB2与ITS结合,才能获得较为准确结果。Myung等[36]分别利用ITS、LSU、RPB2序列鉴定了韩国的Amoneula亚属的绒紫红菇(R.mariae)和微紫柄红菇(R.violeipes)两个种。这两个种用形态学方法和LSU序列分析法均难以区分,但用ITS和RPB2鉴定发现其互为单源。因此,作者认为在鉴定Amoneula亚属时,ITS和RPB2是最好的方法。国内也有利用ITS构建系统发育树分析亲缘关系,利用RPB2验证其结果正确性的研究报道。肖冬来等[37]构建的ITS序列系统发育树表明福建正红菇(R.vinosa)与云南大红菌(R.griseocarnosa)间序列差异较小,亲缘关系较近,与欧洲红菇(R.vinosa)、玫瑰红菇(R.rosea) 间序列差异较大,亲缘关系较远,在系统发育树上位于不同的分枝上,这与基于RPB2构建的系统发育树得到的结果一致。
2.2.2 进化关系
研究红菇属的进化关系时,可将采集的不同样品的ITS区段分别进行克隆测序,并将ITS序列在数据库中进行同源性比对,构建系统发育树。但也有研究表明,一定的地理因素并不会导致ITS序列发生改变。王桂文等[38]对采集自广西浦北县、容县和上思县的红菇子实体样本的ITS序列进行比对,并在GenBank/EMBL/DDBJ三大数据库进行同源性检索,得出结论:这三个县的食用红菇样本没有地理类群差异。这可能是由于相对于不同类型的北极森林和植物多样性较大的热带雨林,广西的3个不同县之间的环境差异较小,因此还没有形成地理类群差异。如果将范围扩大到中国云南与北美及欧洲,那么地理种群差异将明显增大。Cao等[39]用ITS序列分析了云南的红菇样本,结果表明云南的变绿红菇(R. virescens)与北美及欧洲的红菇样本有3%~6%的序列差异,并形成明显的进化枝。
除此之外,ITS序列分析还可用于红菇的特殊性状的进化地位分析,但这一方面的研究尚处于起步阶段。Peter等[40]利用ITS-rDNA综合分析检测发现亲硝基在臭红菇亚属中是一个普遍的特性,并处于较早的进化地位。
2.3 遗传多样性
ITS序列在研究红菇的近缘属乳菇属的遗传多样性时亦有使用。李河等[41]利用ITS序列分析来自湖南省道县、张家界市、芷江县、麻阳县和贵州省玉屏县以及泰国6个橙黄乳菇(Lactarius akahatsu)种群38个菌株的遗传结构和遗传多样性,结果表明橙黄乳菇种群具有丰富的遗传多样性。薛振文等[42]对4株红汁乳菇(L.hatsudake)菌株用HKY85距离测量法分析其遗传距离,结果表明,相比采自丽水市缙云与丽水城郊百果园的菌株遗传距离为0,湖南的标本与上述标本存在一定的遗传距离,推测地理隔离可能会对红汁乳菇遗传产生一定的影响。郭亮等[43]比较了中国湖南省、贵州省、云南省的松乳菇(L.deliciosus)的rDNA ITS区碱基序列的差异,群体遗传分析结果表明松乳菇自然种群间有一定程度的基因漂流发生,还存在显著的遗传分化,但遗传分化程度与地理距离没有显著的线性关系。
3 问题与展望
红菇具有显著的药用价值和食用价值。目前,对红菇属的形态学研究、营养学研究、药理学研究已经逐渐成熟。而另一方面,对红菇属的鉴定、分类和遗传学方面的研究却仍然处于初始阶段,具有很大的发展空间。传统的形态学鉴定需要采集新鲜的样本,而且容易受到环境及其观察者的主观性因素影响,加之红菇属有些菌株不易从形态特征上识别。DNA作为遗传物质,理论上比形态特征能更加清晰准确的反应菌株的遗传信息及进化地位。因此在形态学鉴定的基础上,可以结合ITS和LSU对红菇属的新种进行鉴定,用ITS、LSU、RAPD和RPB2对其进行亲缘关系及进化关系的分析,用ITS、RPB2进行遗传多样性分析,使结果更加准确。
除了上述4种分子标记方法外,还有一些鉴定其它大型真菌常用的分子标记方法,例如张微思等[44]利用ISSR引物,对来自云南香格里拉、大理、楚雄和昆明的4个松口蘑(Tricholoma matsutake)居群的遗传多样性进行了研究,亦有一些在其它大型真菌中被统计过的序列位点。再如王莹等利用生物信息学方法对美味牛肝菌(Boletus edulis)全基因组中的SSR位点进行了统计分析,并与其它3种担子菌基因组灰盖鬼伞(Coprinus cinereus)、裂褶菌(Schizphylhls commne)、糙皮侧耳(Pleurotus ostreatus)进行了比较[45]。这些分子标记方法常见于研究得比较成熟的大型真菌中,而在红菇属的分子生物学研究中却比较少,有待应用。
近年来,随着分子标记方法的不断成熟和改进,基因库的不断完善,一些类似红菇属的形态多样的大型真菌的鉴定方法,从传统的形态学分类鉴定,将逐渐转向基于DNA分子标记方法的分类鉴定,但亦有少数学者对此提出质疑和反对。主要原因有以下两个方面:一是形态学上的特征可以肉眼分辨或是通过显微镜观察得到,而分子数据则较为抽象,不同分子标记方法得出的结论有时不尽相同;二是形态学分类发展的历史较长,而分子生物学技术是近百年来的一种新兴技术,有些方面还不成熟。但无论如何,DNA辅之以形态学特征,这是分类学和系统学发展的方向。
[1]Kirk PM,Cannon PF,Minter DW,et al.Dictionary of the fungi[M].UK:CABI,2008:1-784.
[2]宋斌,李泰辉,吴兴亮,等.中国红菇属种类及其分布[J].菌物研究,2007,5(1):20-42.
[3]周新平,芦琴,王小平,等.红菇研究进展[J].食用菌,2010,32(3):1-2.
[4]陈旭健,甘耀坤,吴慧慧,等.红菇子实体对小鼠血糖、血脂的影响[J].食品科技,2008,33(4):237-239.
[5]张云波,夏爱军,梁园,等.红菇的药理作用研究进展[J].中国药业,2013,22(2):95-96.
[6]徐绍业,金美华,赵灵颖,等.广东梅县一野生红菇化学成分及急性毒性研究[J].食品研究与开发,2014(15):24-27.
[7]刘惠娜,陈新华.广东梅州灰肉红菇子实体形态结构特征[J].广东农业科学,2011(7):140-144.
[8]李国杰,文华安.中国红菇属分类研究进展[J].菌物学报,2009,28(2):303-309.
[9]陈剑山,郑服丛.ITS序列分析在真菌分类鉴定中的应用[J].安徽农业科学,2007,35(13):3785-3792.
[10]燕勇,李卫平,高雯洁,等.rDNA-ITS序列分析在真菌鉴定中的应用[J].中国卫生检验杂志,2008,18(10):1958-1961.
[11]燕勇,李卫平,高雯洁,等.rDNA-ITS序列分析在真菌鉴定中的应用[J].中国卫生检验杂志,2008,18(10):1958-1961.
[12]田慧敏,刘铁志.DNA分子标记技术在红菇分子鉴定中的应用进展[J].江苏农业科学,2013,41(4):28-31.
[13]蒋丽娟,周立平,嘉晓勤.DNA分子标记技术在红曲菌分类中的研究进展[J].酿酒科技,2009(4):96-100.
[14]赵瑞琳.RAPD分子标记在真菌研究中的应用[J].热带农业科技,2004,27(3):23-26.
[15]张建博,桂明英,刘蓓,等.分子生物学在大型真菌遗传多样性研究中的应用[J].中国食用菌,2008,27(6):3-7.
[16]Zheng XM,Vincent M,Jean ME,et al.A general transcription factor forms a stable complex with RNA polymerase B (II) [J].Cell,50(3):361-368.
[17]刘文丛,刘颖,郭相,等.滇西北地区羊肚茵的分子鉴定及ITS序列分析[J].江苏农业科学,2012,40(3):31-34.
[18]田慧敏,刘铁志,连静,等.基于形态特征及ITS序列对内蒙古红菇的分类鉴定[J].食用菌学报,2014(4):15-19.
[19]Li GJ,Li SF,Liu XZ,et al.Russula jilinensis sp.nov.(Russulaceae)from northeast China[J].Mycotaxon,2012,120 (10):49-58.
[20]Li GJ,Zhao Q,Zhao D,Yue SF,et al.Russula atroaeruginea and R.sichuanensis spp.nov.from southwest China[J].Mycotaxon,2013,124(2):173-188.
[21]Steven LM,MCatherine A,Terry WH.Russulaceae of the Pakaraima Mountains of Guyana 2.New species of Russula and Lactifluus[J].Mycotaxon,2012,121(3):233-253.
[22]颜军,李涛,马绍宾,等.两个牛肝菌分离株的鉴定与培养[J].食用菌学报,2010,17(2):26-31.
[23]王桂文,孙文波.广西红菇子实体及分离株的rDNA ITS序列分析[J].广西科学,2004,11(3):261-265.
[24]孙文波,李海鹰,王桂文,等.应用RAPD鉴定红菇组织分离菌株的探索试验[J].广西科学,2000,7(3):222-224.
[25]吴学谦,李海波,付立忠,等.几种森林大型真菌纯培养菌种的RAPD及ITS分子标记鉴定[J].林业科学,2007,43(12):94-100.
[26]尹军,华张平,龚庆芳,等.亚稀褶黑菇和稀褶黑菇的ITS序列分析[J].菌物学报,2008(2):237-242.
[27]陈新华.广东商品红菇形态和分子鉴定、营养成分分析及其生物活性研究[D].长沙:中南大学,2010.
[28]Somanjana K,Arun KD,Krishnendu A.Prospecting Russula senecis:a delicacy among the tribes of West Bengal[J]. PeerJ,2015,3(4):810.
[29]李雪玲.贝盖侧耳的系统发育地位—基于nrDNA-LSU和ITS序列分析的研究[J].北京林业大学学报,2005,27(3):67-71.
[30]Razaq A,Ilyas S,Khalid AN,et al.Russula foetentoides (Russulales,Russulaceae)-a new species from Pakistan[J]. Mycology,2014,66 (2):289-298.
[31]Looney BP.Molecular annotation of type specimens of Russula species described by WA Murrill from the southeast U-nited States[J].Mycotaxon,2014,129 (2):255-268.
[32]曹政,张鑫,伍建榕,等.红菇属一中国新记录种—榄色红菇[J].山西农业大学学报:自然科学版,2014,34(6):564-567.
[33]陈玉华,刘君昂,周国英,等.松乳菇ITS序列比较及其在乳菇属中的系统发育分析[J].食用菌学报,2013,20(1):18-24.
[34]付立忠,李海波,魏海龙,等.四个红菇科菌株的rDNA ITS序列分析和系统发育研究[J].食用菌学报,2007,14(2):23-28.
[35]Myung SP,Hyun L,Seung YO,et al.Species delimitation of three species within the Russula subgenus compacta in Korea:R.eccentrica,R.nigricans,R.subnigricans[J].Journal of Microbiology,2014,52(8):631-638.
[36]Myung SP,Jonathan JF,Hyun L,et al.Delimitation of Russula subgenus Amoenula in Korea using three molecular markers[J].Mycobiology,2013,41(4):191-201.
[37]肖冬来,陈宇航,杨菁,等.福建正红菇遗传多样性分析[J].福建农业学报,2013,28(9):902-905.
[38]王桂文,孙文波.广西红菇子实体及分离株的rDNA ITS序列分析[J].广西科学,2004,11(3):261-265.
[39]Cao Y,Zhang Y,Yu Z F,et al.Structure,gene flow and recombination among geographic populations of a Russula virescens ally from southwestern China[J].Plos One,2013,8 (9):e73174.
[40]Peter GA,Ectomycorrhizal iconoclasts:the ITS rDNA diversity and nitrophilic tendencies of fetid Russula[J].Mycologia, 2012,104 (5):998-1007.
[41]李河,徐建平,郭亮,等.橙黄乳菇遗传多样性分析[J].食用菌学报,2012,19(3):29-36.
[42]薛振文,梁俊峰,应国华,等.红汁乳菇及近缘种的分子鉴定及遗传距离分析[J].中国食用菌,2013,32(6):36-37.
[43]郭亮,刘君昂,周国英,等.中国南方地区松乳菇遗传多样性分析[J].菌物学报,30(3):384-391.
[44]张微思,王浚,李宗菊,等.云南松口蘑的ISSR遗传多样性研究[J].云南农业大学学报,2011,26(5):593-597.
[45]王莹,陈明杰,汪虹,等.美味牛肝菌全基因组SSR位点的分布规律研究[J].菌物学报,2015,(2):204-214.
Research Advances on the Genus Russula using Molecular techniques
WANG Bi-han,LI Zong-ju,ZUO Kui,YUAN Xiao-qin,MA Shao-bin
(College of Life Science,Yunnan University,Kunming 650091,China)
The research progress on the genus Russula at home and abroad were summarized in recent years from two aspects: species confirmation and phylogenetic relationships.Among them,four common molecular biology techniques,such as ITS,LSU, RPB2 and RAPD sequence analysis,used in the study of Russula were emphatically discussed,and the study prospects of molecular phylogenetic on Russula were also given.
Russula;new species;genetic relationship;DNA molecular markers
S646.9
A
1003-8310(2016)02-0001-06
10.13629/j.cnki.53-1054.2016.02.001
*项目来源:国家“十二五”科技支撑计划课题(2013BAD16B01)。
王碧涵(1991-),女,在读硕士研究生,主要从事中国云南产红菇属的分子系统学研究。E-mail:bihanwang@foxmail.com
**通信作者:马绍宾(1965-),男,博士,教授,主要从事大型真菌的分类及系统发育研究。E-mail:msbkm@126.com
2016-01-06
