一株分离自“生命营养液”细菌菌株的分类鉴定
2016-03-06张欣亚森克力木姚粟刘洋翟磊程池
张欣,亚森·克力木,姚粟,刘洋,翟磊,程池,*
(1.中国食品发酵工业研究院中国工业微生物菌种保藏管理中心,北京 100015; 2.新疆埃杜升生物开发有限公司,新疆乌鲁木齐 830000)
一株分离自“生命营养液”细菌菌株的分类鉴定
张欣1,亚森·克力木2,姚粟1,刘洋1,翟磊1,程池1,*
(1.中国食品发酵工业研究院中国工业微生物菌种保藏管理中心,北京 100015; 2.新疆埃杜升生物开发有限公司,新疆乌鲁木齐 830000)
从新疆地区特色饮料“生命营养液”中分离到一株优势菌株CICC 10820。旨在明确菌株CICC 10820的分类地位,为研究饮料“生命营养液”的营养功效及该菌的生物学功能和应用奠定基础。依次采用16S rRNA和磷酸丙糖异构酶(tpi)基因序列分析的分子分类鉴定工具对分离菌株进行分类鉴定,鉴定结果,菌株CICC 10820为第三梭菌(Clostridium tertium)。
生命营养液;第三梭菌;CICC 10820;鉴定
“生命营养液”是我国新疆地区的一种特色自然发酵功能性饮料,以新疆特产干果(巴旦木、核桃、杏仁等)、豆类(黄豆、绿豆、黑豆等)、谷类(小麦、大米、黑米等)及多种干鲜果类和蔬菜类等多种天然食材为原料,按照特定的配比,经水蒸煮、过滤,滤液再在特定的温度下进行无氧发酵、有氧发酵等复杂自然发酵过程,整个发酵过程历时4个月[1]。“生命营养液”富含蛋白质、维生素、矿物质等营养成分和功能因子,提高人体免疫力,对心血管疾病、糖尿病、多种炎症的控制和辅助治疗具有积极的效果[2]。赵婷等[3]发现其含有天然的抑菌活性成分,对大肠埃希氏菌、沙门氏菌、铜绿假单胞菌、志贺氏菌等多种致病菌具有抑菌活性。陈建国等[4]发现“生命营养液”具有体外抗HIV-1活性,其作用机制可能抑制病毒的整合。
菌种是微生物发酵食品的重要组成部分。发酵菌种代谢产生各种更易人体吸收的营养元素和功能因子,传统发酵菌种又往往是益生菌,通过在人体中定植,发挥有益作用。目前还未见关于对这种特色发酵制品微生物方面的研究报道。本课题组对“生命营养液”的微生物组成和活性成分进行了解析,获得了有价值的微生物资源和功效组分数据。本研究选取自“生命营养液”中分离到的优势菌株CICC 10820为研究对象,利用16S rRNA和tpi基因基因条形码序列(bar-coding)对其进行鉴定,以明确其分类学地位,为下一步深入研究该发酵菌种的生物功能与“生命营养液”功效性成分间的关系,以及发酵菌剂的研制奠定基础。同时,明确的分类名称也是未来国内对食品微生物菌种的管理的基本要求。
1 材料与方法
1.1 材料与试剂
供试菌株分离自“生命营养液”样品(样品由新疆埃杜升生物开发有限公司提供),并保藏于中国工业微生物菌种保藏管理中心(CICC),保藏编号为CICC 10820。NA培养基购自北京陆桥技术有限责任公司。细菌基因组DNA提取试剂盒、Taq DNA聚合酶、DL 2000 Marker均购自天根生化科技有限公司。
1.2 实验仪器
5415 R型高速冷冻离心机,日本LUBOTA公司; Professional TRIO 48型基因扩增仪,美国ABI公司; LDZX-50KBS型立式压力蒸汽灭菌器,上海申安医疗器械厂;DK-12型电热恒温水浴锅,上海森信实验仪器有限公司。
1.3 实验方法
1.3.1 菌株分离
采用稀释平板涂布法进行菌株的分离。在无菌条件下,吸取“生命营养液”样液25 mL于装有玻璃珠的225 mL无菌水的三角瓶中,充分振荡混匀。用移液器取混合液1 mL加入到9 mL具塞玻璃试管中,充分振荡混匀,依次进行梯度稀释,制备成1× 10-3~10 mL/L的系列稀释液。分别取0.1 mL涂布到NA培养基平板,置于30℃下培养48 h。用接种环挑取优势菌落于NA培养基平板划线,置于30℃下培养,待长出形态统一的菌落后,挑取单菌落转接到新鲜的NA斜面培养后于4℃保藏,保藏编号CICC 10820。
1.3.2 形态学观察
将供试菌株接种于NA固体培养基,置于30℃培养48 h后,观察其菌落形态特征并利用光学显微镜进行菌体形态特征观察[5]。
1.3.3 16S rRNA基因序列分析
利用细菌基因组DNA提取试剂盒提取菌株CICC 10820基因组DNA。采用刘洋等[6]的方法以基因组DNA为模板,扩增16S rRNA基因序列并用ABI 3700基因测序仪测序,测序由北京诺赛基因组研究中心有限公司完成。测序结果在EzBioCloud数据库中进行比对[7],采用CLUSTAL W对CICC 10820及其若干近缘种16S rRNA基因序列进行多序列比对[8],并利用N-J法通过MEGA 5软件进行系统发育分析[9]。
1.3.4tpi基因序列分析
以菌株CICC 10820基因组DNA为模板,利用tpi基因序列的特异性引物及条件进行PCR扩增[10-11]。扩增条件为94℃5 min;94℃30 s;55℃30 s;72℃30 s;40个循环;72℃10 min;4℃保存。产物用ABI 3700基因测序仪测序,测序得到的结果在GenBank数据库中进行对比。
2 结果与分析
2.1 形态学观察结果
菌株CICC 10820在NA培养基上,37℃培养2 d,见图1。由图1a可见,该菌落较小,呈白色,圆形,不透明;由图1b可见,利用光学显微镜观察显示菌体为杆状,大小为0.6 μm×(1.6~5.0)μm,单个排列,革兰氏阳性。
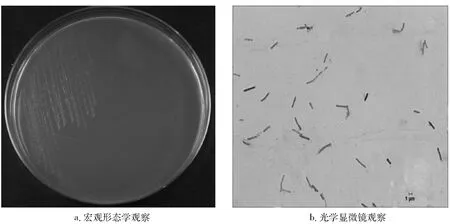
图1 菌株CICC 10820形态学观察结果Fig.1Morphological observation of strain CICC 10820
2.2 16S rRNA基因序列分析
菌株CICC 10820的基因组DNA提取结果见图2,所提取的DNA片段约为23.1 kb,利用引物27 f和1 492 r扩增得到16S rRNA基因全长序列,扩增产物经测序及分析处理后,将序列信息提交至Gen-Bank,登录号为KU324429。以Bacillus subtilisDSM 10T(AJ276351)为外群,构建CICC 10820与相关近缘模式菌株的系统发育树,见图3。由系统发育分析确定菌株CICC 10820归属为梭菌属(Clostridiumsp.)。
2.3 tpi基因序列分析结果
以菌株CICC 10820的基因组DNA为模版,利用特异性引物(tpiA/tpiB)扩增菌株tpi基因序列,产物经测序及分析处理后,将序列信息提交至Gen-Bank,登录号为KU324430。将序列在GenBank中进行Blast比对,与第三梭菌(Clostridium tertium)的基因序列的相似性为99%,与梭菌属中的其他近缘种的相似性均低于95%。图4为以ClostridiuminnocuumATCC 14501T(AY700148)为外群,构建CICC 10820与相关近缘模式菌株的系统发育树。由系统发育分析确定菌株CICC 10820被鉴定为第三梭菌(Clostridium tertium)。
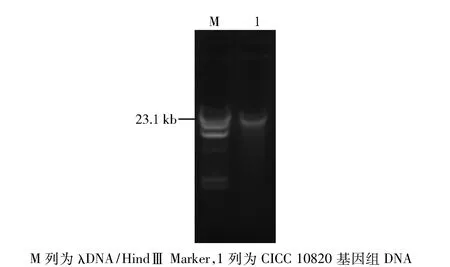
图2 CICC 10820基因组DNAFig.2CICC 10820 genomic DNA
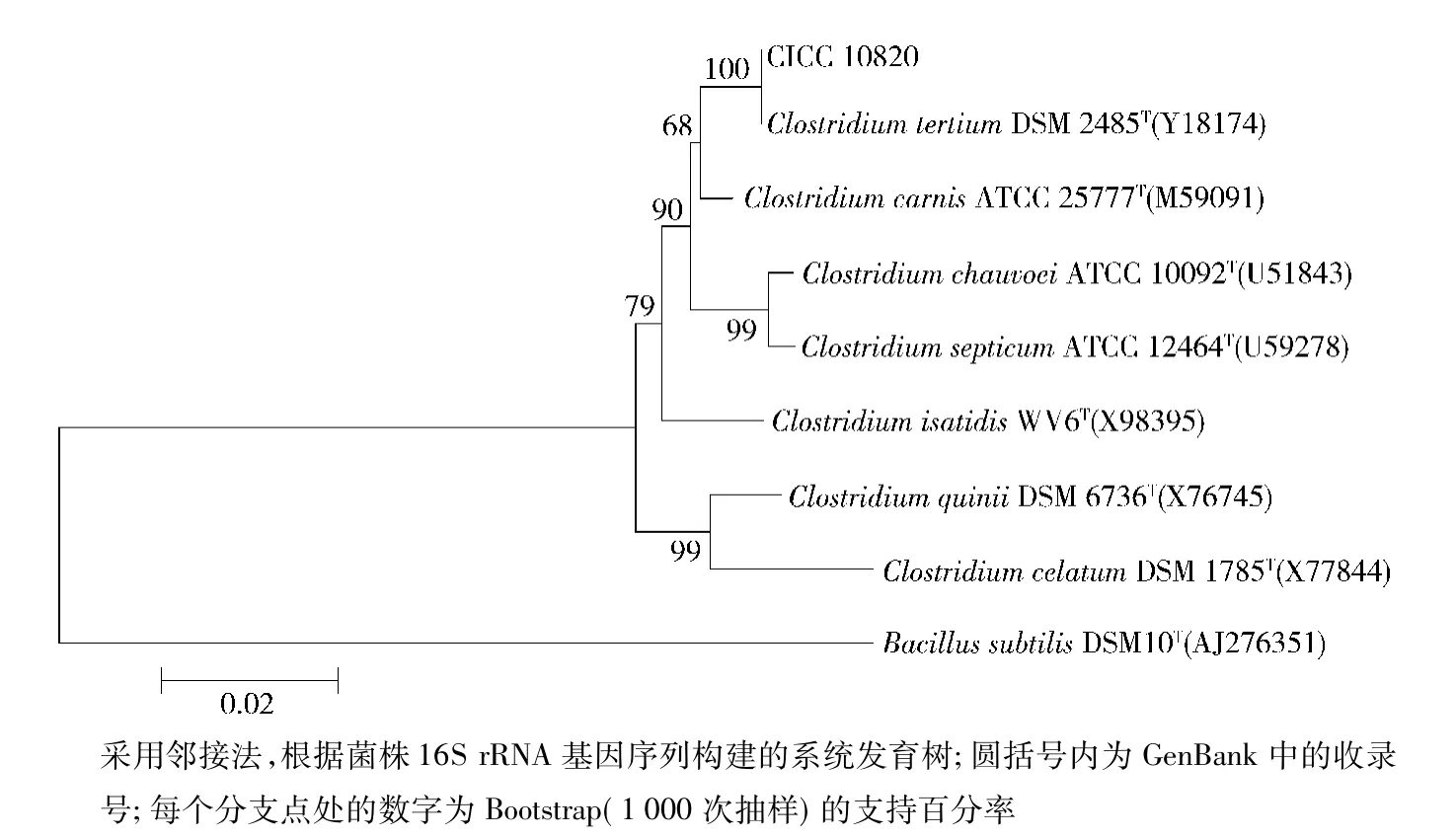
图3 CICC 10820 16S rRNA基因系统发育分析Fig.3Phylogenetic analysis of CICC 10820 16S rRNA gene
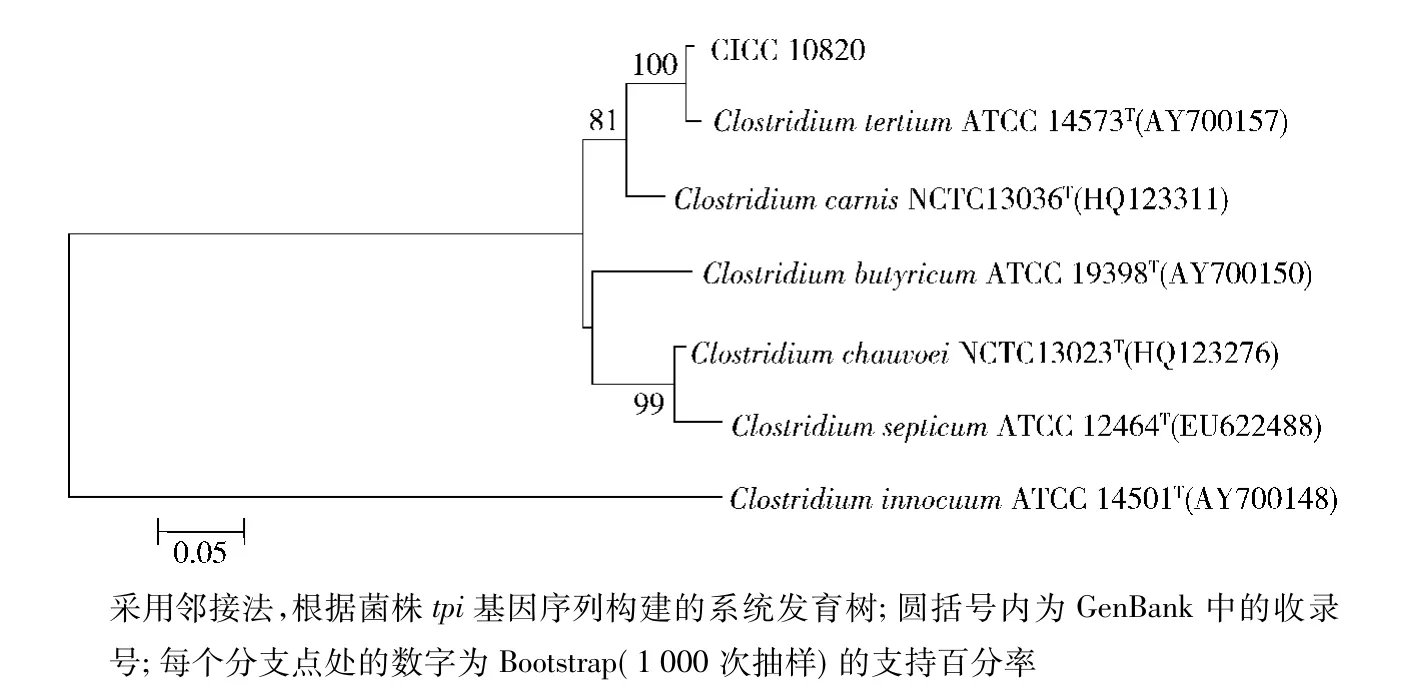
图4 CICC 10820 tpi基因序列系统发育分析Fig.4Phylogenetic analysis of CICC 10820 tpi gene
2.4 菌株CICC 10820分类鉴定结论
通过形态学观察,16S rRNA基因序列系统发育分析及tpi基因序列的比对分析,分离自“生命营养液”样品中的菌株CICC 10820被鉴定为第三梭菌(Clostridium tertium)。
3 结论
与表型鉴定的传统微生物鉴定方法相比,分子生物学方法更加准确、快捷,已广泛用于微生物的分类鉴定。16S rRNA基因已是公认的细菌分类鉴定的基本标尺,常用于微生物属、群水平的鉴定,与其他看家基因、功能基因等结合使用可将微生物鉴定至更小的分类单元。磷酸丙糖异构酶(tpi)基因是梭菌鉴定的有力工具,能有效区分梭菌属内不同菌种[10-11]。本文通过测定16S rRNA基因和tpi基因序列,与权威核酸序列数据库模式菌株进行相似性比对,并结合系统发育分析的分子生物学方法,将菌株CICC 10820准确鉴定为第三梭菌(Clostridium tertium)。
第三梭菌由Henry等[12]于1917年首次发现,最初命名为Bacillus tertius。随着研究的深入和分类学的发展,该菌的分类地位发生了很大变化,由最初的芽孢杆菌属(Bacillussp.)转移到梭菌属(Clostridiumsp.)[13]。1980年国际微生物委员会正式将其更名为第三梭菌(Clostridium tertium),隶属于硬壁菌门(Firmicutes),梭菌纲(Clostridia),梭菌目(Clostridiales),梭菌科(Clostridiaceae),梭菌属(Clostridium)[14]。目前,梭菌属(Clostridiumsp.)共209个种。代表种为酪酸梭菌(Clostridium butyricum),产丁酸,并能以芽胞形式到达肠道,并发挥调节肠道微生态平衡的作用,作为益生菌微生态产品,已商业化应用于保健品、药品、食品添加剂、饲料添加剂等领域[15-16]。目前,关于第三梭菌(Clostridium tertium)研究方面的报道较少。Kataoka等[17]从植物莲花根部土壤中分离到第三梭菌(Clostridium tertium),该菌能够利用纤维二糖、果糖、明胶、糖原、乳糖、麦芽糖、甘露醇、蜜二糖、棉子糖、核糖、淀粉、蔗糖、木糖、海藻糖等多种碳源和牛肉膏、蛋白胨、(NH4)2HPO4、(NH4)2CO3等多种有机、无机氮源,并产生醋酸盐、丁酸盐等有机酸[17]。第三梭菌(Clostridium tertium)能分泌大量的胞外酶,尤其是能分解半纤维素的甘露糖酶,在食品、饲料添加剂领域有广泛的应用价值[17-19]。“生命营养液”发酵原料中含有的豆类和干果,其占很大比例。第三梭菌(Clostridiumtertium)分泌的甘露糖酶对这些豆科植物种子、干果中富含的半乳甘露聚糖、葡甘露聚糖、阿拉伯半乳糖等半纤维素的分解可能产生积极作用。因此,猜测该菌种可能来源于这些干果、豆类植物的生长环境(如土壤、植物表面)或者属于植物内生菌,内生于干果或豆科类植物的种子。第三梭菌(Clostridium tertium)作为兼性厌氧菌,能够适应“生命营养液”的无氧、有氧二次发酵环境条件。另外,第三梭菌(Clostridium tertium)也有作为益生作用的报道。Borody[20-21]利用包括第三梭菌(Clostridium tertium)在内的梭菌、双歧杆菌、乳酸菌等组成益生菌制剂,用于治疗肠道菌群异常引起的慢性疾病,如肠易激综合征(irritable bowel syndrome,IBS)。本文从新疆特色发酵饮品“生命营养液”中分离到优势菌株,通过分子生物学方法将其鉴定为第三梭菌(Clostridium tertium),为下一步深入研究该菌的生物学功能、菌剂的开发以及“生命营养液”饮品的营养保健功效等方面奠定基础。
[1]亚森·克力木.一种保健营养液及其制备方法: 201110111492.8[P].2011-11-02.
[2]新疆埃杜升生物开发有限公司.埃杜升生命营养液[EB/OL].[2014-06-01].http:www.aldost.com/ cn/product_show.php?id=1.
[3]赵婷,吾布利·卡地尔,翟磊,等.新疆“生命营养液”抑菌能力评价及其菌种分离纯化[J].食品科学技术学报,2015,33(2):48-53.
ZHAO T,UBUL·Kadir,ZHAI L,et al.Evaluation of antimicrobial ability and isolation of strains from Xinjiang“life nutrient liquid”[J].Journal of Food Science and Technology,2015,33(2):48-53.
[4]陈建国,梁寒峭.新疆“生命营养液”体外抗HIV-1活性的分析[J].安徽农业科学,2015,43(19):8-9.
CHEN J G,LIANG H Q.Analysis of in vitro anti-HIV-1 activity of Xinjiang“life nutrient liquid”[J].Journal of Anhui Agricultural Sciences,2015,43(19):8-9.
[5]LIU Y,LIU L,QIU F B,et al.Paenibacillus hunanensis sp nov isolated from rice seeds[J].International Journal of Systematic and Evolutionary Microbiology,2010,60 (6):1266-1270.
[6]刘洋,曹艳花,姚粟,等.一株西沙群岛野生诺尼种子内生细菌CICC 10599的分离与鉴定[J].食品与发酵工业,2014,32(6):65-69.
LIU Y,CAO Y H,YAO S,et al.Identification on endo-phytic bacterial strain CICC 10599 isolated from Paracel islands wild noni(Morinda citrifolia L.)Seeds[J].Food and Fermentation Industries,2014,32(6):65-69.
[7]CHUN J,LEE J H,JUNG Y,et al.EzTaxon:a webbased tool for the identification of prokaryotes based on 16S ribosomal RNA gene sequences[J].Int J Syst Evol Micr,2007,57(10):2259-2261.
[8]THOMPSON J D,HIGGINS D G,GIBSON T J.Clustal w:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,positionspecific gap penalties and weight matrix choice[J].Nucleic Acids Res,1994,22(22):4673-4680.
[9]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011,28(10):2731-2739.
[10]DHALLUIN A,LEMEE L,PESTEL-CARON M,et al.Genotypic differentiation of twelve Clostridium species by polymorphism analysis of the triosephosphate isomerase (tpi)gene[J].Syst Appl Microbiol,2011,26(1):90 -96.
[11]GAROFOLO G,GALANTE D,SERRECCHIA L,et al.Development of a real time PCR taqman assay based on the TPI gene for simultaneous identification of Clostridium chauvoei and Clostridium septicum[J].J Microbiol Methods,2011,84(2):307-311.
[12]HENRY H.An investigation of the cultural reactions of certain anaerobes found in wounds[J].Journal of pathology and Bacteriology,1917,21(3),344-385.
[13]JOHNSON J L,FRANCIS B S.Taxonomy of the clostridia:ribosomal ribonucleic acid homologies among the species[J].Journal of General Microbiology,1975,88 (2):229-244.
[14]SKERMAN V B D,MCGOWAN V,SNEATH P H A.Approved lists of bacterial names[M].Washington: ASM Press,1989:225-240.
[15]张善亭,史燕,张淑丽,等.丁酸梭菌的研究应用进展[J].生物技术通报,2013(9):27-33.
ZHANG S T,SHI Y,ZHANG S L,et al.Progress on the studies and application of Clostridium butyricum[J].Biotechnology Bulletin,2013(9):27-33.
[16]KANAI T,MIKAMI Y,HAYASHI A.A breakthrough in probiotics:Clostridium butyricum regulates gut homeostasis and anti-inflammatory response in inflammatory bowel disease[J].Journal of Gastroenterology,2015,50(9):1-12.
[17]KATAOKA N,TOKIWA Y.Isolation and characterization of an active mannanase-producing anaerobic bacterium,Clostridium tertium KT-5A,from lotus soil[J].Journal of Applied Microbiology,1998,84(3):357-367.
[18]JUTURU V,JIN C W.Insight into microbial hemicellulases other than xylanases:a review[J].Journal of Chemical Technology and Biotechnology,2013,88(3): 353-363.
[19]SONI H,KANGO N.Microbial mannanases:properties and applications[M].India:Springer,2013.
[20]BORODYTJ.Probioticrecolonisationtherapy: US8460648[P].2013-06-11.
[21]BORODY T J.Compositions for probiotic recolonisation therapy:US9040036[P].2015-05-26.
Identification of Bacterial Strain Isolated from Life Nutrition Liquid
ZHANG Xin1,Yasin Kirim2,YAO Su1,LIU Yang1,ZHAI Lei1,CHENG Chi1,*
(1.China Center of Industrial Culture Collection,China National Research Institute of Food and Fermentation Industuies,Beijing 100015,China;2.Xinjiang ALDOS Biological Development Co Ltd,Urumqi 830000,China)
The bacterial strain CICC 10820 isolated from life nutrition liquid was identified via 16S rRNA and tpi gene sequences analysis.According to the result,strain CICC 10820 was identified as Clostridium tertium.This research will provide the foundation for the further study.
life nutrition liquid;Clostridium tertium;CICC 10820;identification
李宁)
TS275.4
A
10.3969/j.issn.2095-6002.2016.06. 006
2095-6002(2016)06-0031- 05
2016-01- 04
国家微生物资源平台专项(NIMR2015-4)。
张欣,男,工程师,硕士,主要从事微生物分类学研究;
*程池,男,教授级高工,主要从事微生物学研究。
。
张欣,亚森·克力木,姚粟,等.一株分离自“生命营养液”细菌菌株的分类鉴定[J].食品科学技术学报,2016,34 (6):31-35.
ZHANG Xin,Yasin Kirim,YAO Su,et al.Identification of bacterial strain isolated from life nutrition liquid[J].Journal of Food Science and Technology,2016,34(6):31-35.
