五株猪流行性腹泻病毒的分离鉴定及序列分析
2016-02-07王贵华于萍萍满坤吴珊珊刘明明孟萌赵亚荣
王贵华,于萍萍,满坤,吴珊珊,刘明明,孟萌,赵亚荣*
(1. 北京大北农科技集团股份有限公司动物医学研究中心,北京100195;2. 北京市畜禽生物制品工程技术研究中心,北京100195)
五株猪流行性腹泻病毒的分离鉴定及序列分析
王贵华1,2,于萍萍1,2,满坤1,2,吴珊珊1,2,刘明明1,2,孟萌1,2,赵亚荣1,2*
(1. 北京大北农科技集团股份有限公司动物医学研究中心,北京100195;2. 北京市畜禽生物制品工程技术研究中心,北京100195)
为研究猪流行性腹泻病毒的流行情况并分析该病毒的培养特性,对甘肃、北京、河北、山西、浙江五个猪场送检的疑似猪流行性腹泻病猪小肠内容物,用RT-PCR方法进行检测,证明为猪流行性腹泻病毒核酸阳性。然后将阳性病料进行传代培养、病毒含量测定以及特异性检验,证明获得五株猪流行性腹泻病毒,分别命名为Ningxia9、Beijing5、Gaocheng8、Shanxi1、Zhejiang08。对分离的毒株进行S基因全长测序及氨基酸序列分析。结果表明:五株分离毒株在盲传19代开始出现明显的细胞病变,盲传到24代,出现稳定的细胞病变且五株分离毒株的24代病毒含量在104.5~5.0TCID50/mL之间;用S蛋白进行进化树分析表明,五株分离毒株与近年来国内流行毒株亲缘关系很近,在同一个进化分支上;分离的五株野毒株之间的氨基酸同源性高达99.2%~99.9%,而这五株野毒与GenBank上提交的2011年、2012年分离的野毒株的之间的氨基酸同源性为98.4%~99.9%;实验室分离毒株及国内近年流行毒株均与国内外经典毒株存在一定的共性,即特定位置氨基酸的插入、缺失或置换。
猪流行性腹泻病毒;分离鉴定;序列分析
猪流行性腹泻病毒( porcine epidemic diarrhea virus, PEDV) 为猪的一种冠状病毒,可引起猪的急性、高度接触性肠道传染病猪流行性腹泻( porcine epidemic diarrhea, PED)。该病主要特征为脱水、呕吐和腹泻。各种年龄和品种的猪均易感,尤其是对哺乳仔猪危害最为严重,发病率高达100%,死亡率为30%~80%,是导致仔猪早期死亡的主要疾病之一[1-2]。1971年英国首次报道了本病,随后在日本、德国和比利时等许多国家均有报道,我国1976年首次报道[3],2013年美国爆发疫情,2014年德国、加拿大报道了PED的流行[4-5],目前该病已经成为影响养猪业的重要腹泻病之一。
PEDV为单股正链RNA病毒,S糖蛋白位于病毒粒子表面,在刺激机体产生中和抗体过程中发挥重要作用,其中的S1结构域包含主要中和抗原表位[6-7]。S基因序列分析发现,在不同地区不同时间分离的毒株之间变异都很大,近几年的流行毒株和2010年以前的毒株以及传统疫苗毒株的差异主要存在于S基因[8],因此S基因是分析PEDV流行毒株的遗传变异和流行特点的主要切入点。
本研究对甘肃、北京、河北、山西、浙江五个猪场疑似猪流行性腹泻病猪采集小肠内容物,RT-PCR方法进行检测确定为猪流行性腹泻病毒感染。采用细胞培养法分离五株猪流行性腹泻病毒,通过对S基因序列进行了分析,结果表明该分离株与国内当前流行株同源性很高,为进一步疫苗的研究和猪流行性腹泻的防控奠定了基础。
1 材料
1.1 病料 疑似猪流行性腹泻病猪小肠内容物,收集于甘肃、北京、河北、山西、浙江五个地方猪场。
1.2 细胞和培养基 Vero细胞由北京大北农集团动物医学研究中心保管和供应,DMEM购自Hyclone公司。
1.3 主要试剂 PCR反应用试剂、DNA Marker、RNA 提取试剂盒、琼脂糖凝胶DNA回收试剂盒、T载体等购自天根生化公司;胎牛血清和胰蛋白酶均购自GIBCO公司;PEDV特异性血清由大北农集团动物医学研究中心制备。
1.4 引物合成和基因测序 由生工生物工程(上海)股份有限公司完成。
2 方法
2.1 病料的处理 取病猪小肠适量,刮取肠粘膜和内容物,充分研磨,按照1︰5的比例(重量︰体积)加入PBS,混匀,反复冻融3次,离心取上清,0.22 μm滤膜过滤,滤液分装备用。
2.2 RT-PCR检测 将病料进行RT-PCR检测,根据病毒S蛋白编码基因的序列设计引物:正向引物为5’-GGATACTTTGGCCTCTTGTG-3’;反向引物5’-CGCACTCGGATTACTCACAGC-3’。 PEDV预计扩增片段为484 bp。取处理好的病料上清液,按照QIAamp柱式RNA提取试剂盒(Qiagen)提取RNA。用M-MLV合成cDNA,总反应体系20 μL:RNA 3 μg、Oligo(dT)18 Primer (10 μmol/L) 0.5 μL、dNTPs1 μL、5×First-Stand Buffer 4 μL、RNAsin 0.5 μL、M-MLV 0.5 μL、RNase-free Water补足至20 μL。轻轻混匀,37 ℃水浴60 min,95 ℃水浴5 min后,立即冰浴。以cDNA为模板,建立50 μL PCR反应体系:10×PCR Buffer 5 μL、dNTPs 2 μL、正向引物1 μL、反向引物1μL、cDNA2 μL、Taq酶0.5 μL、ddH2O补足至50 μL。将PCR反应体系置PCR仪内,95 ℃预变性5 min,95 ℃ 45 s、50 ℃ 1 min、72 ℃ 2 min进行32个循环后,72 ℃延伸5 min。PCR产物在1% 琼脂糖凝胶电泳中进行检测。
2.3 接种细胞和传代 取长满单层的Vero细胞,用pH7.4的PBS洗三次,按照10%的比例接种上述处理物,添加胰蛋白酶至终浓度10 μg/mL,37 ℃吸附1 h,然后加入含终浓度10 μg/mL胰蛋白酶的无血清DMEM维持液,37 ℃5%CO2培养箱培养,逐日观察,若无CPE则72 h收获,反复冻融3次后按上述方法传代。如此操作盲传,观察是否产生CPE。同时设置空白细胞作为对照。
2.4 病毒含量测定 将分离毒株第24代毒用细胞维持液进行10倍系列稀释,取10-4、10-5、10-6、10-7四个稀释度,每个稀释度分别接种长满Vero细胞单层的96孔细胞板6孔,100 μL/孔,同时设阴性对照细胞6孔,37 ℃ 5%CO2培养箱培养72~120 h,观察细胞病变,细胞颗粒增多、圆缩、细胞破损、脱落判为感染。同时阴性对照组细胞孔应不出现细胞病变。采用Reed-Muench法,计算TCID50。
2.5 病毒的特异性检验 将第24代毒用DMEM液稀释至103.0TCID50/mL与等量的PEDV特异性血清混合,置37 ℃中和1 h,接种长满Vero细胞单层的96孔细胞板4孔,同时设不中和的病毒阳性对照细胞4孔和仅接种细胞维持液的阴性对照细胞4孔,37 ℃ 5% CO2培养箱培养72~120 h,观察细胞病变,中和组和阴性对照组均应不出现细胞病变,而病毒对照组应出现细胞病变。
2.6 S基因序列分析 参照GenBank中的PEDV-S基因序列设计三对特异性引物。S1-p1:5′-GTTGAAGAATGGTAAGTTGCTAG-3′;S1-p2:5′-ATAAAGAATACGCTGAATGGC-3′;
S2-p1:5′- GGCACTGACGATGACGTTTC-3′;S2-p2:5′-CCATTAGAACAGCGCTTATAGTCT-3′;S3-p1:5′-TTTTTAATAAAGTGGTTACTAATGG-3′;S3-p2:5′-CTGTGTCAATCGTGTATTGAAAAAG-3′。
按照2.2的方法分别提取五株分离毒株24代病毒的RNA并进行反转录和PCR,S1片段PCR反应条件为:95 ℃预变性5 min,95 ℃ 50 s 、52 ℃ 1 min、72 ℃ 2 min进行32个循环后,72 ℃延伸5 min。S2片段PCR反应条件为:95 ℃预变性5 min,95 ℃ 50 s 、50 ℃ 1 min、72 ℃ 2 min进行32个循环后,72 ℃延伸5 min。S3片段PCR反应条件为: 95 ℃预变性5 min,95 ℃ 50 s、54 ℃ 1 min、72 ℃ 2 min,进行32个循环后,72 ℃延伸5 min。扩增产物经电泳和回收后连接到T载体进行测序分析,对S1、S2、S3序列进行拼接,将拼接完全的S基因进行氨基酸序列推导,并进行遗传进化分析和序列比对。
3 结果
3.1 RT-PCR检测结果 将病料提取物进行RT-PCR检测,扩增S基因片段为484 bp,结果见图1。

M:DNA Marker;1-5:样品;6:阴性对照图1 病料中PEDV的RT-PCR检测结果
3.2 病毒的分离培养结果 将上述样品处理后接种于Vero细胞,经过19代盲传后开始出现细胞病变,再继续传5代以后,细胞病变出现时间和病变类型基本稳定,约在接种后48-52 h出现明显细胞病变,主要表现为细胞颗粒增多,圆缩,细胞破损,脱落(图2)。
3.3 病毒含量测定 将第24代病毒进行病毒含量测定,Ningxia9为104.5TCID50/mL,Beijing5为104.75TCID50/mL,Gaocheng8为105.0TCID50/mL,Shanxi1为104.5TCID50/mL,Zhejiang08为105.0TCID50/mL。
3.4 病毒的特异性检验 病毒中和试验结果表明,分离的PEDV具有特异性,病毒被特异性血清中和后接种细胞没有出现CPE,阴性对照也没有出现CPE,接种病毒组细胞出现典型的CPE。3.5 S基因序列分析结果 五株分离毒株24代病毒的S蛋白全长氨基酸序列进化树分析结果见图3。
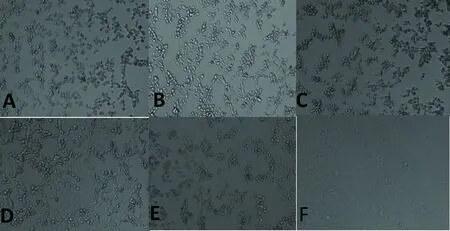
A-E:5个分离株;F:阴性对照图2 猪流行性腹泻病毒病变图(400×)
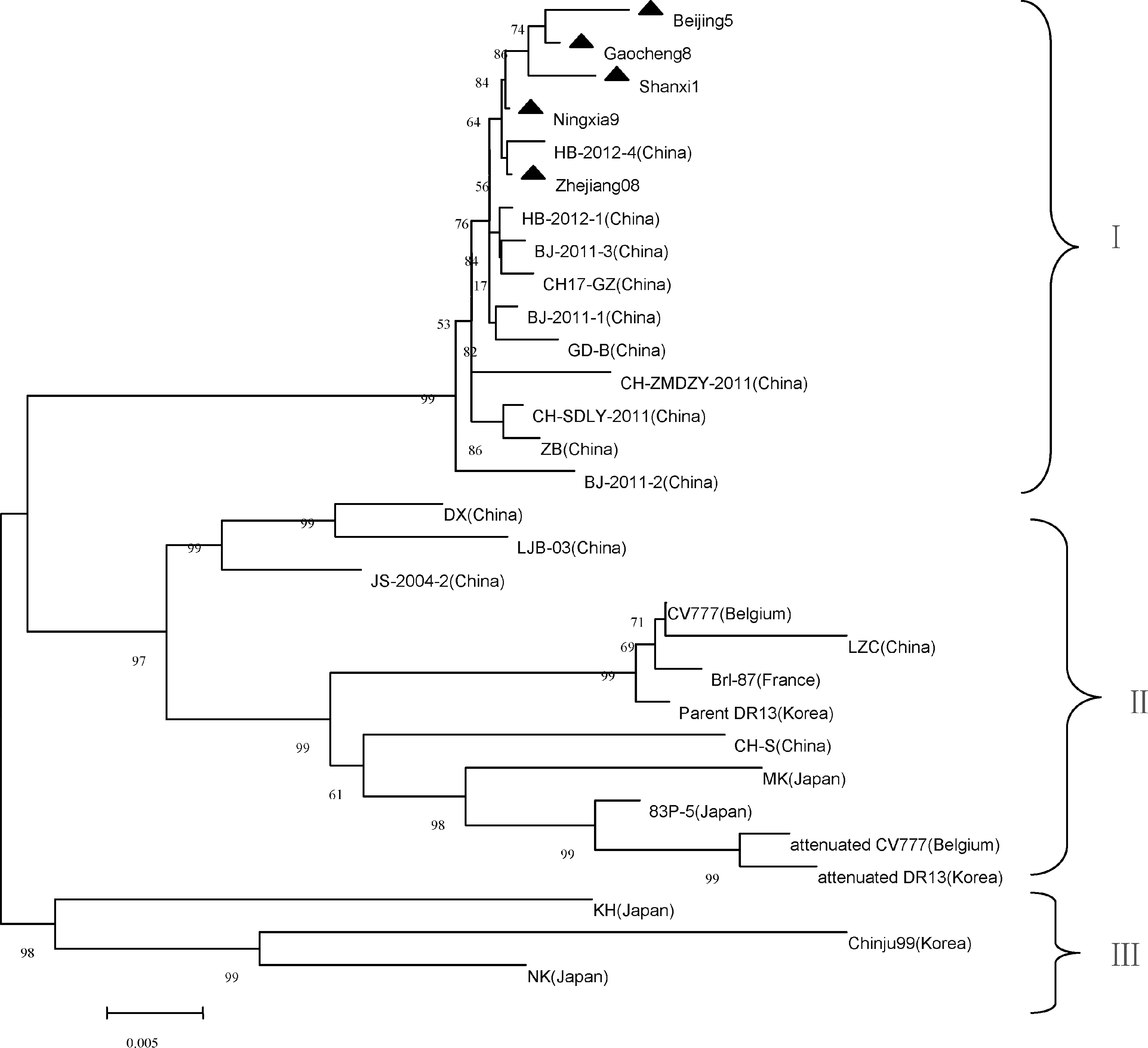
图3 S蛋白全长氨基酸序列进化树分析
PEDV主要分为三个进化群,群Ⅰ由本实验室分离的五株野毒株(Ningxia9、Beijing5、Gaocheng8、Shanxi1、Zhejiang08)以及2011年腹泻病爆发后各大高校和科研院所分别于2011年、2012年分离的野毒株(HB-2012-4、HB-2012-1、BJ-2011-1、BJ-2011-2、BJ-2011-3、GD-B、CH17-GZ、CH-SDLY-2011、ZMDZY-2011、ZB)组成;群Ⅱ主要由国内外经典毒株组成,包括中国2004年到2007年分离毒株(DX、LJB-03、JS-2004-2、LZC、CH-S),韩国毒株(Parent DR13、attenuated DR13),比利时毒株(CV777),法国毒株(Brl-87),日本毒株(MK、83P-5);群Ⅲ由一株韩国分离株(Chinju99)和两株日本分离株(KH、NK)组成。
根据推导的氨基酸序列进化树结果将群Ⅰ中本实验室分离的五株野毒株以及GenBank上提交的2011年、2012年分离的五个野毒株(HB-2012-4、HB-2012-1、BJ-2011-1、BJ-2011-2、BJ-2011-3)与群Ⅱ中国内外经典毒株(attenuated CV777、attenuated DR13、CV777、Parent DR13、DX、LJB-03、JS-2004-2、LZC)的全长S蛋白进行氨基酸序列比对分析,采用ClustalW法比对,有如下结果:实验室分离的五株野毒株Ningxia9、Beijing5、Gaocheng8、Shanxi1、Zhejiang08之间的氨基酸同源性高达99.2%~99.9%,而这五株野毒与GenBank上提交的2011年、2012年分离的野毒株之间的氨基酸同源性为98.4%~99.9%,其中BJ-2011-2与Beijing5、Shanxi1、Gaocheng8之间的氨基酸同源性稍低,分别为98.5%、98.8%、98.7%,CH-ZMDZY-2011与本实验室分离的五株野毒同源性也较低为98.4%~99.1%,说明本实验分离的野毒株与2011年腹泻大流行的毒株具有很高的同源性(表1)。

表1 群Ⅰ中流行毒株与群Ⅱ中国内外经典毒株之间S蛋白氨基酸序列比较
群Ⅰ中的十个毒株与群Ⅱ中的国内外经典毒株,无论是野毒株还是疫苗株,氨基酸序列变化存在如下共性:存在两个部位的氨基酸插入,一个是位于59~62aa位,插入了QGVN四个氨基酸,另一个是位于140 aa位,插入了N一个氨基酸;存在一个部位氨基酸的缺失,位于163~164位的两个氨基酸缺失,N(D)I(T);存在26处氨基酸置换,氨基酸置换个数由1个到5个不等(图4、表2)。
4 讨论
PEDV对细胞、培养条件的要求较高,自20世纪70年代报道以来,直到1988年Hoffman等通过在培养基中添加胰酶的方法才首次利用细胞培养方法成功分离到一株PEDV[9],此后陆续有采用相同的方法成功分离到PEDV的报道[10-11]。本研究参考国内外文献,在维持液中加入终浓度10 μg/mL的胰蛋白酶,利用Vero细胞成功分离到五株PEDV。
已知PEDV为冠状病毒,可将其S蛋白人为划分为S1区(1~789aa)和S2区(790~1387aa)。其中,S1区为主要的抗原表位存在区[12-13],序列分析表明26处置换全位于S1蛋白氨基酸序列之中,主要位于前369 aa中,且氨基酸的插入和缺失也位于该区域中。位于中和抗原表位83~276 aa处的的替换有19处;位于线性表位248~280为2处。

表2 群Ⅰ中变异毒株与群Ⅱ中国内外经典毒株之间S蛋白全长的氨基酸差异

变异情况氨基酸个数氨基酸变化位置替换3QST→SAN27~294SMNS→IGEN55~581S→T645GTGIE→AGQHP68~721L→V821Y→H842DS→RG86~871Q→H891I→T1202DN→SI130~1311V→A1383DGK→EHS161~1621A→S1791H→Y1841I(L)→F1871R→K1973K(R)RS→SGG201~2031T→E2111Y→S2281E→Q2301S(T)→I2382DS→EP247~2481L→V2711M→I3051L→F3471T→I369插入4QGVN59~621N140缺失2N(D)I(T)163~164
冠状病毒S1基因的变化能体现病毒的变异情况。由氨基酸序列比对分析表明流行毒株的氨基酸序列变化主要存在于S1区,氨基酸序列的变化有可能是导致病毒变异的原因[14-17]。S基因氨基酸进化树分析表明,本实验室分离的五株猪流行性腹泻病毒株与国内当前流行株同源性很高,将其致弱作为候选疫苗株,对于猪流行性腹泻防控具有重要的实践意义。
[1] Wood E N. An apparently new syndrome of porcine epidemic diarrhea[J]. Vet Rec, 1997, 100: 243-244.
[2] 陈建飞,冯力,时洪艳,等.猪流行性腹泻疫苗的研究[J].猪业科学,2010, 12: 50-51.
[3] 蔡宝祥. 家畜传染病学(第三版)[M]. 北京: 中国农业出版社, 2002: 213-214.
[4] Vlasova A N, Marthaler D, Wang Q,etal.Distinct characteristic sand complex evolution of PEDV strains North America, May 2013-Feburary 2014 [J]. Emerging Infectious Disease, 2014, 20 (10): 1620-1628.
[5] Wang L Y, Beverly B, Zhang Y. New variant of porcine epidemic diarrhea virus,United States, 2014[J]. Emerging Infectious Diseases, 2014, 20 (5): 917-919.
[6] Duarte M, Tobler K, Bridgen A,etal. Sequence of the spike protein of the porcine epidemic diarrhea virus [J]. J Gen Virol, 1994, 75: 1195-1200.[7] Follis K E, York J, Nunberg J H. Furin cleavage of the SARS coronavirus spike glycoprotein enhances cell-cell fusion but does not affect virion entry [J] . Virology, 2006 , 350 : 358-369.
[8] Sun R, Leng Z, Zhai S L,etal. Genetic variability and phylogeny of current Chinese porcine epidemic diarrhea virus strains based on spike, ORF3, and membrane genes [J]. The Scientific World Journal, 2014, (2014): 8.
[9] Hoffmann M, Wyler R. Propagation of the virus of procine epidemic diarrhea in cell culture[J]. J Clin Microbiol,1988,11:2235-2239.
[10] 李树根, 李力施, 霍燕琼, 等. 使用细胞培养单层分离猪流行性腹泻病毒[J]. 中国兽医科技, 1991, 10 : 24-25.
[11] 童昆周,李力复,林志雄,等.猪流行性腹泻弱毒疫苗的研究[J].中国兽医科技, 1996,26(l):3-4.
[12] Chang S H,Bae J L,Kang T J,etal.Identification of the epitope region capable of inducing neutralizing antibodies against the pocine epidemic diarrhea virus[J].Mol Cells,2002,14:295-299.
[13] Sun D,Feng L,Chen J,Shi H,etal.Cloning and sequence analysis of the spike protein gene of porcine epidemic diarrhea virus CH/JL strain and identification of its antigenic region containing linear epitopes[J].Chinese Journal of Virology, 2007,23:224-229.
[14] ChenJ F,Liu X Z, Shi H Y,etal.Detection and molecular diversity of spike gene of porcine epidemic diarrhea virus in China[J].Viruses, 2013,5:2601-2613.
[15] Wang J B,Zhao P J,Guo L H,etal.Porcine epidemic diarrhea virus variants with high pathogenicity[J].Emerging Infectious Diseases, 2013,19:2048-2049.
[16] Li W T,Li H,Liu Y B,etal.New variants of porcine epidemic diarrhea virus[J]. Emerging Infectious Diseases,2012,18:1350-1353.
[17] 刘孝珍, 陈建飞, 时洪艳, 等.2011年猪流行性腹泻病毒的遗传变异分析[J]. 中国预防兽医学报,2012, 3: 180-183.
(编辑:李文平)
Isolation and Identification of Five Porcine Epidemic Diarrhea Virus Strains and Sequence Analysis
WANG Gui-hua1,2, YU Ping-ping1,2, MAN Kun1,2, WU Shan-shan1,2,LIU Ming-ming1,2, MENG Meng1,2, ZHAO Ya-rong1,2*
(1.VeterinaryMedicineResearchCentreofBeijingDaBeiNongGroup,Beijing100195,China;2.BeijingEngineeringResearchCenterofBiologicalproductsinLivestockandPoultry,Beijing100195,China)
In order to research epidemiology and culture specificity of porcine epidemic diarrhea virus(PEDV), five feces samples from piglets suffering severe diarrhea in farms from Gansu, Beijing, Hebei, Shanxi and Zhejiang, were confirmed as PEDV positive by RT-PCR. Then these five samples were cultured by vero cell line to 24 passages, the five cultures were titrated and sequenced full long of S gene. Results showed that cytopathic effect (CPE) appeared from 19thto 24thpassages, and virus titer were between 104.5~5.0TCID50/mL.Sequence analysis showed that the amino acid homology was 99.2%~99.2%among five wild strains, 98.4%~99.9% among these five strains and the wild strains on GenBank from 2011 to 2012. Furthermore, the isolates, domestic pandemic strains and classic strains had certain commonality such as specific amino acid insertions and deletions or replacement.
porcine epidemic diarrhea virus; isolation and identification;sequence analysis
王贵华,博士,从事动物疫苗与诊断制品研究。
赵亚荣。E-mail: zhaoyarong@live.cn
2015-04-18
A
1002-1280 (2016) 07-0013-07
S852.65
