番茄品种SSR指纹图谱的构建
2016-01-18王溪桥文朝慧王军平
尤 佳,王 晋,王溪桥,文朝慧,王军平
(甘肃出入境检验检疫局, 兰州730010)
番茄(Solanum lycopersicum L.)是我国的主栽蔬菜之一。随着种子产业的发展,植物新品种的保护变得越来越重要。在我国培育和推广新的优良品种是番茄的主要发展方向,因此亟需建立一套快捷、准确、方便的番茄品种鉴定技术方法。
如今DNA指纹图谱已成为准确鉴定品种的有力工具[1],而SSR分子标记因其重复性好、呈共显性遗传等特点广泛应用于构建遗传图谱[2-4]、分析遗传多样性[5-7]、筛选目标性状基因标记[8-9]等方面。
本试验采用SSR标记技术对21份番茄品种构建指纹图谱,拟开发适用于番茄品种分子鉴定的SSR技术体系,为今后进行番茄品种DUS测试及真实性鉴定奠定基础。
1 材料与方法
1.1 试验材料
选7个番茄品种及其亲本共21份材料作为试验材料,每个杂交种的前2份材料为其父母本(见表1)。2014年将试验材料种植于试验田,采集叶片并进行田间纯度统计。另外,随机取T 223和T 255的F1材料100粒于培养皿内的无菌滤纸上在室内催芽,7d后取全株幼苗提取DNA。
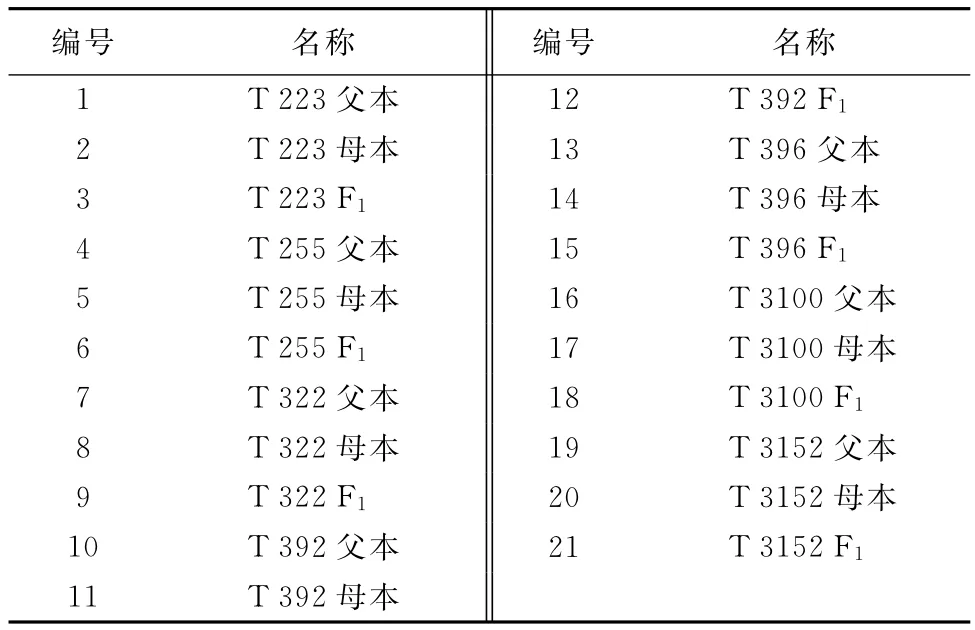
表1 供试番茄材料
1.2 处理方法
1.2.1 基因组 DNA 提取
田间取幼嫩叶片,采用CTAB法[10]提取基因组DNA,用紫外分光光度计法检测其质量和浓度,稀释至50ng/μL,-20℃下保存备用。
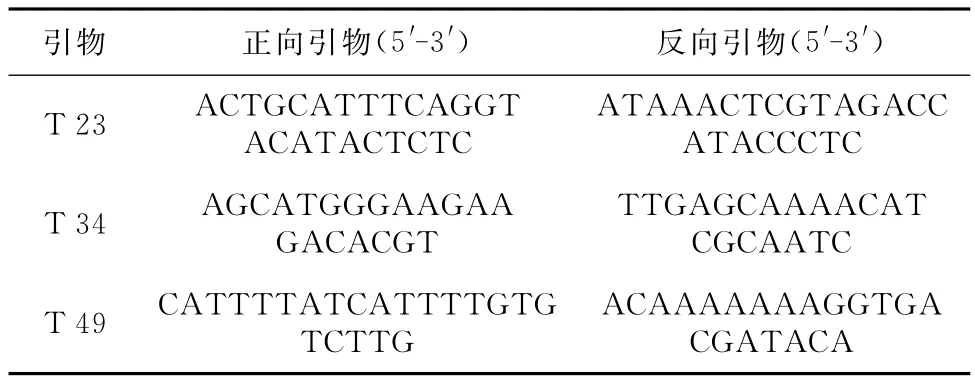
表2 SSR引物序列
1.2.2 SSR分析
试验所用SSR引物序列来自公开发表的文献[11-13],初步选用50对多态性好的引物对21种材料进行扩增分析,从中选取多态性高、条带清晰稳定、重复性好的用于构建指纹图谱(见表2)。SSR扩增体系为12.5μL(见表3)。SSR扩增程序(见表4)。

表3 12.5μL的扩增反应体系
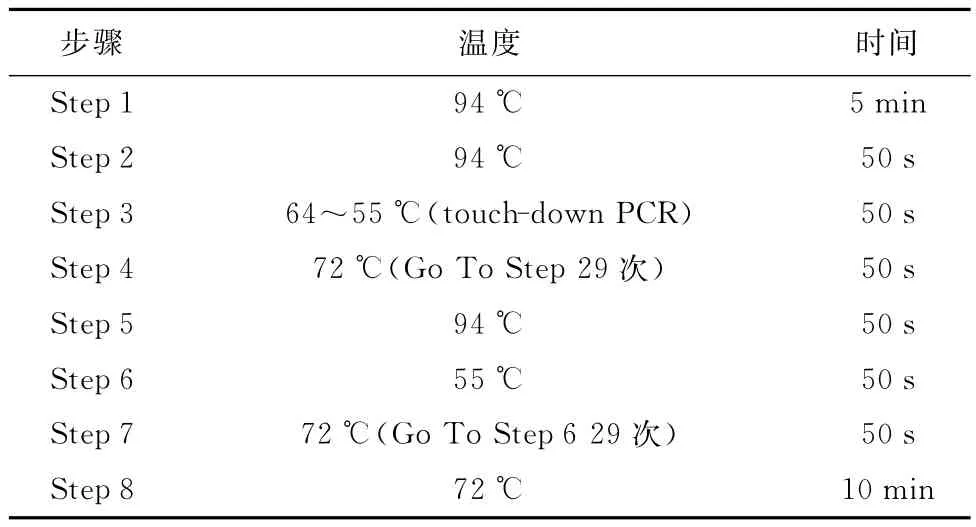
表4 扩增程序
SSR扩增产物用8%变性聚丙烯酰胺凝胶电泳,银染显色后对结果进行记录并分析。
1.2.3 农艺性状鉴定
于田间统计种子品种纯度。
1.2.4 数据统计分析
以二进制和基因型记录SSR分析结果,扩增条带在相同迁移率位置上,有带记为1,无带记为0。
2 结果与分析
2.1 指纹图谱构建
通过50对SSR引物筛选出多态性高、扩增带型稳定、清晰及重复性好的T 23、T 34和 T 49三对引物对21份番茄材料构建指纹图谱(图1)。电泳结果显示,21份番茄材料,不能单独用1对SSR引物区分,需要多对引物组合。引物T 23可以区分12份材料,再结合引物T 34可区分18份材料,最后结合引物T 49可将全部材料一一鉴别。以此3对引物组合产生的标记信息,以“1”和“0”分别表示扩增条带的有无,将3对引物组合产生的多态性位点依次排序,建立品种计算机化的数字DNA指纹(表5)。
2.2 杂交种纯度鉴定
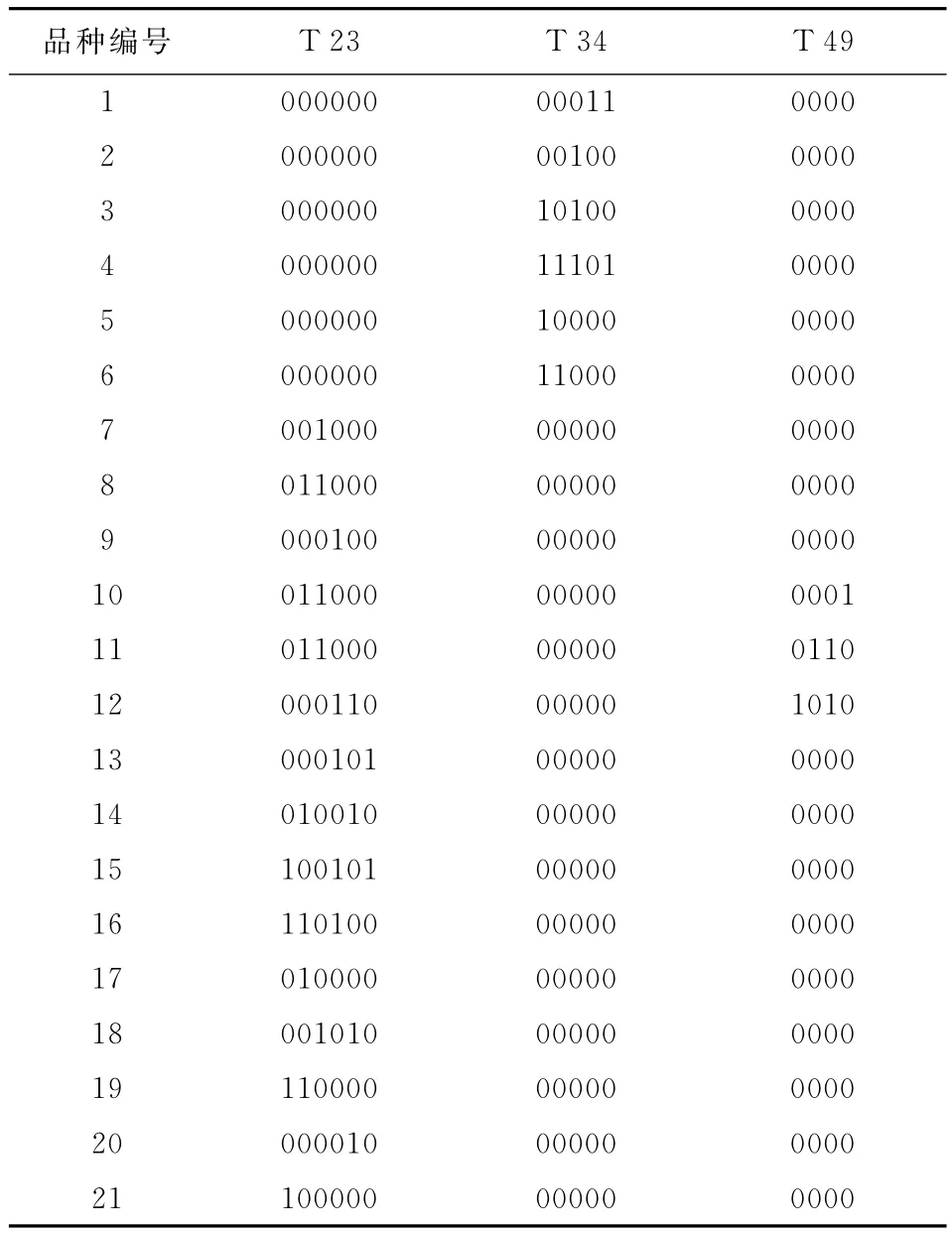
表5 7个番茄品种及其亲本的SSR指纹图谱
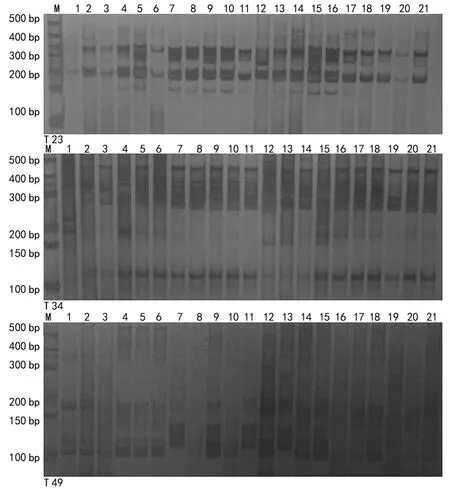
图1 3个SSR引物对7个番茄杂交种及其亲本的扩增
为进一步验证试验结果的可靠性,选择材料T 223和T 255的F1各100粒种子,进行种子纯度检测,统计带型结果显示种子纯度分别为93.22%和93.75%,与田间性状统计结果93.8%和93.8%相符。图2、图3为部分扩增图。
3 讨 论
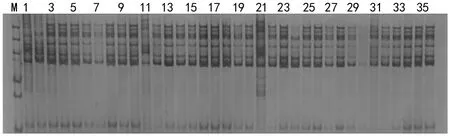
图2 SSR特征引物T 34对番茄品种T 255F1的纯度鉴定(1~36)
相比其它分子标记技术,SSR标记技术具有多态性高、易检测、共显性和特异性强等特点[14]。因此,SSR技术已广泛应用于马铃薯[15]、油菜[16]、大白菜[17]等多种作物的指纹图谱构建。本研究从50对多态性较好的番茄SSR引物中选出3对扩增稳定、条带清晰的引物,成功构建了T 223、T 255等7个番茄品种的指纹图谱,为这些品种在分子方面快速、准确的鉴定奠定了基础。利用引物T 34对T 223、T 255品种的杂交种进行分子鉴定,纯度分别为93.22%和93.75%,与田间性状统计结果(93.8%和93.8%)相符。表明可利用本研究构建的指纹图谱进行番茄品种纯度的快速鉴定。
国内用于番茄育种的亲本材料遗传变异幅度小,很多国内的番茄品种不易找到多态性的差异。目前,开展番茄品种纯度鉴定工作首先要自主开发更多的SSR标记。
本研究利用SSR分子标记技术得到T 23、T 34、T 49共3对引物,构建了21份材料的指纹图谱,实现了对这些材料的分子鉴定,为SSR技术在番茄种子纯度鉴定中的应用和推广打下了坚实的基础。
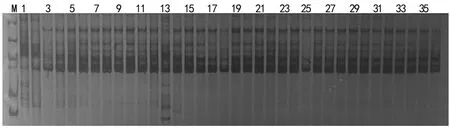
图3 SSR特征引物T 34对番茄品种T 223F1的纯度鉴定(1~36)
[1]Róder M S,Wendehake K,Korzun V,et al.Construction and analysis of a microsatellite-based database of European wheat varieties[J].Theor Appl Genet,2002,106:67-73.
[2]Cregan P B,JARVIK T,Bush A L,et al.An integrated genetic linkage map of the soybean genome[J].Crop Sci.,1999,39:1 464-1 490.
[3]赵姝华,李钥莹,邹剑秋,等.高粱分子遗传图谱的构建[J].杂粮作物,2005,25(1):11-13.
[4]张帆,万雪琴,潘光堂,等.玉米分子遗传图谱的构建[J].玉米科学,2006,14(3):6-9.
[5]梅铭凤,胜国旗,江玉梅,等.一粒小麦种质遗传多样性分析[J].麦类作物学报,2005,25(1):20-25.
[6]杨勇,王鹏文,张树光,等.SSR标记在糯玉米遗传多样性研究上的应用[J].玉米科学,2006,14(6):62-65.
[7]王一平,魏兴华,华蕾,等.不同地理来源旱稻种质资源的遗传多样性分析[J].作物学报,2007,33(12):124-130.
[8]张海泉,贾继增,杨虹,等.来自粗山羊草抗条锈病基因的SSR标记[J].遗传,2008,30(4):491-494.
[9]周新力,吴会杰,张如佳,等.来自簇毛麦抗条锈病新基因的SSR标记[J].植物病理学报,2008,38(1):69-74.
[10]Paterson A H,Brubaker C L and Wendel J F.A rapid method for extraction of cotton(Gossypium spp.)genomic DNA suitable for RFLP or PCR analysis[J].Plant Molecular Biology Reporter,1993,11:122-127.
[11]C.He·V.Poysa·K.Yu.Development and characterization of simple sequence repeat(SSR)markers and their use in determining relationships among Lycopersicon esculentum cultivars[J].Theor Appl Genet,2003,106:363-373.
[12]王柏柯,余庆辉,杨生保,等.4个加工番茄新品种SSR指纹图谱的构建与品种鉴定[J].新疆农业科,2010,47(7):1 474-1 478.
[13]王日升,李杨瑞,黄伟雄,等.利用SSR标记鉴定番茄种质资源[J].西北植物学报,2005,25(12):2 426-2 430.
[14]Powell W,Machray G C,Provan J.Polymorphism revealed by simple sequence repeats[J].Trends Plant Science,1996(7):215-222.
[15]Provan J,Powell W,Waugh R.Microsatellite analysis of relationships within cultivated potato(Solanum tuberosum)[J].Theor Appl Genet,1996,92:1 078-1 084.
[16]许鲲,张冬晓,伍晓明,等.国家冬油菜区试新品种SSR指纹图谱构建与遗传关系分析[J].中国油料作物学报,2008,30(1):29-34.
[17]李丽,何伟明,马连平,等.用EST-SSR分子标记技术构建大白菜核心种质及其指纹图谱库[J].基因组学与应用生物学,2009,28(1):76-88.
