玻璃质壳类底栖有孔虫卷转虫属Ammonia spp.的DNA保存方式和提取效能比较研究*
2016-01-15类彦立李铁刚
吕 曼 类彦立 李铁刚
(1. 中国科学院海洋研究所海洋生物分类与系统演化实验室 青岛 266071; 2. 国家海洋局第一海洋研究所海洋沉积与环境地质国家海洋局重点实验室 青岛海洋科学与技术国家实验室海洋地质过程与环境功能实验室 青岛 266061;3. 中国科学院大学 北京 100049)
有孔虫(Foraminifera)门隶属于原生动物界, 目前已知化石种约36000种, 现生种约4000—6000种,大部分为海洋底栖种类(Sen Gupta, 1999)。有孔虫因其壳体可长期保存在海洋水体及沉积物中, 因此在古海洋学等学科研究中有重要价值(汪品先等, 1980,1988)。另外在环境指示(Eichler et al, 2012)、生态评估(Boucheta et al, 2012)以及海洋生态系统的能量流动与物质循环中, 都扮演着重要角色。
现生有孔虫有钙质玻璃壳、钙质瓷质壳、砂质壳(胶结壳)和无壳(蛋白质膜)等多种类型。其中玻璃质类底栖有孔虫 Ammonia(Brünnich, 1772)是中国近海重要的优势类群, 常以优势种出现, 在中国各海域均见报道(汪品先等, 1980; 成鑫荣, 1987; 李保华等,2008; 赵泉鸿等, 2009; 李小艳等, 2010; 类彦立等,2015)。这类有孔虫常被用来指示滨岸和低盐等海洋环境, 具有重要的生态价值(郝诒纯等, 1980; 汪品先等, 1980)。
Ammonia是最早被确定为有孔虫的一个属, 其种类之多、形态变异之大使得这类有孔虫的分类鉴定尤为困难和混乱(Holzmann et al, 2000; Hayward et al,2004), 同样的困难也存在于其他类群的有孔虫。而分子技术的应用为解决这一难题提供了新的思路(Pawlowski et al, 2010, 2014, 2013)。有孔虫分子生物学研究已经有较长的历史(Langer et al, 1993), 对于Ammonia属的分子鉴定多基于对编码核糖体RNA基因的序列分析, 这方面的工作在国外已经有很多报道(Pawlowski et al, 1994b, 1995; Hayward et al, 2004),而国内的开展尚未普及(徐美香等, 2004; 李保华等,2005, 2007): 技术应用的不成熟、相关数据库信息量太少等都是其限制因素。
众所周知, 有孔虫很难在实验室培养并且繁殖,而且野外采样也受到时间和空间的限制。因此, 如何有效保存有孔虫的 DNA以用于后续研究, 是有孔虫分子生物学研究能够广泛开展的前提。海洋生物常用的保存方式有冷冻、乙醇固定及福尔马林固定等。但有研究指出后两种化学固定方法并不适用于有孔虫(Holzmann et al, 1996)。
本工作通过参考前人研究结果(徐美香等, 2004;李保华等, 2005, 2007; Pawlowski et al, 1994b, 1995;),最终选出3种保存方法, 即烘干法、冷冻法和裂解液法, 进行了更为系统的有孔虫 DNA保存方法的实验研究。另外, 由于不同壳质类型的有孔虫所适用的DNA提取方法不尽相同(Pawlowski, 2000)。本实验还比较了 DOC裂解液法和 Guanidine裂解液法对Ammonia属DNA提取效能的不同, 以期筛选出最适合玻璃质壳类底栖有孔虫的 DNA保存和提取方法,提供适合野外采集及实验室等不同环境下的最优方案。进而为分子生物学在这类有孔虫研究中的应用提供技术支持。
1 材料与方法
1.1 材料
1.1.1 活体有孔虫材料准备 实验所用的有孔虫材料是2014年2月份采自青岛湾潮间带的底栖优势类群, 卷转虫属 Ammonia spp. 经形态鉴定, 主要为Ammonia aomoriensis (Asano, 1951)与 Ammonia beccarii(Linnaeus, 1758)的混合种。把沉积物样品置于14°C左右的培养箱中孵化 2天, 挑选爬到沉积物表面、壳体完整、大小均一、房室呈棕黄色、体表黏附有颗粒物的 Ammonia spp., 这样的有孔虫活性较好(Holzmann et al, 1996; Schweizer et al, 2005)。放到盛有过滤海水的培养皿中, 投喂硅藻, 在实验室培养 1周。将有孔虫小心转移到灭菌过滤海水中, 在体视显微镜下, 用毛笔仔细清洗壳体表面的杂质, 准备进行DNA提取和保存实验。
1.1.2 试剂准备 DOC裂解液(0.1mol/LTris-HCl pH8.5, 4mmol/L EDTA pH8.0, 1%DOC, 0.2%TX-100);Guanidine裂解液(4mol/L Guanidinium isothiocyanate,0.05mol/L Tris-HCl pH7.6, 0.01mol/L EDTA,0.068mol/L Sarkosyl=N-Lauroylsarcosine sodium salt,1%β-巯基乙醇)(Pawlowski, 2000); 异丙醇; 70%乙醇;1×TE Buffer; 琼脂糖; TAE 电泳缓冲液; 2×PCR Mix(TIANGEN Golden Easy PCR System); ddH2O;PCR引物由北京六合华大基因科技有限公司合成。
1.1.3 仪器准备 PCR仪; 凝胶成像分析系统;电泳仪; 干燥箱; 高速冷冻离心机; –20°C、–80°C 冰箱; 恒温水浴锅。
1.2 DNA提取方法
采用DOC裂解液和Guanidine裂解液两种处理方法提取Ammonia DNA, DOC裂解液法设置2个平行组, Guanidine裂解液法设置3个平行组, 每个平行组均为单个样本。实验操作如下:
1.2.1 DOC裂解液法 取一只清洗过的Ammonia实验样品放置在含50μL DOC裂解液的1.5mL无菌离心管中, 研磨使壳破碎。于60°C水浴孵化1h。10000 r/min离心5min, 去除不溶物。所得即为基因组DNA,于 –20°C 保 存 (Holzmann et al, 1996; Pawlowski,2000)。
1.2.2 Guanidine裂解液法 取一只清洗过的Ammonia实验样品放置在含 50μL Guanidine裂解液的 1.5mL无菌离心管中, 研磨使壳破碎。60°C加热15min, 或室温过夜。8000 r/min离心 1min, 取上清液。加入与第一步中Guanidine裂解液等量的异丙醇,混匀。在–20°C沉淀至少2h, 或过夜。最大转速离心15min, 移去上清液, 注意不要扰动沉淀。用 100μL 70%乙醇洗涤沉淀物, 最大转速离心1min, 移去上清液。将沉淀物真空干燥5—10min, 或者室温下干燥更长时间。用15—20μL 1×TE Buffer溶解沉淀物, 在4°C放置至少1h, 或过夜, 期间取出涡旋数次。所得即为基因组 DNA, 于–20°C 保存(Pawlowski, 2000)。
1.3 DNA保存方法
对实验样品分别进行烘干、冷冻和裂解液保存等3种方法处理。鉴于预实验以及 Holzmann等(1996)的实验结果, 加之实验样品数量限制, 为了最大限度进行实验研究, 进行如下实验设计: 烘干法共 11个处理组, 未设平行组; 冷冻法共 4个处理组, 每个处理设2个平行组; 裂解液法共6个处理组, 每个处理设2个平行组。采用1.2.1中介绍的DOC法提取DNA。具体操作如下:
1.3.1 烘干保存法 取清洗好的 Ammonia实验样品放置在 1.5mL无菌离心管中, 每管一只。分别在温度为 20、30、40、50、60、70、80、90、100、110和 120°C烘干 30min, 每个温度组烘干 11只。每个温度组各取一只, 立即提取 DNA。其余的在室温下保存, 之后每周取每个温度组的一个样本提取DNA, 连续进行10周实验。每次实验均取一只活的Ammonia提取DNA作为阳性对照, 另用无菌水做空白对照。
1.3.2 冷冻保存法 取清洗好的 Ammonia实验样品放置在1.5mL无菌离心管中, 每管一只。设以下4个冷冻组: 在无水和 50μL无菌过滤海水中分别在–20°C和–80°C下冷冻。之后每周对每个冷冻组提取DNA, 连续进行8周实验, 提取前需要解冻并排出海水。设置阳性和空白对照。
1.3.3 裂解液保存法 为比较裂解液中是否含有EDTA对于Ammonia DNA保存效果的差异, 进行如下实验。取清洗好的Ammonia实验样品放置在1.5mL无菌离心管中, 每管一只。设以下6个实验组: 分别在2种裂解液(含4mmol/L EDTA的DOC以及不含EDTA 的 DOC)、3 种温度(室温、4°C、–20°C)下对样品进行保存实验。之后每周对每个实验组提取DNA,连续进行8周实验。设置阳性和空白对照。
1.4 DNA检测方法
基于核糖体 RNA(rRNA)基因大亚基(large subunit, LSU)片段的序列分析在 Ammonia属种类鉴定等方面的研究中已经有很好的应用(Pawlowski et al, 1994a, 1995; Holzmann et al, 1996, 2000; Schweizer et al, 2011)。
本实验运用 PCR技术, 对所有实验组提取的DNA, 扩增其 LSU rRNA基因片段(大约 750bp), 取4μL扩增产物, 经 1.0%琼脂糖凝胶(TAE缓冲液,110V)电泳 20min、EB染色 10min, 然后用凝胶成像分析系统拍照。通过分析电泳条带质量判断所提取的DNA质量。DNA提取方法组的比较, 是直接观察电泳图条带的质量并结合后期测序结果; DNA保存方法组的比较, 是在获得电泳图之后, 用 ImageJ软件对电泳图中目的条带的亮度进行定量分析, 用“灰度值”表示定量分析的结果, PCR条带的亮度或者灰度值可用于间接反映DNA保存的质量(宋锋等, 2010)。
PCR 引物: 正向 2TA(5′CACATCAGCTCGAGT GAG3′), 反向 L1F(5′ACTCTCTCTTTCACTCC3′)。反应体系: 25μL (2×PCR Mix 12.5μL, 10μmol/L 的正反向引物各 0.5μL, DNA 模板 2μL, ddH2O 9.5μL)。反应条件: 95°C 预变性 60s; 94°C 变性 30s、52°C 退火 30s、72°C延伸90s, 共34个循环; 72°C延伸3min。
1.5 数据统计分析
对电泳条带的亮度量化后, 用 SPSS19.0软件包中有关的方差分析做数据统计分析, 采用LSD检验。比较Ammonia DNA的保存效果在不同处理方法、不同保存时间组之间的差异。
2 结果与结论
2.1 DNA提取方法比较
由图1可以看出, 经DOC 裂解液法和Guanidine裂解液法提取到的DNA, 在PCR扩增之后都能得到整齐、单一、明亮的、与目标条带大小一致的条带, 且后续测序结果证实是目标条带。证明这两种方法所提取的Ammonia属有孔虫DNA的浓度和纯度都能满足后续PCR的需求。但是DOC裂解液法比Guanidine法操作更简单, 试剂更便宜, 所以前者更适合Ammonia属有孔虫DNA的提取。

图1 两种DNA提取方法的PCR扩增电泳图谱Fig.1 Gel electrophoreses of PCR products obtained from two methods of DNA extraction
2.2 DNA保存方法比较
2.2.1 烘干保存法 经 ImageJ软件分析各组的PCR条带所得灰度值见表1。用SPSS19.0做单因素方差分析, 采用LSD检验。结果显示20°C组和30°C显著高于40°C组(P<0.05), 从均值图看 20°C组结果最好, 但未与30°C组产生显著差异(图2)。综合考虑实验的简便性和保存结果的优异性, 建议 20°C为最佳烘干温度。另外, 比较第0、1、2周的结果发现, 三组间没有显著差异(P>0.05), 表明烘干后立即提取DNA和保存一段时间之后提取的结果无显著差异。到第10周灰度值有显著下降(P<0.05), 说明保存时间超过10周样品DNA可能会有显著降解。
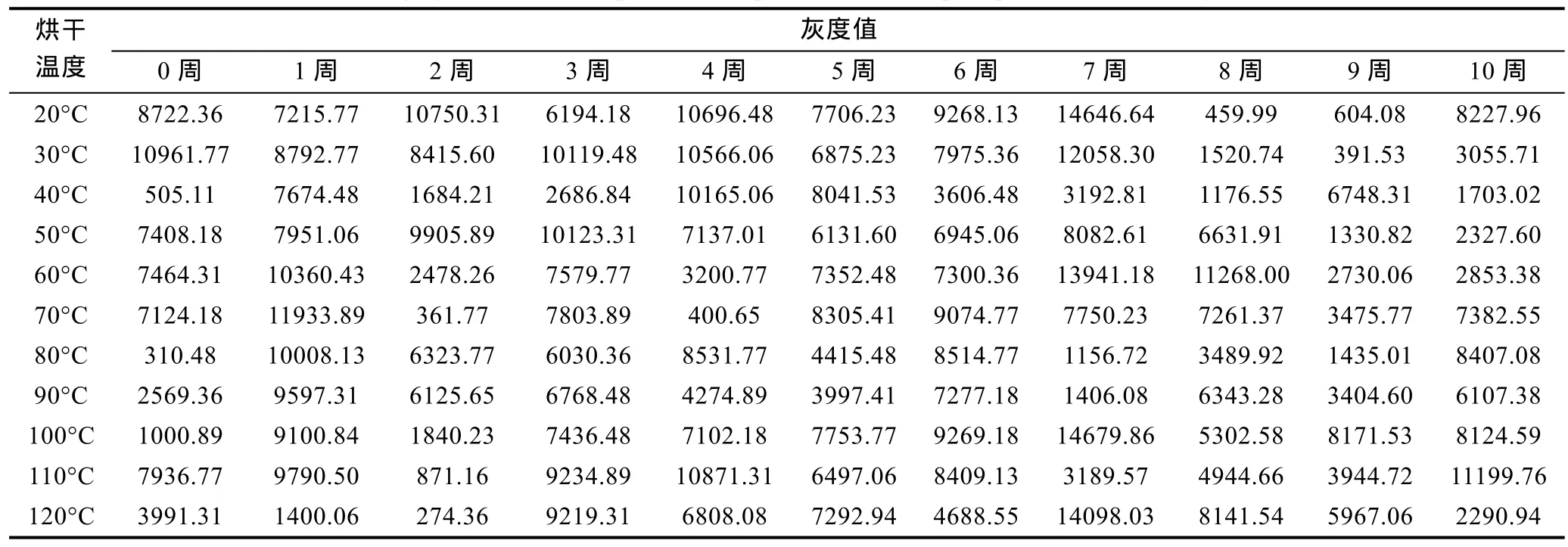
表1 烘干保存各实验组PCR条带的灰度值Tab.1 The gray value of PCR products amplified from samples preserved in air-dry method
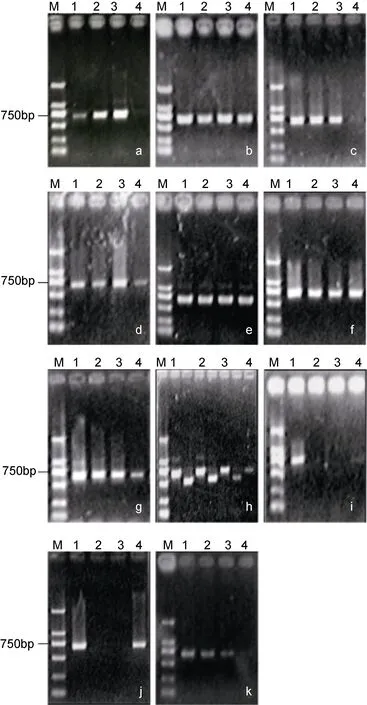
图2 烘干保存法的PCR扩增电泳图谱Fig.2 Gel electrophoreses of PCR products preserved in air-dry method
2.2.2 冷冻保存法 各组的PCR条带灰度值见表2(以均值表示)。用SPSS19.0作3因素单变量方差分析, 不同时间组采用LSD检验。结果显示–80°C组均值高于–20°C组, 但未达到统计上的显著水平(P>0.05)。无水组结果显著好于有水组(P<0.05)。不同保存时间之间没有显著差异, 直到第8周, 各组依然有较好的结果(图 3)。因此, 采用–20°C 或–80°C 无水条件对Ammonia属有孔虫进行冷冻处理, 至少 8周以内其DNA都有较好的保存效果。
2.2.3 裂解液保存法 各组的 PCR条带灰度值见表 2(以均值表示)。统计分析方法与“冷冻保存组”相同。均值比较结果显示4°C组效果比室温和–20°C组好, 含EDTA组比不含EDTA组效果好, 但都没有达到统计上的显著水平(P>0.05)。不同保存时间之间没有显著差异, 直到第8周, 各处理组依然有较好的结果(图 4)。因此, 若采用 DOC裂解液保存Ammonia属有孔虫的DNA, 建议选择4°C含EDTA的裂解液,至少8周以内对DNA都有较好的保存效果。
2.2.4 三种方法总体比较 分别选取烘干法、冷冻法和裂解液法中保存效果最好的组, 比较它们之间的差异。烘干法选取 20°C, 1—8周组; 冷冻法选取–80°C无水, 1—8周组; 裂解液法选取4°C含EDTA,1—8周组。结果显示组间有显著差异: 冷冻法优于烘干法, 烘干法优于裂解液法(P<0.05)。因此, 最适合钙质玻璃壳底栖有孔虫的 DNA保存方法为–80°C无水条件下冷冻; 若–80°C 冷冻条件不可得, 可用–20°C冷冻代替, 但对样品的有效保存时间可能不如–80°C条件下长。
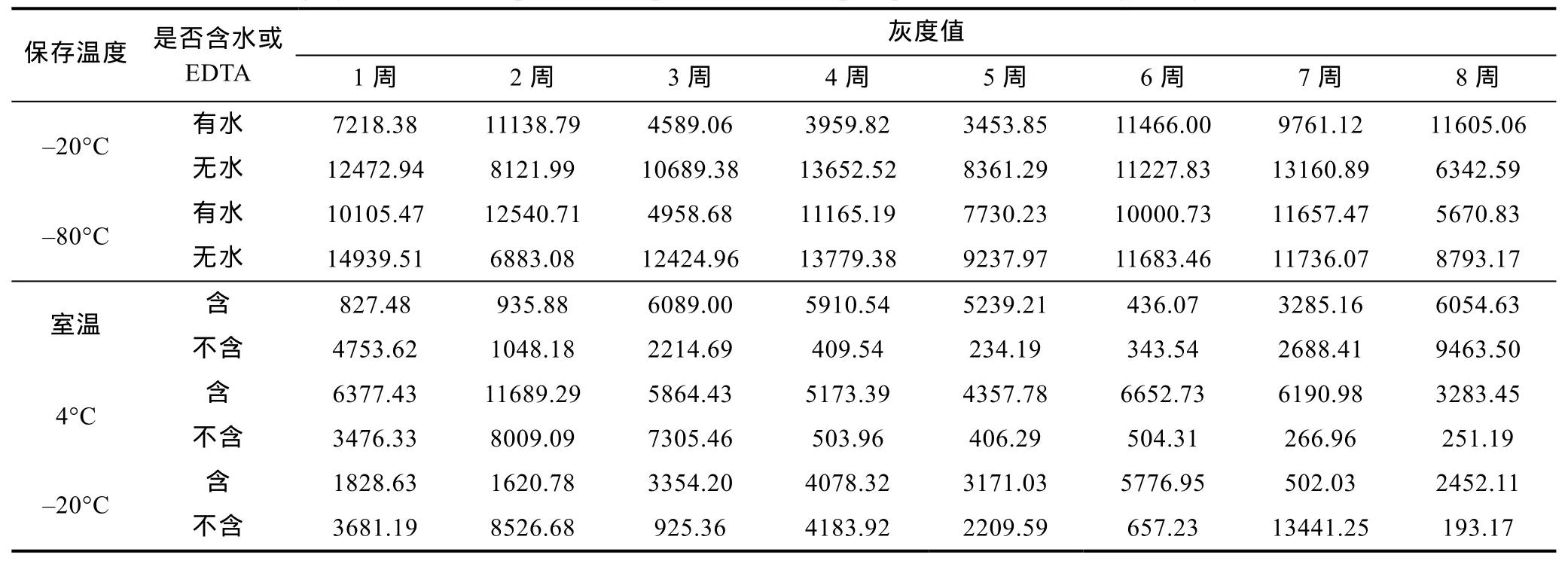
表2 冷冻保存及裂解液保存各实验组PCR条带的灰度值Tab.2 The gray value of PCR products amplified from samples preserved in freezing and lysis buffer method
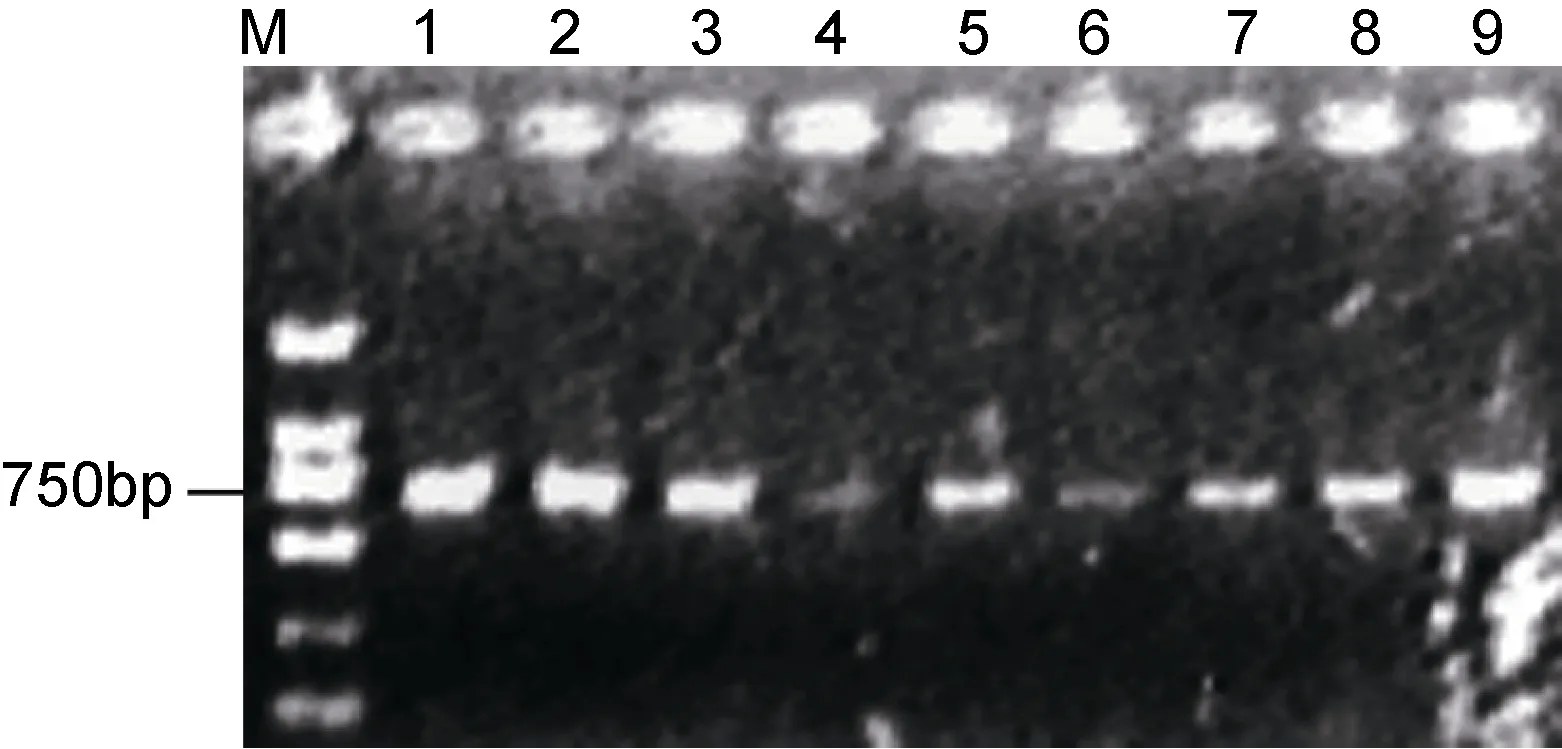
图3 冷冻保存8周的PCR扩增电泳图谱Fig.3 Gel electrophoreses of PCR products preserved in freezing method for 8 weeks
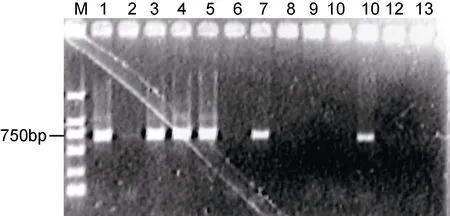
图4 裂解液保存8周的PCR扩增电泳图谱Fig.4 Gel electrophoreses of PCR products preserved in lysis buffer for 8 weeks
3 讨论
玻璃质类底栖有孔虫是我国近海最常见的优势有孔虫类群之一, 在潮间带该类群可占整体有孔虫群落的30%—70%, 其群落结构有重要的生态指示意义(Murry, 1991; 李保华等, 2008; 李小艳等, 2010)。对有孔虫准确的分类鉴定是研究其群落结构的前提;尤其是对Ammonia属为代表的玻璃质有孔虫, 形态鉴定较为混乱(类彦立等, 2015), 分子鉴定手段的应用对弥补传统形态学分类的不足具有重要作用(Pawlowskiet al, 1994b, 1995)。然而国内在现生有孔虫分子生物学方面的研究还几乎处于空白期。工作的一大难点在于分子技术的应用, 例如DNA提取方面,由于经常有微生物与有孔虫共生等原因导致很难获得纯的有孔虫DNA(Holzmannet al, 1996; Pawlowski,2000)。加之作为单细胞原生动物, 有孔虫DNA拷贝量比起其他多细胞动物要小得多, 使用单个样本提取出满足PCR要求的基因组DNA困难较大; 而对多样本提取 DNA时很难保证所有样本是同一个种, 尤其对于像Ammonia这样的形态鉴定疑难类群。研究中常常需要在对样品提取DNA的同时, 借助SEM等技术进行形态鉴定。因此, 干燥之后的样品还能稳定提取 DNA, 并且满足后续 PCR扩增, 对于有孔虫分子生物学研究非常重要。另外, 某些有孔虫类群的rRNA基因有高度的细胞内变异, 给 DNA测序工作带来了一定的困难(Pawlowski, 2000)。
对有孔虫 DNA进行有效保存是进行后续分子鉴定的前提。Holzmann等(1996)对Ammonia的DNA保存方法进行了一些研究, 但除了 30°C干燥保存外, 其余方法所探索的保存时间都不超过5周。国内未见有专门研究有孔虫 DNA保存方法的报道。本研究进行了更多的保存方法以及更长的保存周期(8至 10周)的实验探索, 各种方法各有优缺点,以期满足不同保存时间以及不同条件下(如野外或实验室等)的需求。
有孔虫基因结构的一些独特之处, 例如 SSU rRNA和LSU rRNA基因序列不仅拥有不同寻常的长度, 而且存在很大的种内变异(Pawlowski, 2000), 使得一些种类的核糖体基因很难成功扩增。另外个体大小、虫体活性、有壳等因素都限制了有孔虫DNA提取的成功率。因此, 即使是对新鲜样品直接提取DNA,也不一定能够成功进行 PCR扩增。这也可能是导致相同实验条件下结果相差很大的原因(例如图3中4、5号条带以及图4中2、3号条带)。
由于本实验中使用了2TA-L1F和2TA-L7两对引物进行巢式PCR扩增(Schweizer et al, 2011), DOC裂解液法提取DNA后PCR结果出现双带(如图1a中1、2号条带所示)。有研究显示Ammonia属LSU rRNA基因的扩增可能不需要巢式 PCR(Holzmann et al,1996)。本研究通过对扩增出的双带进行测序, 表明它们分别是引物2TA-L1F及2TA-L7扩增的产物, 从而证实Ammonia属LSU rRNA基因的扩增不需要巢式PCR。
有报道指出 EDTA对有孔虫的壳有破坏作用(Seears et al, 2014)。在本实验过程中, 每次提DNA之前均对样品镜检, 发现用含 EDTA裂解液保存的Ammonia样品的壳溶解程度比不含EDTA组的更严重; 在室温和4°C保存的Ammonia样品的壳溶解程度比–20°C组的更严重。推测随着壳的逐渐溶解, 原本由壳保护着的核物质可能会受到某种影响。为了探索这种影响对Ammonia DNA保存效果造成的差异, 本实验分别检验了含与不含EDTA的裂解液对于Ammonia DNA的保存效果, 前者效果普遍比后者好。
综上所述, 本研究以玻璃质壳类底栖有孔虫的代表类群Ammonia spp.为材料, 比较了3种DNA保存方法(烘干、冷冻、DOC裂解液)以及2种DNA提取方法(DOC 裂解液法、Guanidine裂解液法)。实验结果表明: (1) 烘干保存法中 20°C为最佳烘干温度,保存时间超过10周样品DNA可能会有显著降解; 冷冻保存法中–20°C和–80°C冷冻保存直至第8周样品DNA依然较完好; DOC裂解液保存法中样品用含EDTA的裂解液在4°C保存效果较好, 至少可以保存8周。(2) 从对有孔虫DNA保存效果来看, –80°C无水条件下冷冻效果最佳; 烘干保存法使得有孔虫形态鉴定与分子鉴定可以更好地结合; 裂解液法保存则可以很方便地直接进行后续 DNA提取实验, 较适合短期保存。3种保存方法各有优缺点, 可根据不同实验需求进行选择。(3) DOC裂解液法和 Guanidine裂解液法对玻璃质类有孔虫 DNA提取效果都很好,但前者更经济且操作简便。
4 结论
玻璃质壳类底栖有孔虫DNA保存和提取优化方案如下:
DNA保存:
保存法1——烘干法:
A. 设备器皿准备:
干燥箱、1.5mL无菌离心管。
B. 操作步骤:
(1) 取单只实验样品, 于无菌过滤海水中洗净;
(2) 虫体置于1.5mL无菌离心管中, 于20—30°C干燥30min;
(3) 在提取DNA之前于室温、密封保存。
保存法2——冷冻法:
A. 设备器皿准备:
–20°C冰箱、–80°C超低温冰箱、1.5mL无菌离心管。
B. 操作步骤:
(1) 取单只实验样品, 于无菌过滤海水中洗净;
(2) 虫体置于1.5mL无菌离心管中, 密封;
(3) 在提取 DNA 之前于–20°C 或–80°C 冷冻保存。
保存法3——裂解液法:
A. 设备和试剂准备:
(1) 设备器皿: 4°C冷藏箱、1.5mL无菌离心管;
(2) 试剂: DOC裂解液(Pawlowski, 2000)。
组分:
TRIS 1mol/L pH 8.5 100μL
EDTA 0.5mol/L (pH 8.0) 8μL
DOC (Na deoxycholate) 10% 100μL
TX-100 10% 20μL
H2O 780μL
B. 操作步骤:
(1) 取单只实验样品, 于无菌过滤海水中洗净;
(2) 虫体置于 1.5mL无菌离心管中, 加入含4mmol/L EDTA的DOC裂解液;
(3) 在提取DNA之前于4°C保存。
DNA提取:
A. 设备和试剂准备:
(1) 设备器皿: 水浴锅、高速离心机、1.5mL无菌离心管;
(2) 试剂: DOC裂解液, 组分同“保存法3”。
B. 操作步骤:
(1) 取单只实验样品, 于无菌过滤海水中洗净;
(2) 虫体置于 1.5mL无菌离心管中, 加入 50μL DOC裂解液;
(3) 将虫体彻底研磨碎, 于60°C水浴孵化1h;
(4) 10000r/min离心5min, 去除不溶物;
(5) 上清液中即为提取的基因组 DNA, 于–20°C保存。
致谢 感谢同济大学海洋与地球科学学院的翦知湣教授给予类彦立在有孔虫研究中的无私建议和指导, 另外中国科学院南京地质与古生物研究所李保华研究员曾共同探讨, 感谢 Paul Brönniman Foundation 2015基金以及Prof. Dr. Jan W. Pawlowski(University of Geneva, Switzerland)给予方法学讨论,中国科学院海洋研究所实验员王雪皎、郑萌萌、曹丽娜协助工作。谨致谢忱。
成鑫荣, 1987. 长江口表层沉积物中活有孔虫分布的初步研究.海洋地质与第四纪地质, 7(1): 73—79
李小艳, 石学法, 程振波等, 2010. 渤海莱州湾表层沉积物中底栖有孔虫分布特征及其环境意义. 微体古生物学报,27(1): 38—44
李保华, Ertan K T, Hemleben C, 2005. 有孔虫分子生物学研究进展. 自然科学进展, 15(5): 534—538
李保华, 王晓燕, 龙江平, 2008. 海南岛近岸沉积物中的有孔虫特征与分布. 微体古生物学报, 25(3): 225—234
李保华, 李铁刚, Ertan K T等, 2007. 西北太平洋浮游有孔虫的 SSU rDNA序列及其古海洋学意义. 自然科学进展,17(3): 391—395
汪品先, 章纪军, 闵秋宝, 1980. 东海表层沉积物中有孔虫的分布. 见: 海洋微体古生物论文集. 北京: 海洋出版社,20—38
汪品先, 章纪军, 赵泉鸿等, 1988. 东海底质中的有孔虫和介形虫. 北京: 海洋出版社. 1—307
宋 锋, 孙 敏, 罗克明, 2010. 一种基于PCR技术鉴定单拷贝转基因烟草的方法. 中国生物工程杂志, 30(4): 83—88
赵泉鸿, 湣翦知, 张在秀等, 2009. 东海陆架泥质沉积区全新世有孔虫和介形虫及其古环境应用. 微体古生物学报,26(2): 117—128
郝诒纯, 裘松余, 林甲兴等, 1980. 有孔虫. 北京: 科学出版社,1—224
类彦立, 李铁刚, 2015. 奥茅卷转虫 Ammonia aomoriensis(Asano, 1951)与毕克卷转虫Ammonia beccarii (Linnaeus,1758) (有孔虫)的分类学以及在黄东海分布的温盐深特征比较研究. 微体古生物学报, 32(1): 1—19
徐美香, 赵泉鸿, 肖 湘, 2004. 南海沉积物中有孔虫分子生物学鉴定的初步研究. 台湾海峡, 23(3): 354—359
Boucheta V M P, Alvea E, Rygg B et al, 2012. Benthic foraminifera provide a promising tool for ecological quality assessment of marine waters. Ecological Indicators, 23:66—75
Eichler P P B, Eichler B B, Gupta B S et al, 2012. Foraminifera as indicators of marine pollutant contamination on the inner continental shelf of southern Brazil. Marine Pollution Bulletin, 64(1): 22—30
Hayward B W, Holzmann M, Grenfell H R et al, 2004.Morphological distinction of molecular types in Ammonia-towards a taxonomic revision of the world’s most commonly misidentified foraminifera. Marine Micropaleontology, 50(3—4): 237—271
Holzmann M, Pawlowski J, 1996. Preservation of foraminifera for DNA extraction and PCR amplification. Journal of Foraminiferal Research, 26(3): 264—267
Holzmann M, Pawlowski J, 2000. Taxonomic relationships in the genus Ammonia (Foraminifera) based on ribosomal DNA sequences. Journal of Micropalaeontology, 19(1): 85—95
Langer M R, Lipps J H, Piller W E, 1993. Molecular paleobiology of protists: Amplification and direct sequencing of foraminiferal DNA. Micropaleontology, 39(1): 63—68
Murry J W, 1991. Ecology and Palaeoecology of Benthic Foraminifera. New York: John Wiley and Sons Inc., 397
Pawlowski J, 2000. Introduction to the molecular systematics of foraminifera. Micropaleontology, 46(S1): 1—12
Pawlowski J, Bolivar I, Fahrni J et al, 1994b. Taxonomic identification of foraminifera using ribosomal DNA sequences. Micropaleontology, 40(4): 373—377
Pawlowski J, Bolivar I, Farhni J et al, 1995. DNA analysis of“Ammonia beccarii” morphotypes: one or more species?.Marine Micropaleontology, 26(1—4): 171—178
Pawlowski J, Bolivar I, Guiard-Maffia J et al, 1994a.Phylogenetic Position of foraminifera inferred from LSU rRNA gene sequences. Molecular Biology and Evolution,11(6): 929—938
Pawlowski J, Holzmann M, 2014. A plea for DNA barcoding of foraminifera. The Journal of Foraminiferal Research, 44(1):62—67
Pawlowski J, Holzmann M, Tyszka J, 2013. New supraordinal classification of Foraminifera: molecules meet morphology.Marine Micropaleontology, 100: 1—10
Pawlowski J, Lecroq B, 2010. Short rDNA barcodes for species identification in foraminifera. The Journal of Eukaryotic Microbiology, 57(2): 197—205
Schweizer M, Pawlowski J, Duijnstee I A P et al, 2005.Molecular phylogeny of the foraminiferal genus Uvigerina based on ribosomal DNA sequences. Marine Micropaleontology, 57(3—4): 51—67
Schweizer M, Polovodova I, Nikulina A et al, 2011. Molecular identification of Ammonia and Elphidium species(Foraminifera, Rotaliida) from the Kiel Fjord (SW Baltic Sea) with rDNA sequences. Helgoland Marine Research,65(1): 1—10
Seears H A, Wade C M, 2014. Extracting DNA from within intact foraminiferal shells. Marine Micropaleontology, 109:46—53
Sen Gupta B K, 1999. Modern Foraminifera. Dordrecht: Kluwer Academic Publishers, 1—371
