基因组编辑技术及其在昆虫遗传转化中的应用
2015-12-17吕志创张桂芬刘万学万方浩
吕志创, 严 盈,2,3, 张桂芬, 武 强, 刘万学, 万方浩,4*
1中国农业科学院植物保护研究所,植物病虫害生物学国家重点研究室,北京 100193; 2Department
of Entomology, North Carolina State University, Campus Box 7613, Raleigh, NC 27695-7613, USA;
3Genetic Engineering and Society Center and W.M. Keck Center for Behavioral Biology,
North Carolina State University, Raleigh, NC 27695-7613, USA;
4农业部外来入侵生物预防与控制研究中心,北京 100193
基因组编辑技术及其在昆虫遗传转化中的应用
吕志创1, 严盈1,2,3, 张桂芬1, 武强1, 刘万学1, 万方浩1,4*
1中国农业科学院植物保护研究所,植物病虫害生物学国家重点研究室,北京 100193;2Department
of Entomology, North Carolina State University, Campus Box 7613, Raleigh, NC 27695-7613, USA;
3Genetic Engineering and Society Center and W.M. Keck Center for Behavioral Biology,
North Carolina State University, Raleigh, NC 27695-7613, USA;
4农业部外来入侵生物预防与控制研究中心,北京 100193
摘要:锌指核酸酶、类转录激活因子式核酸酶和CRISPR/Cas技术是近几年发展起来的3种主要基因组编辑技术,其原理都是通过在生物基因组特定位点制造DNA双链断裂损伤,从而激活机体自身的DNA 损伤修复机制,在此过程中引发各种变异。基因组编辑技术已在研究基因功能和基因修复中成功应用,基于基因组编辑技术的诸多优点,如CRISPR/Cas技术能对基因组中多个特定位点进行编辑,其有望成为昆虫遗传转化的主要策略。本文就锌指核酸酶、类转录激活因子式核酸酶和CRISPR/Cas技术的基本原理及其在昆虫中的应用做一简介,为今后利用基因组编辑技术进行昆虫遗传转化提供些许参考。
关键词:CRISPR/Cas技术; 遗传转化技术; 基因组编辑; 类转录激活因子式核酸酶技术; 锌指核酸酶技术
Genome editing techniques and their application
in insect genetic transformation
Zhi-chuang LÜ1, Ying YAN1,2,3, Gui-fen ZHANG1, Qiang WU1, Wan-xue LIU1, Fang-hao WAN1,4*
害虫遗传防治主要经历了2个发展阶段:基于辐射的昆虫不育技术(Sterile insect technology,SIT)和基于基因组编辑技术的遗传调控体系。虽然传统SIT是一种环境友好的害虫防治方法,但在实际运用中存在释放的昆虫竞争力降低、大规模饲养成本太高、难于对释放到野外的种群进行有效监测等问题。基于基因组编辑技术的害虫遗传调控策略不但可以有效地避免上述问题,并且能通过对靶标害虫基因组特定位点的精细调控,使整个控制体系更加科学高效和透明可控。目前,基因组编辑技术主要依靠2大类核酸酶:一类是天然存在的能够识别较长序列的核酸酶,如归巢核酸酶(Homing endonucleases),归巢核酸酶有严格的序列识别特异性,且难以通过工程化手段来改变其靶向特异性,因此应用较为局限(Smithetal.,2006);另一类是人工构建的改造核酸酶(Engineered nucleases),即借助遗传工程手段,将天然存在并能够靶向DNA的蛋白和核酸内切酶组装,如锌指核酸酶(Zinc finger nucleases,ZFNs)、类转录激活因子式核酸酶(Transcription activator-like effector nucleases,ALENs)和CRISPR/Cas(Clustered regularly interspaced short palindromic repeats,CRISPR; CRISPR associated protein,Cas)系统。这3种改造核酸酶的基因组编辑技术近年来发展迅猛并得到了广泛应用,其原理都是通过在生物基因组特定位点制造DNA双链断裂(DNA double-strand break,DSB)损伤,从而激活机体自身的DNA 损伤修复机制,在此过程中引发各种变异。DNA损伤修复方式主要有非同源末端连接修复(Non-homologous end joining,NHEJ)和同源介导修复(Homology-directed repair,HDR)(Hsuetal.,2014)。NHEJ能够快速有效地重新连接断裂末端,在该过程中于连接位点引入小的插入或缺失,从而造成目的基因移码突变并失去原有功能。HDR需要一个携带有断裂位点两端遗传信息的同源DNA臂,以实现少数核苷酸改变基因定点矫正或定点插入一个新基因。这2种高度保守的DNA损伤修复方式可被用于ZFNs技术、TALENs技术和CRISPR/Cas技术为导向的基因组靶向编辑。本文着重介绍这3种技术的原理及其在昆虫转化中的应用(表1)。
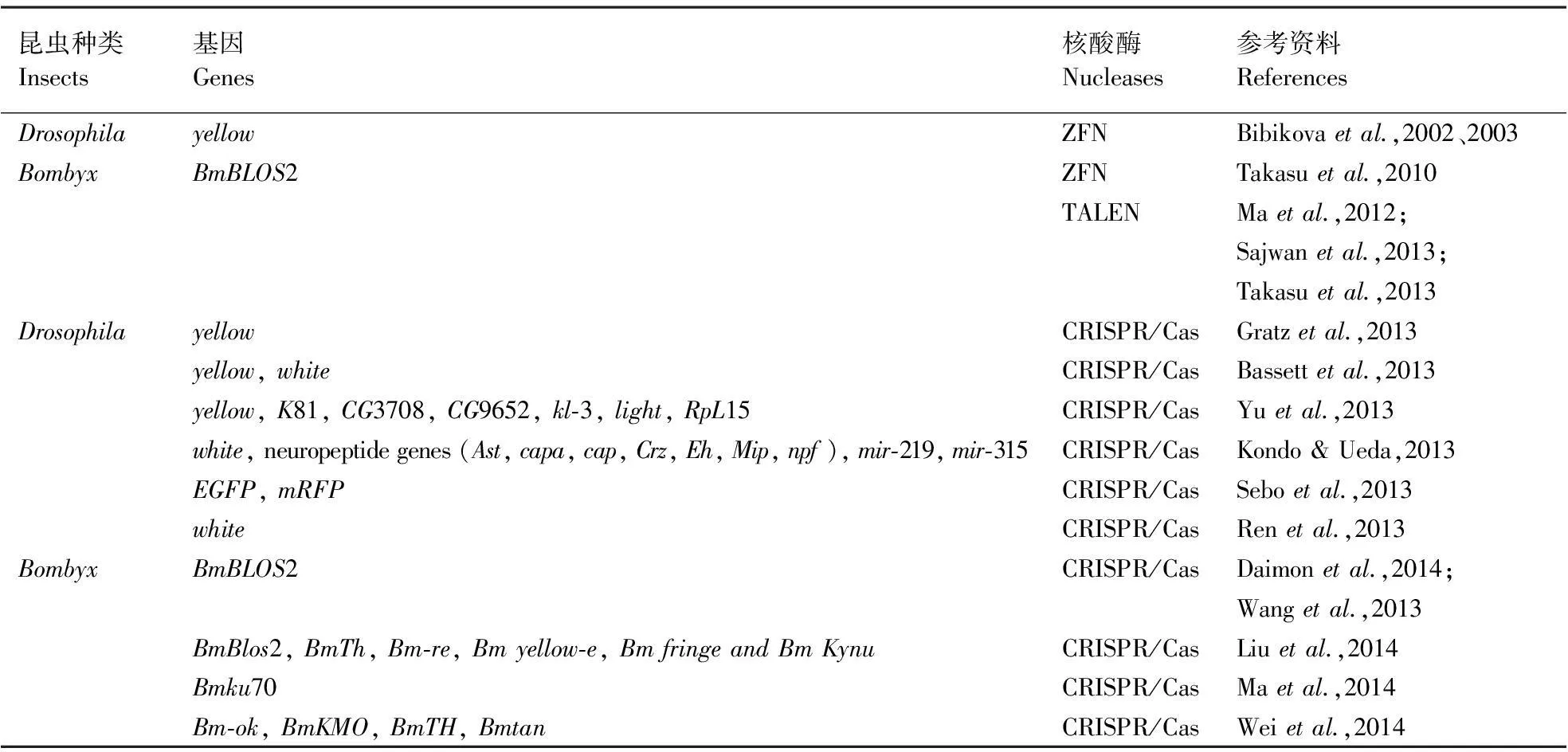
表1 ZFN, TALEN and CRISPR/Cas介导的基因组编辑技术在果蝇和家蚕中的应用
1 锌指核酸酶(ZFNs)技术
锌指(Zinc finger,ZF)是一种常见的DNA结合蛋白结构基元,每个锌指可直接特异识别3个连续的核苷酸。锌指蛋白(Zinc finger protein,ZFP)在自然界广泛存在,从简单的真核生物酵母到高级的哺乳动物小鼠,以及人基因组中都含有许多编码锌指蛋白的基因。锌指蛋白是一类天然存在的具有与特定DNA序列结合的特性蛋白(Klug,1999),是重要的转录调控因子。少量锌指蛋白还能作用于RNA(Brown,2005; Hall,2005)、蛋白或其他小分子(Gamsjaegeretal.,2007),具有重要的生物学功能。不同的锌指蛋白具有类似的结构框架,框架外关键位点的氨基酸变异会改变锌指蛋白结合DNA的特异性(Klug,2010)。锌指蛋白DNA结合域能够使锌指核酸酶结合特定DNA序列,并在基因组的特定位点产生双链断裂,随后,通过非同源末端连接修复(NHEJ)或同源介导修复(HDR)的DNA损伤修复方式进行基因组靶向编辑(图1A)。针对已知靶基因,目前构建人工锌指蛋白的策略主要有模块组装(Modular assembly,MA)、寡聚体库工程化筛选构建法(Oligomerized pool engineering,OPEN)、下文依赖组装法(Context-dependent assembly,CoDA)和双锌指筛选法。
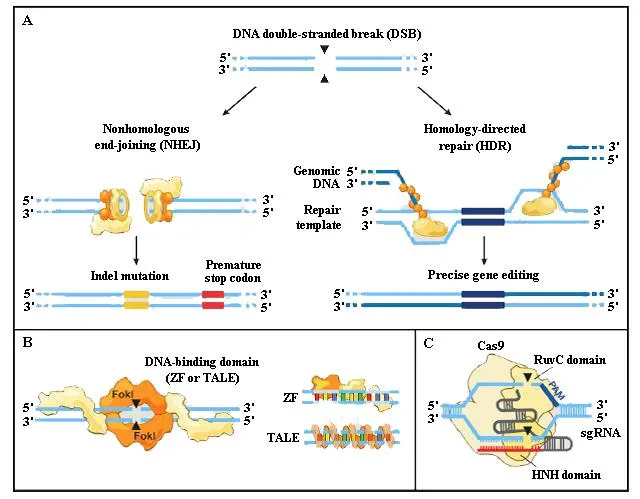
图1 内源性DNA修复基因组编辑技术原理(Hsu et al.,2014)
经遗传工程改造后的限制性内切酶FokI消除了其序列识别特异性,提高了切割效率(Bitinaiteetal.,1998)。特异锌指核酸酶的形成包括:选择靶标基因的靶位点,构建识别靶序列DNA的活性锌指蛋白,然后将锌指蛋白和FokI亚基融合即可。锌指核酸酶的切割特异性取决于锌指蛋白识别DNA靶序列的能力(Handel & Cathomen,2011)。Kimetal.(1996)首次将人工构建的多个锌指串联形成的锌指蛋白结构域与改造后的FokI限制性内切酶的切割结构域连接,形成具有切割特异 DNA活性的人工核酸内切酶(Zinc finger nucleases,ZFn),实现对靶序列的切割。串联锌指的数目与可识别的靶序列长度成正相关关系,串联锌指数目增加的同时也增加DNA靶向修饰的特异性(Milleretal.,1985; Wolfeetal.,2000)。基于每个ZF单体能够特异性地靶向3个碱基,因此理论上至少需要64种锌指蛋白单体,以满足针对任意序列构建ZFNs的可能性(王跃强等,2003)。但目前开发的ZF单体不能满足靶向任意序列,提高了ZFNs的设计与构建难度(Carrolletal.,2006)。
由锌指核酸酶介导的基因靶向编辑主要包括基因组突变和基因靶向敲入,广泛应用于基因治疗中。基因组突变主要指锌指核酸酶介导产生DNA断裂,细胞修复DNA断裂时随机引入或删除几个碱基,从而导致单个基因或基因组序列的紊乱,实现基因突变。 Bibikovaetal.(2002)首次利用模块组装法设计了含有3锌指的锌指核酸酶,并在热激蛋白启动子控制下诱导表达锌指核酸酶,结果发现雄性果蝇体细胞的y基因靶位点处发生了突变,同时,突变个体后腹部出现黄色斑点,说明突变破坏了靶基因功能。目前通过锌指核酸酶技术在基因组水平上获得目标靶基因突变的研究已广泛应用于不同的生物,如在黑腹果蝇Drosophilamelanogaster(Meigen)(Beumeretal.,2008)、斑马鱼Daniorerio(Doyonetal.,2008; Mengetal.,2008)、大鼠Rattusnorregicus、小鼠Musmusculus(Cuietal.,2010)、拟南芥Arabidopsisthaliana(L.) Heynh.(Zhangetal.,2010)和家蚕BombyxmoriL.(Takasuetal.,2010)等模式生物中成功实现了靶基因的敲除或定点修饰。Beumeretal.(2006)利用锌指核酸酶作用于果蝇的胚胎,对ry和bw基因进行突变试验,结果表明,2.4%的雄性果蝇发生bw基因突变。在38 ℃的刺激下,89%的雌性和67%的雄性产生ry基因突变,子代中雌性突变率为14.0%,雄性突变率为6.8%。随后,Beumeretal.(2008)通过在果蝇胚胎早期注射锌指核酸酶mRNA,导致靶基因coil和pask的靶向修饰效率达到10%以上,所获得的每个基因敲除的纯合子个体完全丧失了靶基因的表达能力。Takasuetal.(2010)使用ZFNs技术, 在家蚕基因组水平上破坏了BmBLOS2基因, 突变个体表现出油蚕表型,并且这种变异成功遗传到下一代。
2 类转录激活因子式核酸酶(Transcriptionactivator-like effector nucleases,TALENs)技术
Bonasetal.(1989)在植物病原菌黄单胞菌Xanthomonas中分离、克隆得到类转录激活因子(Transcription activator-like effector nucleases,TALE)家族中的第一个成员AvrBs3基因。TALE蛋白类似于真核生物的转录因子,通过识别并结合特异的DNA序列调控生物内源基因的表达,使得宿主生物对外来物的敏感性提高(Boch & Bonas,2010)。周金伟等(2013)对TALEs的结构做了详细明了的综述。C-端、N-端和中间区域组成了TALE的3个主要部分(Boch & Bonas,2010; Bogdanoveetal.,2010),通常地,C-端含有一个核定位信号(Nuclear localization signal,NLS)和转录激活结构域(Activation domain,AD),N-端含有转运结构域(translocation domain,TD),中间区域则是能够和DNA进行特异性结合的结构域。中间的DNA结合结构域是一段由1.5~33.5个TALE单元组成的重复氨基酸序列,每个TALE单元由33~35个氨基酸残基组成(Bochetal.,2010)。TALE蛋白的每个重复单元高度保守,除了第12和13位的氨基酸残基可变以外, 其他的氨基酸残基都是一样的,称为重复可变双残基(Repeat variable di-residue,RVD),第12位氨基酸残基主要起稳定RVD环的作用,第13位氨基酸残基决定TALE蛋白所识别的核苷酸(Dengetal.,2012; Maketal.,2012)。RVD决定了TALE蛋白对DNA序列的特异性识别,不同的RVD能够特异性地识别碱基A、T、C、G中的一种或者多种。目前发现的RVD种类有5种,分别是氨基酸HD特异识别碱基C、氨基酸NI识别碱基A、氨基酸NN识别碱基G或A、氨基酸NG识别碱基T和氨基酸NK识别碱基A、T、C、G中的任一种(周金伟等,2013; Bochetal.,2009; Morbitzeretal.,2010; Moscou & Bogdanove,2009)。
Meckleretal.(2013)发现RVD对识别DNA序列的特异性能力为NG>HD~NN>>NI>NK,此外,不同的RVD识别不同碱基的能力也不一样,如NN对G的亲和性大于A,表明TALE蛋白的重复单元跟DNA碱基之间有很好的一一对应关系。TALENs技术这种根据一个重复单元对应一种碱基,将多个重复单元串联在一起来决定识别序列的特异性,理论上可以人为设计能够识别并结合任何DNA序列的TALEs蛋白。由于FokI需要形成二聚体才能发挥切割作用,因此需要一对TALEN共同作用。2个TALEN识别的DNA位点之间的长度为14~18 bp的DNA序列片段称为spacer(Milleretal.,2011)。2个TALEN结合到各自的DNA序列上,此时,2个TALEN中ForkI核酸内切酶结构域可在spacer处形成二聚体,对特异的DNA进行识别和定点切割,能在特定位点产生DSB作用机制,产生的DSB能够通过非同源末端连接修复(NHEJ)和同源介导修复(HDR)的高度保守的修复方式进行修复(图1B),使TALENs技术在基因组靶向编辑中起作用。TALEN切割位点的特异性由2个TALEN的识别序列共同决定。因此,TALEN切割DNA的特异性高,在基因组编辑方面有广阔的应用前景。
成功构建TALENs是TALE蛋白成功应用于基因组工程学方面的前提。TALENs的构建主要包括3个步骤:
第一,TALENs靶位点的选择:从选定的目的基因序列中找出适合TALE蛋白结合的目标靶点,此步骤是进行TALE蛋白设计的关键。在选择TALENs靶点时,Cermaketal.(2011)、Christianetal.(2011)和 Reyonetal.(2012)均提出了需要遵循的基本原则,如靶位点5′端的第一位碱基不能是T、靶位点的第二位碱基不能是腺嘌呤A等。此外,可借助公开网站帮助预测和设计TALEs靶位点,目前公开网站有https:∥tale-nt.cac.cornell.edu/、http:∥zifit. partners.org/ZiFiT/和http:∥idtale.kaust.edu.sa/等。
第二,TALE蛋白重复序列的构建:制备针对目标靶点的特异TALE蛋白,此步骤是TALENs构建中最复杂和重要的环节。可以使用全序列人工合成构建TALE蛋白重复序列,也可以采用酶切连接法(Restriction enzyme and ligation,REAL)(Lietal.,2012; Sanderetal.,2011)、基于质粒载体或PCR的GG法(Lietal.,2011; Sanjanaetal.,2012; Zhangetal., 2011)、高通量TALENs合成技术FLASH(Fast ligation-based automatable solid-phase high-throughput)(Reyonetal.,2012)和组装法(沈延等,2013; Briggsetal.,2012; Schmid-Burgketal.,2013)等方法进行构建。
第三,TALE蛋白序列与FokI的组装:在TALE蛋白上选择合适的FokI结构域,利用分子生物学方法把TALE和FokI内切酶序列进行组装,就可以构建出完整的能够特异性识别目的基因并进行切割的TALENs序列。在构建TALENs时,要考虑重复序列跟FokI内切酶之间的距离(Milleretal.,2011),以及TALE蛋白N-端重复序列的识别亲和力比C-端强等(Meckleretal.,2013)。
自从Kayetal.(2007)发现TALE能够进入细胞核识别upa20基因的启动子并调控upa20基因的表达,进而能控制细胞的大小, 以及Bochetal.(2009)破译了TALE的一个重复单元一个碱基的“密码”以来, TALENs技术已经得到广泛的应用。Maetal.(2012)使用TALENs技术,破坏了家蚕BmBLOS2基因,其突变个体表现出油蚕表型;同时还使用2组TALENs在2个位点切割,造成了靶标位点中间片段的删除。Watanabeetal.(2012)同时使用ZFNs技术和TALENs技术,针对蟋蟀中的Lac2基因,成功实施了定点敲除。Liuetal.(2012)使用TALENs技术敲除了果蝇yellow基因。
3 CRISPR/Cas技术
Ishinoetal.(1987)在K12大肠杆菌的碱性磷酸酶基因附近发现串联间隔重复序列,随后发现这种间隔重复序列广泛存在于细菌和古细菌的基因组中。这种间隔重复序列被正式命名为成簇的、规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR),CRISPR结构非常稳定,25~50 bp的重复序列被间区序列(Spacers)间隔(Jansenetal.,2002)。研究发现,Cas系统(CRISPR-associated sequences system,CASs)在原核生物中具有某种性免疫功能(Barrangouetal.,2007; Bolotinetal.,2005)。随后的研究表明,Cas系统是一种原核生物特有的针对噬菌体、质粒等外源性遗传物质的免疫保护系统,通过序列特异的RNA介导,切割降解外源性DNA,从而抵抗外源DNA入侵(Terns & Terns,2011; Wiedenheftetal.,2012)。目前使用的CRISPR/Cas系统识别序列为23 bp,靶向序列20 bp,其识别位点最末3位NGG序列被称作PAM(Protospacer adjacent motif)序列,该序列对于DNA切割非常重要(Jiangetal.,2013),也是CRISPR/Cas系统能够区分自身DNA和外源DNA的主要原因之一(图1C)。
CRISPR/Cas系统通常分为Ⅰ型、Ⅱ型和Ⅲ型3种类型,每种CRISPR/Cas系统均包含具有核酸酶活性的Cas基因和决定切割位点特异性的2个非编码RNA:crRNA(CRISPR RNA)和反式激活crRNA(trans-activating crRNA)(Barrangouetal.,2007; Brounsetal.,2008; Makarovaetal.,2011; Marraffinietal.,2008)。Ⅰ型和Ⅲ型CRISPR/Cas系统需要多种蛋白形成复合物才能行使功能,而Ⅱ型CRISPR/Cas系统组成简单,仅需要Cas9核酸酶一种蛋白参与即可发挥功能(Makarovaetal.,2011),目前已被改造成为基因组靶向编辑的工具。发挥作用的Cas9核酸酶复合体由Cas9蛋白、RNA-crRNA前体(RNA pre-crRNA) 和tracrRNA3个组分组成(Congetal.,2013),即在应用CRISPR/Cas系统进行基因组编辑时,需要表达这3个组分。进一步的研究发现,根据crRNA和tracrRNA的结构特性,将crRNA和tracrRNA构建成一条嵌合的向导RNA(Single guide RNA,sgRNA),sgRNA具备crRNA:tracrRNA复合体的功能,从而将CRISPR/Cas系统简化成Cas9蛋白和sgRNA2个组分(Jineketal.,2012; Malietal.,2013),且sgRNA的构建具有快速、简便和成本低等优点。Cas9蛋白与sgRNA的结合能够实现在特异位点处切割DNA,Cas9蛋白与不同的sgRNA结合能够针对不同的位点进行编辑(图1C)。因此,在对多位点进行编辑时,CRISPR/Cas系统具有更明显的优势。
虽然CRISPR/Cas技术的应用还处于初级阶段,但该技术构建容易、成本低、适宜多位点编辑、具有进行大规模筛选试验的潜力,必然具有广阔的发展前景。目前研究人员已成功利用CRISPR/Cas9系统(Cas9蛋白和sgRNA)对果蝇进行靶标突变,不同之处是如何使Cas9蛋白和sgRNA共同起作用(Bassett & Liu,2014)。Gratzetal.(2013)使用CRISPR/Cas技术,通过向果蝇胚胎联合注射分别表达Cas9基因和sgRNA的质粒,定点敲除了yellow基因并能传递到后代,但该CRISPR/Cas系统仅有5.9%的低突变效率;此外,该系统还能对果蝇进行双位点编辑,从而导致片段删除。Bassettetal.(2013)和Yuetal.(2013)分别将在体外转录合成的Cas9 mRNA和sgRNA注射入果蝇早期胚胎中,分别获得88%和80%的高突变率。由上可见,注射质粒和mRNA获得的突变率存在巨大差别,可能与Cas9和sgRNA表达水平,或生殖细胞在胚胎中的表达时间相关。Kondo & Ueda(2013)利用CRISPR/Cas体系进行果蝇转基因研究,产生分别表达Cas9和sgRNA的2种转基因果蝇,当这2种果蝇杂交时即可获得高效率的突变体,90%以上的果蝇能产生突变后代;进行双位点编辑时可导致长达1.6 kb的片段删除。
利用CRISPR/Cas技术进行转基因果蝇研究,虽然突变率高、重复性好,但分别获得sgRNA转基因和Cas9转基因产品、以及在突变后代中去除Cas9和sgRNA转基因需要耗费大量时间。Seboetal.(2013)将编码sgRNA的质粒注射到表达Cas9的转基因果蝇品系里,发现注射编码sgRNA质粒的果蝇G1后代中获得高的突变率,但大部分不育。Renetal.(2013)注射编码sgRNA质粒,并使用nanos启动子驱使Cas9表达,获得较高比例的可育转基因个体,并在G1后代中产生高的突变率。除了注射表达sgRNA的质粒外,还可直接将体外合成转录本sgRNA注射到胚胎里、进而获得表达Cas9的果蝇品系。但是这种技术的效率如何并未得知,其构建体系并未建立(Bassett & Liu,2014)。Bassettetal. (2014)分别使用Actin5c和U6启动子驱使Cas9和sgRNA的表达,成功地在果蝇细胞系中实现Cas9载体的表达,结果发现高于80%的细胞产生了高效率的突变。
4 总结与展望
随着高通量测序技术的飞速发展,越来越多的物种基因组被测序,因此解读基因组功能显得日益重要。ZFNs技术、TALENs技术和CRISPR/Cas技术各具不同的优势和局限性(王跃强等,2013; 张智辉等,2013):
(1)ZFNs技术的出现使得基因打靶效率大大提高,同时可针对某些特定的序列设计ZFn,减少了随机性进而实现靶基因的修饰。但由于ZFn的识别结构域中存在上下文依赖效应,所以目前尚无法实现对任意一段序列均可设计出满足要求的ZFn,也无法实现在每个基因或其他功能性染色体区段都能够顺利找到适合的ZFn作用位点。由于ZFNs技术对于DNA序列的识别特异性较低,因此,如何构建高特异性的ZFn是目前存在的较大技术难题。此外,由于ZFn的脱靶切割容易引起细胞毒性,使得其在基因治疗领域的应用出现一定的局限性。
(2)相比ZFns技术,TALENs技术因为使用了TALE分子作为人工核酸酶的识别结构域,因此具有较高的特异性;但由于TALE蛋白与DNA 碱基一一对应,且对碱基的识别只由2 个氨基酸残基决定,因此设计较简单。不足的是TALEN构建过程中,TALE分子的模块组装和筛选过程比较繁杂,需要大量的测序工作,对于普通实验室的可操作性较低,而商业化公司构建也需要花费上千美元,使用成本较高。
(3)CRISPR/Cas技术具有精确性高、系统构建简单和使用成本低、能同时对同一细胞多个位点进行切割等优势。CRISPR/Cas9对靶序列的识别是RNA与DNA的碱基配对过程,只要有一个碱基无法配对,就不会实现Cas9对DNA的切割,降低了脱靶切割的几率,减少了细胞毒性。此外,CRISPR/Cas9的构建仅仅需要设计与靶序列互补的RNA即可,过程更为简单和廉价,普通的实验室也可自行完成构建,大大提高了基因操作的效率及简便性。并且,CRISPR/Cas9系统是由RNA介导的DNA切割,若在RNA水平上进行分子操作,可实现精确且瞬时的切割,较易调节切割时间。
但CRISPR/Cas9系统在真核基因组编辑中也存在着一些不足,如,因为Cas9蛋白对于目标序列的切割依靠crRNA序列和前间区序列邻近基序(PAM),若目标序列周围不存在PAM或者无法严格配对,则Cas9蛋白不能行使核酸酶的功能,因此CRISPR/Cas9技术不能对任意序列进行切割。此外,由于CRISPR/Cas9系统所靶向的序列仅需十余个碱基对精确配对,可能降低CRISPR/Cas9系统切割的特异性。
目前这几种基因组编辑技术在昆虫遗传转化研究中的运用还处于初级阶段,但其在基因操作方面已表现出较大的成功,因此可以预测,今后在昆虫遗传转化方面必然具有巨大的潜力和广阔的应用前景,将在昆虫遗传转化技术中产生深远影响,也将在实现害虫遗传控制中发挥重要作用。如目前中国农业科学院在农业害虫桔小实蝇遗传控制方面的研究已取得了较好的前期研究基础,如已经克隆了胚胎分化期特异表达基因sryα及母体效应基因nanos和vasa等胚胎早期高表达基因,并分离了它们的上游潜在启动区序列;克隆了促细胞凋亡基因hid的cDNA全长;克隆了性别决定基因transformer的cDNA全长并得到了其内含子序列,通过分析获得了其雌性特异剪切元件;并已利用以上分子元件构建了双元件胚胎致死品系所需的胚胎早期驱动载体和致死效应载体。在这些基础之上,目前正着手用考虑用 CRISPR/Cas来实现多个位点定点敲除,以满足不同需求的桔小实蝇转化品系。
参考文献
沈延, 黄鹏, 张博. 2013. TALEN 构建与斑马鱼基因组定点突变的实验方法与流程. 遗传, 35(4): 533-544.
王跃强, 郑贵斌, 谭安江, 黄勇平. 2013. 基因组编辑技术及其在昆虫研究中的应用. 中国科学: 生命科学, 43(12): 1105-1111.
张智辉, 董少忠, 寸韡. 2013. 基因组定点编辑技术的研究进展. 生命科学, 25(7): 735-742.
周金伟, 王灵慧, 申义君, 余树民, 曹随忠. 2013. 类转录激活因子效应物核酸酶(TALENs)的构建及其在基因组定点修饰中的应用. 中国细胞生物学学报, 35(11): 1-9.
Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero D A and Horvath P. 2007. CRISPR provides acquired resistance against viruses in prokaryotes.Science, 315: 1709-1712.
Bassett A R and Liu J L. 2014. CRISPR/Cas9 and genome editing inDrosophila.JournalofGeneticsandGenomics, 41: 7-19.
Bassett A R, Tibbit C, Ponting C P and Liu J L. 2013. Highly efficient targeted mutagenesis ofDrosophilawith the CRISPR/Cas9 system.CellReport, 4: 220-228.
Bassett A R, Tibbit C, Ponting C P and Liu J L. 2014. Mutagenesis and homologous recombination inDrosophilacell lines using CRISPR/Cas9.BiologyOpen, 3: 42-49.
Beumer K, Bhattacharyya G, Bibikova M, Trautman J K and Carroll D. 2006. Efficient gene targeting inDrosophilawith zinc-finger nucleases.Genetics, 172: 2391-2403.
Beumer K J, Trautman J K, Bozas A, Liu J L, Rutter J, Gall J G and Carroll D. 2008. Efficient gene targeting inDrosophilaby direct embryo injection with zinc-finger nucleases.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 105: 19821-19826.
Bibikova M, Beumer K, Trautman J K and Carroll D. 2003. Enhancing gene targeting with designed zinc finger nucleases.Science, 300: 764.
Bibikova M, Golic M, Golic K G and Carroll D. 2002. Targeted chromosomal cleavage and mutagenesis inDrosophilausing zinc-finger nucleases.Genetics, 161: 1169-1175.
Bitinaite J, Wah D A, Aggarwal A K and Schildkraut I. 1998. FokI dimerization is required for DNA cleavage.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 95: 10570-10575.
Boch J and Bonas U. 2010.XanthomonasAvrBs3 family-type Ⅲ effectors: discovery and function.AnnualReviewofPhytopathology, 48: 419-436.
Boch J, Scholze H, Schornack S, Landgraf A, Hahn S, Kay S, Lahaye T, Nickstadt A and Bonas U. 2009. Breaking the code of DNA binding specificity of TAL-type Ⅲ effectors.Science, 326: 1509-1512.
Bogdanove A J, Schornack S and Lahaye T. 2010. TAL effectors: finding plant genes for disease and defense.CurrentOpinioninPlantBiology, 13: 394-401.
Bolotin A, Quinquis B, Sorokin A and Ehrlich S. 2005. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin.Microbiology, 151: 2551-2561.
Bonas U, Stall R E and Staskawicz B. 1989. Genetic and structural characterization of the avirulence geneavrBs3 fromXanthomonascampestrispv.vesicatoria.MolecularandGeneralGenetics, 218: 127-36.
Briggs A W, Rios X, Chari R, Yang L, Zhang F, Mali P and Church G. 2012. Iterative capped assembly: rapid and scalable synthesis of repeat-module DNA such as TAL effectors from individual monomers.NucleicAcidsResearch, 40: e117.
Brouns S J, Jore M M, Lundgren M, Westra E R, Slijkhuis R J, Snijders A P, Dickman M J, Makarova K S, Koonin E V and van der Oost J. 2008. Small CRISPR RNAs guide antiviral defense in prokaryotes.Science, 321: 960-964.
Brown R S. 2005. Zinc finger proteins: getting a grip on RNA.CurrentOpinioninStructuralBiology, 15: 94-98.
Carroll D, Morton J J, Beumer K J and Seqal D J. 2006. Design, construction and in vitro testing of zinc finger nucleases.NatureProtocols, 1: 1329-1341.
Cermak T, Doyle E L, Christian M, Wang L, Zhang Y, Schmidt C, Baller J A, Somia N V, Bogdanove A J and Voytas D F. 2011. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting.NucleicAcidsResearch, 39: 1-11.
Christian M, Cermak T, Doyle EL, Schmidt C, Zhang F, Hummel A, Bogdanove A J and Voytas D F. 2011. Targeting DNA double-strand breaks with TAL effector nucleases.Genetics, 186: 757-761.
Cong L, Ran F A, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA and Zhang F. 2013. Multiplex genome engineering using CRISPR/Cas systems.Science, 339: 819-823.
Cui X, Ji D, Fisher D A, Wu Y, Briner D M and Weinstein E J. 2010. Targeted integration in rat and mouse embryos with zinc-finger nucleases.NatureBiotechnology, 29: 64-71.
Daimon T, Kiuchi T and Takasu Y. 2014. Recent progress in genome engineering techniques in the silkworm,Bombyxmori.DevelopmentGrowth&Differentiation, 56: 14-25.
Deng D, Yan C, Pan X, Mahfouz M, Wang J, Zhu JK, Shi Y and Yan N. 2012. Structural basis for sequence-specific recognition of DNA by TAL effectors.Science, 335: 720-723.
Doyon Y, McCammon J M, Miller J C, Faraji F, Ngo C, Katibah G E, Amora R, Hocking T D, Zhang L, Rebar E J, Gregory P D, Urnov F D and Amacher S L. 2008. Heritable targeted gene disruption in zebra fish using designed zinc finger nucleases.NatureBiotechnology, 26: 702-710.
Gamsjaeger R, Liew C K, Loughlin F E, Crossley M and Mackay J P. 2007. Sticky fingers: zinc-fingers as protein-recognition motifs.TrendsinBiochemicalSciences, 32: 63-70.
Gratz S J, Cummings A M, Nguyen J N, Hamm D C, Donohue L K, Harrison M M, Wildonger J and O′Connor-Giles K M. 2013. Genome engineering ofDrosophilawith the CRISPR RNA-guided Cas9 nuclease.Genetics, 194: 1029-1035.
Hall T M T. 2005. Multiple modes of RNA recognition by zinc finger proteins.CurrentOpinioninStructuralBiology, 15: 367-373.
Handel E M and Cathomen T. 2011. Zinc-finger nuclease based genome surgery: it′s all about specificity.CurrentGeneTherapy, 11: 28-37.
Hsu P D, Lander E S and Zhang F. 2014. Development and applications of CRISPR-cas9 for genome engineering.Cell, 157: 1262-1278.
Ishino Y, Shinagawa H, Makino K, Amemura M and Nakata A. 1987. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion inEscherichiacoli, and identification of the gene product.JournalofBacteriology, 169: 5429- 5433.
Jansen R, Embden J, Gaastra W and Schouls L M. 2002. Identification of genes that are associated with DNA repeats in prokaryotes.MolecularMicrobiology, 43: 1565-1575.
Jiang W, Bikard D, Cox D, Zhang F and Marraffini L A. 2013. RNA-guided editing of bacterial genomes using CRISPR-Cas systems.NatureBiotechnology, 31: 233-239.
Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna J A and Charpentier E. 2012. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity.Science, 337: 816-821.
Kay S, Hahn S, Marois E, Hause G and Bonas U. 2007. A bacterial effector acts as a plant transcription factor and induces a cell size regulator.Science, 318: 648-651.
Kim Y G, Cha J and Chandrasegaran S. 1996. Hybrid restriction enzymes: zinc finger fusions to FokⅠcleavage domain.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 93: 1156-1160.
Klug A. 1999. Zinc finger peptides for the regulation of gene expression.JournalofMolecularBiology, 293: 215-218.
Klug A. 2010. The discovery of zinc fingers and their applications in gene regulation and genome manipulation.AnnualReviewofBiochemistry, 79: 213-231.
Kondo S and Ueda R. 2013. Highly improved gene targeting by germlinespecific Cas9 expression inDrosophila.Genetics, 195: 715-721.
Li T, Huang S, Zhao X, Wright D A, Carpenter S, Spalding M H, Weeks D P and Yang B. 2011. Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes.NucleicAcidsResearch, 39: 6315-6325.
Li T, Liu B, Spalding M H, Weeks D P and Yang B. 2012. High-efficiency TALEN-based gene editing produces disease-resistant rice.NatureBiotechnology, 30: 390-392.
Liu J C, Li Z, Yu P, Huang H, Wei C, Zhu N, Shen Y, Chen Y, Zhang B, Deng W M and Jiao R. 2012. Efficient and specific modifications of theDrosophilagenome by means of an easy TALEN strategy.JournalofGeneticsandGenomics, 39: 209-215.
Liu Y, Ma S, Wang X, Chang J, Gao J, Shi R, Zhang J, Lu W, Liu Y, Zhao P and Xia Q Y. 2014. Highly efficient multiplex targeted mutagenesis and genomic structure variation inBombyxmoricells using CRISPR/Cas9.InsectBiochemistryandMolecularBiology, 49: 35-42.
Ma S, Chang J, Wang X, Liu Y, Zhang J, Lu W, Gao J, Shi R, Zhao P and Xia Q. 2014. CRISPR/Cas9 mediated multiplex genome editing and heritable mutagenesis of BmKu70 inBombyxmori.ScientificReport, 4: 4489.
Ma S, Zhang S, Wang F, Liu Y, Xu H, Liu C, Lin Y, Zhao P and Xia Q. 2012. Highly efficient and specific genome editing in silkworm using custom TALENs.PLoSONE, 7: e45035.
Mak A N, Bradley P, Cernadas R A, Bogdanove A J and Stoddard B L. 2012. The crystal structure of TAL effector PthXo1 bound to its DNA target.Science, 335: 716-719.
Makarova K S, Haft D H, Barrangou R, Brouns S J, Charpentier E, Horvath P, Moineau S, Mojica F J, Wolf Y I, Yakunin A F, van der Oost J and Koonin E V. 2011. Evolution and classification of the CRISPR-Cas systems.NatureReviewsMicrobiology, 9: 467-477.
Mali P, Yang L, Esvelt K M, Aach J, Guell M, DiCarlo J E, Norville J E and Church G M 2013. RNA-guided human genome engineering via Cas9.Science, 339: 823-826.
Marraffini L A and Sontheimer E J. 2008. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA.Science, 322: 1843-1845.
Meckler J F, Bhakta M S, Kim M S, Ovadia R, Habrian C H, Zykovich A, Yu A, Lockwood S H, Morbitzer R, Elsäesser J, Lahaye T, Segal D J and Baldwin E P. 2013. Quantitative analysis of TALE-DNA interactions suggests polarity effects.NucleicAcidsResearch, 41: 4118-4128.
Meng X, Noyes M B, Zhu L J, Lawson N D and Wolfe S A. 2008. Targeted gene inactivation in zebrafish using engineered zinc-finger nucleases.NatureBiotechnology, 26: 695-701.
Miller J, McLachlan A D and Klug A.1985. Repetitive zinc-binding domains in the protein transcription factor IIIA fromXenopusoocytes.EMBOJournal, 4: 1609.
Miller J C, Tan S, Qiao G, Barlow K A, Wang J, Xia D F, Meng X, Paschon D E, Leung E, Hinkley S J, Dulay G P, Hua K L, Ankoudinova I, Cost G J, Urnov F D, Zhang H S, Holmes M C, Zhang L, Gregory P D and Rebar E J. 2011. A TALE nuclease architecture for effcient genome editing.NatureBiotechnology, 29: 143-148.
Morbitzer R, Römer P, Boch J and Lahaye T. 2010. Regulation of selected genome loci using denovo-engineered transcription activator-like effector (TALE)-type transcription factors.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 107: 21617-21622.
Moscou M J and Bogdanove A J. 2009. A simple cipher governs DNA recognition by TAL effectors.Science, 326: 1501.
Ren X, Sun J, Housden B E, Hu Y, Roesel C, Lin S, Liu L P, Yang Z, Mao D, Sun L, Wu Q, Ji J Y, Xi J, Mohr S E, Xu J, Perrimon N and Ni J Q. 2013. Optimized gene editing technology forDrosophilamelanogaster using germ line-specific Cas9.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 110: 19012-19017.
Reyon D, Tsai S Q, Khayter C, Foden J A, Sander J D and Joung J K. 2012. FLASH assembly of TALENs for high-throughput genome editing.NatureBiotechnology, 30: 460-465.
Sajwan S, Takasu Y, Tamura T, Uchino K, Sezutsu H and Zurovec M. 2013. Efficient disruption of endogenousBombyxgene by TAL effector nucleases.InsectBiochemistryandMolecularBiology, 43: 17-23.
Sander J D, Cade L, Khayter C, Reyon D, Peterson R T, Joung J K and Yeh J R. 2011. Targeted gene disruption in somatic zebrafsh cells using engineered TALENs.NatureBiotechnology, 29: 697-698.
Sanjana N E, Cong L, Zhou Y, Cunniff M M, Feng G and Zhang F. 2012. A transcription activator-like effector tool box for genome engineering.NatureProtocols, 7: 171-192.
Schmid-Burgk J L, Schmidt T, Kaiser V, Honing K and Hornung V. 2013. A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes.NatureBiotechnology, 31: 76-81.
Sebo Z L, Lee H B, Peng Y and Guo Y. 2013. A simplified and efficient germline-specific CRISPR/Cas9 system forDrosophilagenomic engineering.Fly(Austin), 8: 52-57.
Smith J, Grizot S, Arnould S, Duclert A, Epinat J C, Chames P, Prieto J, Redondo P, Blanco F J, Bravo J, Montoya G, Pques F and Duchateau P. 2006. A combinatorial approach to create artificial homing endonucleases cleaving chosen sequences.NucleicAcidsResearch, 34: e149.
Takasu Y, Kobayashi I, Beumer K, Uchino K, Sezutsu H, Sajwan S, Carroll D, Tamura T and Zurovec M. 2010. Targeted mutagenesis in the silkwormBombyxmoriusing zinc finger nuclease mRNA injection.InsectBiochemistryandMolecularBiology, 40: 759-765.
Takasu Y, Sajwan S, Daimon T, Osanai-Futahashi M, Uchino K, Sezutsu H, Tamura T and Zurovec M. 2013. Efficient TALEN construction forBombyxgene targeting.PLoSONE, 18: e73458.
Terns M P and Terns R M. 2011. CRISPR-based adaptive immune systems.CurrentOpinioninMicrobiology, 14: 321-327.
Wang Y, Li Z, Xu J, Zeng B, Ling L, You L, Chen Y, Huang Y and Tan A. 2013. The CRISPR/Cas system mediates efficient genome engineering inBombyxmori.CellResearch, 23: 1414-1416.
Watanabe T, Ochiai H, Sakuma T, Horch HW, Hamaguchi N, Nakamura T, Bando T, Ohuchi H, Yamamoto T, Noji S and Mito T. 2012. Non-transgenic genome modifications in a hemimetabolous insect using zinc-finger and TAL effector nucleases.NatureCommunication, 3: 1017.
Wei W, Xin H, Roy B, Dai J, Miao Y and Gao G. 2014. Heritable genome editing with CRISPR/Cas9 in the silkworm,Bombyxmori.PLoSONE, 9: e101210.
Wiedenheft B, Sternberg S H and Doudna J A. 2012. RNA-guided genetic silencing systems in bacteria and archaea.Nature, 482: 331-338.
Wolfe S A, Nekludova L and Pabo C O. 2000. DNA recognition by Cys2His2 zinc finger proteins.AnnualReviewofBiophysicsandBiomolecularStructure, 29: 183-212.
Yu Z, Ren M, Wang Z, Zhang B, Rong Y S, Jiao R and Gao G. 2013. Highly efficient genome modifications mediated by CRISPR/Cas9 inDrosophila.Genetics, 195: 289-291.
Zhang F, Cong L, Lodato S, Kosuri S, Church G M and Arlotta P. 2011. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription.NatureBiotechnology, 29: 149-153.
Zhang F, Maeder M L, Unger-Wallace E, Hoshaw J P, Reyon D, Christian M, Li X, Pierick C J, Dobbs D, Peterson T, Joung J K and Voytas D F. 2010. High frequency targeted mutagenesis inArabidopsisthalianausing zinc finger nucleases.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 107: 12028-12033.
(责任编辑:郭莹)
+同等贡献作者(The two authors contributed equally to this work)。
1StateKeyLaboratoryforBiologyofPlantDiseasesandInsectPests,InstituteofPlantProtection,ChineseAcademyofAgricultural
Sciences,Beijing100193,China;2DepartmentofEntomology,NorthCarolinaStateUniversity,CampusBox7613,Raleigh,
NC27695-7613,USA;3GeneticEngineeringandSocietyCenterandW.M.KeckCenterforBehavioralBiology,
NorthCarolinaStateUniversity,Raleigh,NC27695-7613,USA;4CenterforManagementofInvasive
AlienSpecies,MinistryofAgriculture,Beijing100193,China
Abstract:The technologies of zinc finger nucleases (ZFNs), transcription activator-like effector nucleases (TALENs) and CRISPR/Cas (clustered regularly interspaced short palindromic repeats, CRISPR; CRISPR associated protein, Cas) are three major genome editing techniques that have being developed in recent years. The basic principle is to make DNA double-strand break (DSB) damaged in the genome specific sites and to activate the body′s own DNA damage repair mechanism, then causing all sorts of variation in the process. The genome editing techniques have important research prospect in the research of gene function and gene repair. They are also expected to become the main strategy of insect genetic transformation since they have many merits. For example, CRISPR/Cas can be used to edit multiple specific loci in the genome. In this paper, the basic principles of the ZFNs, activation of transcription factor type nucleic acid enzymes and CRISPR/Cas technologies and their applications in insects are briefly introduced. The information provides some references for the use of genome editing techniques in insect genetic transformation.
Key words:CRISPR/Cas technology; genetic transformation; genome editing; transcription activator-like effector nucleases; zinc finger nucleases
通讯作者*(Author for correspondence), E-mail: wanfanghao@caas.cn
作者简介:王玉生, 男, 硕士研究生。 研究方向: 入侵昆虫遗传控制。 E-mail: yushengwang01@163.com; 蔡玉音, 女, 硕士研究生。 研究方向: 昆虫生物化学与分子生物学。 E-mail: caiyuyin1990@163.com
基金项目:环保公益性行业科研专项(201409061); 中德合作科研项目[PPP项目,留金欧(2012)6014和留金欧(2014)6013)]; 农业部2014年农作物病虫鼠害疫情监测与防治(外来入侵生物防治)项目; 科技导报社博士生创新研究资助计划(kjdb201001-3)
收稿日期(Received): 2015-01-10接受日期(Accepted): 2015-02-07
DOI:10.3969/j.issn.2095-1787.2015.02.006
