分子生物学技术在蚧虫分类鉴定中的应用
2015-12-16魏亦寒郑斯竹詹国辉
魏亦寒,郑斯竹,蔡 平,詹国辉,高 渊
(1.苏州大学金螳螂建筑与城市环境学院,江苏苏州,215123;2.苏州出入境检验检疫局,江苏苏州, 215000)
蚧虫又称介壳虫,属于昆虫纲Insecta 半翅目Hemiptera 蚧总科Coccoidea。据ScaleNet 统计,除化石昆虫14个科之外,全世界共记录48 科1050 属7500 余种,我国已知116 科249 属830种,是一类分布广、寄主多的植食性昆虫,除少部分为资源昆虫外,多为农林业中发生普遍且为害严重的害虫(汪永庆,2001;Kozar,2008;武三安,2009;Ben-Dov et al.,2012;罗梅等,2012)。该类群的成虫和若虫主要在寄主植物的幼嫩部位取食危害,导致树势衰弱;亦可传播病原微生物导致植物病害大面积的发生;另外虫体排出的蜜露常诱致煤烟病的产生,影响光合作用,对松林资源、自然景观等的生态影响巨大。
长期以来,基于介壳虫形态特征的分类研究具有局限性:首先,介壳虫体形较小或微小,有些种类鲜见雄虫,可供比较的形态学特征有限;其次,形态分类特征不稳定,如有些粉蚧的个体大小、刚毛的长短、透明孔的有无等都可能受外部环境的影响(齐晓丰,2008);再者,传统形态分类通常对专业知识和经验要求较高,只有分类专家或者经过专门培训的人员才能开展此项工作;此外,口岸检疫部门在实际工作中,截获的入境蚧虫不一定为成虫,有时是若虫,甚至是不完整的虫体残体、蜕皮等(Kondo et al.,2008;Utsugi,2011),无法通过形态特征进行快速有效地鉴定。
近年来,昆虫分子生物学迅速发展起来,在分子鉴定和系统演化两个方面的应用较为广泛,通过研究昆虫核酸分子结构探求各类群之间的亲缘关系和进化关系(唐克轩等,2002;Robertson,2003;晏慧君等,2006)。利用分子生物学技术,可以有效地揭示自然种群中遗传结构、基因流动和选择作用等问题,为昆虫的分类鉴定提供新的技术和方法(Downie et al.,2004;Frézal and Leblois,2008;刘明辉等,2009)。本文针对分子生物学技术在蚧虫系统发育以及种类鉴定的应用予以综述,并对其存在的问题进行讨论,以期为蚧虫类昆虫的分类鉴定提供帮助。
1 蚧虫分子生物学研究基础
目前蚧虫类昆虫分子生物学的研究,以粉蚧科Pseudococcidae 和盾蚧科Diaspididae 居多,研究内容主要是对整个基因组DNA(genome DNA),mtDNA(mitochondrial DNA,线粒体DNA)、rDNA(ribosomal DNA,核糖体 DNA)及satDNA(satellite DNA,卫星DNA)等特定部分DNA 的研究(Aljanabi and Martinez,1997;Ashfaq et al.,2010;Hosseini and Hajizadeh,2011),其中以rDNA 和mtDNA 应用居多。
核糖体DNA 是核DNA 基因组中度重复簇的一个组分,18S 基因是rDNA 中保守性最好的部分,ETS、ITS 和IGS 等非编码区有高度的多态性,这些特征使rDNA 常用来鉴别蚧虫种下至高级分类阶元的分析(Park et al.,2010),已有研究证明,18S、28S、EF-lα 基因是盾蚧亚科下属级分类单元分子系统发育的有效基因标记之一,适于远缘物种关系,也可用于推断早期的进化事件(Annstrong et al.,1997;Cook et al.,2002;钟涛,2002;Downie et al.,2004;魏久锋等,2009;何衍彪,2012),5.8S 基因包含的信息量最少,目前极少应用。
线粒体DNA 是共价闭合的环状双链DNA 分子,广泛存在于昆虫体内富含线粒体的飞行肌和卵等组织细胞中,大小一般为15.4-16.3 kb,以高拷贝数目存在于线粒体内,因其属于细胞器基因组,缺乏细胞核基因组中的保护机制,因而变异较为明显,能够反映较短时间内的进化事件,被广泛应用于蚧虫种间及种下的鉴别。蚧虫分子研究最多的线粒体基因有12S rRNA、16S rRNA、COI(cytochrome coxidase subunit I,线粒体细胞色素C 氧化酶亚基I)和COII,而COIII、cytb(Cytochrome b)、ND4(NADHdehydroge-nase4)和ND 5 研究报道较少,ND1 和ND 2 则甚少应用(赵鸿等,2004)。一般情况下,12S rRNA 和16S rRNA 比较适合于研究属间、不同种团以及分化时间较早的系统关系,COI、COII、ND1 和ND2则经常用于分析亲缘关系密切的种、亚种及地理种群之间的系统关系。
截至2014年12月29 日,NCBI 上共记载了蚧总科昆虫核苷酸序列286744 条,主要集中于18S基因、28S 基因、COI 基因和COII 基因,其中18S基因763 条,28 S 基因2168 条,COI 基因1810 条,COII 基因266 条。
2 分子生物学技术在蚧虫分类鉴定中的应用
近年来迅速发展起来了一系列分子生物学方法,如RFLP、RAPD、SSR、ISSR 和DNA 条形码技术等,并很快被应用于昆虫的物种鉴定和系统进化等的研究中,主要解决蚧虫分类系统存在的问题,集中于其功能基因、亲本起源和协同进化等方面的研究。
2.1 各种分子生物学技术的应用
2.1.1 限制性片段长度多态性技术
RFLP(Restriction Fragment Length Polymorphism,限制性内切酶切片长度多态性)技术是基于生物在长期进化过程中因突变、重组等原因引起核苷酸发生替换、插入或缺失等,使同源生物限制性内切酶识别位点发生变化,用限制性内切酶处理不同生物个体的DNA,依据所产生的大分子片段的长度和数目差异,反映限制性内切酶的切割位点在DNA 分子上的分布,计算DNA 间的遗传距离,据此来判定个体间、群体间或种间的差异(闫华超等,2006;黄映萍,2010)。鉴于RFLP 的来源于自然变异,稳定性高,且特定的切割位点使其具有较高的可靠性,日前在蝗虫、线虫、蚊类、蚁类、棉铃虫等昆虫中应用较为广泛,而且多选择mtDNA,而蚧虫研究中虽有涉及,但多是结合PCR 技术联合分析,以确定其种类所属。
针对蚧虫属、族、种内不易确定分类地位的物种,Chiu 等(2004)运用RFLP 技术对苏铁白轮盾蚧Aulacaspis yasumatsui、月橘白轮盾蚧A.murrayae 和樟白轮盾蚧A.yabunikkei 的rDNA 的18S、ITS 1 及部分的5.8 S 区域扩增,苏铁白轮盾蚧和月橘白轮盾蚧均产生500 bp 的DNA 片段,而樟白轮盾蚧则产生约450 bp 的DNA 片段;之后对扩增出的ITS1 区域进行限制性内切酶切割,结果以内切酶TaqI 的酶切图谱鉴别效果最佳,因此rDNA/PCR-RFLP 技术可用于区别这3种盾蚧,白轮盾蚧属Aulacaspis spp.昆虫为广西、台湾等地的重要入侵种,在苏铁Cycansrevoluta Thunb.植物上危害日趋严重,该研究为检疫口岸蚧虫种级分类阶元的快速鉴定提供了技术支持。Rung 等(2009)利用RFLP-PCR 技术成功区分大洋臀纹粉蚧Planococcus minor 和柑橘粉蚧P.citri,并能将其与来自夏威夷的柑橘粉蚧种群相区分,研究表明RFLP 技术能够确定蚧虫地理种间的亲缘关系,为蚧虫种属特异性鉴定提供有效手段,是蚧虫分类和进化可靠的证据。Burban 等(1999)用PCR-RFLP-SSCP 技术研究日本松干蚧Matsucoccns matsumurae mtDNA 的多态性,并将其与已有的寄主树种的遗传信息相比较,结果出现三条不重叠的条带,由此指出日本松干蚧的种群变异与寄主植物的遗传结构有关,当地种大多表现为协同进化,而其他的则因为种内寄主转换而有所不同,这可能与人为设定树种有关,此次研究为构建蚧虫基因组连锁图谱、蚧虫基因变异影响因素的奠定了基础,尤其对蚧虫属级分类阶元的界定进行了补充。RFLP 技术与SSCP、PCR 技术联合的发展方向,对RFLP 技术的不足之处是一种技术层次上的弥补,目前国内外单独利用RFLP 技术对目标昆虫开展的研究已经不能满足需求,若能借鉴植物病原菌和同翅目其他昆虫如蚜虫、飞虱等的鉴定技术,并以随后兴起的AFLP 技术(Amplified Fragment Length Polymorphism,扩增片段长度多态性)为参考,做出改进,通过Southern 杂交与放射性显影技术相结合,检测与探针DNA 有同源性的片段(黄可辉等,2005;李献梅等,2009;黄映萍,2010),则更能拓宽该技术在蚧虫分子研究方面的应用范围,突显其优势。
2.1.2 随机扩增多态性DNA 技术
RAPD(Random Amplified Polymorphism DNA,随机扩增多态性DNA)技术是1990年Williams 提出的DNA 分子多态检测技术,其原理是利用一系列随机排列的碱基组成的寡聚核苷酸(通常为9-10个碱基)单链作为引物,对所研究的基因组DNA 进行PCR 扩增,扩增产物通过聚丙烯酸胺或琼脂糖凝胶电泳分离,EB 显色或放射性自显影来检验DNA 片段的多态性,借此反映基因组相应区域 的 DNA 的多态(Williams and Kubelik,1990)。此技术最早应用在蚜虫的寄生蜂种群的鉴定和区分上,随后在按蚊、寄生蜂、舞毒蛾、蝗虫、蚜虫、粉虱等昆虫中均有应用(Black et al.,1992;刘春林等,2003)。Ben-Dov 等(2000)利用RAPD 技术结合形态学特征,对1872年定名的一种星蜡蚧Ceroplastes vinsonii Signoret 重新确定,发现其是1881年定名的龟蜡蚧(C.floridensis Comstock)的同种异名,对来自Reunion 的蜡蚧属新种C.reunionensis 与红蜡蚧C.Rubens 进行比较研究,发现它们虽然在形态特征上相似,但在分子水平上两个种的种间和种内都有差异,说明RAPD技术对蚧虫分类地位和传统分类学的标准进行了修正。
总结以上研究结果得出,磁共振在乳腺癌诊断中具有较显著的临床价值,确诊率较高,误诊率及漏诊率较低,可为临床医师诊断病情提供依据,值得各医疗机构推广应用。
昆虫的种群遗传分化与种群本身的适应性有关,而且受环境因素的影响,据研究,蚧虫种群分化同时受到寄主因素和地理因素的双重影响。李梦等(2006)利用5 条随机引物(S61、S64、S67、H9、H16)对河北不同地区种群的10个蚧虫种群进行研究,结果显示不同地理来源的皱大球蚧 Eulecanium kuwanai 和瘤坚大球蚧 E.gigantean 之间已经产生了一定程度的遗传分化,遗传差异与地理距离呈正相关;寄生于白蜡Fraxinus chinensis 和刺槐Robinia pseudoacacia 两种不同寄主植物上的扁平球坚蚧Parthenolecanium corni种群之间存在一定程度的遗传差异,随后,高宝嘉等(2007)也取得了相同的结果;刘全超(2012)根据前人经验检以相同的方法和设计思路检测了来自河北邯郸、石家庄和保定3个地区16个不同寄主白蜡Fraxinus chinensis、紫叶碧桃Amygdalus persicaf pendula、月季Rosa chinensis、紫叶李Prunus cerasifera ehrhar f.atropurpurea、二球悬铃木Platanus acerifolia、稠李Psdus racemosa、杜梨Pyrus betulaefolia、枫杨Pterocarya stenoptera、木槿Hibiscus syriacus、垂丝海棠Malus halliana、紫薇Lagerstroemia indica 和华桑Morus cathayana 的草履蚧Drosicha corpulenta种群之间的DNA 多态性,结果显示不同寄主来源的草履蚧已产生明显的种群分化,而同种寄主不同地理种群也存在遗传差异。上述三项研究结果趋同,这可能与蚧虫的生态习性、地理位置和生态环境有密切关系。RAPD 技术稳定性差的问题一直是争议的焦点,此实验分别对扩增体系中各种反应物梯度和反应条件进行了优化,摸索了适合实验材料的理想的退火温度,同时利用酯酶同工酶标记技术对RAPD 标记的结果进行验证,建立了RAPD 对蚧虫的最适反应体系,但作者认为必须使用一定数量的引物以获得足够的基因用于分析。
部分蚧虫在系统发育上和虫种问题方面长期存在歧义,松干蚧是松树上一类比较古老的昆虫,针对我国松干蚧的虫种问题,蚧虫学界分类主张各不相同,汤枋德(1978)认为分布于辽宁的松干蚧是一个新种,但是杨平澜等(1976)认为分布于辽、鲁、江、浙的松干蚧均为日本松干蚧,杨钤等(2006)采用优化的RAPD 技术和筛选出的4 条长度为10 bp 的随机引物(S500、S1415、11N010145 和11N010146)对来自浙江金华、山东青岛、辽宁抚顺的3个松干蚧地理种群进行DNA片段扩增,证明这3个地理来源的松干蚧均为日本松干蚧,并由UPGMA 聚类分析推测浙江金华的松干蚧极有可能是从山东青岛传过去,此研究重新评估了松干蚧属,为解决我国松干蚧的分布和物种问题提供了借鉴。陈航(2007)对采自不同寄主的7种紫胶虫进行多态性分析,聚类分析结果表明,田紫胶虫Kerria ruralis 与尼泊尔紫胶虫K.nepalensis、普萨紫胶虫K.pusana 亲缘关系紧密,与云南紫胶虫K.yunnaensis、中华紫胶虫K.chinensis 关系较远,其中田紫胶虫与中华紫胶虫的亲缘关系较远,信德紫胶虫K.sindica 和紫胶蚧K.lacca 与其它5种紫胶虫亲缘关系较远。该论文澄清了云南紫胶虫与紫胶蚧、中华紫胶虫三者之间,证实了信德紫胶虫与紫胶蚧紧密的姐妹群,但还需要筛选出更多独立进化的基因标记进行分析和比较,才能彻底澄清紫胶虫系统发育研究中存在的疑点。
2.1.3 微卫星DNA 技术
SSR(Simple Sequence Repeat,简单重复序列)是指由1-6个核苷酸串联重复而形成的核苷酸序列,也被称为“微卫星”,Spritz(1981)首先在珠蛋白中发现,根据微卫星序列两端的互补序列设计引物,通过PCR 反应扩增微卫星片段,将扩增片段进行聚丙烯酞胺凝胶电泳,依据微卫星序列拷贝数的差异决定基因型,并计算等位基因发生的频率(闫华超,2006)。NCBI 登陆的数据中,以双翅目、鳞翅目和膜翅目昆虫的微卫星数量最多,占75%,而半翅目昆虫的研究相对偏少。
谢映平等采用3 条微卫星引物对日本龟蜡蚧Ceroplastes japonicus、角蜡蚧C.ceriferus、白蜡绵粉蚧Phenacoccus fraxinus、瘤坚大球蚧和朝鲜球蚧Didesmococcus koreanus 进行扩增,结果显示种内遗传分化程度从大到小为日本龟蜡蚧>朝鲜毛球蚧>角蜡蚧>瘤坚大球蚧>白蜡绵粉蚧;种间的遗传分化程度明显低于种内(石晶,2005;谢映平等,2007),确定了一种大规模提取蚧虫基因组DNA 的方法,印证了朝鲜毛球蚧和瘤大球坚蚧的遗传关系比一直同在蜡蚧亚科的角蜡蚧和日本龟蜡蚧的遗传关系还近,是对坚蚧类分类地位提升的支持,也进一步将毛球蚧类从软蚧类分离出来归入坚蚧类,虽然试验不可能完全代表所在属或科的遗传多态性特征,但总体支持了汤氏分类系统和Hodgson 的蚧科分类系统,此外增加蚧虫分子研究中的单微卫星位点数量是对该技术一个很有必要的补充。继上述研究之后,李慧等(2014)利用5'锚定简并引物对扶桑绵粉蚧、桃小食心虫Carposina sasakii、稻水象甲Lissorhoptrus oryzophilus等10种重要农业害虫进行微卫星DNA 筛选,发现扶桑绵粉蚧冗余率(重复序列的数量/总序列的数量)最高为71.7%,扶桑绵粉蚧微卫星类型为完全型,验证了SSR 技术在扶桑绵粉蚧微卫星引物筛选的有效性,为进一步挖掘其微卫星位点奠定基础。罗梅等(2014)利用高通量搜索的方法对扶桑绵粉蚧转录组中28120 条unigenes 的数据进行搜索,共找到1781个SSR 位点,发生频率(含有SSR 的unigene 数量与总unigene 数量之比)为5.79%,SSR 的分布频率(SSR个数与总unigene数量比)为6.33%,SSRs 的主要重复类型是单核苷酸重复,SSR 重复单元的重复次数分布在5-19次之间,单核苷酸重复基序的扩增多态性比三核苷酸重复基序的丰富,该结论验证了SSR 技术在低级单元中的多态性普遍比高级单元的高,为扶桑绵粉蚧遗传多样性分析、进化分析及入侵生物学等提供了理论依据。
2.1.4 简单重复序列间扩增技术
ISSR(Inter Simple Sequence Repeat,简单重复序列间扩增)技术是在SSR 的5'或3'端加锚1-4个嘌呤或嘧啶碱基组成16-18个碱基序列,然后以此为引物,对两侧具有反向排列SSR 的一段基因组DNA 序列进行扩增(黄可辉,2005)。马力等(2007)利用ISSR-PCR 方法实现了对枣大球蚧、朝鲜毛球蚧Didesmococcus koreanus、日本龟蜡蚧 Ceroplasts japonicus 和白蜡绵粉蚧Phenacoccus fraxinus 的快速鉴定,同年又筛选出了多态性丰富的13个ISSR 引物,提供了一个适合蚧虫的ISSR-PCR 最佳反应体系和反应条件的标准化程序,增强了ISSR 标记技术在蚧虫种间遗传多样性分析的稳定性;Chen(2013)等运用ISSR 和RAPD 相结合的技术对20种紫胶虫的COI、28S 和EF-1α 进行系统发育分析,发现印度次大陆是转移到欧亚大陆的紫胶虫的分布中心,大陆漂移理论和现有的化石记录表明紫胶虫是从印度次大陆漂向欧亚大陆的,随着温度的升高,紫胶虫可能进入热带地区。此研究是在明确7种紫胶虫的系统发育和进化关系后开展的一项工作(陈航,2007),虽然当时取得了一些建设性的成果,但云南紫胶虫、中华紫胶虫、田紫胶虫、尼泊尔紫胶虫与普萨紫胶虫5 者之间的核基因分析结果并不一致,还无法对其扑朔迷离的关系形成定论,本研究增添的ISSR 技术更加深入地比较和分析了紫胶虫的遗传规律和进化机制。朱新帅等(2014)用ISSR 引物对来自新疆哈密、巴州、阿克苏、喀什、和田5个地区共计15个枣球蜡蚧地理种群进行多态分析,筛选出46 条ISSR 引物,并证明种群间的遗传背景存在明显分化,种群的遗传多样性与年均温度、年均湿度呈正相关,与寄主树龄和纬度无相关性,而与海拔呈负相关;为枣球蜡蚧遗传变异、发生与环境的关系提供了理论支撑,ISSR 技术对评估和区分种群间的变异取得较好的成果,我国运用ISSR 技术对蚧虫的鉴定和系统发育的研究仅山西大学实验室稍显成熟,但也仅仅局限于紫胶虫,有关蚧虫其他种类都还比较笼统,甚至还未涉及,需要进一步的探讨。
2.1.5 DNA 条形码技术
DNA 条形码(DNA Barcoding)也称DNA 条形编码,类似于超级市场商品包装上用于识别商品的粗细、间隔不同的黑白条纹图案,是利用COI的前部长约650 bp 序列作为标记来实现快速、准确和自动化地对物种进行鉴定和分类,DNA 条形码最初由加拿大圭尔夫大学(University of Guelph)的动物学家Hebert 等(2003)提出。其技术流程包括样品获取、DNA 提取、PCR 扩增、序列测定、序列比对及根据序列差异进行判别。该技术在鳞翅目、鞘翅目、膜翅目和双翅目等昆虫中均有研究(Monaghan et al.,2005;Nelson et al.,2007;Fisher and Smith,2008)。
蚧虫的DNA 条形码研究多是将COI 基因与核基因联合分析,例如,可将18S rDNA、16S rDNA、EF-1α 和COI 联合作为蚧虫分子系统学研究中的标准序列,以增加同源序列之间的可比性(Caterino et al.,2000;Zhang et al.,2000;Hajibabaei,2006)。Gullan 等(2003)通过扩增COI、28S、EF-1α 基因序列及其比对分析,并结合外部形态特征发现了粉蚧1 新种Ferrisia gilli,并证明双条弗粉蚧为物种复合体;随后,又对粉蚧科腺刺粉蚧属Ferrisia Fullaway 的6种粉蚧进行研究,发现这些粉蚧平均遗传距离均大于4%,并建议将双条弗粉蚧细分为6个不同的支系(Gullanet et al.,2010),因此结合形态特征上的差异可将其分为几个不同的亚种,这无疑是对粉蚧科昆虫分类区系的再次完善;对大洋臀纹粉蚧、柑橘臀纹粉蚧、无花果臀纹粉蚧Planococcus ficus等3种近缘种粉蚧的mtDNA COI 序列以及18S 和28S 基因序列的研究结果同样说明蚧虫COI 基因序列变异程度较其他基因高(何衍彪等,2007),此结论是蚧虫DNA 条码研究开展的先决条件。
随着蚧虫的mtDNA COI 受到越来越多的重视,Park 等(2011)通过重新设计COI 基因序列的正向引物,对盾蚧科和粉蚧科进行了物种鉴定的有效性研究,揭示了介壳虫DNA 条形码序列核苷酸组成的特性(Deng et al.,2012);Saccaggi 等(2008)通过mtDNA COI 基因序列特征分析成功将葡萄绵粉蚧Planococcus ficus、柑橘臀纹粉蚧和长尾粉蚧Pseudococcus longispinus 区别开来;同时,利用引物PcoF1/LepR1 扩增mtDNA COI 基因,实现了对新菠萝灰粉蚧Dysmicoccus neobrevipes、大洋臀纹粉蚧和南洋臀纹粉蚧Planococcus lilacius 等蚧虫的快速检测(徐浪,2010;徐浪等,2013)。由此可知该技术在蚧虫的鉴定可行性方面已经毋庸置疑,除此之外,也证明了DNA 条形码在同属蚧虫鉴定中的优势。
为进一步开展蚧虫DNA 条形码系统研究,何衍彪等随后又初步明确了热区的菠萝洁粉蚧、新菠萝灰粉蚧在我国的分布,海南万宁种群可能是菠萝洁粉蚧的1个隐存谱系的结论,并发现mtDNA COI 基因序列有33个变异位点(何衍彪等,2011;何衍彪,2012),这与Rung 等(2008)对大洋臀纹粉蚧和柑橘臀纹粉蚧的鉴定结果一致;此外Ball 和Armstrong(2008)利用该技术证实了珠蚧科Margarodidae 不同地理种群介壳虫复合种的存在;褚栋等(2009)对来自海南三亚和陵水地区的扶桑绵粉蚧mtDNA COI 测序,并与美国佛罗里达州的扶桑绵粉蚧序列进行比较,推测扶桑绵粉蚧可能是至少含有2个隐存谱系或姊妹种的复合种,新入侵我国海南三亚和陵水地区的扶桑绵粉阶是该复合种内的1个隐存谱系或物种,且可能不是来自美国佛罗里达州;陈哲等(2012)对中国、巴基斯坦、美国的扶桑绵粉蚧mtDNA COI基因序列的碱基差异比较和遗传距离分析进一步验证了褚栋等的结论,而对于扶桑绵粉蚧另外一遗传支系是否也入侵中国有待于扩大样本采集点和样本数来进一步深入验证。以上结果说明DNA条形码可作为粉蚧近缘种鉴别的依据,同时可有效地发现和揭示蚧虫中的隐存分类单元和复合种。
继DNA 条形码概念提出之后,一个专门获取、储存、分析和发表DNA 条形码记录的数据库—生物DNA 条形码数据库(DNA Barcode of Life Data,BOLD,http://www.boldsystems.org)成立,并于2007年获得官方认可。田虎(2013)以来自全国7个省12个不同采集区域的21种介壳虫为靶标构建了介壳虫类昆虫DNA 条形码数据库,目前该系统可对40种介壳虫类昆虫进行鉴定识别;截止到2014年12月,BOLD 中与条形码有关的序列已达4201240 条,其中,可作为鉴定物种水平的DNA 条形码,即每个物种有三个以上且序列长度不低于500 bp 的序列为3654244 条序列(http://www.barcodinglife.org/)。其中,同翅目蚧总科下含有来自11个国家的449 条48种蚧虫的DNA 条码序列,见表1。其中,439 条有物种名称,分别代表295个种类。
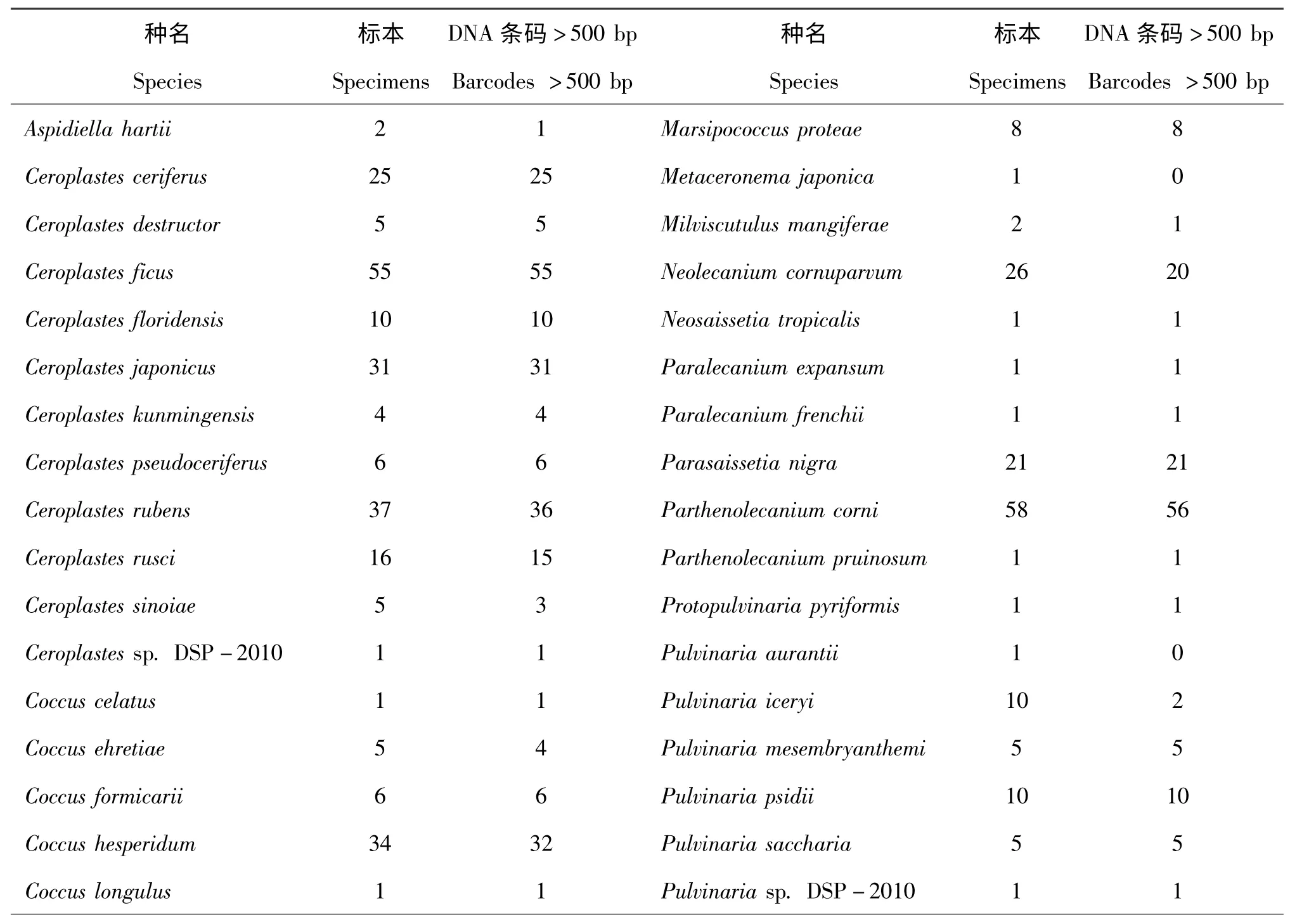
表1 蚧总科含DNA 条码昆虫种类Table 1 Insect species of Coccoidea with DNA barcoding

(续上表)
2.2 各种分子生物学技术的比较
针对不同的靶标基因匹配有不同的分子生物学技术,而且各有其利弊。针对蚧虫低级阶元,即种内或者近缘种之间的系统发生关系的分析,以上几种分子生物学技术均可获得预期效果。
RFLP 技术中的限制性内切酶可以将DNA 片段消化成很小的DNA 片段,但这只是对较小的基因组,在切割大分子基因时,各种片段在凝胶上相互重叠,很难区分。目前通常将RFLP-PCR 技术与杂交技术结合,但这样造成RFLP 技术操作复杂繁琐和放射性危害,而且RFLP 技术用于相距较远种(岐化值大于10%-15%)时,因为较远种间的酶切片段同源性差,则不能清晰地推演种间进化关系。
RAPD 技术在一定程度上弥补了RFLP 技术的缺陷,其操作简便易行的特点受到诸多学者的偏爱,而且所用的引物没有严格的种属界限,能在短时间内进行大量样品分析(黄可辉,2005)。不足之处在于,RAPD 技术重复性差,对于不同试验对象基因组来说,究竟进行多少引物的RAPD 筛选才能获得足够的基因仍然没有统一的标准。
相对于RFLP 和RAPD 技术,SSR 技术凝胶电泳时具有单碱基的高分辨率,SSR 序列广泛存在于真核生物基因组的非编码区和编码区,遗传信息量大,一个SSR 座位可以检测26个等位基因(孙荆涛等,2012),微卫星分子标记在昆虫迁飞习性的研究中作用显著,但是微卫星具有较复杂变异模型,很难将种群间的遗传距离转化为种群分化的时间尺度,而且必须依赖每类微卫星两段序列设计引物,而不像RAPD 引物是随机合成的,需前期研究基础,而且该技术对整个实验操作过程的准确度和精确度要求非常高,稍许差错就会对最终结果造成很大影响(Ellegren,2004)。
随后ISSR-PCR 技术应运而生,该技术是在SSR 基础上发展起来的,保证了可重复性,常常采用1个SSR 序列和1个引物对,从而保证了引物与基因组DNA 中的SSR 的5'或3'末端结合,通过PCR 反应扩增SSR 之间的DNA 片段,以增强多态性(张立荣,2002;黄映萍,2010),无需预先克隆和测序,无需知道DNA 序列即可用引物进行扩增,也正因如此,一些ISSR 引物可能在特定基因组中没有配对区域而无扩增产物。
随着研究的不断深入,DNA 条形码技术在蚧虫研究中越来越受到重视,除了具有其他分子标记手段所不具备的功能外,DNA 条码技术的优点还包括:引物通用性,积累广泛的生物分类信息及分析进化关系,其鉴定所需样品量少,只需一小块或一小片材料即可鉴定一个物种;同时可鉴定出许多群体中普遍存在的隐存分类单元(Nelson et al.,2007),这对部分形态特征不稳定的蚧虫的鉴定具有很好的参考价值;另外DNA 条形码具有数字化的数据库,因而提供了信息化的分类学标准和有效的生物分类学手段,成为学科进展最迅速的前沿之一。
3 存在的问题及发展前景
3.1 存在的问题
3.1.1 与传统形态分类学的兼容问题
分子生物学技术对于大多数动物类群都具有良好的鉴别力,其快速、准确、便捷的鉴定能力,弥补了传统形态学鉴定方法的不足,突破了以往对经验的过分依赖的局限,为物种识别鉴定提供了新的研究方法和手段。然而,某些蚧虫的模式标本已经遗失,有的也因年代过于久远而无法取样,并且这些标本通常具有重要的分类学价值,分子学方法对于标本具有破坏性,因此从珍贵的模式标本上获取目的基因是不合适的。虽可以采用一般标本的方式作为折衷,但这种处理方式可能导致分子系统与林奈分类系统在对应上出现偏差。利用分子系统学对蚧虫进行鉴定,选用rDNA还是mtDNA 的技术分析的可靠性,必须借由传统分类学的验证,完全脱离形态学的DNA 分类是没有意义的,需要形态学与标准基因的联合分析。
3.1.2 假基因的干扰问题
运用不同DNA 提取方法所获得的基因组细胞内含有大量的DNA 序列,包括自身序列和非自身序列,核基因组里存在线粒体基因的同源序列,如果这些序列被误用作目的基因,便会造成电泳杂带、测序双峰以及背景噪音模糊等,进而影响DNA 条形码的种群分类鉴定。假基因(pseudogene)在基因表达、基因调控以及产生基因多样性等方面可能都扮演着极为重要的角色,科学研究发现假基因并不是“垃圾基因”,但其确切的作用机制仍不清楚(Moulton et al.,2004)。假基因可分为以下两种:一类是重复假基因,这类假基因是由于基因组DNA 重复,或染色体不均等交换过程中,基因编码区或调控区发生突变如碱基置换、插入或缺失,导致复制后的基因丧失正常功能而形成的,第二类是加工假基因或返座假基因(processed pseudos),这类假基因是由mRNA 转录本反转录成cDNA 后重新整合到基因组,由于插入位点不合适或序列发生突变而失去正常功能形成的,由于假基因不被表达,所以细胞中不存在相应的RNA,可通过RT-PCR 的方法予以解决(Frohman et al.,1988;Collura et al.,1996;王关林和方宏筠,1998;Lorenz et al.,2005)。诸多研究显示提取的DNA 样品中存在PCR 抑制物,影响扩增效率,可加入少量牛血清蛋白或Q-solution 等洗涤样品,或者稀释DNA 等降低抑制,进行二次PCR 和设计特异性引物等也是解决此问题的有效途径(Moulton et al,2004;Juen and Traugott,2006;李凯等,2010)。
3.2 发展前景
早期传统的形态分类法为蚧虫的分类鉴定奠定了基础,但目前仅仅依靠形态学资料还不能完全解决蚧虫的系统分类问题。80年代以来分子生物学的迅速发展,并渗入系统与进化研究中而形成了一门新兴交叉学科-昆虫分子生物学,为昆虫系统学的研究和发展增添了新的活力。分子生物学技术主要针对于形态特征极其相似的近缘种、复合种及种以下的亚种、生物型、地理种群的识别和鉴定,能解决传统的形态学分类方法难以解决的问题。如何从丰富的蚧虫DNA 信息中筛选出准确有效地鉴别物种的片段是很关键的。鉴于DNA条形码相对于其他分子生物学技术的优势,在鳞翅目、鞘翅目等昆虫中已有很成熟的DNA 条形码数据库建立,蚧虫的COI 基因长度约为1500 bp,究竟哪一段可以作为蚧虫的基因条形码尚无定论。
部分蚧虫属种间差异率很小,不同物种需要使用不同的基因序列以完成其DNA 条形码的构建,单一的COI 序列不能够完全做到区分的目的,增加分析基因的数量是确保准确分类的有效途径,因此需要联合一些其他基因比如核内基因来增加标记数(Moritz and Cicero,2004)。对于不同地理来源的昆虫,DNA 条形码在建立鉴定技术和物种系统进化关系时并不能准确区分地区,即其具有区域性研究的限制;另有学者指出DNA 条形码对于新分化的种群也存在欠缺之处,只能作为分类学研究中发现新物种的线索;对于长度变异较大的条形码序列(如非编码基因PsbA-trnH),在比对方法的确定以及对插入和缺失的处理方面仍存在较大争议,分类非常困难,虽然有一些解决途径被提出,但仍需改进。
总体来说,相对国外逐渐发展的分子体系,国内蚧虫DNA 条形码研究尚处于起步阶段,有关蚧虫类昆虫的分子分类研究体系较弱,作为一种标准的物种鉴定方法,DNA 条形码技术有着巨大的科学应用潜力,尤其在蚧虫分类学和系统发育学研究等领域应用前景更为显著,随着生物技术的发展,构建蚧虫完整的DNA 条形码序列数据库将是很好的发展方向。
References)
Annstrong K,Cameron C,Frampton E.Fruitfly(Diptera:TepMtidae)species identification:A rapid molecular diagnostic technique for quarantine application[J].Bulltin of Entomological Reaearch,1997,87:111-118.
Aljanabi SM,Martine Z.Universal and rapid salt extraction of high quality genomic DNA f or PCR based techniques[J].Nucleic.Acids.Res.,1997,25(22):4692-4693.
Ashfaq M,Noor AR,Mansoor S.DNA-based characterization of an invasive mealybug(Hemiptera:Pseudococcidae)species damaging cotton in Pakistan[J].Applied Entomology and Zoology,2010,45(3):395-404.
Ball SL,Armstrong KF.Rapid,one-step DNA extraction for insect pest identification by using DNA barcodes[J].Journal of Economic Entomology,2008,101(2):523-532.
Black IVWC,Duteart NM,Puterka GJ.Use of random amplified polymorphic DNA polymerase chain reaction(RAPD-PCR)to detect DNA polymorphisms in a aphids[J].Bull.Entomol.Res.,1992,82:151-159.
Ben-Dov Y,Miller DR,Gibson GAP.Scale Net:A database of the scale insects of the world.United States Department of Agriculture(USDA),2002,Available online:http://www.sel.barc.usda.gov/scalenet/scalenet.htm.
Ben-Dov Y,Matile-Ferrero D,Gafny R.Taxonomy of Ceroplastes rubens Maskell with description of a related new species(Hemiptera:Coccoidea:Coccidae)from Reunion,including DNA polymorphism analysis[J]Annales de la Societe Entomologique de France,2000,36(4):423-433.
Beuning L,Murphy P,Wu E.Molecular-based approach to the differentiation of mealybug(Hemiptera:Pseudococcidae)species[J].Economic Entomology,1999,92(02):463-472.
Burban C,Petit RJ,Carcreff E,et al.Rangewide variation of the maritime pine bast scale Matsucoccus feytaudi Duc.(Homoptera:Matsucoccidae)in relation to the genetic structure of its host[J].Molecular Ecology,1999,10(8):1593-1602.
Chen H.Phylogenetic Relationships And Molecular Evolution of 7 Species In Lac Insects(Tachardiidae:Kerria)[D].Beijing:Chinese Academy of Forestry Offcial,2007.[陈航.7种紫胶虫的系统发育与分子进化分析(Tachardiidae:Kerria)[D].北京:中国林业科学研究院,2007]
Collura RV,Auerbach MR,Stewart CB.A quick,direct method that can differentiate expressed mitochondrial genes from their nuclear pseudogenes[J].Current Biology,1996,6(10):1337-1339.
Chen H,Chen X,Feng Y,et al.Molecular phylogeny and biogeography of lac insects(Hemiptera:Kerriidae)inferred from nuclear and mitochondrial gene sequences[J].Molecular Biology Reports,2013,40(10):5943-5952.
Caterino MS,Cho S,Sperling FAH.The current state of insect molecular systematics:a thriving tower of babed[J].Annu.Rev.Entomol.,2000,45(1):1-54.
Cook LG,Gullan PJ,Trueman HE.A preliminary phylogeny of the scale insects(Hemiptera:Stemorrhyncha:Coccoidea)based on nuclear small subunit ribosomal DNA[J].Molecular Phylogenetics and Evolution,2002,25(1):43-52.
Chu D,Liu GX,Fu HB,et al.Phylogenetic analtsis of mt COI reveals the cryptic lineages in Phenacoccus solenopsis complex(Hemiptera:Pseudococcidae)[J].Acta Entomologica,2009,52(11):1261-1265.
Chiu YC,Wu WJ,Wong CY,et al.Application of the PCR-RFLP technique for the rapid diagnosis of scale insects(Homoptera:Coccoidea)on imported agricultural products in Taiwan[J].Formosan Entomol.,2004,24(4):365-375.
Chen Z,Zhang J,Fu HF,et al.On the validity of the species Phenacoccus solenopsis based on morphological and mitochondrial COI data,with the description of a new body color variety[J].Biodiversity Science,2012,20(4):443-450.[陈哲,张姜,傅杭飞,等.基于形态特征和线粒体COI 基因探讨扶桑绵粉蚧物种的有效性并记述一体色变异型扶桑绵粉蚧[J].生物多样性,2012,20(4):443-450]
Dayrat B.Towards integrative taxonomy[J].Biological Journal of the Linnean Society,2005,85(3):407-415.
Downie DA,Gullan PJ.Phylogenetic analysis of mealybugs(Hemiptera:Coccoidea:Pseudococcidae)based on DNA sequences from three nuclear genes,and a review of the higher classification[J].Systematic Entomology,2004,29(2):238-260.
Deng J,Yu F,Zhang TX,et al.DNA barcoding of six Ceroplastes species(Hemiptera:Coccoidea:Coccidae)from China[J].Molecular Ecology Resources,2002,12(5):791-796.
Frohman MA,Dush MK,Martin GR.Rapid production of full-length cDNAs from rare transcripts:Amplification using a single gene-specific oligonucleotide primer[J].Proc.Natl.Acad.Sci.USA,1988,85:8998-9002.
Frézal L,Leblois R.Four years of DNA barcoding:Current advances and prospects[J].Infect Genet.Evol.,2008,8:727-736.
Fisher BL,Smith MA.A revision of Malagasy species of Anochetus mayr and Odontomachus latreille(Hymenoptera:Formicidae)[J].PLoS ONE,2008,3:e1787.
Gullan PJ,Downie DA,Steffan SA.A new pest species of the mealybug genus Ferrisia Fullaway(Hemiptera:Pseudococcidae)from the United States[J].Annals of the Entomological Society of America,2003,96(6):723-737.
Gullan PJ,Kaydan MB,Hardy NB.Molecular phylogeny and species recognition in the mealybug genus Ferrisia Fullaway(Hemiptera:Pseudococcidae)[J].Systematic Entomology,2010,35(2):329-339.
Gao BJ,Li M,Wang CH,et al.RAPD analysis of genetic divergence among populations in Eulecanium kuwanai Kanda and Eulecanium gigantean Shinji[J].Acta Ecologica Sinica,2007,27(3):918-923.[高宝嘉,李梦,王春和,等.皱大球蚧(Eulecanium kuwanai)和瘤坚大球蚧(Eulecanium gigantean)地理种群的RAPD 遗传分化[J].生态学报,2007,27(3):918-923]
He YB.Molecular Identification,Genetic Structure and Control Strategy of Pink Pineapple Mealybug,Dysmicoccus Brevipes(Cockerell)[D].Chongqing:Southwest University,2012.[何衍彪.菠萝洁粉蚧的分子鉴定、遗传结构及其控制基础研究[D].重庆:西南大学,2012]
Huang YP.Advance on DNA molecular maker technigue[J].Journal of the Graduates Sun Yat-sen University(Natural Sciences.Medicine),2010,31(2):27-36.[黄映萍.DNA 分子标记研究进展[J].中山大学研究生学刊(自然科学·医学版),2010,31(2):27-36]
Huang HK,Guo QX,Yu B.The appliton of molecularmarker methods in entomology studies[J].Entomological Journal of East China,2005,14(2):109-114.[黄可辉,郭琼霞,虞赟,等.分子标记法在昆虫学研究中的应用[J].华东昆虫学报,2005,14(2):109-114]
Hosseini R,Hajizadeh J.Molecular identification of three of the most important mealybug species(Hemiptera:Sternorrhyncha:Coccoidea:Pseudococcidae)on ornamental plants in Guilan province,Iran[J].Zootaxa,2011,3009:46-54.
Hajibabaei M,Janzen DH,Burns JM,et al.DNA barcodes distinguish species of tropical Lepidoptera[J].Proc.Natl.Acad.Sci.USA,2006,103:968-971.
He YB,Wan XW,Zhan RL,et al.Genetic relationship of 12 species of mealybugs(Hemiptera:Pseudococcidae)based on DNA sequences[J].Chinese Journal of Tropical Crops,2011,32(12):2324-2330.[何衍彪,万宣伍,詹儒林,等.基于DNA 序列的12种粉蚧亲缘关系分析[J].热带作物学报,2011,32(12):2324-2330]
He Yb,Zhan RL,Zhao YL.Research progress on Dysmicoccus brevipes(Cockerell)and mealybug wilt of pineapple[J].Guangdong Agricultural Sciences,2007,2:44-49.[何衍彪,詹儒林,赵艳龙.菠萝粉蚧及菠萝凋萎病研究进展[J].广东农业科学,2007,2:44-49]
Juen A,Traugott M.Amplification facilitators and multiplex PCR:Tools to overcome PCR-inhibition in DNA-gut content analysis of soil living invertebrates[J].Soil Biology and Biochemistry,2006,38(7):1872-1879.
Kondo T,Gullan PJ,Williams DJ.Coccidology:The study of scale insects(Hemiptera:Sternorrhyncha:Coccoidea)[J].Revista Corpoica-Cienciay Tecnología Agropecuaria,2008,9(2):55-61.
Li M.Study on the Population Differentation and Genetic Diversity of the Patial Coccoidea[D].Hebei:Agricultural University of Hebei Province,2006.[李梦.部分蚧科昆虫种群分化及遗传多样性的研究[D].河北:河北农业大学,2006]
Liu QC.Studyon The Population Differentiation and Genetic Diversity of Drosicha Corpulenta Populations on Different Hosts and Geography[D].Hebei:Agricultural University of Hebei Province,2012.[刘全超.草履蚧不同寄主和地理种群分化及遗传多样性研究[D].河北:河北农业大学,2012]
Luo M,Dong ZY,Bin SY,et al.Molecular cloning,prokaryotic expression and expression at different developmental stages of cathepsin B gene in mealybug Phenacoccus solenopsis Tinsley(Hemiptera:Pseudococcidae)[J].Acta Entomologica Sinica,2012,55(3):276-283.[罗梅,董章勇,宾淑英,等.扶桑绵粉蚧组织蛋白酶B 基因的克隆、原核表达和不同发育阶段表达分析[J].昆虫学报,2012,55(3):276-283]
Luo M,Dong ZY,Bin SY,et al.Molecular cloning and bioinformatics analysis of calmodulin genes in Phenacoccus solenopsis Tinsley[J].Journal of Huazhong Agricultural University,2012,31(3):320-324.[罗梅,董章勇,宾淑英,等.扶桑绵粉蚧钙调蛋白基因的克隆与生物信息学分析[J].华中农业大学学报,2012,31(3):320-324]
Lorenz JG,Jackson WE,Beck JC,et al.The problems and promise of DNA barcodes for species diagnosis of primate biomaterials[J].Philosophical Transactions of the Royal Society of London.Series B,Biological Sciences,2005,360(1462):1869-1878.
Li H,Lang KL,Shen CP,et al.Isolation and characterization of microsatellite DNA loci from ten important agricultural pest using anchored PCR method[J].Journal of Biosafety,2014,23(1):60-65.[李慧,郎坤玲,沈长朋,等.基于锚定PCR 技术对10种重要农业害虫微卫星DNA 位点的筛选及其特征分析[J].生物安全学报,2014,23(1):60-65]
Li XM,Wang XF,Cui ZJ.Applications and optimizations of T-RFLP in fingerprinting environmental microbial populations[J].Joural of China Agricultural University,2009,14(4):1-9.[李献梅,王小芬,崔宗均.末端限制性片段长度多态性技术(T-RFLP)在微生物群体分析上的应用与技术优化[J].中国农业大学学报,2009,14(4):1-9]
Liu CL,You LS,Luo QH,et al.Random amplified polymorphic DNA of the similar broconid wasps cotton field andpaddy field from China[J].Acta Zootaxonomica Sinica,2003,28(4):568-572.[刘春林,游兰韶,罗庆怀,等.中国棉田和稻田茧蜂近缘种的RAPD 分子标记研究[J].动物分类学报,2003,28(4):568-572]
Liu MH,Yang RP,Zeng CH,et al.Application and prospect of biological control to diseases and insect pests with modern biotechnology[J].Jiangxi Plant Protection,2009,32(1):14-18.[刘明辉,杨荣萍,曾春辉,等.现代生物技术在病虫害生物防治中的应用途径和前景[J].江西植保,2009,32(1):14-18]
Luo M,Zhang H,Bin SY,et al.High-throughput discovery of SSR makers in the mealybug,Phenacoccus solephsis(Hemiptera:Pseudococcidae),from its transcriptome database[J].Acta Entomologica Sinica,2014,57(4):395-400.[罗梅,张鹤,宾淑英,等.基于转录组数据高通量发掘扶桑绵粉蚧微卫星引物[J].昆虫学报,2014,57(4):395-400]
Li K,Zhao LY,Tian JC,et al.Research progress in DNA-based approach tracking trophic links[J].Acta Phytophylacica Sinica,2010,37(1):83-88.[李凯,赵丽亚,田俊策,等.食物链网络DNA 跟踪技术研究进展[J].植物保护学报,2010,37(1):83-88]
Monaghan MT,Balke M,Gregory TR,et al.DNA-based species delineation in tropical beetles using mitochondrial and nuclear markers[J].Philosophical Transactions of the Royal Society of London.Series B,Biological Sciences,2005,360(1462):1925-1933.
Moritz C,Cicero C.DNA barcoding:promise and pitfalls[J].PLoS Biol.,2004,2(10):1529-1531.
Moulton MJ,Song H,Whiting MF.Assessing the effects of primer specificity on eliminating numt coamplification in DNA barcoding:A case study from Orthoptera(Arthropoda:Insecta)[J].Mol.Ecol.Resour.2010,10(4):615-27.
Ma L,Xie YP,Xue JL.Rapid identification of scale insect by total DNA extraction and PCR[J].Journal of Anhui Agricultural Sciences,2007,35(5):145-147.[马力,谢映平,薛皎亮.蚧虫DNA 的提取及PCR 法虫种快速鉴定技术[J].安徽农业科学,2007,35(5):145-147]
Nelson LA,Wallman JF,Dowton M.Using COI barcodes to identify forensically and medically important blowflies[J].Med.Vet.Entomol.,2007,21:44-52.
Park DS,Leem YJ,Hahn KW,et al.Molecular identification of mealybugs(Hemiptera:Pseudococcidae)found on Korean pears[J].Journal of Economic Entomology,2010,103(1):25-33.
Park DS,Suh SJ,Hebert PDN,et al.DNA barcodes for two scale insect families,mealybugs(Hemiptera:Pseudococcidae)and armored scales(Hemiptera:Diaspididae)[J].Bulletin of Entomological Research,2011,101(4):429-434.
Qi XF.The Researchof Coccoidea Faunas in Beijing[D].Beijing:Beijing Forestry University,2008.[齐晓丰.北京地区蚧虫区系的研究[D].北京:北京林业大学,2008]
Rung A,Miller DR,Scheffer SJ.Polymerase chain reaction-restriction fragment length polymorphism method to distinguish three mealybug groups within the Planococcus citri-P.minor species complex(Hemiptera:Coccoidea:Pseudococcidae)[J].Journal of Economic Entomology,2009,102(1):8-12.
Rung A,Scbeffer SJ,Evans G,et al.Molecular identification of two closelv related species of mealybugs of the genus Planococcus(Homoptera:Pseudococcidae)[J].Annals of the Entomological Society of America,2008,101(3):525-532.
Robertson HM,Warr CG,Carlson JR.Molecular evolution of the insect chemoreceptor gene superfamily in Drosophila melanogaster[J].Proceedings of the National Academy of Sciences,2003,100(2):14537-14542.
Shi J.Genomic DNA Isolationof Scale Insect and Microsatellite Primers Polymorphism PCR[D].Shanxi:Shanxi University,2005.[石晶.蚧虫基因组DNA 提取及微卫星引物多态性扩增研究[D].山西:山西大学,2005]
Saccaggi DL,Krüger K,Pietersen G.A multiplex PCR assay for the simultaneous identification of three mealybug species(Hemiptera:Pseudococcidae)[J].Bulletin of Entomological Research,2008,98(1):27-33.
Tang FD.Discusstion on Matsucoccus matsumurae(KUW.)with descrition of a new species[J].Acta Entomologica Sinica,1978,21,164-169.[汤枋德.关于松干蚧的讨论及一新种描记-兼与《中国的松干蚧》文商榷[J].昆虫学报,1978,21:164-169]
Tian H.DNA Barcoding for Species Identification of Scale Insects[D].Beijing:Chinese Academy of Agricultural Sciences,2013.[田虎.介壳虫类昆虫DNA 条形码识别技术研究[D].北京:中国农业科学院,2013]
Tang KX,Kai GY,Zhang L,et al.Advances in RACE and its application in plant gene cloning[J].Journal of Fudan University(Natural Science),2012,41(6):704-709.[唐克轩,开国银,张磊,等.RACE 的研究及其在植物基因克隆上的应用[J].复旦学报(自然科学版),2012,41(6):704-709]
Utsugi JB,Toshihide KT,Motomi ITO.Current progress in DNA barcoding and future implications for entomology[J].Entomological Science,2011,14(2):107-124.
Wu SA.Checklist and faunistic analysis of scale insect pests(Hemiptera:Coccoidea)in Chinesemainland[J].Journal of Beijing Forestry University,2009,31(4):55-63.[武三安.中国大陆有害蚧虫名录及组成成分分析(半翅目:蚧总科)[J].北京林业大学学报,2009,31(4):55-63]
Wang GL,Fang HJ.Principleand Technology of Plant Genetic Engineering[M].Beijing:Science Press,1998,84-108.[王关林,方宏筠.植物基因工程原理与技术[M].北京:科学出版社,1998,84-108]
Wei JF,Feng JN,Wang P,et al.Study on the phylogeny of seven genera of Diaspidinae based on EF-1a sequences[J].Journal of Northwest A & F University(Nat.Sci.Ed.),2009,37(11):149-156.[魏久锋,冯纪年,王 鹏,等.基于核基因EF-lα序列的盾蚧亚科7个属的系统发育[J].西北农林科技大学学报(自然科学版),2009,37(11):149-156]
Williams JGK,Kubelik AR,Livak KJ,et al.DNA polylnorphisms amplified by arbitrary Primers are useful as genetie markers[J].Nuel.Aeids.Res.,1990,18(22):6531-6535.
Wang YQ,Wang XG,Xu LX.A new rapid method for extraction of high quality of genomic DNA from animal tissues[J].Chinese Journal of Zoology,2001,36(1):27-29.[汪永庆,王新国,徐来祥.一种动物基因组DNA 提取方法的改进[J].动物学杂志,2001,36(1):27-29]
Xie YP,Ma L,Xue JL,et al.The genetic relationship of the five species of scale insects by using the microsatellite primers approach[J].Journal of Shanxi Agricultural University(Natural Science Edition),2007,27(4):400-404.[谢映平,马力,薛皎亮,等.应用微卫星技术分析五种蚧虫的亲缘关系[J].山西农业大学学报(自然科学版),2007,27(4):400-404]
Xie YP,Ma L,Xue JL,et al.Advances in molecular systematics of Coccoidea.In:Li DM.Dynamic of Entomological Research,Representative Conference of National Member of the Entomological Society of China and Academic Annual Symposium of 2007[C].Beijing:China Agricultural Scientech Press,2007,33-36.[谢映平,马力,薛皎亮,等.蚧虫分子系统学研究进展.见:李典谟.昆虫学研究动态-中国昆虫学会第八次全国会员代表大会暨2007年学术年会论文集[C].北京:中国农业科学技术出版社,2007,33-36]
Xu L,Yu DJ,Jiao Y,et al.TaqMan real-time qualitative PCR for the inspection and identification of Planococcus minor and P.lilacius(Homoptera:Pseudococcidae)[J].Plant Quarantine,2010,24(2):24-28.[徐浪,余道坚,焦懿,等.大洋臀纹粉蚧和南洋臀纹粉蚧TaqMan 实时荧光PCR 检测方法[J].植物检疫,2010,24(2):24-28]
Xu L,Yu DJ,Jiao Y,et al.Molecular identification of Dysmicoccus neobrevipes and related species through DNA barcoding[J].Plant Quarantine,2013,27(3):43-45.[徐浪,余道坚,焦懿,等.新菠萝灰粉蚧及其近似种的DNA 条形码鉴定[J].植物检疫,2013,27(3):43-45]
Yan FC,Gao L,Li GL.Progress in molecular maker technigue and its application[J].Bulletin of Biology,2006.41(2):17-18.[闫华超,高岚,李桂兰.分子标记技术的发展及应用[J].生物学通报,2006.41(2):17-18]
Yan HJ,Huang XQ,Cheng ZQ,et al.Advances of the studies on constructionstrategy and analysis of cDNA library[J].Journal of Yunnan Agricultural University,2006,21(1):1-6.[晏慧君,黄兴奇,程在全.cDNA 文库构建策略及其分析研究进展[J].云南农业大学学报,2006,21(1):1-6]
Yang PL,Hu JL,Ren ZY.Pine bast scales from China[J].Acta Entomologica Sinica,1976,19,199-204.[杨平澜,胡金林,仁遵义.中国的松干蚧[J].昆虫学报,1976,19:199-204]
Yang Q,Xie YP,Fan XH,et al.Genetic differentiation of Matsucoccus matsumurae from three geographic populations in China[J].Scientia Silvae Sinicae,2013,49(12):88-96.[杨钤,谢映平,樊金华,等.日本松干蚧3个地理种群的遗传分化[J].林业科学,2013,49(12):88-96]
Zhong T.New advance of rapid amplification of cDNA ends[J].Foreign Medical Science(Molecular Biology Section),2002,24(1):7-11.[钟涛.cDNA 末端快速扩增技术新进展[J].国外医学(分子生物学分册),2002,24(1):7-11]
Zhang QH,Mao Y,Chen ZH.Strategy of intact cDNA cloning genome study[J].Journal of Chinese Biotechnology,2000,20(4):3-5.
Zhao H,Peng DL,Zhu JL,et al.RDNA-ITS-PCR in the molecular identification of plant-parasitic nematode[J].Plant Quarantine,2004,18(2):100-105.[赵鸿,彭德良,朱建兰.rDNA-ITS-PCR 技术在植物寄生线虫分子诊断中的应用[J].植物检疫,2004,18(2):100-105]
Zhu XS,Wang DY,Yu JN.Genetic variation of Eulecanium giganteum(Shinji)geographical populations and its correlation with ecological factors[J].Chinese Journal of Ecology,2014,33(5):1267-1273.[朱新帅,王登元,于江南.新疆枣球蜡蚧地理种群的遗传变异与生态因子的相关性[J].生态学杂志,2014,33(5):1267-1273]
Zhang LR,Xu DQ,Liu DQ.SSR marker,ISSR marker and their application to plant genetics and breeding[J].Journal of Agricultural University of Hebei,2002,25(1):90-93.[张立荣,徐大庆,刘大群.SSR 和ISSR 分子标记及其在植物遗传育种研究中的应用[J].河北农业大学学报,2002,25(1):90-93]
