离腹寡毛果实蝇性别决定基因transformer 2及其在昆虫中的进化
2015-12-09刘桂清万方浩
刘桂清,万方浩
(1.中国农业科学院植物保护研究所,植物病虫害生物学国家重点实验室,北京 100193;2.广东省昆虫研究所,广东省野生动物保护和利用公共实验室,广东省农业害虫综合治理重点实验室,广州 510260;3.青岛农业大学农学与植物保护学院,山东青岛 266109)
离腹寡毛实蝇属Bactrocera Macquart、小条实蝇属Ceratitis Macleay 和按实蝇属Anastrepha Schiner是实蝇科Tephritidae 中寄主范围最广,最具经济重要性的三大类群。世界已知的离腹寡毛果实蝇有561种,有记载的为害经济植物的种类有53种,其中具有经济重要性的种类36种,除橄榄实蝇B.oleae (Rossi)原产于南欧,现扩至非洲、亚洲印度北部和巴基斯坦西北部外,多数种主产旧大陆的亚洲热带、亚热带和暖温带,以及澳大利亚和南太平洋地区,少数种类分布于非洲南部和夏威夷群岛。该属包含许多世界性果蔬植物的检疫害虫,如桔小实蝇B.dorsalis (Hendel)、橄榄实蝇、瓜实蝇B.cucurbitae (Coquillett)、番石榴实蝇B.correcta (Bezzi)、桔大实蝇B.minax (Enderlein)、辣椒实蝇B.latifrons (Hendel)、昆士兰果实蝇B.tryoni (Froggatt)、扎氏果实蝇Bactrocera jarvisi(Tryon)、桃实蝇B.zonata (Saunders)、南瓜实蝇B.tau (Walker)等,可危害水果类寄主植物有2l 科37 属,蔬菜类寄主植物有4 科16 属(梁广勤等,1996)。
鉴于离腹寡毛实蝇属危害范围广及对国内外贸易造成的严重损失,世界上许多国家和地区都将离腹寡毛果实蝇列为重要的危险性检疫害虫,我国农业部2007年5月28 日颁布实施的《中华人民共和国进境植物检疫性有害生物名录》中,离腹寡毛实蝇属所有物种都被列为检疫性对象。除加强检疫外,昆虫不育技术 (Sterile insect technique,SIT)是根除重大实蝇害虫或预防其在新地区定殖的有效方法 (Klassen and Curtis,2005),该技术的核心是通过大规模释放不育雄虫与野生雌虫发生不育交配从而达到控制该地区害虫种群的目的(Knipling,1955)。在辐照处理蛹之前将靶标雌、雄虫分离是不育雄虫释放的前提,不育雄虫释放不仅可以提高SIT 的防治效果(Rendón et al.,2004),而且是病媒昆虫如蚊虫昆虫不育技术项目实施的先决条件 (Catteruccia et al.,2005)。
通过分析性别决定通路关键基因的进化,有助于阐明昆虫性别分化的分子机制,而且为发展雌、雄虫性别分离策略,提高昆虫不育昆虫技术的应用效果和开发新的害虫种群遗传控制方法奠定基础。目前,黑腹果蝇Drosophila melanogaster Meigen 性别决定分子机制研究相对成熟,为离腹寡毛果实蝇性别决定机制的比较研究提供了依据。在果蝇中,遗传级联 (sex lethal >transformer +transformer 2 >double sex)上游基因通过性别特异剪接方式产生不同的产物控制级联下游基因的剪接,从而控制雌雄分化 (Bell et al.,1991;Sánchez,2008)。Tra-2 基因是性别决定通路关键基因,tranformer (tra)需要与trasformer 2 (tra-2)相互结合形成Tra/Tra-2 复合体,调控下游基因double sex (dsx)pre-mRNA 以雌性特异方式剪接产生雌性特异Dsx 蛋白。胚胎显微注射tra-2 基因双链RNA 会导致XX 染色体胚胎发育为雄虫表型(Salvemini et al.,2009;Schetelig et al.,2012),这种性别反转有利于只释放不育雄虫的害虫防治项目。目前,有关离腹寡毛果实蝇tra-2 基因生物信息学方面的系统研究尚未见报道,故本研究首先克隆得到瓜实蝇tra-2 基因,对7种离腹寡毛果实蝇tra-2 基因结构进行比较研究,并对18种昆虫tra-2 基因进行系统发育和分子进化分析。
1 材料与方法
1.1 瓜实蝇基因组DNA 和总RNA 的提取和文库构建
瓜实蝇为广东出入境检验检疫局检验检疫技术中心室内饲养虫源。成虫以人工饲料饲养,幼虫以南瓜饲养。饲养条件为27℃±1℃、RH 75%±5%,光周期为14 h∶10 h (光照∶黑暗)。选取初羽化瓜实蝇雌、雄虫提取基因组DNA 和总RNA。
参照Bender et al.(1983)的方法提取瓜实蝇成虫基因组DNA。采用TriZol Reagent (Invitrogen)分别提取单头瓜实蝇雌、雄成虫总RNA,1.2%琼脂糖凝胶电泳检测 RNA 样本的完整性,NanophotometerTMP-Class (IMPLEN,德国)检测RNA 的纯度和浓度,每个样本取2 μg 总量的合格RNA,按 照 TransScript TM One-Step gDNA Removal and cDNA Synthesis SuperMix (北京全式金生物技术有限公司)第一链cDNA 合成试剂盒说明书构建cDNA 文库;按照BD SMARTer RACE cDNA Amplification Kit (BD Biosciences)试剂盒说明书分别构建雌、雄虫5'-RACE cDNA 和3'-RACE cDNA 文库,具体步骤及注意事项参照试剂盒说明书。
1.2 引物设计
根据地中海实蝇Ceratitis capitata、橄榄实蝇、加勒比按实蝇Anastrepha suspensa 同源tra-2 基因的氨基酸序列比对结果,设计简并引物F30/R530 扩增瓜实蝇tra-2 的中间片段,在获得的中间片段序列基础上,分别设计5' RACE 和3' RACE 特异引物,扩增tra-2 的cDNA 全长。参照番石榴实蝇同源tra-2 基因的分子结构,设计3 对外显子特异引物F13/R337,F327/R711,F686/R1140 用于扩增瓜实蝇tra-2 的内含子序列。本研究所用引物均使用Primer Premier 5.0 设计,引物序列详细信息见表1,所有引物均由生工生物工程(上海)股份有限公司合成。

表1 瓜实蝇tra-2 基因克隆引物Table 1 Primers for cloning Bactrocera cucurbitae homolog tra-2
1.3 瓜实蝇同源tra-2 基因克隆
以单头雌、雄虫第一链cDNA 为模板,采用简并引物F30/R530 扩增tra-2 的中间片段,PCR 反应体系为:灭菌ddH2O 水19.5 μL,10×PCR reaction buffer 2.5 μL,dNTP Mix (10mM)0.5 μL,上、下游引物(10 μmol/L)各0.5 μL,TranStart Taq DNA 聚合酶 (2.5U)0.5 μL,cDNA 模板1 μL。反应程序为95℃变性3 min;94℃,30 s,55℃,30 s,72℃,1 min,共35 个循环;72℃延伸7 min。PCR 产物经1 %琼脂糖凝胶电泳检测,割取目的大小条带并用 EasyPure Quick Gel Extraction Kit 琼脂糖凝胶DNA 回收试剂盒(北京全式金生物技术有限公司)说明书回收PCR 产物,参照pEASY-T1 Simple Cloning Kit 克隆试剂盒(北京全式金生物技术有限公司)说明书将回收产物连接到pEASY-T1 克隆载体,重组质粒转化Trans1-T1 感受态细胞,然后涂布在含有Ampicillin/X-gal/IPTG 的LB 平板上,37℃倒置培养12 h,蓝白斑筛选,挑取5 个白色菌落进行菌液PCR,并将菌落PCR 阳性克隆株挑入LB 液体培养基(含 Amp) 中过夜培养,按照 AxyPrepTMPlasmid Miniprep Kit 质粒提取试剂盒(Axygen)说明书提取质粒,送至生工生物工程(上海)股份有限公司进行测序。
为扩增tra-2 基因的cDNA 全长,以5' RACE Ready cDNA 为模板,5' RACE out/in 为引物扩增tra-2 5' UTR;以3' RACE Ready cDNA 为模板,3'RACE out/in 扩增tra-2 3' UTR,RACE-PCR 反应程序如下:95℃预变性3 min;94℃,30 s,72℃,2 min 扩增5 个循环;94℃,30 s;70℃,30 s;72℃,1 min 扩增5 个循环;94℃,30 s;68℃,30 s;72℃,1 min 扩增35 个循环;72℃延伸10 min。最后,以基因组DNA 为模板,分别以引物F13/R337、F327/R711、F686/R1140 扩增该基因的内含子,PCR 反应程序为:95℃退火3 min,94℃,30 s,60℃,45 s,72℃,1 min 扩增35 个循环,72℃延伸7 min。所有PCR 产物经1 %琼脂糖凝胶电泳检测,割胶回收,并将回收产物连接至pEASY-T1 克隆载体,重组质粒转化Trans1-T1 感受态细胞,筛选阳性克隆株,送测序,具体步骤同上。
1.4 序列分析
利用Jemboss 软件进行核苷酸序列翻译,并预测蛋白质的分子量、等电点和跨膜结构域。NCBI网站Blast 工具对序列进行相似性比对分析,利用Spidey 分析基因外显子和内含子的边界(http://www.ncbi.nlm.nih.gov/spidey/)。利 用 Bioedit(Hall,1999)和Clustal W (Thompson et al.,1994)软件进行氨基酸序列比对分析,参照地中海实蝇Tra-2 蛋白结构特征确定Tra-2 蛋白的功能结构域,如N 端和C 端精氨酸/丝氨酸富集区(Arginine/Serine rich region,RS)、RNA 识别基序 (RNA recognition motif,RRM) 等 (Salvemini et al.,2009)。
1.5 分子进化分析
本文中引用了18种昆虫的Tra-2 序列进行分析,物种及登录号分别为:双翅目:瓜实蝇(GenBank:KR056217)、桔小实蝇 (GenBank:KP342061)、番石榴实蝇(GenBank:KM658207)、橄榄实蝇(GenBank:AJ547623)、昆士兰果实蝇(GenBank:KJ443723)、扎式果实蝇 B.jarvisi(Tryon)(GenBank:KJ443722)、西印度按实蝇Anastrepha obliqua (Macquart) (GenBank:FN658607)、加勒比按实蝇A.suspensa (Loew)(GenBank:JN597290)、地中海实蝇 Ceratitis capitata (Wiedemann)(GenBank:EU999754)、家蝇Musca domestica L.(GenBank:AY847518)、铜绿蝇 Lucilia cuprinia (Wiedemann) (GenBank:FJ461620)、黑腹果蝇 Drosophila melanogaster Meigen (GenBank:M23633)、黑果蝇D.virilis Sturtevant (GenBank:XM_ 002049663)、拟暗果蝇D.pseudoobscura Frolova & Astaurov (GenBank:XM_ 001360568);鳞翅目:家蚕Bombyx mori L.(GenBank:NM_ 001126233);膜翅目:意大利蜜蜂Apis mellifera L.(GenBank:XM_ 001121070)、丽蝇蛹集金小蜂 Nasonia vitripennis Walker(GenBank:XP_ 001601106)。
利用MEGA 5 软件进行分子进化分析,采用Kimura 2 参数模型(Kimura,1980)估算核苷酸序列分异 (pNT),并计算转换与颠换的比值(transition/transversion ratio,R)。采用差异位点占所有比较位点的百分数(p-distance)估算Tra-2蛋白及其RS 和RRM 保守结构域的氨基酸序列分异(pAA)(Nei and Kumar,2000)。采用Nei-Gojobori 修正算法 (Zhang et al.,1998),假设transition/transversion ratio 为1,计算核苷酸同义突变率(pS)和非同义突变率(pN)。采用竞争删除选项(Compete deletion option)计算进化距离,邻接法(Neighbor-joining method)(Tamura et al.,2011)构建系统发育树,重复构建1000 次。同时利用DnaSP v.5 软件(Librado and Rozas,2009)分析核苷酸序列分异,所有突变位点经Tajima D检验。并通过计算Effective Number of Codons(ENC)、Codon BiasIndex (CBI)和Scaled Chisquare (Schi2)分析该基因的密码子偏好性。
2 结果与分析
2.1 瓜实蝇tra-2 基因结构
以第一链cDNA 为模板,以F30/R530 为引物,RT-PCR 扩增得到片段长度为501 bp 的产物,BlastX 分析发现推断的氨基酸序列与橄榄实蝇、暗色实蝇A.serpentina (Wiedemann)(GenBank:FN658613)Tra 2 蛋白序列的相似度为97%,推断得到的501 bp 的片段为瓜实蝇tra-2 基因序列。通过RACE 技术扩增得到tra-2 cDNA 全长1467 bp,开放读码框753 bp,共编码250 个氨基酸残基,分子量为295.59 kD,等电点PI 为6.87,有2 个跨膜结构域。瓜实蝇tra-2 与桔大实蝇同源tra-2 基因的序列一致性最高,达到90%,其次是与其他离腹寡毛果实蝇,与地中海实蝇tra-2 基因的序列一致性为83.6%,与黑腹果蝇的最低,仅为51.6%(表2)。7种离腹寡毛果实蝇tra-2 基因结构类似,均包含8 个外显子和7 个内含子,外显子和内含子大小及其翻译起始位点见图1,翻译起始密码子均位于第1 个外显子末端,与地中海实蝇tra-2 基因结构相似(Salvemini et al.,2009),与黑腹果蝇tra-2 (翻译起始密码子位于第2 个外显子)不同。

图1 瓜实蝇tra-2 基因结构及与同属其他种tra-2 基因结构比较Fig.1 Comparison of genomic organization of Bactrocera cucurbitae tra-2 with that from other Bactrocera species
2.2 瓜实蝇和其他离腹寡毛果实蝇Tra-2 蛋白
以该基因推导的Tra-2 氨基酸序列具有SR 蛋白家族的典型特征,如RNA 结合基序 (RNA Recognition Motif,RRM)及其侧翼的2 个精氨酸/丝氨酸富集结构域(Arginine/Serine rich domain,RS),暗示Tra-2 蛋白可以与其他蛋白结合发挥其功能。7种离腹寡毛果实蝇之间的序列一致性最高,其中扎式果实蝇与昆士兰果实蝇的氨基酸序列一致性最高,达到99.6%,橄榄实蝇和瓜实蝇的最低,为94%,番石榴实蝇与桔小实蝇的核苷酸序列一致性最高,达到97.6%,番石榴实蝇与瓜实蝇的最低,为87.4%。7种离腹寡毛果实蝇与地中海实蝇的序列一致性较高,与黑腹果蝇的最低,只有45.7% (表2)。在氨基酸残基数量上,瓜实蝇和桔大实蝇的Tra-2 蛋白均有250 个氨基酸残基数,比桔小实蝇、番石榴实蝇和扎实果实蝇的Tra-2 蛋白少1 个,比昆士兰果实蝇的Tra-2 蛋白少5 个,但氨基酸差异主要发生在RS1 和RS2结构域,所有物种的RNA 识别基序RRM 结构域均有相同的氨基酸残基数(图2)。
2.3 Tra-2 系统发育和分子进化分析
以家蚕、意大利蜜蜂、丽蝇蛹集金小蜂的Tra-2 蛋白作为系统进化树的外围支,重构双翅目Tra-2 蛋白的进化关系(图3),结果表明,家蚕、意大利蜜蜂、丽蝇蛹集金小蜂Tra-2 蛋白代表1 个基础分支,双翅目昆虫Tra-2 代表另1 个分支。在双翅目昆虫中,果蝇单独聚为一支,蝇科的家蝇和丽蝇科的铜绿蝇以及实蝇科果实蝇聚为另一支,在实蝇科果实蝇中,按实蝇属与小条实蝇属先聚在一起,然后与离腹寡毛实蝇属聚在一起。离腹寡毛实蝇属Tra-2 蛋白与小条实蝇属和按实蝇属的最为接近,其次是丽蝇科和蝇科的Tra-2 蛋白,这些物种Tra-2 蛋白表现为单一起源,而果蝇Tra-2 蛋白表现出实质性的分化。
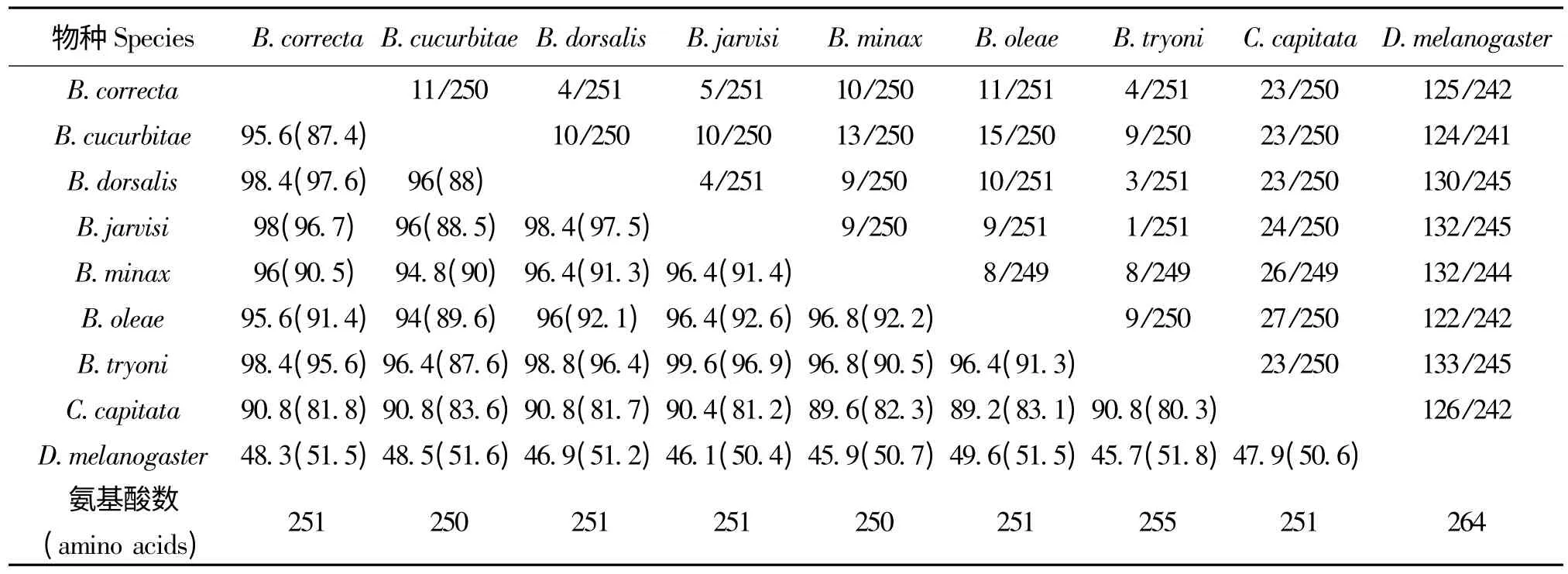
表2 Tra-2 氨基酸和cDNA 序列一致性Table 2 Tra-2 amino acids and cDNA sequence percentage identity

图2 双翅目昆虫Tra-2 氨基酸序列比较Fig.2 Comparison of the putative Tra-2 polypeptides from Diptera species
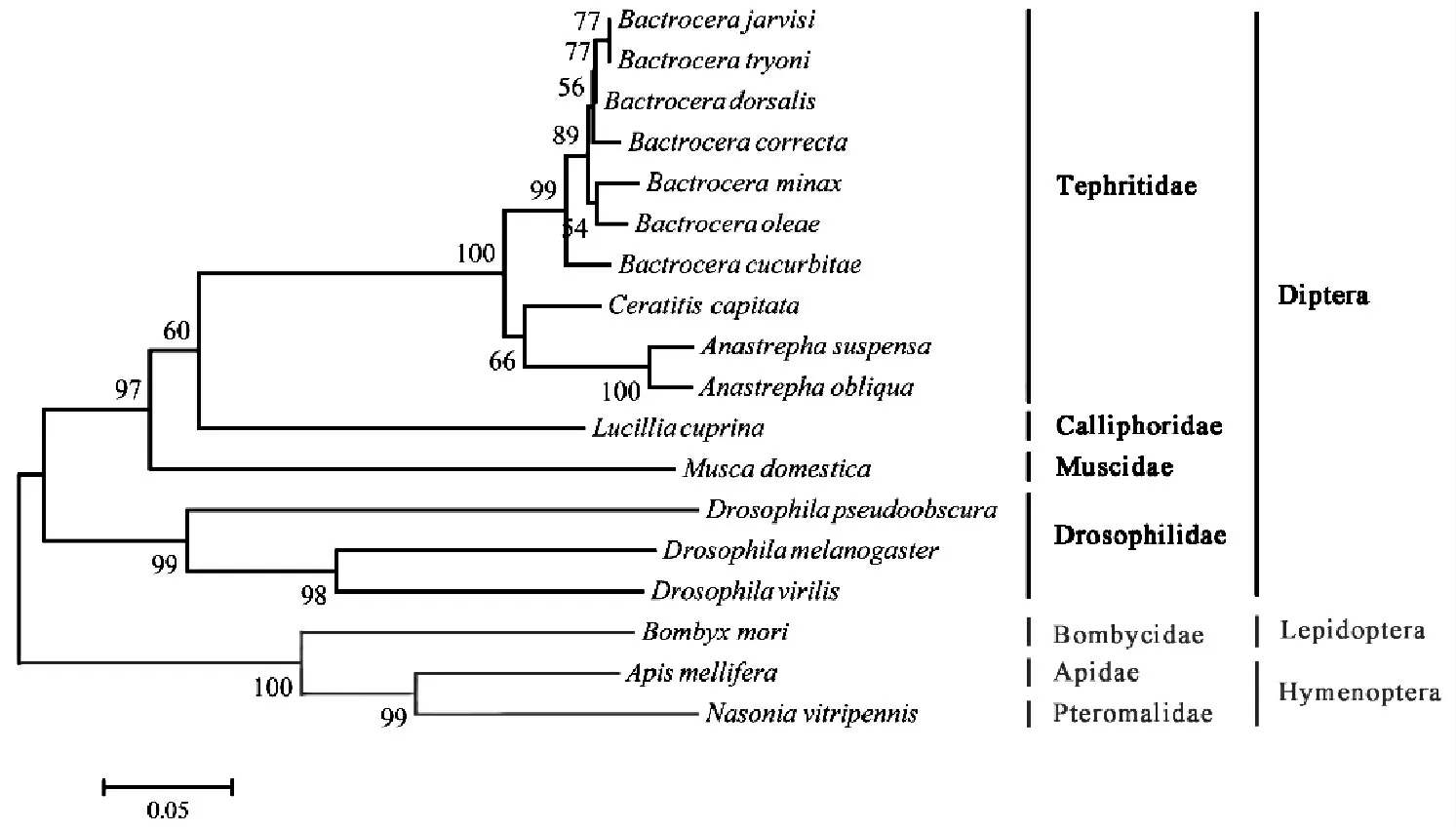
图3 邻接法构建18种昆虫Tra-2 蛋白系统发育树Fig.3 Phylogenetic tree reconstructed from 18 Tra-2 proteins using the neighbor-joining method

表3 Tra-2 基因的同义和非同义分异Table 3 Synonymous and nonsynonymous divergence of tra-2 genes
DnaSP5 分析18种昆虫tra-2 基因核苷酸序列比对的1152 个核苷酸位点,发现425 个核苷酸位点存在变异,其中372 个为简约信息位点(Parsimony information sites),53 个为单突变位点(Singleton sites)。Tra-2 基因序列分异结果如表3所示,同义分异Ks 均大于非同义分异Ka,其中同义替换位点数(SS)为133.33-144.50,同义替换核苷酸多样性均值Pi (s)为0.63599;非同义替换位点数(NSS)为395.50-406.67,非同义替换核苷酸多样性均值Pi (a)为0.25439,表明同义变异引起的tra-2 基因核苷酸变异高于非同义变异,核苷酸多样性在不同的核苷酸区段也存在差异,如图4A 所示,tra-2 基因RS1 和RS2 结构域的核苷酸多样性高于RRM 结构域。所有突变位点经Tajima D 检验,Tajima's D 为-0.94782,差异不显著(P >0.10)(图4B),表明tra-2 基因核苷酸变异符合中性进化模型。
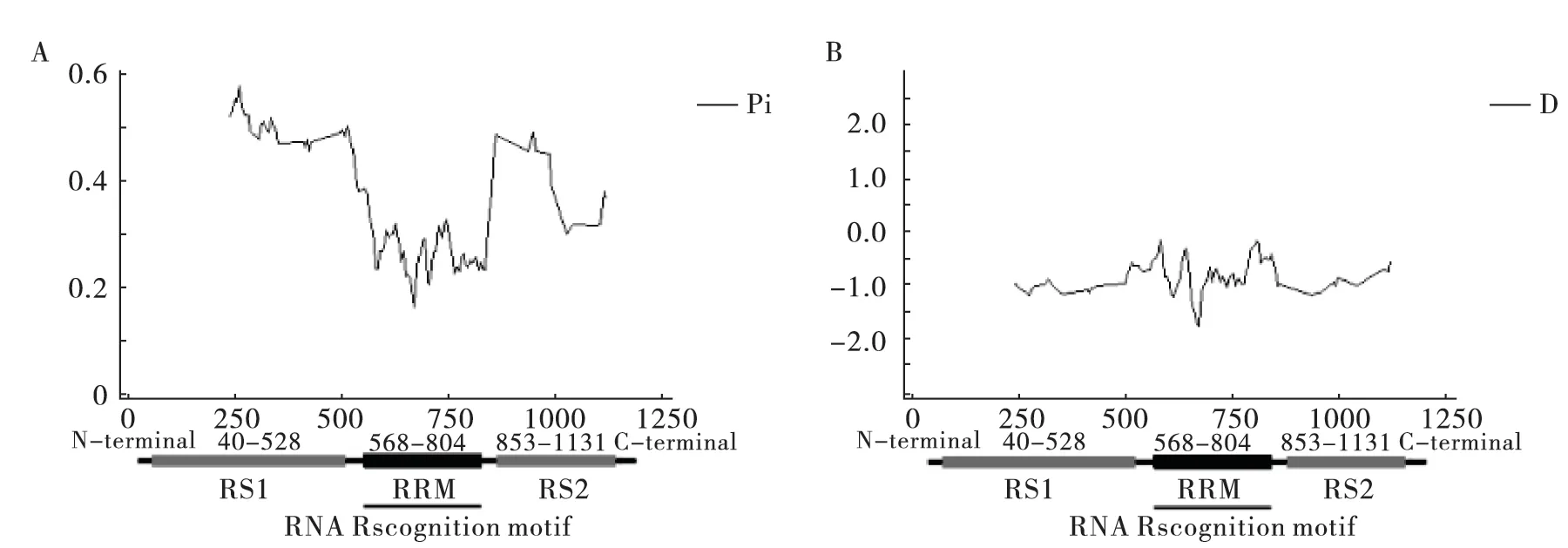
图4 18种昆虫tra-2 基因编码核苷酸多态性和Tajima 中性检测Fig.4 DNA polymorphism of tra-2 coding sequences from 18 insects and Tajima's test of neutrality
在氨基酸和核苷酸水平分析18种昆虫tra-2 基因的变异,同时分析Tra-2 蛋白RRM、RS1、RS2结构域的氨基酸和核苷酸变异,结果如表4 所示,tra-2 基因的总体氨基酸和核苷酸变异分别为0.333±0.019 和0.533±0.022,均高于实蝇科昆虫tra-2 基因 (氨基酸和核苷酸变异分别为0.073±0.010 和0.148± 0.010),实蝇科 虫tra-2蛋白高度保守。Tra-2 蛋白及其RRM、RS1 和RS2 结构域的同义突变水平均显著高于非同义突变水平(Z-test,P<0.001),RS1 结构域变异高于RS2,两者均高于RRM 结构域,说明tra-2 基因变异主要来源于同义突变,受净化选择压力,RRM与RS 结构域相比更为保守。密码子偏好性分析表明Tra-2 RRM 结构域密码子偏好性低于RS1 和RS2结构域。
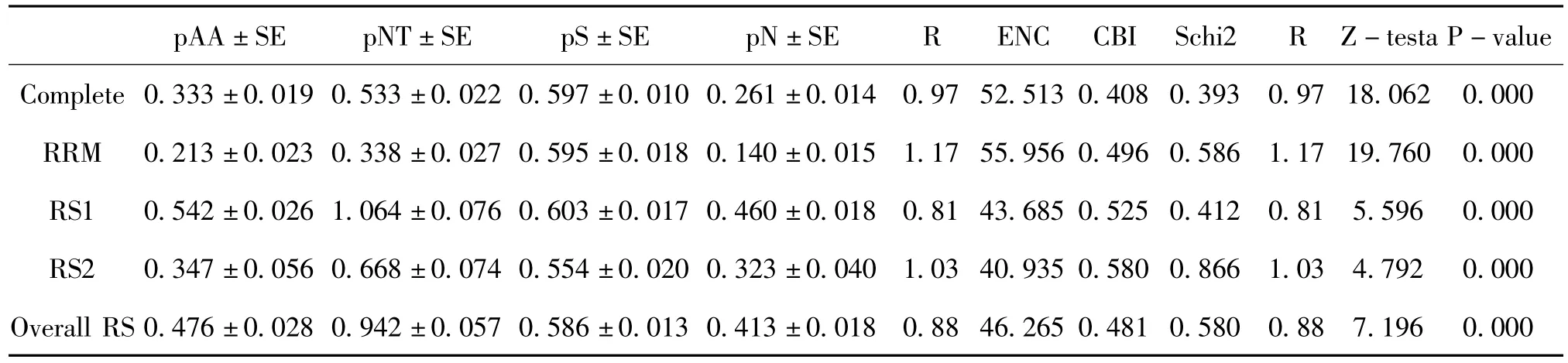
表4 18种昆虫Tra-2 蛋白及其保守结构域进化参数Table 4 Evolutionary parameters of the 18 Tra-2 proteins and their conserved domains
3 结论与讨论
通过同源基因克隆和RACE 技术克隆得到瓜实蝇tra-2 基因组DNA 和cDNA 全长,瓜实蝇tra-2基因与其他6种寡毛果实蝇和地中海实蝇的基因结构一致,均包含8 个外显子和7 个内含子,而且存在内含子剪接抑制ISS 位点和RNA 结合蛋白RBP1 结合位点。瓜实蝇tra-2 基因不存在性别特异剪接,只有1 个转录本,编码250 个氨基酸残基,编码氨基酸具有SR 蛋白家族特征,如RS 和RRM 结构域。与黑腹果蝇tra-2 存在4 个转录本(Amrein et al.,1990;McGuffin et al.,1998)不同,瓜实蝇tra-2 与地中海实蝇(Salvemini et al.,2009)、家蝇 (Burghardt et al.,2005)、铜绿蝇(Concha and Scott,2009)、加勒比实蝇(Schetelig et al.,2012)等同源tra-2 基因均只有1个转录本,并未检测到生殖系特异的转录本。序列分析结果表明瓜实蝇Tra-2 与其他离腹寡毛果实蝇和地中海实蝇的氨基酸和核苷酸的序列一致性均比黑腹果蝇的Tra-2 高,Tra-2 系统发育结果也表明7种离腹寡毛果实蝇与实蝇科的地中海实蝇和按实蝇、蝇科的家蝇和丽蝇科的铜绿蝇的遗传距离更近,为单一起源,而果蝇Tra-2 表现出明显的分异。分子进化分析结果表明tra-2 基因核苷酸变异主要来源于同义变异,Tra-2 蛋白受净化选择压力,以维持RNA 结合蛋白的典型特征,特别是RNA 识别基序RRM 的结构。
在地中海实蝇、橄榄实蝇、铜绿蝇和加勒比按实蝇等昆虫中,Tra-2 通过与Tra 结合形成Tra/Tra-2复合体,在性别决定中发挥双重功能:一方面通过与下游基因dsx 雌性特异外显子结合,调控dsx 雌性特异剪接;另一方面通过与tra 雄性特异外显子结合,抑制这些外显子被剪接翻译为成熟的mRNA (Concha and Scott,2009;Salvemini et al.,2009;Schetelig et al.,2012),Tra-2 内含子剪接抑制ISS 位点在其中发挥关键作用 (Ruiz et al.,2007)。由于Tra 缺乏RNA 结合蛋白结构域,因此,Tra 发挥作用必须与RNA 结合蛋白如Tra-2 相互作用(Black,2003)。tra-2 基因核苷酸序列同义和非同义替换率结果表明该基因受净化选择压力,通过维持Tra-2 蛋白结构的高度保守发挥其在昆虫性别决定中的作用。在果蝇中,Tra 已发生高水平的中性进化(Kulathinal et al.,2003),而Tra-2 精氨酸/丝氨酸富集区RS 是Tra 和Tra-2相互结合的位置,因此,tra-2 基因RS 结构域核苷酸变异很可能是为了适应tra 核苷酸变异,以保证Tra/Tra-2 复合体的形成,Tra-2 通过与Tra 协同进化发挥其在昆虫性别决定中的作用。
References)
Amrein H,Maniatis T,Nöthiger R.Alternatively spliced transcripts of the sex determining gene tra-2 of Drosophila encode functional proteins of different size [J].EMBO Journal,1990,9:3619-3629.
Bell LR,Horabin JI,Schedl P,et al.Positive auto-regulation of sex lethal by alternative splicing maintains the female determined state in Drosophila [J].Cell,1991,65:229-239.
Bender W,Spierer P,Hogness DS.Chromosomal walking and jumping to isolate DNA from the Ace and rosy loci and the bithorax complex in Drosophila melanogaster [J].Journal of Molecular Biology,1983,168:17-33.
Black DL.Mechanisms of alternative pre-messenger RNA splicing[J].Annual Review of Biochemistry,2003,72:291-336.
Burghardt G,Hediger M,Siegenthaler C,et al.The transformer 2 gene in Musca domestica is required for selecting and maintaining the female pathway of development [J].Development Genes and Evolution,2005,215:165-176.
Catteruccia F,Benton JP,Crisanti A.An anopheles transgenic sexing strain for vector control [J].Nature Biotechnology,2005,23:1414-1417.
Concha C,Scott MJ.Sexual development in Lucilia cuprina (Diptera,Calliphoridae)is controlled by the transformer gene[J].Genetics,2009,182:785-789.
Hall TA.BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT [J].Nucleic Acids Symposium Series,1999,41:95-98.
Kimura M.A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences[J].Journal of Molecular Evolution,1980,16:11-120.
Klassen W,Curtis CF.History of the sterile insect technique.In:Dyck V,Hendrichs J,Robinson AS,eds.Sterile Insect Technique[M].Netherlands:Springer,2005.
Knipling EF.Possibilities of insect control or eradication through the use of sexually sterile males [J].Journal of Economic Entomology,1955,48:459e462.
Kulathinal RJ,Skwarek L,Morton RA,et al.Rapid evolution of the sex determining gene,transformer:Structural diversity and rate heterogeneity among sibling species of Drosophila[J].Molecular Biology Evolution,2003,20:441-452.
Liang GQ,Yang GH,Liang F,et al.Bactrocera Species in Asia-Pacific Area[M].Guangzhou:Guangdong Science and Technology Press,1996.[梁广勤,杨国海,梁帆,等.亚太地区寡毛实蝇.广州:广东科技出版社,1996]
Librado P,Rozas J.DnaSP v5:A software for comprehensive analysis of DNA polymorphism data [J].Bioinformatics,2009,25 (11):1451-1452.
McGuffin ME,Chandler D,Somaiya D,et al.Auto-regulation of transformer-2 alternative splicing is necessary for normal male fertility in Drosophila[J].Genetics,1998,149:1 477-1 486.
Nei M,Kumar S.Molecular Evolution and Phylogenetics [M].New York:Oxford University Press,2000.
Rendón P,McInnis D,Lance D,et al.Medfly (Diptera:Tephritidae)genetic sexing:large-scale field comparison of males-only and bisexual sterile fly releases in Guatemala[J].Journal of Economic Entomology,2004,97:1547-1553.
Ruiz MF,Milano A,Salvemini M,et al.The gene transformer of Anastrepha fruit flies (Diptera,tephritidae)and its evolution in insects[J].PLoS ONE,2007,2:e1239.
Salvemini M,Robertson M,Aronson B,et al.Ceratitis capitata transformer-2 gene is required to establish and maintain the autoregulation of ctra,the master gene for female sex determination[J].International Journal of Developmental Biology,2009,53:109-120.
Sánchez L.Sex-determining mechanisms in insects[J].International Journal of Developmental Biology,2008,52:837-856.
Schetelig MF,Milano A,Saccone G,et al.Male only progeny in Anastrepha suspensa by RNAi-induced sex reversion of chromosomal females [J].Insect Biochemistry and Molecular Biology,2012,42 (1):51-57.
Tamura K,Peterson D,Peterson N,et al.MEGA 5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods [J].Molecular Biology and Evolution,2011,28:2731-2739.
Thompson JD,Higgins DG,Gibson TJ,et al.CLUSTAL W.Improving the sensitivity of progressive multiple sequence alignments through sequence weighting,position specific gap penalties and weight matrix choice [J].Nucleic Acids Research,1994,22:4673-4680.
Zhang J,Rosenberg HF,Nei M.Positive Darwinian selection after gene duplication in primate ribonuclease genes[J].Proceedings of the National Academy of Sciences of the United States of America,1998,95:3708-3713.
