油橄榄查尔酮合酶与查尔酮异构酶基因全长的克隆及序列分析
2015-12-13陈文拴黄乾明陈华萍杨泽身王安逸苏光灿
陈文拴,黄乾明,*,陈华萍,杨泽身,王安逸,苏光灿
(1.四川农业大学理学院,四川 雅安 625014;2.凉山州中泽新技术开发有限责任公司,四川 西昌 615000)
油橄榄查尔酮合酶与查尔酮异构酶基因全长的克隆及序列分析
陈文拴1,黄乾明1,*,陈华萍1,杨泽身2,王安逸2,苏光灿2
(1.四川农业大学理学院,四川 雅安 625014;2.凉山州中泽新技术开发有限责任公司,四川 西昌 615000)
查尔酮合酶(chalcone synthase,Chs)与查尔酮异构酶(chalcone isomerase,Chi)是植物类黄酮合成代谢途径中的两个关键酶。采用同源克隆、反转 录聚合酶链式反 应(reverse transcription polymerase chain reaction,RT-PCR)、融合引物嵌套PCR(fusion primer and nested integrated PCR,FPNI-PCR)与3’-cDNA末端快速扩增技术(rapid amplification of cDNA end,RACE)相结合,分别 克隆出油橄榄chs与chi基因全长。序列分析表明,油橄榄chs基因全长DNA(GenBank登录号:KF935223.1)和cDNA(GenBank登录号:KF935224.1)序列分别为2 085 bp和1 173 bp,该基因的开放阅读框(open read ing frame,ORF)编码390 个氨基酸,其氨基酸序列与葡萄和番茄全长Chs氨基酸序列的相似度在90%左右。生物信息学分析表明,该基因编码的蛋白属于Chs蛋白家族。油橄榄chi基因全长DNA(GenBank登录号:KF886190)和cDNA(GenBank登录号:KF886191)序列分别为1 373 bp和750 bp,其ORF编码249 个氨基酸,该氨基酸序列与已报道的其他植物Chi氨基酸序列的相似度最高在65%左右,序列中包含一段由198 个氨基酸残基组成的具有分子内裂解酶活性的功能域,符合 Chi蛋白家族特征。
油橄榄;查尔酮合酶;查尔酮异构酶;基因克隆;序列分析
油橄榄(Olea europaea)属于木犀科,木樨榄属,常绿阔叶乔木,其果实主要用来压榨生产有“液体黄金”之称的橄榄油[1-2]。在油橄榄中除脂肪酸外关注较多的是具有抗氧化活性的物质,其中以黄酮类化合物最具代表性[3-4]。研究发现,黄酮在植物遭受辐射损伤、病虫害与病原菌侵染等逆境胁迫过程中发挥着重要的抗逆作用,同时也与植物花朵和果实颜色的形成有着密切的关系[5-6],此外还可以作为药物具有抗氧化、抗癌和改善心血管系统等多种药理作用[7-9],很具开发潜力和研究价值。
查尔酮合酶(chalcone synthase,Chs)与查尔酮异构酶(c halcone isomerase,Chi)是植物类黄酮合成代谢途径中的两个关键酶。1分子黄酮类化合物共同生物合成前体的4-香豆酰CoA(4-coumaroyl CoA)与3分子的丙二酰CoA(malonyl CoA)在Chs的催化下生成黄酮类物质15碳骨架结构的查尔酮,查尔酮在Chi的作用下转变为柚皮素[5,10-12],查尔酮与柚皮素均可以作为底物进入其他不同黄酮类物质的合成代谢支路[5]。目前对植物chs与chi基因的研究多集中在粮食、蔬菜和花卉等草本植物,而在木本植物中则相对较少,虽然有关于油橄榄中这两个基因片段的报道,如国家生物技术信息中心(National Center for Biotechnology Information,NCBI)中已收录了油橄榄chs基因 571 bp的cDNA片段(GenBank登录号:AF384049.1)与chi基因500 bp的cDNA片段(GenBank登录号:GU646679.1),但缺乏对这两个关键酶 基因完整序列以及结构分析等方面的研究。本研究在油橄榄chs与chi基因片段的基础上,采用同源克隆、反转录聚合酶链式反应(reverse transcription polymerase chain reaction,RT-PCR)、融合引物嵌套PCR(fusion primer and nested integrated PCR,FPNI-PCR)与3’-cDNA末端快速扩增技术(rapid amplification of cDNA end,RACE)相结合,分别克隆出油橄榄chs与chi基因的全长DNA与cDNA,并分析了基因以及编码蛋白的分子特征与功能属性,以期为今后从分子水平揭示油橄榄黄酮的合成代谢调控机制、抗逆作用机理以及改良种质资源等研究提供参考。
1 材料与方法
1.1 材料与试剂
实验材料取自西昌凉山州中泽新技术开发有限责任公司北河油橄榄种植基地配多灵品种的幼嫩叶片。
植物RNA提取试剂盒 天恩泽基 因科技有限公司;通用型DNA纯化回收试剂盒、质粒DNA小量提取试剂盒、大肠杆菌DH5α感受态细胞 天根生化科技(北京)有限公司;Premix Taq、PrimeSTAR☒Max DNA Polymerase、pMDTM19-T Vector Cloning Kit、PrimeScriptTMRT Reagent Kit 日本TaKaRa公司;实验中所合成的引物 美国Invitrogen公司;其他 化学试剂均为国产或进口分析纯。
1.2 仪器与设备
A100/A200型基因扩增仪 郎基科学仪器有限公司;DYY-6B型稳压稳流电泳仪 北京市六一仪器厂;UV-3000型紫外分析仪 上海嘉鹏科技有限公司;HG303-4电热恒温培养箱 南京电器三厂;HYA恒温摇床 中国科学院武汉科学仪器厂。
1.3 方法
1.3.1 油橄榄总DNA与总RNA的提取及cDNA第一链的制备
采用改良的C T A B法提取油橄榄叶片中的总DNA[13];抽提叶片总RNA后,用PrimeScriptTMRT Reagent Kit和自备引物3’CDS Primer A[14](实验中所用引物见表1)合成油橄榄cDNA第一链。

表1 实验所用引物Table 1 Primers used in this study
1.3.2 基因的克隆与测序
根据同源序列比对结果与NCBI中收录的油橄榄chs基因的cDNA片段序列分别设计扩增油橄榄chs基因片段的上下游引物SU与SD,根据NCBI中收录的油橄榄chi基因的cDNA片段序列设计一对上下游引物IU与ID,以总DNA为模板分别扩增油橄榄chs与chi基因的DNA片段,扩增产物经胶回收后与T载体进行连接,再经转化大肠杆菌DH5α感受态细胞,挑选阳性转化子扩大培养后提质粒测序。
根据测序结果设计扩增油橄榄chs基因5’端未知序列的3 条巢式特异引物AS1、AS2、AS3。以总DNA为模板,用特异引物AS1与 另设计的融合有特异引物接头的随机引物FAD1、FAD2、FAD3、FAD4分别进行第一轮PCR反应,反应参数:94 ℃ 1 min,98 ℃ 1 min;94 ℃ 30 s,55 ℃ 1 min,72 ℃ 2 min,5 个循环;94 ℃ 30 s,25 ℃ 3 min,72 ℃ 2 min;(94 ℃ 30 s,55 ℃ 1 min,72 ℃ 2 min,2 个循环;94 ℃ 30 s,44 ℃ 1 min,72 ℃ 2 min)15 个循环;72 ℃ 10 min,4 ℃结束[15]。以第一轮PCR产物为模板,用特异引物AS2与FSP1进行第 二轮PCR反应,反应参数:94 ℃ 1 min;94 ℃ 30 s,55 ℃ 15 s,72 ℃ 2 min,30 个循环;72 ℃ 10 min,4 ℃结束[15]。以第二轮PCR产物 为模板,用特异引物AS3与FSP2进行第三轮PCR反应,反应参数:94 ℃ 1 min;94 ℃ 30 s,56 ℃ 15 s,72 ℃ 2 min,30 个循环;72 ℃ 10 min,4 ℃结束[15]。对第三轮有明显条带的PCR产物回收连接克隆后进行测序。根据chi基因的测序结果设计扩增其5’端未知序列的3 条巢式特异引物AI1、AI2、AI3,而后操作程序同上,特异引物的Tm见表1。
根据油橄榄chs基因的cDNA片段序列设计用于扩增其3’端未知cDNA的两条巢式特异引物ZS1与ZS2。以反转录cDNA第一链为模板,用特异引物ZS1与通用引物UPML和UPMS(两者物质的量之比为1∶5)一起进行第一轮PCR反应,反应参数:94 ℃ 2 min;94 ℃ 30 s,72 ℃ 2 min,5 个循环;94 ℃ 30 s,66 ℃ 30 s,72 ℃ 2 min,5 个循环;94 ℃ 30 s,61 ℃ 30 s,72 ℃ 2 min,20 个循环;72 ℃ 10 min,4 ℃结束[14]。以第一轮PCR产物为模板,用特异引物ZS2与通用引物NUP进行第二轮PCR反应,反应参数:94 ℃ 1 min;94 ℃ 30 s,57 ℃ 15 s,72 ℃ 2 min,30 个循环;72 ℃ 10 min,4 ℃结束[14]。对第二轮有明显条带的PCR产物回收连接克隆后进行测序。用巢式特异引物ZI1与ZI2扩增油橄榄chi基因3’端未知cDNA,特异引物的Tm见表1,操作程序同上。
确定基因两端的非翻译区(untranslated regions,UTR)后,在基因两端分别设计扩增油橄榄chs基因全长的上下游引物HSU和HSD与扩增油橄榄chi基因全长的上下游引物HIU和HID,分别以油橄榄总DNA与cDNA第一链为模板扩增基因的全长DNA与cDNA。
1.3.3 序列的生物信息学分析
序列比对分析确定基因的外显子、内含子、开放阅读框(open reading frame,ORF)以及所编码的氨基酸序列,并进行相似度分析;采用SignalP-4.1进行信号肽预测;利用SOPMA与SWISS-MODEL进行蛋白质空间结构的预测分析;用SMART进行结构域分析;利用MEGA 5构建系统进化树。
2 结果与分析
2.1 基因克隆
用引物SU与SD扩增油橄榄chs基因片段,电泳检测到一条1 700 bp左右的条带(图1a),用引物IU与ID扩增油橄榄chi基因片段,电泳检测到一条800 bp左右的条带(图1b),经序列分析确定为油橄榄chs与chi基因的DNA片段。
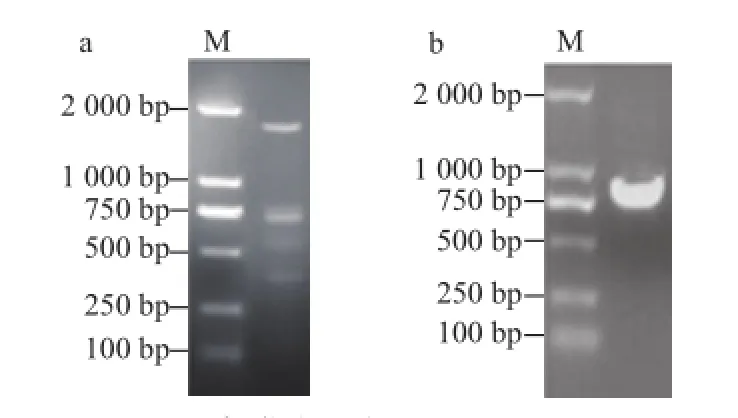
图1 1 chschs(a)与achichi(b)基因片段的扩增结果Fig.1 PCR amplified products of chs (a) and chi (b) gene fragments
扩增油橄榄chs基因的5’端未知序列时,用引物FAD1~FAD4进行的巢式PCR均扩增出了明亮的条带(图2a),但FAD4的终产物分子质量最大,有700 bp左右,最终选取该条带测序。扩增油橄榄chi基因的5’端未知序列时,只有用引物FAD3进行的巢式PCR扩增出一条约500 bp的明亮条带(图2b)。测序结果经序列比对分析表明,均获得了包括油橄榄chs与chi基因起始密码子ATG在内的5’端未知序列。扩增油橄榄chs与chi基因的3’端未知序列,最终均获得一条550 bp左右的明亮条带(图3),测序结果显示已扩增到以上两个基因包括Poly A在内的3’端cDNA序列。
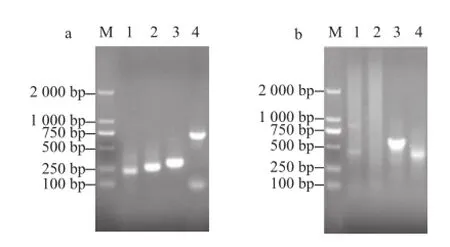
图2 扩增chs(a)与chi(b)基因的5端未知序列Fig.2 Amplification of the 5’-unknown sequences of chs (a) and chi (b) genes

图3 3 chschs(a)与achichi(b)基因的33’-RACE扩增ACEFig.3 3’-RACE products of chs (a) and chi (b) genes
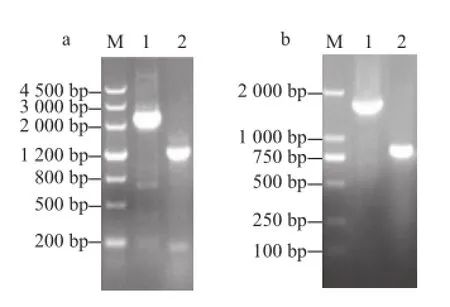
图4 4 chschs(a)与achichi(b)基因全长的扩增Fig.4 Amplification of the full-length chs (a) and chi (b) genes

用引物HSU与HSD扩增油橄榄chs基因的全长DNA与cDNA,分别得到一条2 200 bp左右与一条1 300 bp左右的条带(图4a),测序结果为2 219 bp与1 307 bp。用引物HIU与HID扩增油橄榄chi基因的全长DNA与cDNA,分别得到一条1 400 bp左右与一条800 bp左右的条带(图4b),测序结果为1 397 bp与774 bp。
2.2 基因的核苷酸序列分析
克隆获得的油橄榄chs基因全长DNA(GenBank登录号:KF935223.1)包含有3 个外显子(65~242、884 ~1 576、1 848~2 149 bp),两个内含子核苷酸序列均符合典型的gt-ag剪切方式,全长cDNA(GenBank登录号:KF935224.1)包含一个1 173 bp的完整ORF,编码一条390 个氨基酸的多肽链(图5a)。序列比对发现,油橄榄与NCBI中已收录的金鱼草chs基因相似度最高,两者完整ORF的相似度为78.35%,其次与牵牛花、葡萄和洋地黄的相似度在76%~78%之间。
所克隆的油橄榄chi基因全长DNA(GenBank登录号:KF886190)包含4 个外显子(23~128、221~379、785~1 008、1 135~1 395 bp),3 个内含子均符合gt-ag剪切方式,全长cDNA (GenBank登录号:KF886191)中有一个编码249 个氨基酸多肽链的完整ORF(图5b)。同源比对显示油橄榄与蔷薇科的梨和樱桃相似度较高,完整ORF的相似度分别为68.84%与67.81%,与山茶科的茶树、忍冬科的金银花的相似度分别为66.8%和64.59%。
2.3 编码的氨基酸序列分析
氨基酸序列同源比对发现,油橄榄Chs多肽链中有3 段较长的保守序列(第174~198、254~273、367~386位),其完整多肽链序列与葡萄的相似度最高,相似度为90.26%,与番茄和马铃薯的相似度分别为90.00%和89.49%。经ProtParam在线分析,油橄榄Chs多肽链的分子质量为43.044 5 kD,等电点为6.24,含有44 个带正电荷的氨基酸残基,48 个带负电荷的氨基酸残基,分子式为C1915H3053N519O568S19,不稳定系数为42.98,属于不稳定性蛋白。采用SignalP-4.1预测油橄榄Chs蛋白定位于细胞质。
油橄榄Chi与梨、樱桃、茶树、金银花以及唇形科的黄芩和藿香的相似度相对较高,完整多肽链序列的相似度分别为63.86%、64.66%、64.54%、65.87%、65.06%、 61.78%。该蛋白的分子质量为26.64 kD,等电点为6.01,含有25 个带正电荷的氨基酸残基,27 个带负电荷的氨基酸残基,分子式为C1202H1890N306O368S4,不稳定系数为45.58,属于不稳定性蛋白。蛋白质亚细胞定位预测油橄榄Chi蛋白存在于细胞质。
2.4 油橄榄Chs与Chi蛋白的结构分析
用S O P M A与S W I S S-M O D E L在线进行蛋白质结构分析显示,油橄榄Chs蛋白中有44.36%的α-螺旋区、6.92%的β-转角和33.08%的无规卷曲(图6)。用SMART在线进行结构域分析显示,油橄榄C h s的 N端包含一段具有酰基转移酶活性的结构域,C端包含一段具有氨基酰基转移酶活性的结构域,序列内部包含一段3-氧酰基-[酰基载体蛋白(acyl carrier protein,ACP)]合酶Ⅲ结构域,多肽链中最长的氨基酸保守序列(AKDLAENNKGARVLVVCSEITAVTF)就位于该结构域内,属于Chs蛋白家族中的一员。

图5 油橄榄chschs(a)与achichi(b)基因的核苷酸序列和编码的氨 基酸序列Fig.5 The nucleotide sequences and deduced amino acid sequences of chs (a) and chi (b) gen es from O. eu ropaea
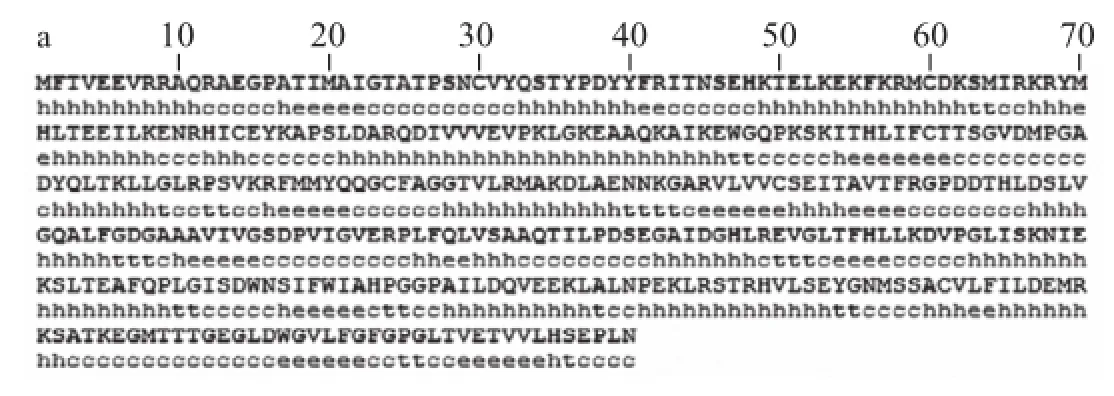

图6 油橄榄Chs蛋白的二级(a)和三级(b)结构分析Fig.6 Protein structure analysis of Chs from O. europaea
蛋白质二级结构分析显示,油橄榄Chi蛋白中有9 处α-螺旋,合计占序列全长的42.57%;有10 处β-转角,占序列全长的8.03%;无规卷曲有19 处,占序列全长的32.53%(图7a);其中6处较大的α-螺旋在同源建模生成的三维条带图中清晰可见(图7b)。结构域分析显示油橄榄Chi蛋白包含一段有198 个氨基酸残基组成的具有分子内裂解酶活性的功能域,该功能域中有一段Bet v 1结构域,符合Chi蛋白家族特征。带有Bet v 1结构域的蛋白质广泛存在于双子叶植物的细胞质中,与植物的发病机理有着密切的联系,其大量表达与病原体感染、创伤或其他外界胁迫因素密切相关[16-17],这也就从分子水平上解释了植物在遭受逆境胁迫时其组织细胞中黄酮含量升高的原因。

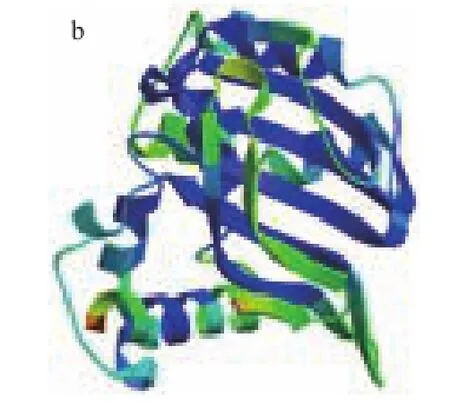
图7 油橄榄Chi蛋白的二级(a)和三级(b)结构分析Fig.7 Protein structure analysis of Chi from O. europaea
2.5 系统进化树的构建
利用MEGA 5软件通过邻接法构建植物Chs氨基酸序列的系统进化树显示(图8a),在植物分类学中同属于菊亚纲的油橄榄、洋地黄、金鱼草、藿香和紫苏在进化树中同处于一个大的分支,表明它们有着共同的起源或进化模式。基于Chi氨基酸序列构建的进化树反映油橄榄与甘薯和花生的亲缘关系要近于其他物种(图8b),但它们在植物分类学中的亲缘关系相差较远,由此可见,依据Chi氨基酸序列构建的进化树不能完全真实地反映不同物种间的自然演化进程,其结果对于明确判断植物间的亲缘关系仅能提供一定的参考。
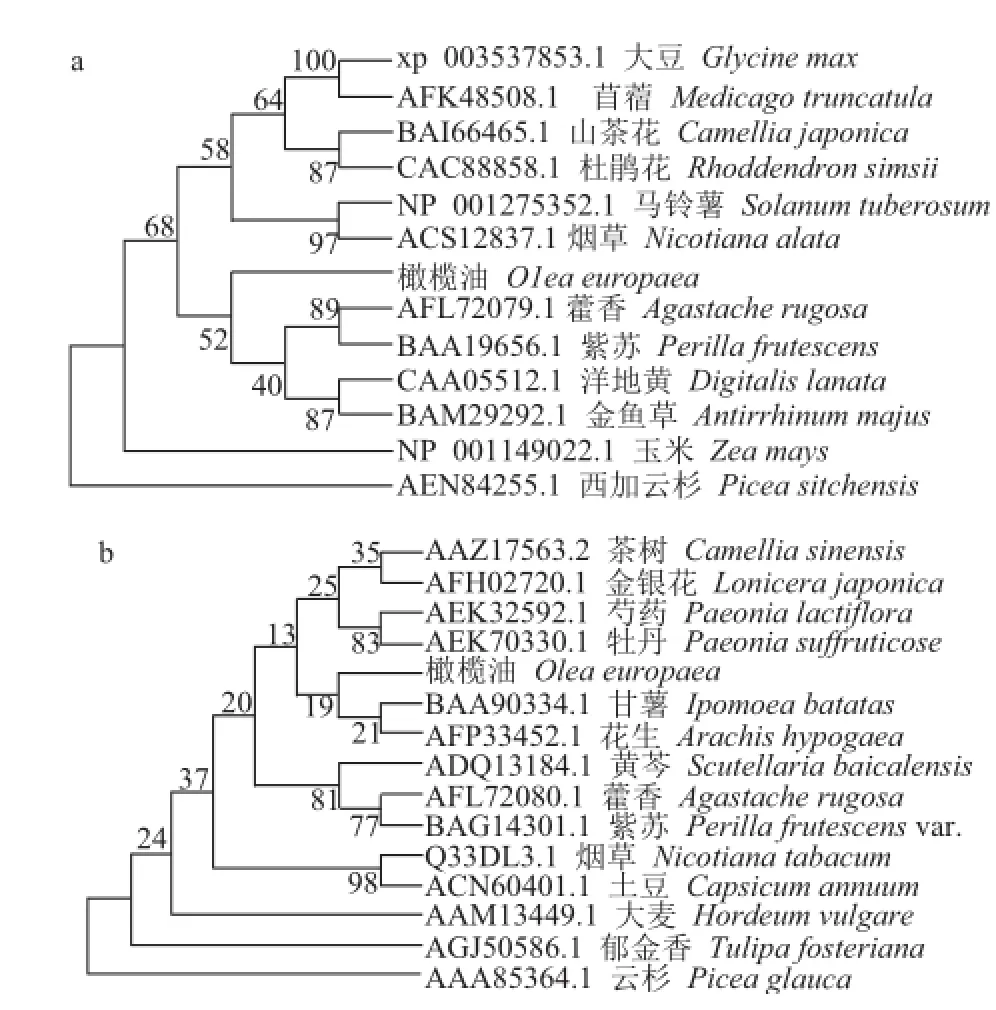
图8 不同来源的Chs(a)与Chi(b)氨基酸序列聚类结果Fig.8 Phylogenetic analysis of amino acid sequences of Chs (a) and Chi (b) from different plants
3 讨 论
植物Chs是Ⅲ型聚酮合酶家族中的一员,分子遗传学研究发现,在进化过程中该酶的功能可能有多次转变[18]。植物chs基因为多基因家族,在不同组织器官中存在着不同的表达模式[19],从而影响着植物组织中黄酮种类与含量的分布。由于植物的花朵发挥着特殊的生理功能,一般情况下植物花朵中chs基因的表达量最高[20]。鉴于chs基因在分化方向与表达模式等方面的多样性,因此非常适合作为研究基因表达调控与调查基因家族起源的模式系统。对已报道的植物chs基因分析发现,除了金鱼草、虎杖与苔藓外[21-22],绝大多数植物chs基因均由2 个外显子和1 个内含子构成,但在本实验中克隆到的油橄榄chs基因和金鱼草中该基因一样有3 个外显子和2 个内含子,且两者编码序列的同源性在所有比对序列中最高,表明它们可能有着相似的演化进程。不过经序列分析发现,油橄榄chs基因的内含子1位于第60位半胱氨酸密码子内部,在第一与第二个碱基之间,外显子1编码59 个氨基酸,其保守性低于外显子2、3,油橄榄Chs蛋白富含可能参与同源二聚体形成的亮氨酸,活性中心由4 个高度保守的氨基酸残基(C164、H303、N336、F215)构成,在多肽链的C端有典型的Chs多基因家族特异标签序列WGVLFGFGPG,从这些高度一致的特征可以看出植物中chs基因的结构趋于保守[23],推测该基因可能有着共同的起源。
Chi是类黄酮合成途径中第一个被认识的酶,在该途径中发挥着重要的 承接作用,由Chi参与的查尔酮异构化反应速率是自发异构化反应速率的107倍,且在酶促反应中有99.999%以上的产物可以直接作为下一步代谢反应的底物[24-25],植物chi基因表达常发生在生命活动旺盛的部位推测与该酶高效的催化活性有关。Chi蛋白具有特殊的三维折叠结构,因此很适合作为植物特有的基因标记[26]。根据作用底物的不同可以把植物中的Chi分为两类:Ⅰ型Chi只能催化6’-羟基查尔酮转化为5’-羟基黄烷酮;Ⅱ型Chi则可以催化6’-羟基查尔酮和6’-脱氧查尔酮分别生成相应的5’-羟基黄烷酮和5’-脱氧黄烷酮[27],此外在某些微生物中发现了与植物直系同源的Chi[28]。Ⅰ型Chi在绝大多数植物中均有存在,在豆科植物中发现的Chi则属于Ⅱ型,仅在刺甘草与百脉根等豆科植物中发现有以上两种类型Chi的存在[27,29],据此推测油橄榄chi基因编码的蛋白属于Ⅰ型,催化底物为6’-羟基查尔酮。蛋白功能位点分析表明油橄榄Chi的氨基酸序列中存在着4 个重要的催化位点T50、Y108、N115、S192与7 个底物结合位点R38、G39、L40、F49、I52、K111、V112,研究发现在豆科植物中Ⅱ型Chi的第192位左右的氨基酸常由T替换了S[24],由此更进一步确定油橄榄Chi属于Ⅰ型。油橄榄chi基因和大多数植物chi基因一样有典型的结构,由4 个外显子和3 个内含子构成,有资料显示大麦chi有3 个外显子[30],白菜的Brchi-2、甘蓝的Bochi-2以及甘蓝型油菜的Bnchi-2均有5 个内含子[31],在豆科植物光叶百脉根5号染色体上串联的4 个chi基因中,除了Ljchi-2含有3 个外显子外,其他均与典型结构相符[27],chi基因内含子表现出的这种多样性有可能会为研究该基因的演化发展提供相关的模型。
对植物球茎、花药与花冠发育过程的研究发现,Chs与Chi的积累和消失受紫外光辐射诱导调控,并且存在协同效应[32]。分子水平上的研究发现植物Chs、Chi与二氢黄酮醇-4-还原酶是以多酶复合体的形式存在于内质网来行使催化功能的[33],在这个多酶复合体中,chi基因的大量表达可受外界损伤、病原菌感染以及辐射等多种因素诱导,因此Chi是类黄酮合成途径中一个重要的节点。科研人员[34]将水母雪莲的chi基因转入烟草,结果提高了烟草总黄酮的含量,将外源chs基因和黄酮醇合酶基因共同转入番茄后提高了番茄中黄酮醇的含量[35],将二氢黄酮醇-4-还原酶基因、chs和chi基因与共同转化至马铃薯后提高了花色素苷的含量[36],最终均改善了植株的抗氧化能力,但是仅将外源chs基因导 入矮牵牛与烟草则出现了共抑制现象,且在矮牵牛中还发现了雄性不育现象[37-38]。因此对基因功能的研究是一个复杂的探索过程,在利用基因工程方法对传统的种质资源进行遗传改良时,有必要对功能基因结构与功能的关系进行深入的探索,并且要充分考虑到它们的生理效应以及遗传效应。
[1] LONG H S, TILNEY P M, van WYK B E. The ethnobotany and pharmacognosy of Olea europaea subsp. africana (Oleaceae)[J]. South African Journal of Botany, 2010, 76(2): 324-331.
[2] CARAMIA G, GORI A, VALLI E, et al. Virgin olive oil in preventive medicine: from legend to epigenetics[J]. European Journal of Lipid Science and Technology, 2012, 114(4): 375-388.
[3] SKERGET M, KOTNIK P, HADOLIN M, et al. Phenols,proanthocyanidins, flavones and flavonols in some plant materials and their antioxidant activities[J]. Food Chemistry, 2005, 89(2): 191-198.
[4] RYAN D, LAWRENCE H, PRENZLER P D, et al. Recovery of phenolic compounds from Olea europaea[J]. Analytica Chimica Acta,2001, 445(1): 67-77.
[5] SCHIJLEN E G, RIC de VOS C H, van TUNEN A J, et al. Modification of flavonoid biosynthesis in crop plants[J]. Phytochemistry, 2004, 65(19): 2631-2648.
[6] NISHIHARA M, NAKATSUKA T. Genetic engineering of flavonoid pigments to modify flower color in floricultural plants[J]. Biotechnology Letters, 2011, 33(3): 433-441.
[7] BEN AMMAR R, BHOURI W, BEN SGHAIER M, et al. Antioxidant an d free radical-scavenging properties of three flavonoids isolated from the leaves of Rhamnus alaternus L. (Rhamnaceae): a structureactivity relationship study[J]. Food Chemistry, 2009, 116(1): 258-264.
[8] LIU Hail, JIANG Wenb, XIE Mengx. Flavonoids: recent advances as anticancer drugs[J]. Recent Patents on Anti-Cancer Drug Discovery,2010, 5(2): 152-164.
[9] YUAN Shimin, JIN G Hua. Insights into the monomers and single drugs of Chinese herbal medicine on myocardial preservation[J]. African Journal of Traditional, Complementary and Alternative Medicines, 2011, 8(2): 104-127.
[10] XU Peng, RANGANATHAN S, FOWLER Z L, et al. Genome-scale metabolic network modeling results in minimal interventions that cooperatively force carbon flux towards malonyl-CoA[J]. Metabolic Engineering, 2011, 13(5): 578-587.
[11] QIN Jianchun, ZHU Lin, GAO Mingjun, et al. Cloning and functional characterization of a chalcone isomerase from Trigonella foenumgraecum L.[J]. Planta Medica, 2011, 77(7): 765-770.
[12] TANNER G J, FRANCKI K T, ABRAHAMS S, et al. Proanthocyanidin biosynthesis in plants purification of legume leucoanthocyanidin reductase and molecular cloning of its cDNA[J]. Jour nal of Biological Chemistry, 2003, 278(34): 31647-31656.
[13] GURUDEEBAN S, RAMANATHAN T, SATYAVANI K, et al. Standardization of DNA isolation and PCR protocol for RAPD analysis of Suaeda sp.[J]. Asian Journal of Biotechnology, 2011, 3: 486-492.
[14] 李娟. 海湾扇贝抗氧化相关基因的克隆、表达和结构分析[D]. 北京: 中国科学院研究生院, 2010: 28-29.
[15] WANG Zhen, YE Shafei, LI Jingjing, et al. Fusion primer and nested integrated PCR (FPNI-PCR): a new high-efficiency strategy for rapid chromosome walking or flanking sequence cloning[J]. BMC Biotechnology, 2011, 11(1): 109-120.
[16] RADAUER C, LACKNER P, BREITENEDER H. The Bet v 1 fold: an ancient, versatile scaffold for binding of large, hydrophobic ligands[J]. BMC Evolutionary Biology, 2008, 8(1): 286-304.
[17] LIU Junjun, EKRAMODDOULLAH A K M. The family 10 of plant pathogenesis-related proteins: their structure, regulation, and function in response to biotic and abiotic stresses[J]. Physiological and Molecular Plant Pathology, 2006, 68(1): 3-13.
[18] DURBIN M L, MCCAIG B, CLEGG M T. Molecular evolution of the chalcone synthase multigene family in the morning glory genome[J]. Plant Molecular Biology, 2000, 42(1): 79-92.
[19] XU Feng, CHENG Shuiyuan, CHENG Shuhan, et al. Time course of expression of chalcone synthase gene in Ginkgo biloba[J]. Journal of Plant Physiology and Molecular Biology, 2007, 33(4): 309-317.
[20] NAKATSUKA T, NISHIHARA M, MISHIBA K, et al. Temporal expression of flavonoid biosynthesis-related genes regulates flower pigmentation in gentian plants[J]. Plant Science, 2005, 168(5): 1309-1318.
[21] ZHANG Hong, GUO Yanwu, YANG Yadong, et al. Cloning of a novel typeⅢ polyketide synthase encoded by a three-intron gene from Polygonum cuspidatum[J]. Journal of Plant Biochemistry and Biotechnology, 2014, 23(1): 104-111.
[22] JIANG Chenguang, SCHOMMER C K, KIM S Y, et al. Cloning and characterization of chalcone synthase from the moss, Physcomitrella patens[J]. Phytochemistry, 2006, 67(23): 2531-2540.
[23] 韩颖颖. 蝴蝶兰查尔酮合酶基因家族成员分子进化和差异表达调控的研究[D]. 上海: 复旦大学, 2005: 1-2.
[24] JEZ J M, BOWMAN M E, NOEL J P. Role of hydrogen bonds in the reaction mechanism of chalcone isomerase[J]. Biochemistry, 2002,41(16): 5168-5176.
[25] BEDNAR R A, HADCOCK J R. Purification and characterization of chalcone isom erase from soybeans[J]. Journal of Biological Chemistry,1988, 263(20): 9582-9588.
[26] JEZ J M, BOWMAN M E, DIXON R A, et al. Structure and mechanism of the evolutionarily unique plan t enzyme chalcone isomerase[J]. Nature Structural & Molecular Bio logy, 2000, 7(9): 786-791.
[27] SHIMADA N, AOKI T, SATO S, et al. A cluster of genes encodes the two types of chalcone isomerase involved in the biosynthesis of general flavonoids and legume-specif i c 5-deoxy (iso) flavonoids in Lotus japonicus[J]. Plant Physiology, 2003, 131(3): 941-951.
[28] GENSHEIMER M, MUSHEGIAN A. Chalcone isomerase family and f old: no longer unique to plants[J]. Protein Science, 2004, 13(2): 540-544.
[29] KIMURA Y, AOKI T, AYABE S. Chalcone isomerase isozymes with different substrate specificities towards 6’-hydroxy and 6’-deoxychalcones in cultured cells of Glycyrrhiza echinata, a leguminous plant producing 5-deoxyflavonoids[J]. Plant Cell Physiology, 2001, 42(10): 1169-1173.
[30] DRUKA A, KUDRNA D, ROSTOKS N, et al. Chalcone isomerase gene from rice (Oryza sativa) and barley (Hordeum vulgare): physical,genetic and mutation mapping[J]. Gene, 2003, 302(1): 171-178.
[31] 曹廷. 芸薹属查尔酮异构酶基因家族的克 隆与功能鉴定[D]. 重庆:西南大学, 2011: 33.
[32] 颜华, 宋云, 李翊云, 等. 查尔酮异构酶基因的克隆序列分析及在大肠杆菌中的表达[J]. 植物学报, 1997, 39(11): 1030-10 34.
[33] BURBULIS I E, WINKEL-SHIRLEY B. Interactions among enzymes of the Arabidopsis flavonoid biosynthetic pathway[J]. Proceedings of the National Academy of Sciences of the United States of America,1999, 96(22): 12929-l2934.
[34] LI F, JIN Z, QU W, et al. Cloning of a cDNA encoding the Saussurea medusa chalcone isomerase and its expression in transgenic tobacco[J]. Plant Physiology and Biochemistry, 2006, 44(7/9): 455-461.
[35] VERHOEYEN M E, BOVY A, COLLINS G, et al. Increasing antioxidant levels in tomatoes through modification of the flavonoid biosynthetic pathway[J]. Journal of Experimental Botany, 2002,53(377): 2099-2106.
[36] LUKASZEWICZ M, MATYSIAK-KATA I, SKALA J, et al. Antioxidant capacity manipulation in transgenic potato tuber by changes in phenolic compounds content[J]. Journal of Agricultural and Food Chemistry, 2004, 52(6): 1526-1533.
[37] 邵莉, 李毅, 杨美珠, 等. 查尔酮合酶基因对转基因植物花色和育性的影响[J]. 植物学报, 1996, 38(7): 517-524.
[38] 夏玉凤, 耿晓娜, 王万双, 等. 转查尔酮合酶基因对烟草花色及花器官的影响[J]. 生物技术通报, 2010, 1(6): 93-98.
Cloning and Characterization of Full-Length Chalcone Synthase and Chalcone Isomerase Genes from Olea europaea
CHEN Wenshuan1, HUANG Qianming1,*, CHEN Huaping1, YANG Zeshen2, WANG Anyi2, SU Guangcan2
(1. College of Science, Sichuan Agricultural University, Ya’an 625014, China; 2. Liang Shan Zhong Ze New Technology Development Co. Ltd., Xichang 615000, China)
Chalcone synthase (Chs) and chalcone isomerase (Chi) are two key enzymes in the biosynthesis pathway of plant flavonoids. The complete genes of chs and chi were isolated from Olea europaea by homology cloning, RT-PCR, FPNI-PCR (fusion primer and nested integrated PCR) and 3’-RACE (rapid amplification of cDNA end). Sequence analysis showed that the full-length DNA (GenBank No: KF 935223.1) and cDNA (GenBank No: KF935224.1) of chs gene from O. europaea were 2 085 bp and 1 173 bp, resp ectively, and the ORF (open reading frame) of chs gene encoded 390 amino acid residues,which showed high similarity (about 90%) with the complete amino acid sequences of Chs from Vitis vinifera an d Solanum lycopersi cum. Bioinformatics analysis showed that the protein encoded by chs gene from O. europaea belonged to the Chs protein family. The full-length DNA (GenBank No: KF886190) and cDN A (GenBank No: KF886191) of chi gene from O. europaea were 1 373 bp and 750 bp, respectively, and the ORF of chi gene encoded 249 amino acids residues. With the highest similarity (about 65%) to the comple te amino acid sequences of Chi from the other plants , the ful l-length amino acid sequence of Chi from O. europaea had an intramolecular lyase domain with 198 amino acids, which was consistent with the characteristics of the Chi protein family.
Olea europaea; chalcone synthase (Chs); chalcone isomerase (Chi); gene cloning; sequence analysis
Q943.2
A
1002-6630(2015)09-0117-08
10.7506/spkx1002-6630-201509022
2014-06-30
四川省科技厅科技支撑计划项目(12ZC2220)
陈文拴(1988—),男,硕士研究生,研究方向为生物化学与分子生物学。E-mail:chenwenshuan@126.com
*通信作者:黄乾明(1964—),男,教授,博士,研究方向为生物化学与分子生物学。E-mail:huangqianming2014@126.com
