入侵植物光梗蒺藜草对土著丛枝菌根真菌群落的影响
2015-12-06陈保冬
向 丹,陈保冬,李 欢,张 莘*
(1城市与区域生态国家重点实验室/中国科学院生态环境研究中心,北京 100085;2青岛农业大学资源与环境学院,山东青岛 266109)
入侵植物光梗蒺藜草对土著丛枝菌根真菌群落的影响
向 丹1,2,陈保冬1,李 欢2,张 莘1*
(1城市与区域生态国家重点实验室/中国科学院生态环境研究中心,北京100085;2青岛农业大学资源与环境学院,山东青岛266109)
运用T-RFLP及定量PCR的方法探讨了内蒙古温带草原上光梗蒺藜草(Cenchrus incertus M.A.)入侵对AM真菌群落的影响。研究结果表明,光梗蒺藜草能够与AM真菌形成良好共生关系,但与本土优势植物相比,光梗蒺藜草根际土壤中的AM真菌丰度显著降低,AM真菌群落结构也发生明显变化。两种植物根际土壤中AM真菌优势种相同,均属于Glomus属,T-RFLP带型为T-RF 524 bp和280 bp,与Maarj AM数据库匹配的AM真菌虚拟种(virtual taxa)为VT109及VT287。此外,Rhizophagus intraradices(VT113,T-RF141 bp)及Diversispora sp.(VT60,T-RF136,141 bp)在入侵植物根际土壤及根系中均显著高于本土植物,而Glomus属的其它T-RFLP带型在本土植物根际土壤和根系中显著高于入侵植物。两种植物根际和根中AM真菌群落结构一致。初步揭示了光梗蒺藜草对本土AM真菌的影响,为探明光梗蒺藜草入侵机制及其防治提供了一定研究基础。
菌根真菌;光梗蒺藜草;植物入侵;T-RFLP;q-PCR
丛枝菌根(AM)真菌是广泛分布于自然和农业生态系统中的一类共生土壤真菌,能够与绝大多数陆生植物形成共生体[1]。菌根共生体系在调节植物个体生长,群落物种构成及演替,以及生态系统稳定性方面起着重要作用[2~4]。
大多数外来入侵植物都是菌根植物,因此,探讨入侵地AM真菌与外来植物的共生关系及其在植物入侵中的作用,是目前外来植物入侵机制研究的热点之一[5]。以往的研究表明,AM真菌对外来植物入侵有特定促进作用[5],或是抑制作用[6],也有报道说明它们之间没有明显的相互作用[7]。另一方面不同入侵植物对AM真菌的影响也存在着明显差异。一些研究表明,入侵植物降低了入侵地AM真菌的丰度并且改变了AM真菌群落组成[8],而另一些研究则表明植物入侵显著提高了土壤中AM真菌的丰度[9]。入侵植物与AM真菌相互关系的复杂性主要是由于二者共生效应常受多种生物因子(宿主植物种类及发育阶段,AM真菌种类)和非生物因子(土壤肥力水平、气候条件等)的影响[10]。鉴于AM真菌与入侵植物相互作用的复杂性及其功能的生态异质性[11~13],非常有必要实地调查入侵植物与AM真菌的共生状况,从而揭示二者的相互作用。
光梗蒺藜草(Cenchrus incertus M.A.)为禾本科蒺藜草属植物,原产美洲,近些年来该种入侵我国北方草原并大面积蔓延,对生态系统造成严重破坏[14,15]。目前对于光梗蒺藜草的研究大多集中在其生物学特性方面[16],而对于土壤功能微生物AM真菌与光梗蒺藜草的相互关系尚未见报道。光梗蒺藜草是否为菌根植物,是否与本地土著AM真菌形成共生关系,其入侵是否改变了AM真菌群落的结构及丰度,这些基本问题仍然未知。
本研究采用末端限制性片段长度多态性(TRFLP)及定量PCR技术,初步探讨光梗蒺藜草的入侵对AM真菌群落的影响,并比较分析入侵植物和本土优势植物狗尾草根际及根系中AM真菌群落结构差异,从而为后续光梗蒺藜草入侵生态学机制及防治奠定基础。
1 材料与方法
1.1采样地概况和样品采集
研究区域位于内蒙古科尔沁沙地中国科学院奈曼沙漠化研究站附近(N42°56′23″/E120°42′41″),海拔358 m,具有明显的温带大陆性半干旱气候特点。年平均气温6.0~6.5℃,年平均降水量366 mm,降水主要集中在6~8月,年均蒸发量1 972.8 mm。样点采集区域位于奈曼围封试验样地的外围区域,是面积大约为200 m×20 m的一个较小的狭长区域。在该区域内,土壤沙化相对较为严重,光梗蒺藜草大量入侵蔓延,而在该区域以外则仍然是本地植物占优势。采样区域的优势植物为光梗蒺藜草与狗尾草,它们的平均覆盖度分别为51%与36%,也有少量其他植物如软毛虫实、蒺藜、地锦草等。该区有的地方已形成光梗蒺藜草的单优种群,有的地方是光梗蒺藜草和当地植物狗尾草共同竞争生长,也有狗尾草占绝对优势的区域和几乎无植物生长的裸露地区域。
在该区域选取3个10 m×10 m的样方进行采样,每一个样方之间的间隔大约50 m。在每个样方内,选取5个光梗蒺藜草的单优群落进行采样。在每个单优群落内以S布点法选取植株3棵,用铁锹挖出深度为20 cm内的植物根系,敲落植株根系上的土壤为根际土,最后把每个样方里的5个单优群落样品混合成1个样品。同样采得3个狗尾草根际土和根系样品。每个土壤样品过2 mm筛后混匀装袋,植物根系样品也混匀后装袋,暂放低温采样箱带回实验室,部分土样保存于-80℃冰箱待用。其余土壤自然风干,用于理化性质分析。
1.2土壤理化性质测定
土壤pH值采用1∶2.5(W/V)土水比,复合玻璃电极检测;有机碳含量测定采用重铬酸钾外加热法[17],速效氮含量测定采用碱解扩散法[18],土壤有效磷含量测定采用碳酸氢钠浸提—钼锑抗比色法[19]。土壤全碳和全氮含量用元素分析仪(Vario ELⅢ,Elementar Company,German)测定。
1.3土壤、根系DNA的提取及定量PCR
取500 mg过2 mm筛的混匀鲜土,按照DNA提取试剂盒说明(Fast DNA spin kit for soil,Bio 101,Vista,CA,USA)进行操作,提取的DNA溶液贮藏于-20℃冰箱待用。植物根系用蒸馏水清洗干净并用纸巾吸去多余水分,称取0.2 g的干净根系于1.5 mL离心管中,用CTAB的方法进行DNA提取[20]。
AM真菌在根系和根际土壤中的基因拷贝数均采用定量PCR的方法(qPCR)测得。所用引物为AMV4.5NF/AMDGR[21]。PCR反应体系为25μL: 12.5μL 2×SYBR Premix Ex Taq(Takara),每个引物各1μL(5μmol/μL),1μL DNA模板(20 ng/μL),10.5μL的灭菌双蒸水。反应参数:95℃变性30 s;95℃5 s,58℃30 s,72℃45 s,35个循环;72℃延伸10 min。定量PCR的标准曲线以相同的AM真菌18S基因为内参。在经过相同的PCR程序扩增后(普通Ex Taq),纯化PCR产物连接到pMD18 -T载体(Promega,Madison,WI,USA)并转化到Escherichia coli JM109感受态细胞中(Takara,Japan),所得克隆子经过测序检验确定为该18S片段后,提取质粒DNA(质粒提取试剂盒,Takara,Japan)并运用NanoDropⓇND-1000紫外—可见分光光度计测定其浓度(NanoDrop,Wilmington,DE,USA)。配制的标准曲线包含从2.01×102到2.01×107基因拷贝数的6个浓度梯度(浓度依次相差10倍)。每个内标及样品在进行定量PCR扩增时都有3个重复。试验所用仪器为BioRad iQ5荧光定量PCR仪(Bio-Rad Laboratories Hercules,CA),数据分析软件为Optical System Software BioRad iQ5。
1.4AM真菌T-RFLP的测定
对菌根真菌18S rDNA进行扩增,选用的引物为菌根真菌分子检测常用引物NS31(5′-TGGAGGGCAGTCTGTC-3′)和AM1(5′-GTTTCCCGTAAGGCGCCGAA-3′)[22,23],其扩增区域为18S rDNA上V4可变区,片段大小约为550 bp。由于该引物具有一定偏扩性,不利于无梗囊霉科和类球囊霉科AM真菌的扩增,并且在土壤中AM真菌的扩增比例较低,为弥补NS31/AM1偏扩带来的误差,选用覆盖度更广的AML1(5′-ATCAACTTTCGATGGTAGGATAGA-3′)和 AML2(5′-′GAACCCAAACACTTTGGTTTCC-3′)[24]作为第一对引物。该引物能覆盖除Archaeospora trappei以外的所有已知AM真菌。
Nested-PCR反应体系包括下列成分:第一次扩增反应体系25μL,包括1μL 20 ng/LDNA模板、0.3μL 25 M前后端引物、2.5μL 10×Ex Taq Buffer、2μL 2.5 mM dNTP混合液、0.3μL 25 M BSA、0.5μL 5 U/L Ex Taq(Takara,Japan),以及17.1μL ddH2O。第二次扩增体系为50μL,包括2μL 20 ng/L DNA模板、0.6μL 25 M引物、5.0μL 10×Ex Taq Buffer、4.0μL 2.5 mM dNTP混合液、0.6μL 5 U/L Ex Taq,以及37.2μL ddH2O。其中第二次扩增引物NS31的5′端用FAM荧光素标记(上海生工生物工程有限公司合成)。
两次PCR反应的程序相同:94℃变性5 min;94℃ 30 s,58℃ 45 s,72℃1 min,循环35次,72℃延伸10 min。第一次扩增产物稀释10倍作为第二次扩增反应的模板。
PCR反应产物的纯度和产量用1%琼脂糖凝胶电泳及核酸蛋白仪NanoDrop 1000(NanoDrop Technologies,Wilmington,DE,USA)同时检测。然后用纯化试剂盒QIA quick PCR Purification Kit(Qiagen,Valencia,CA,USA)切胶纯化。
采用FastDigest Hinf I(Fermentas,Hanover,MD,USA)对纯化后的PCR产物进行酶切。酶切体系为30μL:PCR产物10~15μL,Hinf I酶1μL,10 Buffer 2μL,剩余量由ddH2O补齐。酶切步骤:缓冲液在37℃下水浴锅中温浴15 min使得酶解充分完成,然后在65℃条件下水浴20 min使酶失活。酶切产物用1%琼脂糖凝胶电泳检验是否酶切效果,随后酶切产物脱盐处理,基因扫描由北京诺赛基因公司完成。T-RFLP谱图用GeneMap(Version 3.7)程序进行分析,选择的内标为GS500liz。舍去<40 bp和>550 bp的片段,以及峰面积低于总峰面积1%的T -RFs。然后分别计算图谱中每一个峰的峰面积与所有峰总面积的比值,得到每种T-RFLP的相对含量。以除去噪音的每个T-RFLP带型为一个分类单元,根据图谱中T-RFLP的数目及其丰度进行多样性指数计算及其他统计分析。
1.5克隆文库的建立及分析
为确认T-RFLP指纹图谱中各T-RF代表何种AM真菌,采用T-RFLP带型较多的样品建立克隆文库,再对每个特异性克隆(相似度<97%)进行T-RFLP分析。
克隆文库的具体操作步骤为:利用无荧光标记巢氏PCR引物AML1/AML2+NS31/AM1进行PCR扩增,扩增体系及程序同上。PCR产物经纯化试剂盒QIA quick PCR Purification Kit(Qiagen,Valencia,CA,USA)切胶纯化后连接至质粒载体pGEM-T easy Vector(Promega,Madison,WI,USA),重组质粒通过热击法转化到E.coli JM109(Takara,Japan)中。取200μL转化后的产物均匀涂布于含有IPTG、X-gal和100mg/L氨苄青霉素的LB固体培养基中,在37℃的温度条件下培养16 h后,挑取白色的阳性克隆子进行菌落PCR检验。最后挑出90个阳性克隆,由北京诺赛基因公司完成测序。
测得的序列用DOTOUR软件[25]以97%相似度[26]进行OTU分型,然后用DNAMAN软件(Version 6.0.3.99,Lynnon Biosoft,USA)对这些克隆序列进行模拟酶切,以确定每种OTU对应的T-RFLP带型。同时每种OTU的代表序列与GenBank中的核酸数据库进行序列比对分析(http://maarjam. botany.ut.ee),运用MEGA(version 4.0)[27]构建系统发育树,即可以确定菌根真菌菌种名称。最终把T-RFLP带型与AM真菌菌种名称相对应。本试验中所得每个OTU的代表序列已提交至Genbank(序列号为KC797120-KC797131)。
1.6数据分析
AM真菌а多样性采用Shannon-Wiener指数(H)表示。计算公式为:H=-∑P×In Pi,Pi=ni/n。式中,ni为第i个T-RF带型的数目,n为群落中所有T-RF带型的数据总和。
AM真菌群落结构均基于每个T-RF带型的相对丰度进行计算。群落分类和排序均基于Bray-Curtis差异度指数。排序采用主成分分析法(principle component analysis,PCA)(CANOCO 4.5,Microcomputer Power,Ithaca,NY,USA)。采用Adonis分析进一步检验AM真菌群落在入侵和本土植物间的差异是否显著(R语言,Vegan包,R Development Core Team,2012)。采用非参数检验(SPSS Inc.,Chicago,IL,USA)确定入侵和本土植物间AM真菌а多样性、基因拷贝数、土壤理化性质及RFLP的相对丰度是否差异显著。
为了对T-RF带型进行鉴定,在建立系统发育树之外,把每个T-RF带型的代表序列进一步与Maarj AM数据库比对,得到每个T-RF带型所对应的VT类型(Virtual taxa,虚拟分类单元)。Maarj AM数据库包含所有已报道的球囊菌门(Glomeromycota)真菌18S rDNA序列。VT是Maarj AM数据库中用以区别不同AM真菌序列的新的分类方法[28]。
2 结果与分析
2.1土壤理化性质、AM真菌基因拷贝数及多样性系数
入侵植物和本土植物间根际土壤理化性质总体差异不显著(表1)。根际土壤中AM真菌基因拷贝数在本土植物中显著高于入侵植物光梗蒺藜草(p<0.05),而植物根系中的AM真菌基因拷贝数在两种植物间没有明显差异(表2)。在本实验中总共检测到11个不同的T-RF带型,两种植物根系和根际土中的AM真菌多样性均没有显著差异(表2)。

表1 入侵植物光梗蒺藜草与本土植物狗尾草根际土壤基本理化性质比较

表2 两种植物根系及根际土壤中 AM真菌基因拷贝数及多样性系数比较
2.2 AM真菌群落结构差异
主成分分析(PCA)结果表明,55.4%和22.8%的AM真菌群落差异可以由PC1和PC2解释(图1)。图中两种不同植物明显区分为两个簇,同一植物根系和根际土之间差异则不明显,而两种植物之间,不管是根系还是根际土壤中,AM真菌群落结构均存在明显差异。通过Adonis分析进一步表明,AM真菌群落在入侵和本土植物间存在显著差异(R2=0.5046,p=0.002)。
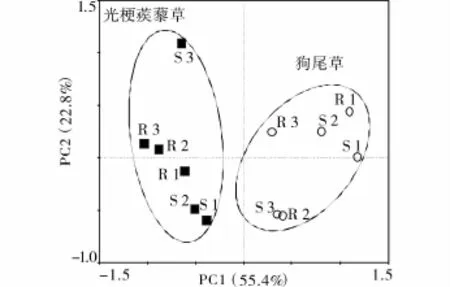
图1 基于 T-RFLP数据的 AM真菌群落结构的主成分分析(PCA)注:R代表根系,S代表土壤。■表示光梗蒺藜草,○表示狗尾草。
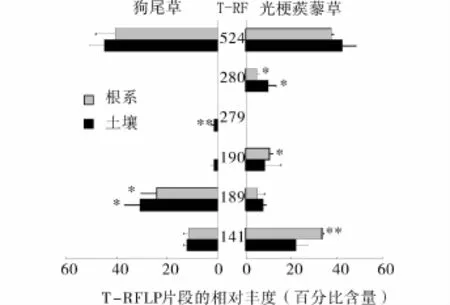
图2 两种植物根系和土壤中 AM真菌 T-RFLP带型的相对丰度(百分比)
图2显示的是优势T-RF带型及两种植物间有显著差异的T-RF带型的相对丰度。其他4个带型(T-RF 136,157,179,301 bp)因为百分比少且仅仅出现在个别样品中,故未在图中展示。由图2可以看出,不管是入侵植物还是本土植物,其根系和根际土壤中的AM真菌群落构成(不同T-RF相对丰度)都基本一致。但是两种植物之间的AM真菌群落构成却存在显著差异。具体来说,T-RF 524 bp为两种植物的共同优势带型,而带型190 bp,141 bp和280 bp的相对丰度在光梗蒺藜草根系中均显著高于狗尾草根系,280 bp的相对丰度在光梗蒺藜草根际土壤中也显著高于狗尾草根际土。与此相反,带型279 bp和189 bp在本土植物狗尾草根系及根际土中均显著高于光梗蒺藜草。此外,280 bp是光梗蒺藜草的特有带型,而279 bp是狗尾草的特有带型。
总体上,除了两个相对丰度较低且仅在个别样品中出现的片段(179 bp和301 bp)没有被检测到以外,剩下的8个HInfI酶切带型都成功地被克隆文库所检测到(图3)。其中有6个Glomus属的TRF带型,其余带型为Rhizophagus intraradices及Diversispora sp.。系统发育树把未知AM真菌鉴定到分子种属水平相对粗糙,且仅仅局限于单个实验内的比较,而Maarj AM数据库中VT(虚拟种)的分类系统已经在世界范围内被广泛接受及使用,因此我们基于VT分类系统对所得T-RF带型进行分析。由图3可以看出,两种植物共同的优势AM真菌(T -RF 524 bp)可以被鉴定为Maarj AM数据库中的VT109及VT287,属于Glomus属。狗尾草的优势带型(189,279 bp)为VT 156,也属于Glomus属。光梗蒺藜草的优势带型T-RF 190 bp、141 bp和280 bp,可以分别被鉴定为VT 214,VT 60,VT 287及VT 64,分别属于Glomus、Rhizophagus和Diversispora属。在属水平,本土植物狗尾草仅仅与Glomus属的AM真菌形成共生体系,而入侵植物能够与3个不同属的AM真菌形成共生关系。

图3 AM真菌 18S rDNA的系统发育树
3 讨论
外来植物与入侵地AM真菌的相互作用是影响外来植物入侵力和生态系统可入侵性的一个重要方面。已有大量研究表明,外来入侵植物能够与土著AM真菌形成共生体系,并对其成功入侵起到积极作用[29~31]。本研究表明,光梗蒺藜草根系和本土植物根系的AM真菌基因拷贝数是一致的,并且与土壤中AM真菌的基因拷贝数在同一个数量级上。这个研究结果明确说明了光梗蒺藜草为菌根植物,它与土著的AM真菌形成了较好的共生体系。虽然本实验不能够明确说明AM真菌在促进光梗蒺藜草个体生长或者入侵中的具体作用,但是至少揭示了AM真菌在光梗蒺藜草入侵过程中的潜在作用。
与植物根系相比,光梗蒺藜草根际土壤中AM真菌的基因拷贝数显著低于本土植物狗尾草,此结果说明光梗蒺藜草入侵可能降低了入侵地土壤中AM真菌丰度。与此相似,有研究表明在受到干扰的生态系统中,外来入侵植物显著降低了土壤中AM真菌的丰度(菌丝密度)及多样性[32,33]。此外本研究结果也表明光梗蒺藜草显著改变了入侵地土壤中的AM真菌群落结构。由于AM真菌与土著植物间长期的适应进化使得它们之间存在较强的相互选择性,一旦这种关系被干扰,则可能反馈影响外来植物与本地植物的竞争关系[34]。例如,入侵植物斑点矢车菊对入侵地AMF群落的影响即不利于本土植物生长[35]。Hawkes等研究表明,入侵植物通过改变土壤中AM真菌的群落结构,使得本土植物根系中AM真菌群落结构发生明显改变,并且变得与入侵植物根系中AM真菌群落结构一致,明确说明了入侵植物对土著AM真菌与本土植物共生关系产生干扰[36]。此外,也有研究表明本土植物对于土著AM真菌的依赖性强于入侵植物[10,32],因此当AM真菌群落被干扰或者抑制时,高菌根依赖性植物将比低菌根依赖性植物遭受更强的生长抑制[8]。由此推测,这也可能是光梗蒺藜草入侵机制之一:入侵植物通过改变入侵地土壤的微生物群落结构,破坏土著植物与土壤微生物之间长期的平衡关系,从而影响本地种的生长,最终实现成功入侵[10,37]。关于光梗蒺藜草与AM真菌的相互作用及其入侵反馈机制还需要后续研究进一步验证。
同一植物根系和根际土壤中AM真菌的群落结构可能存在着显著差异[38],因而那些在根系中实际发挥作用的AM真菌很可能并不是土壤中占优势地位的AM真菌。我们同时检测了入侵植物和本土植物根系和土壤中的AM真菌群落结构,结果表明无论是入侵植物还是本土植物,其根系中AM真菌群落结构都与根际土壤中高度一致,但是两种植物之间AM真菌群落结构存在着显著的差异。这说明光梗蒺藜草对本土AM真菌群落结构具有明显的选择作用,在其入侵地土壤中选择性富集那些在其自身根系中占据优势地位的AM真菌菌种,而这些占据主导地位的AM真菌很可能就是在根系中真正发挥共生作用的AM真菌。与此类似,有研究表明,入侵植物加拿大一枝黄花能够在入侵地土壤中专性富集那些能够显著促进其自身生长的AM真菌菌种,却明显减少那些能够促进本土植物生长的AM真菌菌种[11]。在本研究中,光梗蒺藜草对AM真菌群落结构的特异选择性也可能是其成功入侵的原因之一。
需要说明的是,AMF与外来入侵植物的相互作用不仅受到入侵植物和AMF本身(如AMF种类、起源等)的影响,而且受多种外在生物因子或者非生物因子的影响[30,31]。因此,明确AMF在外来植物入侵中的作用,不仅需要野外的实地调查及实验,还需要综合考虑各种环境因子的影响,开展在不同生态环境条件下的入侵机制研究,以更好地揭示其入侵机制,制定全面的防范对策。在本实验中,我们采用限制性酶切的方法揭示了入侵植物和本土植物AM真菌的群落结构差异,可能受制于分子方法的限制,所检测到的AM真菌多样性不太高,但本实验还是在一定程度上明确说明了两种植物间AM真菌的差异,以及光梗蒺藜草对土著AM真菌群落结构的影响,后续研究可以采用更加先进的分子生物学方法以进一步揭示其差异及机制。
4 结论
本研究表明,入侵植物光梗蒺藜草为菌根植物,能够与AM真菌形成共生关系。此外,光梗蒺藜草对AM真菌具有特异选择性,能够在入侵地土壤中富集那些与其自身根系形成共生关系的AM真菌,却显著减少土壤中与本土植物形成共生的AM真菌。这些结果暗示,AM真菌可能在光梗蒺藜草的入侵过程中起到重要作用。此外,实验揭示了与本土植物和入侵植物共生的AM真菌群落结构差异,与本土植物形成共生的土著AM真菌有可能用于后续的生态恢复。
[1] Smith SE,Read DJ.Mycorrhizal Symbiosis[M].3rd edn. London,UK:Academic Press Ltd,2008.30.
[2] Whiteside MD,Digman MA,Gratton E,et al.Organic nitrogen uptake by arbuscularmycorrhizal fungi in a boreal forest[J].Soil Biology and Biochemistry,2012,55:7-13.
[3] Veresoglou SD,Rillig MC.Suppression of fungal and nematode plant pathogens through arbuscularmycorrhizal fungi[J].Biology Letters,2012,8(2):214-217.
[4] W illis A,Rodrigues B,Harris P.The ecology of arbuscular mycorrhizal fungi[J].Critical Reviews in Plant Sciences,2013,32(1):1-20.
[5] Pringle A,Bever JD,Gardes M,et al.Mycorrhizal symbioses and plant invasions[J].Annual Review of Ecology,Evolution,and Systematics,2009,40:699-715.
[6] Walling SZ,Zabinski CA.Defoliation effects on arbuscular mycorrhizae and plant growth of two native bunchgrasses and an invasive forb[J].App lied Soil Ecology,2006,32(1):111-117.
[7] Kempel A,Nater P,Fischer M,et al.Plant-microbeherbivore interactions in invasive and non-invasive alien plant species[J].Functional Ecology,2013,27(2):498 -508.
[8] Stinson KA,Campbell SA,Powell JR,et al.Invasive plant suppresses the growth of native tree seedlings by disrupting belowground mutualisms[J].PLoSBiology,2006,4(5):e140.
[9] Lekberg Y,Gibbons SM,Rosendahl S,et al.Severe plant invasions can increase mycorrhizal fungal abundance and diversity[J].The ISME Journal,2013,7:1424-1433.
[10] Inderjit,van der Putten WH.Impacts of soil microbial communities on exotic plant invasions[J].Trends in E-cology&Evolution,2010,25(9):512-519.
[11] Zhang Q,Yang R,Tang J,et al.Positive feedback between mycorrhizal fungiand plants influences plant invasion success and resistance to invasion[J].PLoSOne,2010,5(8):e12380.
[12]De Deyn GB,Quirk H,Bardgett RD.Plant species richness,identity and productivity differentially influence key groups ofmicrobes in grassland soils of contrasting fertility[J].Biology Letters,2011,7(1):75-78.
[13]Oliveira RS,Boyer LR,Carvalho MF,et al.Genetic,phenotypic and functional variation within a Glomus geosporum isolate cultivated with orwithout the stress ofa highly alkaline anthropogenic sediment[J].Applied Soil E-cology,2010,45(1):39-48.
[14] Chen SL,Phillips SM.Flora of China[M].Vol 22.Beijing:Science Press,2006.552-553.
[15]Kole C.W ild Crop Relatives:Genomic and Breeding Resources(Millets and Grasses)[M].New York:Springer Press,2011.34-35.
[16]贾鲜艳,李青丰,徐 军.外来入侵植物光梗蒺藜草开花与结实特性研究[J].内蒙古农业大学学报,2012,33(2):83-88.
[17]Walkley A.A critical examination of a rapid method for determining organic carbon in soils-effect of variations in digestion conditions and of inorganic soil constituents[J].Soil Science,1947,63(4):251-264.
[18]Cornfield A.Ammonia released on treating soils with N sodium hydroxide as a possible means of predicting the nitrogen-supplying power of soils[J].Nature,1960,187:260-261.
[19]Olsen SR,Cole CV,Watanabe FS,et al.Estimation of available phosphorous in soils by extraction with sodium bicarbonate[R].Washington,USA:USDep Agric Circ,1954.939.
[20] Doyle JJ.A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J].PhytochemBull,1987,19:11-15.
[21]Sato K,Suyama Y,Saito M,et al.A new primer for discrim ination of arbuscularmycorrhizal fungiwith polymerase chain reaction-denature gradientgel electrophoresis[J].Grassland Science,2005,51(2):179-181.
[22]Simon LM,Lalonde TD,Bruns TD.Specific amplification of 18S fungal ribosomal genes from vesicular arbuscular endomycorrhizal fungi colonising roots[J].Applied and Environmental Microbiology,1992,58:291-295.
[23]Helgason T,Daniell TJ,Husband R,et al.Ploughing up the wood-wide web[J].Nature,1998,394:431.
[24]Lee J,Lee S,Young JPW.Improved PCR primers for the detection and identification of arbuscular mycorrhizal fungi[J].FEMS Microbiology Ecology,2008,65(2): 339-349.
[25]Schloss PD,Handelsman J.Introducing DOTUR,a computer program for defining operational taxonomic unitsand estimating species richness[J].App lied and Environmental Microbiology,2005,71(3):1501-1506.
[26]Dumbrell AJ,Ashton PD,Aziz N,et al.Distinct seasonal assemblages of arbuscular mycorrhizal fungi revealed by massively parallel pyrosequencing[J].New Phytologist,2011,190(3):794-804.
[27]Tamura K,Dudley J,NeiM,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Molecular Biology and Evolution,2007,24(8):1596-1599.
[28]Pik M,Metsis M,Daniell T,et al.Large-scale parallel 454 sequencing reveals host ecological group specificity of arbuscularmycorrhizal fungi in a boreonemoral forest[J].New Phytologist,2009,184(2):424-437.
[29] Fumanal B,Plenchette C,Chauvel B,et al.Which role can arbuscularmycorrhizal fungi play in the facilitation of Ambrosia artemisiifolia L.invasion in France[J].Mycorrhiza,2006,17(1):25-35.
[30] Shah MA,Reshi Z,Rashid I.Mycorrhizal source and neighbour identity differently influence Anthemis cotula L.invasion in the Kashmir Himalaya,India[J].Applied Soil Ecology,2008,40(2):330-337.
[31]Shah MA,Reshi Z,Rashid I.Mycorrhizospheremediated mayweed chamomile invasion in the Kashmir Himalaya,India[J].Plant and Soil,2008,312(1-2):219-225.
[32]Bever J,Schultz P,Miller R,etal.Inoculation with prairie mycorrhizal fungimay improve restoration of native prairie plant diversity[J].Ecological Restoration,2003,21: 311-312.
[33]Broz AK,Manter DK,Vivanco JM.Soil fungal abundance and diversity:another victim of the invasive plant Centaureamaculosa[J].The ISME Journal,2007,1(8): 763-765.
[34]Bever JD,Morton JB,Antonovics J,et al.Host dependent sporulation and species diversity of arbuscularmycorrhizal fungi in a mown grassland[J].Journal of Ecology,1996,84(1):71-82.
[35]Mummey DL,Rillig MC,Holben WE.Neighboring plant influences on arbuscular mycorrhizal fungal community composition as assessed by T-RFLP analysis[J].Plant and Soil,2005,271(1-2):83-90.
[36]Hawkes CV,Belnap J,D’Antonio C,et al.Arbuscular mycorrhizal assemblages in native plant roots change in the presence of invasive exotic grasses[J].Plant and Soil,2006,281(1-2):369-380.
[37]于兴军,于 丹,卢志军,等.一个可能的植物入侵机制:入侵种通过改变入侵地土壤微生物群落影响本地种的生长[J].科学通报,2005,5(9):896-903.
[38]Chen YL,Zhang X,Ye JS,et al.Six-year fertilization modifies the biodiversity of arbuscular mycorrhizal fungi in a temperate steppe in Inner Mongolia[J].Soil Biology and Biochemistry,2014,69:371-381.
Effects of Exotic Plant Cenchrus incertus on Local Arbuscular Mycorrhizal Fungal Community
XIANG Dan1,2,CHEN Bao-dong1,LIHuan2,ZHANG Xin1*
(1 State Key Laboratory of Urban and Regional Ecology,Research Center for Eco-Environmental Sciences,Chinese Academy of Sciences,Beijing 100085,China;2 College of Resources and Environment,Qingdao Agricultural University,Qingdao,Shandong 266109,China)
The AM fungal community composition both in roots and rhizosphere soils of the invasive plant Cenchrus incertus and the dominantnative plant Setaria viridis in a typical steppe in InnerMongoliawereexamined by using terminal restriction fragment length polymorphism analyses(T-RFLP).The results showed that AM fungal abundance in the rhizosphere soil of C.incertus was significantly lower than that of S.viridis.The AM fungal community composition in the rhizosphere soil of the two plant species also largely differed.In general,AM fungal community structures in roots corresponded very well to that in rhizosphere soils for both plant species.The dominant AM fungal type both in invasive and in native plants was T-RFLP 524 bp,which represents Glomus sp.(Virtual taxa 109 and 287).Three specific T-RF types(280,190 and 141 bp)were significantly higherwith C.incertus,representing three clusters in Glomus which also named as VT(virtual taxa)287,64 and 214,Rhizophagus intraradices(VT 113)and Diversispora sp.(VT 60).While the specific T-RF types,189 and 279 bp,for S.viridis,only existed in Glomus cluster 1(VT 156),were significantly lower with C.incertus.These results indicated that AM fungimay play an important role in the invasion process of C.incertus,which still need further investigations.
arbuscularmycorrhiza fungi;Cenchrus incertus;plant invasion;T-RFLP;q-PCR
S154.3
A
1001-5280(2015)05-0534-08
10.3969/j.issn.1001-5280.2015.05.18
2015-06-10
向 丹(1984-),女,湖北宜昌人,博士,主要从事土壤微生物分子生态方面研究,Email:smilingxiangdan@163.com。*通信作者,Email:xinzhang@rcees.ac.cn。
国家自然科学基金面上项目(41371264)。
