基于Illumina HiseqTM 2000高通量转录组测序的里氏拟石磺SSR标记开发
2015-12-02沈和定王冬凤杨铁柱
吴 欣, 沈和定, 王冬凤, 刘 宸, 杨铁柱, 刁 亚
(上海海洋大学 省部共建水产种质资源发掘与利用教育部重点实验室, 上海 201306)
石磺是软体动物门(Mollusca) 、腹足纲(Gastropoda) 、肺螺亚纲(Pulmonata)、石磺总科(Onchidioidea) 、石磺科(Onchidiidae)的一群贝类, 主要分布于热带或亚热带地区的海岸、滩涂、岩礁、红树林、潮间带地区, 是海洋与陆地的过渡带生物[1-2]。我国的石磺主要分布在东南沿海的潮间带高潮区, 已报道易采集的主要有4种, 包括瘤背石磺(Onchidium struma)、平疣桑葚石磺(Platevindex mortoni)、紫色疣石磺(Peronia verruculata)、里氏拟石磺(Paraoncidium reevesii), 学者对于石磺的研究主要集中在瘤背石磺,对后3种研究报道较少。其中, 里氏拟石磺的研究主要集中在形态学[1-3]、化学成分测定[4-6]、线粒体基因组及群体多样性的分析[7-9]几个方面, 未见利用分子标记研究其遗传多样性的报道。
微卫星, 又称简单重复序列(SSR)[10], 具有高稳定性、高多态性、引物通用性、位点特异性、检测方便和呈共显性遗传等特点, 在水产动物中主要用于遗传连锁图谱的制备、性状分析、QTL 定位、群体遗传学、进化遗传、种质鉴定与亲权分析, 品种培育等方面[11]。里氏拟石磺作为海洋向陆地进化的过渡物种之一, 在研究海洋无脊椎动物向陆地辐射进化中具有重要作用[12-13], 本研究利用 Illumina HiseqTM 2000高通量转录组测序对里氏拟石磺进行微卫星(SSR)分子标记开发, 为研究其群体遗传学和石磺科不同种贝类间的系统发生提供基础数据。
1 材料和方法
1.1 实验材料
实验材料于2015年5月采自广东湛江海滩, 共37个, 样本经鉴定后均保存于 95%无水乙醇并置于–20℃冰箱中备用。
1.2 实验方法
1.2.1 里氏拟石磺转录组测序结果
随机挑选6只于2012年10月采自湛江的里氏拟石磺个体进行活体解剖, 将解剖得到的不同组织保存于RNA later(Qiagen: 76104)中用于构建转录组测序文库并送晶能公司(Genergy Biotechnology)进行Illumina HiseqTM 2000双末端测序及生物信息学分析, 共获得原始数据9.161 GB, 有效数据8.617 GB。通过de novo拼接获得了长度大于100 bp的转录物233 636条; Unigene N50长度485 bp, 经GO注释分析有 124 598(53.33%)条归入生物学过程, 51 891(22.21%)条归入分子功能, 57 136(24.46%)条归入细胞组分, 对大于200 bp的拼接序列与Nr数据库进行比对, 22 558条序列得到蛋白注释, 匹配蛋白数目为2 910个, 对注释后的数据进行分析, 共涉及物质及能量代谢、信号传导、转录调控及免疫应答等诸多生理生化过程。
1.2.2 微卫星序列的来源
根据晶能公司(Genergy Biotechnology)基于Illumina HiseqTM 2000测序平台所得的拼接序列,经 MISA(http: //pgrc.ipk-gatersleben.de/misa/)对微卫星位点进行分析, 对于非混合型位点筛选时, 设置条件为单碱基重复至少10次, 2碱基重复至少6次,3~6碱基重复至少5次, 在此基础上, 如同一序列中两个SSR位点间离小于50 bp, 则认为这两个SSR位点组成一个混合型 SSR位点, 在设计引物前利用SSR Hunter1.3[14]对经MISA筛选过的含有微卫星位点的序列进行再次筛选, 保证序列的前后侧翼有足够的长度用于设计引物, 筛选条件和第一次一样,最终筛选出51条含有微卫星位点并适合设计引物的序列。
1.2.3 SSR引物设计与合成
利用Primer Premier 5.0(http: //www.premierbio soft.com/)软件对上一步所得的51条拼接序列进行引物设计, 引物长度为19~26 bp, GC含量在40%~60%之间, 也可放宽至30%~70%, 引物Tm值小于72℃,且上下游引物Tm值差别不能大于2℃, 扩增引物长度在100~300 bp, 所设计引物在侧翼保守序列内。本实验共设计51对引物, 所有引物均由生工生物工程(上海)股份有限公司合成。
1.2.4 样品DNA的提取
取出之前保存于–20℃冰箱样品, 根据天根生化科技(北京)有限公司的海洋动物基因组提取试剂盒(DP324)的要求, 在每只样品腹足处各取 30mg组织块用于DNA提取, 提取后的DNA置于–20℃冰箱暂存备用。
1.2.5 PCR扩增及产物检测
PCR反应体系为10μL, 包括上下游引物各0.3 μL,2×Taq PCR Master Mix 5μL, ddH2O 4μL, DNA 模板0.4 μL。PCR反应条件为: 94℃预变性5 min; 94℃ 30 s,退火30 s, 72℃ 30 s, 共40个循环; 72°C延伸10 min。产物保存于4℃。引物初筛PCR产物使用1%的琼脂糖凝胶电泳检测, God view染色, 电泳条件为: 115 V,35 min, 电泳缓冲液为1×TAE, 凝胶成像系统拍照。引物进行群体检测时, PCR扩增产产物8%的聚丙烯酰胺凝胶电泳进行分离, 电泳条件为: 电压 110 V,电泳时间6 h, 电泳缓冲液为1×TBE; 电泳结束后用硝酸银对凝胶染色并使用相机进行拍照。
1.2.6 里氏拟石磺SSR引物的筛选及群体检测
随机选取5个里氏拟石磺个体, 提取其DNA并将其混合作为引物特异性和最适退火温度筛选时的模板DNA, 根据每对引物的Tm值设置间隔为1.5℃的温度梯度, 共设置 8个温度梯度, 用1%的琼脂糖凝胶电泳进行检测, 将扩增清晰, 条带单一的引物留用, 确定每对引物的最适退火温度, 以便进行后续检测。
选取 32个湛江群体的里氏拟石磺个体, 提取DNA作为模板, 使用初筛后留用的引物进行PCR扩增, PCR反应条件及产物检测方法遵照上述方法, 并对产物进行拍照留存。
1.2.7 数据统计和分析
对湛江群体里氏拟石磺的 PCR扩增结果进行统计时, 将每个位点的扩增条带按从小到大的顺序标记为A, B, C…, 并统计出每个位点在进行群体检测时的基因型, 使用 Cervus3.0[15]软件计算每个微卫星位点的等位基因数(number of allele,Na), 观测杂合度(observed heterozygosity,Ho), 期望杂合度(expected heterozygosity,He), 多态性信息含量(polymorphic information content, PIC), 使用Genepop4.2[16]进行每个位点的 Hardy–Weinberg平衡的检测。
2 结果分析
2.1 转录组中SSR位点的软件分析结果
利用 MISA软件在里氏拟石磺转录组中查找SSR位点的结果, 如表1所示。
2.2 里氏拟石磺SSR筛选结果
51对引物在经过琼脂糖凝胶电泳的初筛之后有24对扩增出清晰单一的条带, 扩增率为 47%。将这24对引物在湛江群体32个个体中做进一步的聚丙烯酰胺凝胶电泳筛选, 有 14个位点表现为多态性, 其余位点表现为单态性。在表现出多态性的14个位点中, 7个位点的等位基因为4, 4个位点的等位基因为3, 3个位点表现为2等位基因。14个位点中, 1个位点为五碱基重复, 2个位点为四碱基重复, 11个位点为三碱基重复, 且四, 五碱基重复的次数均为 5, 三碱基的重复次数均为 6。14对引物中最低退火温度为 50.5℃, 最高退火温度为 61℃, 扩增片段的长度在90~370 bp。
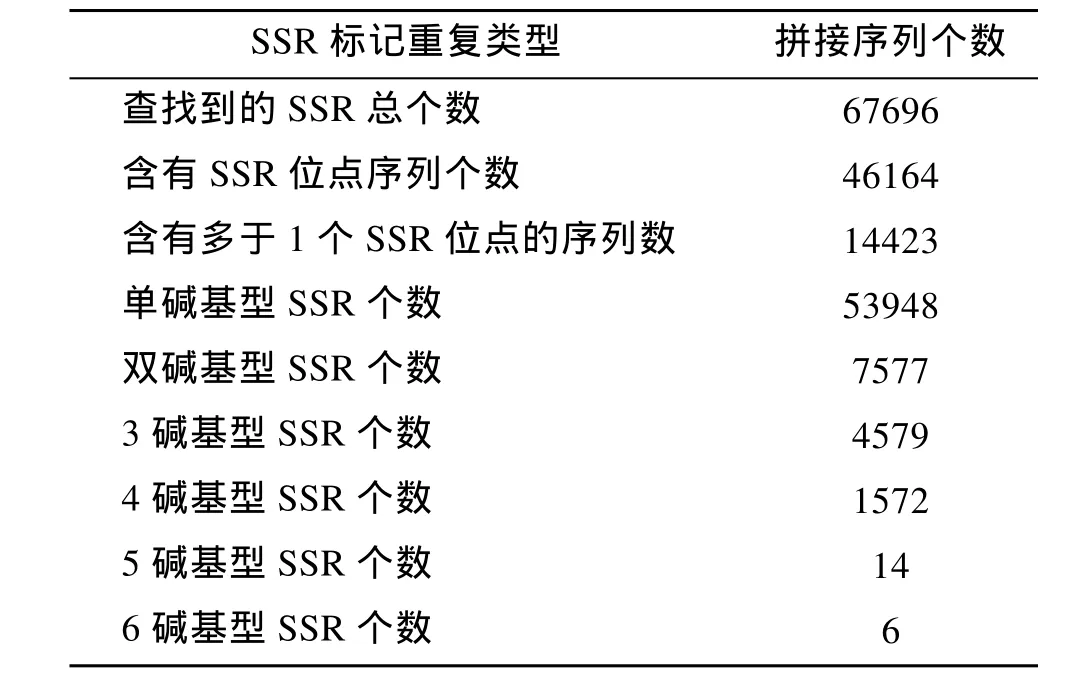
表1 SSR分析结果Tab.1 Output statistics of SSR
2.3 里氏拟石磺湛江群体遗传多样性分析
对14对SSR引物在取自湛江群体的32个体扩增片段进行统计分析, 14个位点的等位基因数在2~4之间, 其中Ho和He分别在0.031 3~0.714 3和0.031 3~0.708 4之间, 多态性信息含量(PIC)在0.089 5~0.640 5之间, 其中 2个微卫星位点表现为高度多态性(PIC>0.5), 10个位点表现为中度多态性(0.5>PIC>0.25), 2个位点表现为低度多态性(PIC<0.25)。经Sequential Bonferroni校正的Hardy-Weinberg 平衡检验, 9个位点不偏离平衡(P>0.05), 其余位点均显著偏离平衡(0.05>P, 表2)。
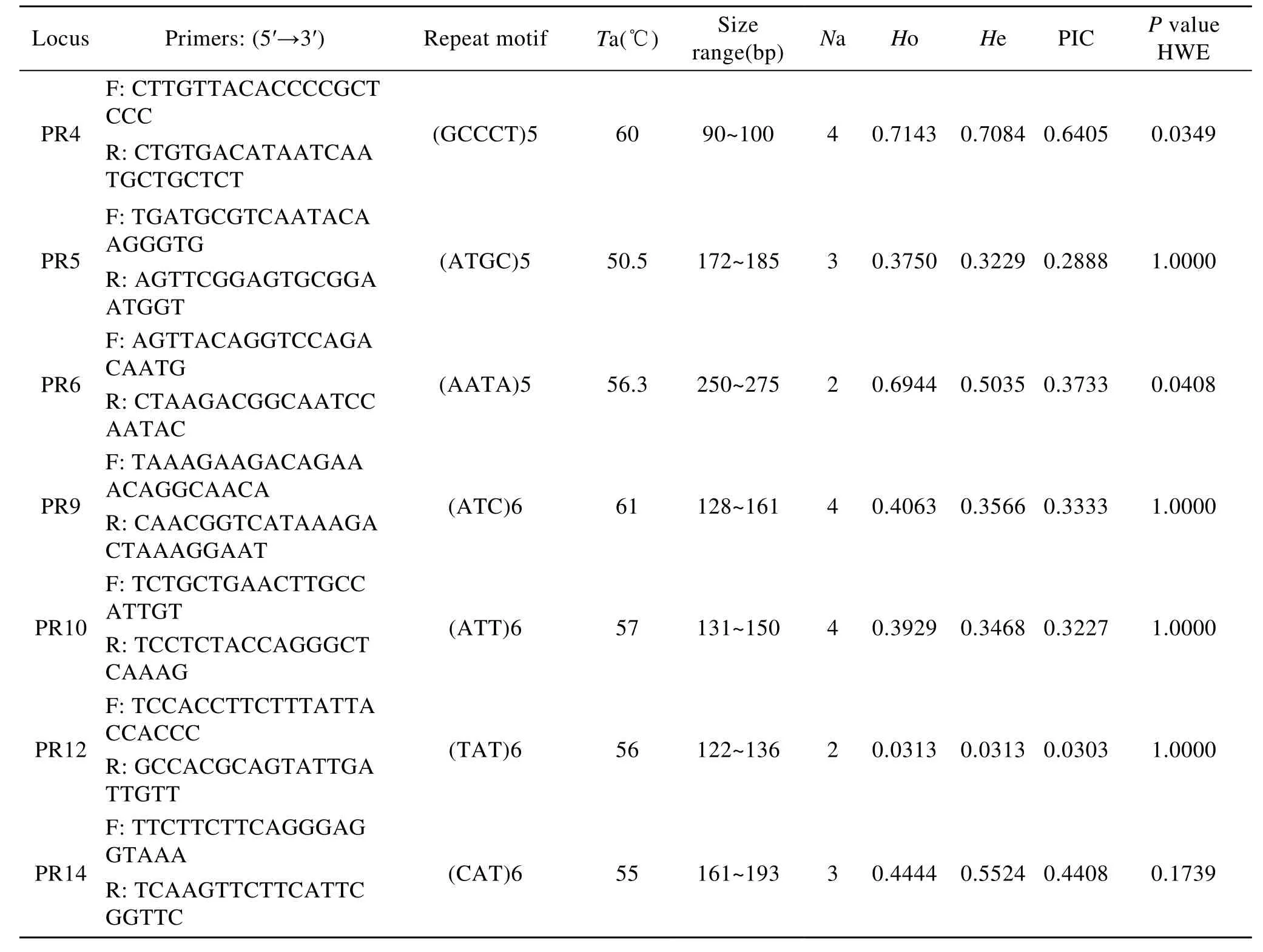
表2 14对SSR引物在32个里氏拟石磺扩增特性的描述Tab.2 Primer sequences and characterization of 14 polymorphic microsatellite loci in 32 Paraoncidium reevesii

续表
3 讨论和结论
SSR是一种应用广泛的分子标记。利用高通量测序技术对里氏拟石磺进行微卫星分子标记的开发不仅比传统方法更为快捷, 花费更少, 而且在获得微卫星位点的同时可以查找对应EST序列的功能性状, 与基因关联性较强。相比于基因组SSR, 基于转录组EST序列开发的SSR多态性较低[17], 本实验共获得14个具有多态性的微卫星位点, 其中2个微卫星位点表现为高度多态性(PIC>0.5), 10个位点表现为中度多态性(0.5>PIC>0.25), 2个位点表现为低度多态性(PIC<0.25), 这表明湛江里氏拟石磺野生群体的遗传多态性处于中度多态性水平。14个位点中有5个位点偏离平衡(P<0.05), 可能的原因是种群中有个体产生基因漂变, 或者出现了无效等位基因[18]。
作为海洋无脊椎动物向陆地延伸的过渡物种,石磺科贝类认为是研究海洋贝类向陆地辐射生活的最好代表。而利用微卫星分子标记来研究水产动物的系统进化已经得到广泛引用: 汪桂玲[19]等曾利用微卫星分析五大湖野生三角帆蚌群体的遗传多样性和种间发生关系; 董秋芬[20]等利用13个微卫星位点对 9种石斑鱼的种系发生关系进行研究。这些都为利用SSR标记研究石磺科贝类间种系发生提供了借鉴。本研究中利用高通量测序技术开发的14个微卫星位点, 不仅为研究里氏拟石磺的遗传多样性提供了基础数据, 也可用于石磺科贝类间种系发生关系的研究中。
[1] 吴旭峰, 沈和定, 吴文健, 等.我国华东沿海4种石磺形态学比较[J].动物学杂志, 2010, 6: 92-100.
[2] Bouchet P, Rocroi J. Classification and nomenclator of gastropod families [J]. Malacologia, 2005, 47(1): 2.
[3] 王冬凤, 沈和定, 吴欣. 石磺科 3种贝类皮肤显微结构比较[J].动物学杂志, 2015, 50(3): 437-444.
[4] 黄金田, 王爱民. 瘤背石磺营养成分分析及品质评价[J]. 海洋科学, 2008, 32(11): 29-35.
[5] 孙变娜, 沈和定, 吴洪喜, 等.四种石磺科贝类的胆甾醇含量测定[J].海洋科学, 2015, 39(1): 24-28.
[6] Sun B, Shen H, Wu H, et al. Determination of Chemical Constituents of the Marine Pulmonate Slug,Paraoncidium reevesii [J]. Tropical Journal of Pharmaceutical Research, 2015, 13(12): 2071-2074.
[7] Zhu H C, Wei L L, Shen H D, et al. Complete mitochondrial genome ofParaoncidium reevesii(Gastropoda, Pulmonata, Systellommatophora,Onchidiidae)[J]. Mitochondrial DNA, 2012, 23(5):379-381.
[8] Sun B, Chen C, Shen H, et al. Species diversity of Onchidiidae (Eupulmonata: Heterobranchia) on the mainland of China based on molecular data[J].Molluscan Research, 2014, 34(1): 62-70.
[9] 周娜, 沈和定, 陈诚. 基于线粒体 COⅠ基因的中国沿海和泰国普吉岛里氏拟石磺群体遗传结构分析[J].水产学报, 2014, 01: 33-40.
[10] Tautz D. Hypervariability of simple sequences as a general source for polymorphic DNA markers[J].Nucleic Acids Research, 1989, 17 ( 16): 6463-6471.
[11] 孙效文, 张晓锋, 赵莹莹, 等. 水产生物微卫星标记技术研究进展及其应用[J]. 中国水产科学, 2008,15(4): 689-703.
[12] Tillier S. A new mountain Platevindex from Philippine islands (Pulmonata: Onchidiidae) [J]. Journal of Molluscan Studies, 1983, 49(supp12A): 198-202.
[13] Klussmann-Kolb A, Dinapoli A, Kuhn K, et al. From sea to land and beyond–new insights into the evolution of euthyneuran Gastropoda (Mollusca)[J]. BMC Evolutionary Biology, 2008, 8(1): 57.
[14] 李强, 万建民. SSRHunter, 一个本地化的 SSR位点搜索软件的开发[J]. 遗传, 2005, 05: 808-810.
[15] Kalinowski S T, Taper M L, Marshall T C. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment [J]. Molecular Ecology, 2007, 16(5):1099-1106.
[16] Rousset F. genepop’007: a complete re implementation of the genepop software for Windows and Linux [J].Molecular Ecology Resources, 2008, 8(1): 103-106.
[17] 程小毛, 黄晓霞. SSR标记开发及其在植物中的应用[J]. 中国农学通报, 2011, 27(5): 304-307.
[18] Liu N, Li S, Zhang J. Isolation and characterization of 16 polymorphic microsatellite loci in the leopard coral grouperPlectropomus leopardus[J]. Conservation Genetics Resources, 2013, 5(4): 1067-1069.
[19] 汪桂玲, 袁一鸣, 李家乐.中国五大湖三角帆蚌群体遗传多样性及亲缘关系的 SSR分析[J].水产学报,2007, 31(2): 152-158.
[20] 董秋芬, 刘楚吾, 郭昱嵩, 等. 9种石斑鱼遗传多样性和系统发生关系的微卫星分析[J]. 遗传, 2007, 29(7):837-843.
