产褐藻胶裂解酶菌种的筛选、鉴定及发酵条件优化
2015-11-02朱玥明韩文佳贾士儒孙媛霞
陈 朋,朱玥明,韩文佳,门 燕,贾士儒,孙媛霞,*
(1.天津科技大学生物工程学院,天津 300457;2.中国科学院天津工业生物技术研究所, 天津 300308)
产褐藻胶裂解酶菌种的筛选、鉴定及发酵条件优化
陈 朋1,2,朱玥明2,韩文佳1,2,门 燕2,贾士儒1,孙媛霞2,*
(1.天津科技大学生物工程学院,天津 300457;2.中国科学院天津工业生物技术研究所, 天津 300308)
以 褐藻酸钠为唯一碳源,经多次富集和驯化,从养殖场腐烂海带中筛选到一株高产褐藻胶裂解酶的菌株Alg07。依据菌体形态、生理生化特征和16S rRNA基因序列分析,归属于芽孢杆菌属(Bacillus),命名为Bacillus weihaiensis Alg07。通过单因素试验和正交试验,确 定菌株Alg07的最佳发酵产酶培养基成分:褐藻酸钠9 g/L、蛋白胨1 g/L、酵母粉3 g/L、NaCl 5 g/L、MgSO4·7H2O 1 g/L、KCl 5 g/L、CaCl24 g/L;最佳发酵产酶条件为:250 mL三角瓶装液量40 mL、培养温度30 ℃、初始发 酵pH 6.5、接种量0.5%、摇床转速180 r/min、培养时间24 h。在优化后的培养条件下,褐藻胶裂解酶活力由35 U/mL提高到563 U/mL。
褐藻胶裂解酶;筛选;鉴定;芽孢杆菌;发酵条件优化
褐藻胶是由β-D-甘露糖醛酸(β-D-1,4-mannuronic acid,简称M)和其5位差向异构体α-L-古罗糖醛酸(α-L-1,4-guluronic acid,简称G)以C-1,4糖苷键非均聚形成的线性分子。根据两种糖的组合方式不同,褐藻胶具有3 种结构单元,聚甘露糖醛酸片段(polymannuronic acid,PM)、聚古罗糖醛酸片段(polyguluronic acid,PG)以及甘露糖醛酸与古罗糖醛酸的交替片段(PMG)[1]。研究发现褐藻胶的降解产物--褐藻寡糖稳定性高、水溶性强、安全无毒,具有抗凝血[2]、抗肿瘤[3]、促进肠道内双歧杆菌生长[4]、增强植物抗逆性[5]等众多生理活性,其开发与应用研究已经成为新的热点。与传统的酸降解的方法制备褐藻寡糖不同,褐藻胶裂解酶酶解生产褐藻寡糖具有催化效率高、过程可控、降解条件温和、节约能源、产物收率高等优点。此外,褐藻胶裂解酶可以作为工具酶用于治疗囊性纤维化(cysticfibrosis,CF)患者的肺部感染[6],褐藻胶精细结构的分析[7],海藻单细胞及原生质体的制备。2012年Wargacki等[8]在Science上发表用褐藻胶裂解酶降解褐藻胶来生产生物乙醇的论文,为解决能源危机提供了新的思路。
褐藻胶裂解酶通过β-消除反应催化降解褐藻胶单体间的1,4糖苷键,在C4、C5之间形成不饱和双键,并伴随4-O-糖苷键的消除,且在非还原端生成4-脱氧-L-erthro-hex-4-烯醇式吡喃糖醛酸(4-deoxy-L-erythro-hex-4-enopyranosyl uro nic acid)[9]。褐藻胶裂解酶按其降解褐藻多糖作用方式的不同在EC数据库中被分为多聚β-D-1,4-甘露糖醛酸裂解酶(EC 4.2.2.3)和多聚α-L-1,4-古洛糖醛酸裂解酶(EC 4.2.2.11)[10]。在自然界中能够产生褐藻胶裂解酶的生物分布广泛,已报道产褐藻胶裂解酶的物种有海洋软体动物[11]、棘皮动物[12]、细菌[13]、真菌[14]等,其中由于细菌生长快、产酶量高,因此相关研究最多。目前褐藻胶裂解酶的产品开发依然面临酶活力较低、酶稳定性差、酶的底物谱窄等问题。目前,国际市场上只有Sigma公司的一种产自黄菌属(Flavobacterium sp.)的褐藻胶裂解酶酶制剂产品A1603问世,价格昂贵,主要以试剂形式出售,这极大地限制了褐藻胶裂解酶在食品、农业及医药等领域的广泛应用。
本实验以褐藻酸钠为唯一碳源,经过多次富集和驯化,从腐烂海带中分离筛选得到一株高产褐藻胶裂解酶的菌株,通过形态观察、生理生化实验和16S rRNA基因序列分析进行菌种鉴定,并对其产酶条件进行优化,以期为褐藻胶裂解酶的分离纯化、酶学性质研究及褐藻寡糖的酶法制备奠定基础。
1 材料与方法
1.1材料、试剂与培养基
筛选样品来源于威海青渔滩海珍品养殖场腐烂海带。
海藻酸钠 国药集团化学试剂有限公司;二硝基水杨酸(dinitrosaliccylic acid,DNS) 生工生物工程(上海)股份有限公司;细菌基因组DNA提取试剂盒、琼脂糖凝胶DNA回收试剂盒 天根生化科技(北京)有限公司;扩增16S rRNA基因引物由金唯智生物科技有限公司合成,其他试剂均为国产分析纯。
富集培养基:海藻酸钠5 g、(NH4)2SO45 g、NaCl 19.45 g、MgCl2·6H2O 12.6 g、Mg SO4·7H2O 6.64 g、KCl 0.55 g、NaHCO30.16 g、柠檬酸铁(FeC6H5O7)0.1 g、蒸馏水1 000 mL,pH 7.2。初筛培养基在富集培养基的基础上添加15 g琼脂。121 ℃湿热灭菌20 min。
初始发酵培养基:海藻酸钠5 g、蛋白胨5 g、酵母膏1 g、NaCl 19.45 g,蒸馏水1 000 mL,pH 7.2。
1.2仪器与设备
UV-1800紫外分光光度计 日本岛津公司;HZQ-C空气浴振荡器 哈尔滨市东联电子技术有限公司;聚合酶链式反应(polymerase chain reaction,PCR)仪 德国Eppendorf公司;恒温培养箱 上海福玛实验设备有限公司;生物显微镜 日本Olympus公司。
1.3方法
1.3.1产酶菌株的筛选
取5 g样品放入盛有45 mL富集培养基的250 mL三角瓶中,在30 ℃、180 r/min条件下摇瓶发酵培养48 h。培养液用无菌生理盐水按照10-3到10-7进行梯度稀释,取0.1 mL的稀释液涂布于初筛固体平板上,30 ℃恒温培养48 h后观察,将先生长的或有透明圈的菌落用平板划线法进行菌株的分离纯化,将分离纯化的菌株用甘油管保存。将分离纯化的菌株接入初始发酵培养基,30 ℃、180 r/min培养48 h,取发酵液,12 000 r/min离心15 min,测定发酵上清液酶活力。
1.3.2褐藻胶裂解酶酶活力测定
取0.2 mL粗酶液加入1.8 mL用pH 8.0 Tris-HCl缓冲液配制的1 g/100 mL海藻酸钠底物中,空白对照只加入1.8 mL底物,40 ℃条件下反应20 min,100 ℃灭活5 min,在空白管中加入0.2 mL已经灭活的粗酶液,加入DNS试剂1.5 mL,沸水浴5 min,冷却至室温,定容至10 mL,于540 nm波长处测定吸光度。每次做3 个平行。
酶活力单位(U)定义为在实验条件下,每分钟催化底物产生1 ☒g还原糖所需的酶量。
相对酶活力:将实验组中最高的酶活力定义为100%,其他条件下的酶活力与最高酶活力的比值为相对酶活力。
1.3.3菌种鉴定
1.3.3.1形态学鉴定及生理生化实验
取发酵培养24 h的菌液稀释至合适浓度,涂布于初筛培养基,30 ℃倒置培养48 h,观察菌落形态及细胞形态(革兰氏染色法)。生理生化鉴定参照《伯杰细菌鉴定手册》[15]。
1.3.3.216S rRNA基因鉴定
用细菌基因组D N A提取试剂盒提取该菌的全基因组,以1 6 S r R N A基因的通用引物2 7 F(5'-AGAGTTTGATCCTGGCTCAG-3')、1492R(5'-GGTTACCTTGTTACGACTT-3')进行扩增,扩增片段送至金唯至生物科技公司测序。将测序结果的基因序列输入美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)核酸数据库进行BLAST分析,采用Clustal X进行完全序列比对切割,将结果输入MEGA6.0软件中,以Neighbor-Joining方法构建系统发育树[16]。
1.3.4初始培养条件
在250 mL三角瓶中装入50 mL初始发酵培养基,121 ℃灭菌20 m in,将培养至对数期后期的种子液无菌接种到三角瓶,接种量为2%,30 ℃、180 r/min条件下培养48 h,测发酵液酶活力,所有实验设计3 次平行。前一步优化的结果用于后续实验。
1.3.5培养基成分优化
通过单因素试验研究碳源种类及其质量浓度、NaCl质量浓度、氮源种类及各种无机盐对菌株产酶的影响。在单因素试验的基础上,通过正交试验确定对菌株产酶有促进作用的各种组分的最佳组合质量浓度。
1.3.5.1碳源种类对产酶的影响
分别选用5 g/L的葡萄糖、乳糖、甘露糖、D-甘露醇、麦芽糖、淀粉和海藻酸钠作为唯一碳源配制发酵培养基,确定最佳碳源。
1.3.5.2海藻酸钠质量浓度对产酶的影响
在初始培养基中添加0、3、6、9、12、15 g/L的海藻酸钠,确定菌株发酵产酶的最佳碳源质量浓度。
1.3.5.3氮源种类对产酶的影响
添加9 g/L的海藻酸钠,分别用5 g/L的有机氮源(牛肉膏、蛋白胨、酵母粉、尿素)、无机氮源((NH4)2SO4、NH4Cl、(NH4)3PO4)及2.5 g/L的复合氮源(尿素+蛋白胨、酵母粉+蛋白胨)作为氮源进行菌株的发酵培养,考察不同氮源对产酶的影响。
1.3.5.4无机盐对产酶的影响
在发酵培养基中分别添加0、5、10、15、20、25、30 g/L的NaCl,确定菌株发酵产酶的最佳NaCl质量浓度。在最佳的NaCl质量浓度的基础上,在发酵培养基中分别添加MgSO4·7H2O、KCl、CaCl2、ZnSO4·7H2O、MnSO4·H2O、CuSO4·5H2O、FeSO4·7H2O、FeCl3·6H2O、FeC6H5O7,其中MgSO4·7H2O、KCl、CaCl2的质量浓度均为1 g/L,其他无机盐的质量浓度均为0.05 g/L,确定对菌株的产酶有促进作用的无机盐。
1.3.5.5MgSO4·7H2O、KCl、CaCl2质量浓度对产酶的影响
在发酵培养基中分别添加0、1、2、3、4、5、6 g/L的MgSO4·7H2O、KCl、CaCl2,确定菌株发酵产酶的最佳无机盐质量浓度。
1.3.5.6培养基成分的正交试验
选择海藻酸钠、蛋白胨、酵母粉、N a C l、MgSO4·7H2O、KCl、CaCl2质量浓度7 个因素为主要影响因素,按照七因素三水平进行正交试验(L18(37))。
1.3.6培养条件优化
以优化后的培养基成分配制发酵培养基,通过单因素试验研究培养温度(15~40 ℃)、初始发酵pH值(5.5~10)、接种量(0.1%~3%)、装液量(20~100 mL/250 mL)对菌株产酶的影响。
2 结果与分析
2.1菌种筛选
经富集培养、分离及纯化,共筛选到11 株能在以褐藻酸钠为唯一碳源的初筛培养基上生长且能形成透明圈的菌株。将筛选到的11 株菌进行发酵培养,进行复筛,对其产酶能力进行比较。结果表明,菌株Alg07发酵上清液酶活力最高。经5 代遗传稳定性培养证明,该菌株产酶能力稳定,作为下一步研究的出发菌株。
2.2菌株Alg07的鉴定
2.2.1形态学鉴定
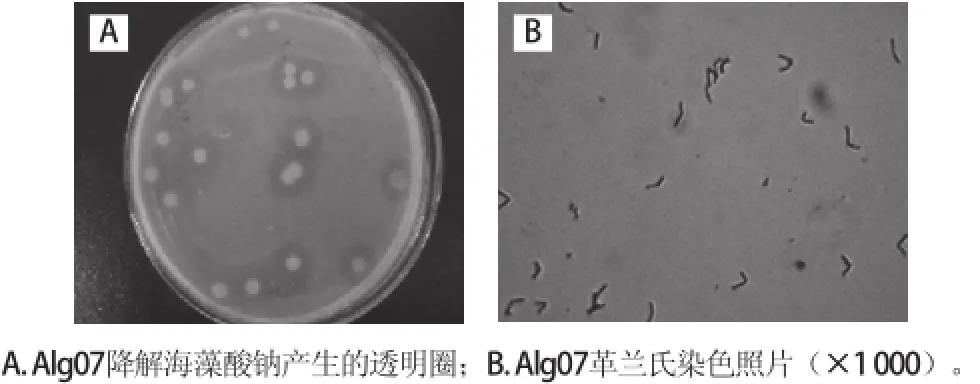
图1 菌株Alg07的形态特征Fig.1 Morphological characteristics of strain Alg07
由图1可知,菌株Alg07在初筛培养基上生长48 h后,菌落呈圆形,淡黄色,菌落较大,中间不透明,边缘半透明,表面粗糙。菌株Alg07在显微镜下为杆状,革兰氏染色阳性。
2.2.2生理生化特征
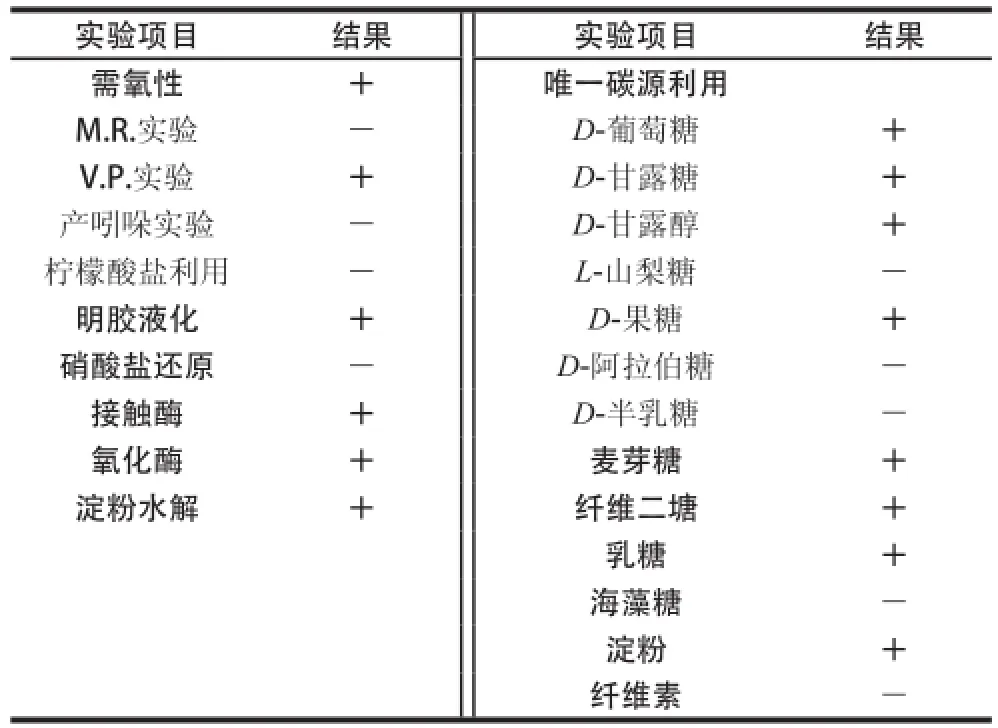
表1 菌株Alg07生理生化特征Table 1 Physiological and biochemical characteristics of strain Alg07
由表1可知,菌株Alg07可在15~45 ℃、pH 5.5~10.5范围内生长,最适生长温度为37 ℃、最适生长pH值为8.5,最高能耐受12 g/100 mL的NaCl。
2.2.316S rRNA基因鉴定
菌株Alg07的16S rRNA基因序列长度为1 497 bp,该序列已提交至NCBI GenBank,其登录号为KM040772。经过比对,与菌株Alg07相似性97%以上的菌株大部分为不可培养的菌株,少数为没有鉴定到种的菌株。与菌株Alg07相似性最高的菌株为B. litoralis S20409(97%)和B. simplex J2S3(97%)。用Mega软件绘制Alg07系统进化树,结果见图2。菌株Alg07与其他已鉴定到种的芽孢杆菌亲缘关系较远,结合形态特征及生理生化特征将其鉴定为芽孢杆菌属(Bacillus),命名为Bacillus weihaiensis Alg07。
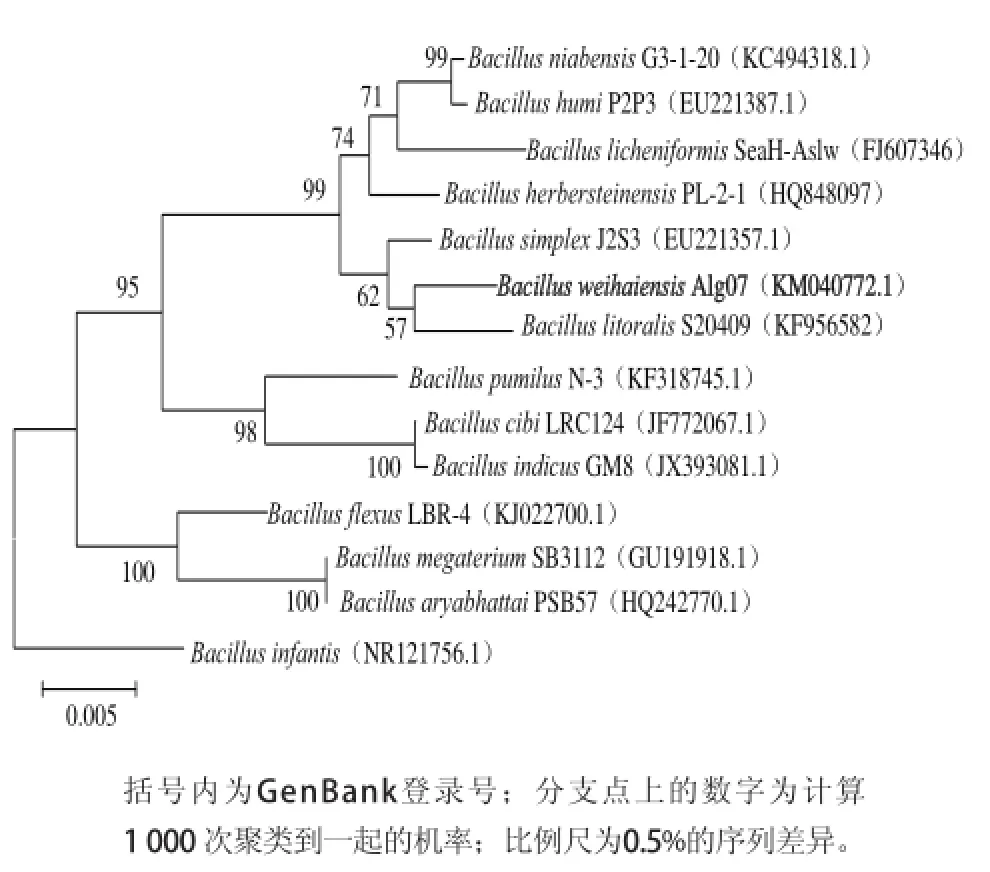
图2 依据16S rRNA基因序列构建的菌株Alg07的系统发育树Fig.2 Phylogenetic tree of strain Alg07 based on 16S rRNA gene sequence
2.3产酶条件优化
2.3.1海藻酸钠质量浓度对产酶的影响
实验发现,菌株Alg07只有以海藻酸钠为碳源时才产酶,说明该菌株产生的褐藻胶裂解酶为诱导酶,这与文献报道的相一致[17-18]。不同海藻酸钠质量浓度对菌株Alg07产酶的影响见图3。
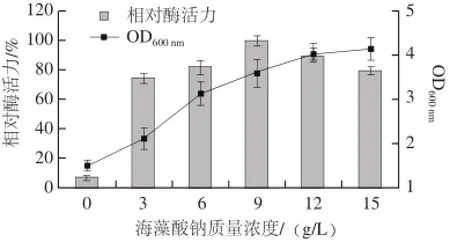
图3 海藻酸钠质量浓度对菌株Alg07产酶的影响Fig.3 Influence of alginate concentration of on enzyme production
由图3可知,当海藻酸钠质量浓度为9 g/L时,其产酶达到最高值,海藻酸钠质量浓度超过9 g/L时,菌株产酶不断降低,原因可能是褐藻胶降解产物会对褐藻酸裂解酶的生物合成产生阻遏。但其菌体生物量(用OD600nm表示)却随着海藻酸钠质量浓度的增加不断增大,可能是海藻酸钠中的色素影响了OD600nm的测定。
2.3.2氮源种类对产酶的影响
氮源是构成生物体蛋白质、核酸及氮素类化合物的原料,是微生物代谢生长的重要营养物质,对菌体的生长及产酶具有重要影响。由表2可知,菌株Alg07能在多种氮源条件下生长,但在以酵母粉与蛋白胨为复合氮源时菌株产酶能力与菌体生物量均明显高于其他氮源。因此,培养基的最适氮源为酵母粉与蛋白胨组成的有机复合氮源。可能是酵母粉与蛋白胨中有许多的小肽及微量元素有利于菌株的生长及产酶。
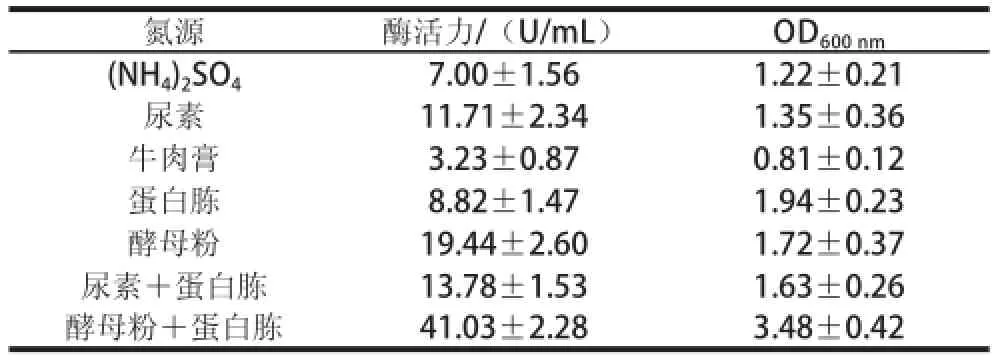
表2 不同氮源对菌株Alg07产酶的影响Table 2 Influence of different nitrogen sources on enzyme production
2.3.3无机盐对产酶的影响
由于菌株Alg07是由海洋附近的腐烂海带中筛选获得的,对盐度的依赖是海洋微生物的基本特征。因此,首先测定了不同质量浓度的NaCl对菌株发酵产酶的影响,实验发现,不同质量浓度的NaCl对菌株发酵产酶影响不显著,当NaCl质量浓度为5 g/L时,菌株产酶达到最大值,选择5 g/L的NaCl质量浓度作为下一步研究的基础。接下来,研究了各种无机盐对菌株发酵产酶的影响,发现MgSO4·7H2O、KCl、CaCl2对菌株Alg07产酶促进作用明显,其余金属离子对菌株的产酶均有不同程度的抑制作用。进一步测定了不同质量浓度的MgSO4·7H2O、KCl、CaCl2对菌种Alg07产酶的影响。实验发现,不同质量浓度的MgSO4·7H2O、KCl对菌株Alg07的产酶无明显的规律性,但不添加这两种无机盐会导致菌株产酶量明显降低。不同质量浓度的CaCl2对菌株的产酶促进作用趋势明显,结果见图4。
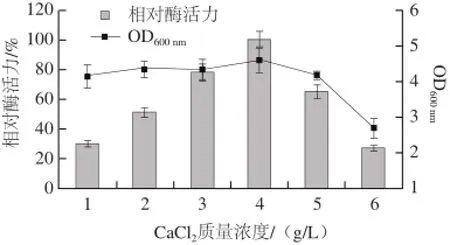
图4 CaaCCll2质量浓度对菌株Alg07产酶的影响Fig.4 Influence ofof CaCl2concentration on enzyme production
由图4可知,当CaCl2质量浓度低于5 g/L时,菌体的生物量变化不大,但不同质量浓度的CaCl2对菌株的产酶影响非常显著。当CaCl2质量浓度为4 g/L时,菌株产酶的能力最高,CaCl2质量浓度过高,过多的CaCl2与海藻酸钠形成沉淀,影响菌体的生长,从而导致菌体产酶量减少。
2.3.4培养基成分正交试验优化

表3 正交试验分析培养基成分对菌株Alg07产酶的影响Table 3 Orthogonal array analysis of the effect of different medium components on enzyme production
在用单因素优化氮源的试验中由于菌株Alg07只有在蛋白胨与酵母粉共同存在时,菌株生长较好,发酵上清液酶活力较高,但单因素试验无法确定蛋白胨与酵母粉的最佳组合质量浓度。另外,培养基中各种成分可能存在相互作用,导致MgSO4·7H2O、KCl对产酶的影响并没有很强的规律性,但不添加这两种无机盐又导致菌株产酶量较低。所以,设计正交试验确定蛋白胨与酵母粉的最佳组合,及最佳的产酶MgSO4·7H2O、KCl质量浓度。由表3可知,菌株Alg07发酵产酶的最佳培养条件为:A2B1C2D1E3F3G2。即海藻酸钠质量浓度为9 g/L、蛋白胨质量浓度为1 g/L、酵母粉质量浓度为3 g/L、NaCl质量浓度为5 g/L、MgSO4·7H2O质量浓度为5 g/L、KCl质量浓度为5 g/L、CaCl2质量浓度为4 g/L。按照极差的大小可以决定因素的主次顺序为:B>G>F>A>C>E>D。因此,B为影响菌株产酶活力的主要因素,即蛋白胨质量浓度对菌株产酶的影响最为显著,由表3可知,氮源的最佳组合质量浓度为蛋白胨1 g/L、酵母粉3 g/L。表3还显示,CaCl2质量浓度对菌株产酶的影响仅次于蛋白胨,不同质量浓度的NaCl、MgSO4·7H2O对菌株产酶的影响很小,且无明显的规律性,与单因素试验所得结果相符,选择5 g/L的NaCl和1 g/L的MgSO4·7H2O,进入下一步试验。
2.3.5培养条件对菌株Alg07产酶的影响
以上述优化的培养基成分制备发酵培养基,考察了培养温度、初始发酵pH值、接种量、装液量对菌株Alg07产酶的影响,结果如图5所示。当培养温度低于20 ℃时,菌体生物量和产酶量都较低。培养温度在20~30 ℃时,酶活力较高,且无明显差异,当培养温度高于30 ℃时,菌体生物量无明显降低,但酶活力降低迅速。考虑到实际生产过程中培养温度过低难以控制,因此培养温度选择30 ℃(图5A)。由图5B可知,培养基的初始pH值对菌体生物量几乎没有影响,对菌体产酶影响也不太显著,当发酵培养基初始pH值为6.5时,菌株产酶达到最大值。
进一步对接种量进行考察,结果表明当接种量为0.5%以下时,菌体生物量与产酶均较低,接种量高于0.5%时,菌种的产酶量相差不大,因此接种量选择0.5%(图5C)。在考察溶氧条件对产酶影响时,结果显示在250 mL三角瓶装液量小于40 mL,菌株Alg07产酶量较高,说明该菌是好氧菌,提高溶氧有利于菌体生长与酶的合成。但由于培养基量过少,可能会使细菌过早进入衰亡期,而且随着发酵时间的延长,最后剩下的发酵液会很少,所以选择了40 mL装液量(图5D)。

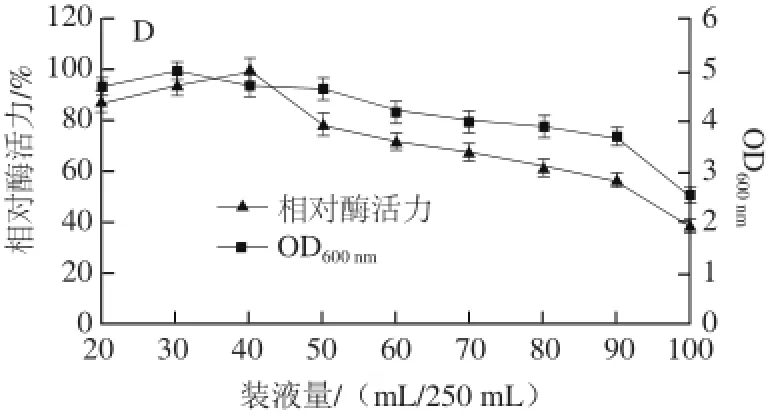
图5 培养条件对菌株Alg07产酶的影响Fig.5 Influence of culture conditions on enzyme production
2.3.6菌株Alg07的发酵产酶曲线
以优化后的培养基成分与培养条件发酵培养菌株Alg07,测定不同发酵时间的菌体生物量与发酵上清液酶活力,结果如图6所示。
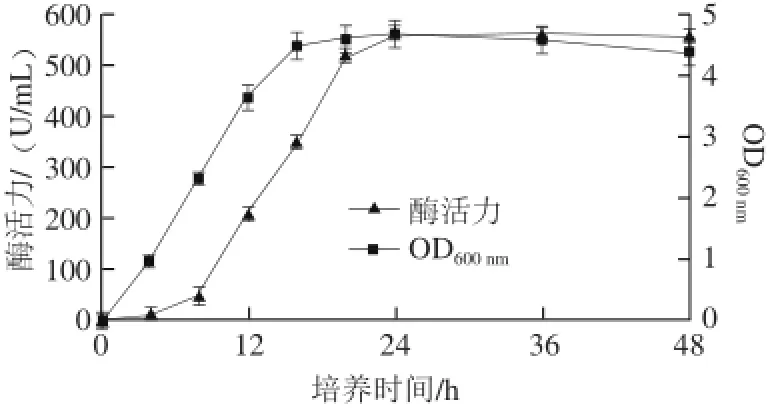
图6 培养时间对菌株Alg07产酶的影响Fig.6 Influence of culture time on enzyme production
由图6可知,菌株Alg07在16 h生物量即达到最大值,24 h发酵上清液酶活力最高,为563 U/mL。菌体的产酶比菌体生长略微滞后,可见是菌体生长后,褐藻酸钠再诱导其产酶,同时,也证明了菌株Alg07所产褐藻胶裂解酶是诱导酶。当酶活力达到最高值后,继续培养24 h,发酵上清液酶活力并没有显著的下降,可能是该酶稳定性较好导致的。目前已报道的产褐藻胶裂解酶细菌主要来源于假单胞菌[19]、弧菌[20]、产黄菌属[13]等。关于芽孢杆菌产褐藻胶裂解酶的报道则较少,1984年,Hansen等[21]首次从土壤和海水中分离出一株产褐藻胶裂解酶的芽孢杆菌JBH2,为革兰氏阳性细菌,好氧,褐藻胶裂解酶在对数生长期出现合成的高峰期,发酵培养48 h其酶活力仅有8.0 U/mL。2010年,刘玉佩等[22]利用响应面法对解淀粉芽孢杆菌生产褐藻胶裂解酶的发酵条件进行了优化,在最优的发酵条件下,发酵培养42 h,理论酶活力为6.48 U/mL。之后,他们对产褐藻胶裂解酶的基因进行了异源表达[23],使酶活力提高到了21 U/mL。Masayuki等[24]从海洋沉淀物中发现一株能够降解海带的Microbulbifer sp. 6532A,培养2 d以后,其褐藻胶裂解酶活力达114.8 U/mL。Li Weijian等[25]筛出Pseudoalteromonas sp. SM0524,产酶条件优化后,发酵周期60 h,酶活力达到62.6 U/mL。An Qingda等[26]从巨藻中筛选出Flavobacterium sp. LXA,培养温度30 ℃,培养时间32 h,酶活力达到30.1 U/mL。
本研究获得的菌株Alg07发酵温度为30 ℃,发酵周期24 h,酶活力为563 U/mL。与菌株JBH2[21]、6532A[24]、SM0524[25]、LXA[26]相比具有酶活力高、发酵周期短等优点。可进一步将对菌株Alg07产生的褐藻胶裂解酶进行分离纯化并进行酶学性质研究,通过克隆褐藻胶裂解酶的基因实现其异源表达或者通过菌种诱变的方式继续提高产酶量,实现褐藻胶裂解酶的工业化生产。
3 结 论
从养殖场腐烂海带中筛选到一株新型的高产褐藻胶裂解酶的菌株Alg07。依据菌体形态、生理生化特征和16S rRNA基因序列分析,鉴定为芽孢杆菌属(Bacillus),命名为Bacillus weihaiensis Alg07。通过单因素试验和正交试验,确定菌株Alg07的最佳发酵产酶培养基成分:褐藻酸钠 9 g/L、蛋白胨1 g/L、酵母粉3 g/L、NaCl 5 g/L、MgSO4·7H2O 1 g/L、KCl 5 g/L、CaCl24 g/L;最佳发酵产酶条件为:装液量40 mL/250 mL、培养温度30 ℃、初始发酵pH 6.5、接种量0.5%、摇床转速180 r/min、培养时间24 h。在优化后的培养条件下,褐藻胶裂解酶活力由35 U/mL提高到563 U/mL。菌株Alg07生产褐藻胶裂解酶具有酶活力高、发酵周期短等诸多优点,是一株新型的极具潜力的褐藻胶裂解酶生产菌株。
[1] HAUG A, LARSEN B, SMIDSRD O. A study of the constitution of alginic acid by partial acid hydrolysis[J]. Acta Chemical Scandinavice,1966, 20: 183-190.
[2] WALL D, DOUGLAS S, FERRO V, et al. Characterisation of the anticoagulant properties of a range of structurally diverse sulfated oligosaccharides[J]. Thrombosis Research, 2001, 103(4): 325-335.
[3] IWAMOTO Y, XU X, TAMURA T, et al. Enzymatically depolymerized alginate oligomers that cause cytotoxic cytodine production in human mononuclear cells[J]. Bioscience, Biotechnology, and Biochemistry,2003, 67(2): 258-263.
[4] AKIYAMA H, ENDO T, NAKAKITA R, et al. Effect of depolymerized alginates on the growth of bifi dobacteria[J]. Bioscience,Biotechnology, and Biochemistry, 1992, 56(2): 355-356.
[5] 刘瑞志. 褐藻寡糖促进植物生长与抗逆效应机理研究[D]. 青岛: 中国海洋大学, 2009: 98-112.
[6] LIN Xiao, FENG Han, ZHAO Yang, et al. A novel alginate lyase with high activity on acetylated alginate of Pseudomonas aeruginosa FRD1 from Pseudomonas sp.QD03[J]. World Journal of Microbiology and Biotechnology, 2006, 22(1): 81-88.
[7] BOYD J, TURVEY J R. Structure studies of alginic acid, using a bacterial poly-α-L-guluronate lyase[J]. Carbohydrate Research, 1988,66(1): 187-194.
[8] WARGACKI A J, LEONARD E, WIN M N, et al. An engineered microbial platform for direct biofuel production from brown macroalgae[J]. Science, 2012, 335: 308-313.
[9] MASAYUKI Y, SATOKO M, OSAMU M, et al. Structure and function of a hypothetical Pseudomonas aeruginosa protein PA1167 classifi edinto family PL-7[J]. The Journal of Biological Chemistry, 2004,279(30): 31863-31872.
[10] KIM H S, LEE C, LEE E Y. Alginate lyase: structure, property, and application[J]. Biotechnology and Bioprocess Engineering, 2011,16(5): 843-851.
[11] BOYEN C, KLOAREG B, POLNE-FULLER M, et al. Preparation of alginate lyases from marine molluscs for protoplast isolation in brown algae[J]. Phycologia, 1990, 29(2): 173-181.
[12] BOYEN C, BERTHEAU Y, BARBEYRON T, et al. Preparation of guluronate lyase from Pseudomonas alginovora for protoplast isolation in laminaria[J]. Enzyme and Microbial Technology, 1990, 12(11): 885-890.
[13] HUANG Lishuxin, ZHOU Jungang, LI Xiao, et al. Characterization of a new alginate lyase from newly isolated Flavobacterium sp. S20[J]. Journal of Industrial Microbiology & Biotechnology, 2013, 40(1): 113-122.
[14] GACESA P, WUSTEMAN F S. Plate assay for simultaneous detection of alginate lyases and determination of substrate specifi city[J]. Applied and Environment Microbiology, 1990, 56(7): 2265-2267.
[15] BUCHANANN B E, BERGEY N E. Bergey's manual of determinative bacteriology[M]. 9th ed. London: Williams and Wilkins Company,1994: 597-635.
[16] LI Changqing, LIU Wenchao, ZHU Ping, et al. Phylogenetic diversity of bacteria associated with the marine sponge Gelliodes carnosa collected from the haina island coastal waters of the South China Sea[J]. Environmental Microbiology, 2011, 62(4): 800-812.
[17] TOHRU K, KOHSUKE U, MASAYUKI M, et al. A new high-alkaline lyase from a deep-sea bacterium Agarivorans sp.[J]. Extremophiles,2009, 13(1): 121-129.
[18] 魏丹, 窦文芳, 李恒, 等. 高效降解褐藻胶新菌种的筛选、鉴定及产酶条件优化[J]. 食品与发酵工业, 2012, 38(7): 26-31.
[19] LI Liyan, JIANG Xiaolu, GUAN Huashi, et al. Three alginate lyases from marine bacterium Pseudomonas fl uorescens HZJ216: purification and characterization[J]. Applied Biochemistry Biotechnology, 2011,164(3): 305-317.
[20] ZHOU Jiushun, CAI Menghao, JIANG Tao, et al. Mixed carbon source control strategy for enhancing alginate lyase production by marine Vibrio sp. QY102[J]. Bioprocess and Biosystem Engineering,2014, 37(3): 575-584.
[21] HANSEN J B, DOUBET R S, RAM J. Alginase enzyme production by Bacillus circulans[J]. Applied and Environment Microbiology, 1984,47(4): 704-709.
[22] 刘玉佩, 汪丽萍, 赵勇, 等. 解淀粉芽孢杆菌产褐藻胶裂解酶的发酵条件优化[J]. 湖南农业科学, 2010(5): 17-20.
[23] 石荣莲, 汪立平, 刘玉佩. 重组褐藻胶裂解酶基因工程菌超声波破碎条件研究[J]. 湖南农业科学, 2010(21): 98-101.
[24] MASAYUKI W, AKIHIRO S, FUMIO N, et al. Isolation and characterization of Microbulbifer species 6532A degrading seaweed thalli to single cell detritus particles[J]. Biodegradation, 2012, 23(1):93-105.
[25] LI Weijian, DONG Sheng, SONG Jie, et al. Purification and characterization of a bifunctional alginate lyase from Pseudoalteromonas sp. SM0524[J]. Marine Drugs, 2011, 9(1): 109-123.
[26] AN Qingda, ZHANG Gongliang, WU Haitao, et al. Production and partial properties of alginase from newly isolated Flavobacterium sp. LXA[J]. Process Biochemistry, 2008, 43(8): 842-847.
Screening and Identification of a Bacterial Stra in and Optimization of Medium Composition and Culture Conditions for the Production of Alginate Lyase
CHEN Peng1,2, ZHU Yueming2, HAN Wenjia1,2, MEN Yan2, JIA Shiru1, SUN Yuanxia2,*
(1. College of Biotechnology, Tianjin University of Science and Technology, Tianjin 300457, China;2. Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China)
A highly efficient alginate-degrading microorganism was isolated from rotten seaweed through several cycles of enrichment and domestication in liquid medium containing alginate as the sole carbon source. According to morphological,physiological characteristics and 16S rRNA gene sequence analysis, the strain was iden tified as Bacillus weihaiensis Alg07. Based on the results of single factor experiments and orthogonal array experiments, the optimal liquid fermentation medium contained 9 g/L of alginate, 1 g/L of peptone, 3 g/L of yeast exact, 5 g/L of NaCl, 1 g/L of MgSO4·7H2O, 5 g/L of KCl and 4 g/L of CaCl2. The strain was cultured at 30 ℃, pH 6.5 and 180 r/min for 24 h in 40 mL of the medium using a 250-mL flask, and the inoculum concentration was 0.5% (V/V). Under these optimal conditions, the activity of alginate lyase was significantly increased from 35 U/mL to 563 U/mL, indicating a 16-fold increase compared with that before optimization.
alginate lyase; screening; identification; Bacillus; fermentation optimization
TS254.4
A
1002-6630(2015)15-0105-07
10.7506/spkx1002-6630-201515020
2014-09-17
国家高技术研究发展计划(863计划)项目(2013AA102105);中国科学院重点部署项目(KSZD-EW-Z-019)
陈朋(1988—),男,硕士研究生,研究方向为酶工程。E-mail:864724133@qq.com
孙媛霞(1963—),女,研究员,博士,研究方向为功能糖与天然活性物质。E-mail:syx0430@hotmail.com
