抗菌肽的定量构效关系研究进展
2015-11-02卢永忠康道乐
卢永忠,康道乐
(青岛科技大学化工学院,山东 青岛 266042)
抗菌肽的定量构效关系研究进展
卢永忠,康道乐
(青岛科技大学化工学院,山东 青岛 266042)
抗菌肽在医药、食品、农业及水产等领域具有潜在的应用价值,定量构效关系(quantitative structureactivity relationships,QSAR)研究对于指导抗菌肽的优化设计具有重要意义。本文介绍抗菌肽在分子表征及建模等方面取得的研究进展,比较抗菌肽与其他小肽在QSAR研究中的异同,指出其中存在的问题及今后的研究发展方向。
抗菌肽;定量构效关系;描述符;氨基酸;数学模型
抗菌肽是生物体抵御病原微生物入侵而产生的一类阳离子多肽,主要通过破坏靶细胞的细胞膜,从而发挥抗菌、抗病毒、抗肿瘤等多种活性[1]。与传统的抗生素相比,抗菌肽具有抗菌谱广、热稳定性好、抗菌机理独特等优点。特别是其抗肿瘤活性引起越来越多的研究者关注[2-3]。这种以细胞膜为靶标的作用机制的优势在于:1)作用速度快(几分钟时间),可以避免或延缓细胞抗药性的形成;2)靶细胞膜组成和结构的差异形成了其选择性作用的基础,可减少毒副作用的产生。因此,抗菌肽有望被开发成新一代抗菌制剂,在医药、食品、农业及水产等领域发挥积极作用[2-5]。
目前发现的抗菌肽大多数存在活性不高或特异性不强的缺陷,在杀伤靶细胞的同时会产生溶血性等毒副作用,限制了其在实践中的应用[2,6]。因此,抗菌肽的优化设计成为该领域一个重要的研究方向[7]。
为了避免传统的“黑箱效应”式的大规模合成与筛选,提高设计效率,定量构效关系(quantitative structureactivity relationships,QSAR)研究开始被应用于抗菌肽分子的优化设计[8]。通过对一系列抗菌肽的结构或理化性质的定量描述,并借助数学和统计学方法以确定化学结构与抗菌活性之间的关系,在此基础上进行新抗菌肽分子的活性预测以及高活性抗菌肽的设计,其中对抗菌肽分子结构的定量描述以及数学模型建立的方法是QSAR研究的重点[8]。
1 抗菌肽化学结构的定量描述
建立QSAR模型的关键之一是合理的分子表征,即分子结构参数化。如何对多肽这种具有柔性结构的复杂分子进行定量描述一直是多肽QSAR研究的重要课题。苦味素、催产素等小肽的QSAR研究为多肽结构表征和数学模型的建立积累了许多值得借鉴的经验[9-10]。但抗菌肽的组成、大小及作用机制不同于小肽,并且可形成α-螺旋、β-折叠等多种空间结构形式,因此又有其特殊性。抗菌肽化学结构的定量描述主要有如下两种方式:
1.1氨基酸描述符
大多数研究采用氨基酸描述符分别表征多肽分子的各个氨基酸,从而将肽的结构转换成结构描述符矩阵中的一个向量,最后建立描述符矩阵与肽活性之间的QSAR模型。氨基酸描述符则是采用多元统计方法从大量氨基酸性质参数中提取得到的少数关键信息成分[11]。由于组成抗菌肽的氨基酸数目较多(一般10~100 个),再加上每种氨基酸的信息涉及理化性质、拓扑结构、局部特征、整体特征等多方面,如果要将所有信息都涵盖,势必会增加计算的复杂性和噪音。因此在用氨基酸描述符表征抗菌肽时,尽可能提取与活性相关的特征,做到既全面精确,又简便易行。氨基酸的性质参数主要有3 个来源:1)收集已知氨基酸的传统物理化学参数,如疏水性、电性、立体特征及氢键性质等;2)通过实验测定或通过结构式的处理和对经验数据或实验数据的概括而获得上述物理化学参数;3)通过量子化学、分子力学和分子动力学等严格的理论计算获得,主要包括量子化学参数、拓扑参数等,这也是近年来研究的方向。在获得这些参数的基础上,利用主成分分析(principal component analysis,PCA)等方法从中获得有代表性的参数,即氨基酸描述符[8]。
涵盖的氨基酸种类、变量种类或计算方法不同,建立描述符的复杂程度也不同。如z-Scale是最早构建的物理化学参数描述符,从20 个编码氨基酸的29 种物化参数中提取与氨基酸的亲水性、立体形状和电性有关的3 个参数[9],后来又进一步扩展到5 个参数[12]。其他研究小组也相继建立了参数数目不等的物理化学描述符[13-17](表1),其中接触能描述符是最简单的一种[18]。
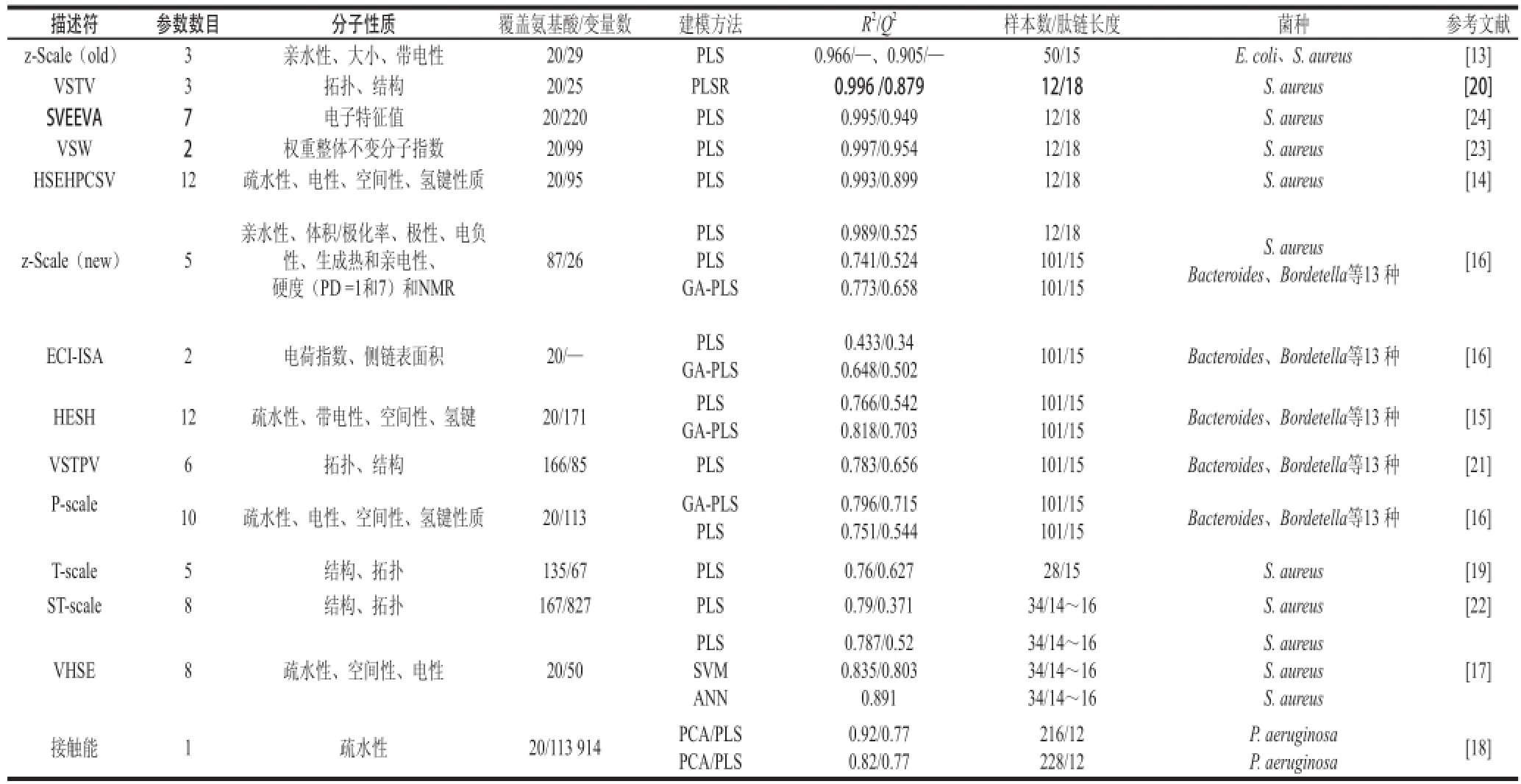
表1 应用于抗菌肽QSAR的氨基酸描述符Table 1 Amino acid descriptors used in QSAR of antimicrobial peptides
拓扑参数描述符的提出则是因为对非编码氨基酸表征的需要。它们往往采用计算的方法获得参数,再进行提取。如T-scale是从135 种氨基酸的67 种结构拓扑参数中提取得到[19]。梅虎[20]、Shu Mao[21]和Yang Li[22]等也分别建立了涵盖不同氨基酸及变量数的拓扑结构描述符[20-22]。
氨基酸的三维结构也是建立描述符的一个重要角度。Collantes等[10]通过计算获得了两种表征分子电荷指数及侧链表面积的描述符ISA-ECI,理应涵盖更丰富的结构信息,但在描述大分子肽时易出错。而Tong Jianbo等[23]构建的三维氨基酸描述符VSW表征抗菌肽的效果明显好于苦味素等小肽。
目前已经建立的氨基酸描述符有30 种左右[11],其中已经在抗菌肽QSAR研究中得到应用的至少有13 种(表1)。这些氨基酸描述符分别属于物理化学描述符(如z-Scale等7 种),三维结构描述符(如VSW、ECIISA),拓扑描述符(如T-scale、ST-scale、VSTV、VSTPV等),量子化学描述符(如SVEEVA)[24]。
在抗菌肽的QSAR研究中,物理化学描述符的种类最多,其中主要成分都涵盖了氨基酸的疏水性、带电性,说明了这些性质与抗菌肽活性有密切的相关性。许航等[11]曾指出,在多肽的QSAR研究中,氨基酸物化描述符的建模效果优于其他种类的描述符。在抗菌肽的QSAR研究中(表1),总体而言这几类描述符对同一组抗菌肽所建立的模型拟合性和预测性都很好,尤其是物理化学描述符HSEHPCSV、三维结构描述符VSW、拓扑描述符VSTV、量子化学描述符SVEEVA,在十八肽的QSAR研究中R2均超过0.99,Q2也在0.9附近[14,16,23-24]。ECI-ISA的建模效果不理想,但采用遗传算法(GA)进行变量筛选后,建模效果有了明显改善,R2从0.433上升到0.648,Q2超过0.5[16];ST-scale建模的预测性还有待提高。
从表1也可以看出,除了氨基酸描述符以外,样本数据也是影响建模效果的重要因素:1)多肽长度的影响。长度不同的多肽集的建模效果不如相同长度的多肽集。如长度为14~16 个氨基酸组成的多肽集,都采用偏最小二乘法(PLS)建模,无论采用哪种描述符,其拟合性和预测力都比长度为12 个或15 个氨基酸的多肽集差。T-scale虽然也是对长度为15 个氨基酸的多肽集建模,但效果不如其他描述符,暗示该拓扑描述符在多肽表征方面不是十分理想。2)多肽作用对象(菌种)的影响。如果抗菌肽活力的测定都以单一菌种为依据,则建模效果优于以多菌种为依据。从常用的这两组抗菌肽数据可以看出,12 个肽的抗菌活性数据来自对金黄色葡萄球菌(Staphylococcus aureus)的检测,而101 个抗菌肽的数据则来自对13 种不同菌种的检测;即使是单一菌种,针对不同菌种的建模效果也不同。如Lejon等[13]构建了同一组抗菌肽分别对大肠杆菌(Escherichia coli)和金黄葡萄球菌的QSAR模型,这两个模型的拟合性存在差异(R2分别为0.966和0.905),反映了作用对象对建模效果的影响。
应该指出的是,氨基酸描述符在表征多肽,尤其是抗菌肽时,未考虑肽链内部各氨基酸残基的交互影响,这是该方法存在的短板[11]。虽然已经构建的多种氨基酸描述符的复杂程度不同,但它们在表现上并没有质的差别。再者,利用氨基酸描述符表征多肽变量过多,过程复杂,且不易表征长度不同的多肽。因此许航等[11]建议不再按照传统思路进一步提出新的氨基酸描述符。
1.2分子整体描述符
鉴于氨基酸描述符存在的问题,从分子整体对多肽进行表征是另一种重要的方式。根据分子结构的维数,分子整体描述符可分为一维、二维、三维描述符;根据物理意义的不同,分子整体描述符可分为组成描述符、分子性质描述符、拓扑描述符、几何描述符等。这些描述符分别基于不同的理论或方法获得,如分子图论、理论或实验数据、原子组成、分子力场、分子形状等[25-31]。有多种软件可用于获取多肽分子的整体参数(表2)。它们采用了不同的计算体系,因此对结构信息提取的类型和数量各不相同,但也可涵盖一些共同的信息。这些软件大多为商业软件,也有一些可免费使用,如Mold2、E-dragon等,前提是多肽长度不超过7~8 个氨基酸[8]。
抗菌肽分子的整体表征是一个值得深入研究的课题,有关这方面的报道近年来逐渐增多。Patel等[25]选择39 个分子结构描述符对29 条氨基酸残基数不等的抗菌肽进行整体表征,并采用神经网络法分别建立了抗金黄色葡萄球菌活性和抗大肠杆菌活性的3D-QSAR模型。Sánchez-Gómez等[26]采用一种新的2D理化描述符PEDES表征多肽分子,成功建立了抗绿脓杆菌(Pseudomonas aeruginosa)的模型。Bhonsle等[27]以多肽分子的三维结构为基础,采用多向偏最小二乘法(multiple partial least squares,MPLS)分别建立了28 条多肽的抗金黄色葡萄球菌活性和抗蛙分枝杆菌(Mycobacterium ranae)活性的3D-QSAR模型。Torrent等[31]证明直接从氨基酸序列可提取足够的分子信息,取得良好的建模效果,并首次在描述符中包含了多肽聚集性这一参数。从上述研究可以看出,整体描述符性能良好,所建模型的拟合性和预测能力都优于氨基酸描述符;尤其在不同长度多肽的表征方面具有一定优势。同时,与氨基酸描述符相似,整体描述符基本上涵盖了抗菌肽分子的带电性、疏水性等特征,也再次证明了这两种性质和生物活性的相关性。上述研究同样也反映了作用对象(靶细胞的种类)对模型质量的影响[31]。
1.3抗菌肽与小肽表征方法的比较
无论是抗菌肽还是几个氨基酸组成的小肽,氨基酸描述符在对它们进行结构表征时原理和方法都是一致的,因此一般来讲氨基酸描述符是可以通用的[14,19-20]。但也有不同之处,毕竟氨基酸越多,表征多肽时涉及的变量越多,建模过程越复杂。许航等[11]系统分析了氨基酸描述符在不同多肽体系QSAR研究中的应用,发现总体上肽序列越短,模型复杂度越低,建模效果越好;肽序列越长,则影响因素越多,建模效果越差。但如果采用正确的建模方法,或多种方法结合,抗菌肽的建模效果甚至好于小肽[14,16]。另外,在分子整体表征时,往往需要涵盖抗菌肽一些特有的性质,如二级结构、聚集性等[31]。
2 建模方法的比较
针对同一组数据,采用不同的建模方法往往会获得不同的结果[17,32]。合理的建模方法(计算方法)的选择是建立抗菌肽分子参数与活性关系的关键。在QSAR研究中,常用的统计方法有多种,变量间存在线性关系时主要采用多元线性回归(multiple linear regression,MLR)、PLSR、PCA等。
2.1多元线性回归
MLR被用来描述一个因变量与多个自变量间的线性依存关系,原理简单易懂,物理意义明了,经常作为首选建模方法。但要求样本数至少是自变量数目的3 倍以上,而抗菌肽的变量数目无论是氨基酸描述符还是整体描述符都较多,但目前可利用的样本容量都不大,因此MLR较少被用于抗菌肽的建模研究,但如果结合逐步回归方法,先进行变量的筛选,以降低模型的复杂性和噪音问题,也可建立可信性和预测性良好的模型[33]。
2.2主成分分析
PCA是一种较经典的多元统计分析技术,它设法将原来的多个指标重新组合成一组新的、无相关性的几个综合指标,以替代原来的多个指标。在抗菌肽的QSAR研究中,PCA的作用主要体现在其对描述符的提取上,以一种优化的方法浓缩综合原始数据的信息,使数据矩阵简化、降低维数,是原来多个指标的线性组合。Jenssen等[18]采用氨基酸的接触能描述符,分别通过PCA法和PLSR法建立了两种数学模型,对绿脓杆菌的抗菌活性预测准确率分别达到了85%和71%。
2.3偏最小二乘回归
PLSR是在PCA的基础上发展起来的一种新型多元统计数据分析方法,集MLR分析、典型相关分析和PCA的基本功能于一体,不仅降低了数据维数,同时也有效地解决了变量间多重共线性的问题,计算结果更为可靠,可解释性也更强。也是抗菌肽QSAR中使用最多的建模方法[13-24]。实际应用时常将变量筛选与建模方法结合,可大大提高模型的质量[15-16]。
随着多肽分子整体表征技术的发展,更为高级的人工智能技术越来越多地应用于抗菌肽的QSAR研究(表3)。
2.4遗传算法
GA的主要思路来源于达尔文的自然选择学说,通过优胜劣汰使得要解决的问题逐渐逼近最优解,尤其适合处理变量筛选等寻优问题。GA法是一种只考虑输入和输出关系的黑箱式结构,不需要明确的数学表达式,通用性强,可广泛应用于离散问题及函数关系不明确的复杂问题。在抗菌肽研究中可用于变量的筛选,和其他建模方法结合,极大地提高了模型的质量[15-16]。GA法在抗菌肽识别方面也表现出极大的优势,其效率比ANN提高了近20 倍[34]。
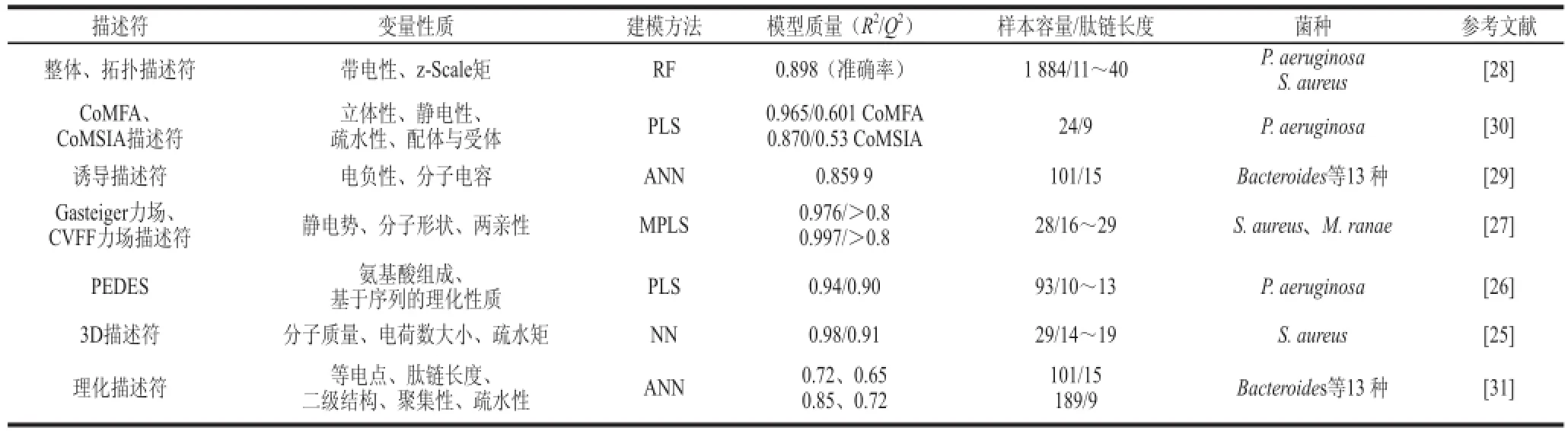
表3 抗菌肽分子整体描述符Table 3 Molecular descriptors for antimicrobial peptides
2.5人工神经网络
ANN是一种模拟人脑处理信息的过程,由大量基本信息处理单元通过复杂的连接而成,是一种非线性动力学系统,具有并行性、容错性、自学习性等特点,特别适合非线性问题的处理。在抗菌肽的QSAR中,其建模效果不亚于PLS等方法,但普及程度却不及PLS,无法解释描述符的贡献可能是其中一个重要原因,这也是ANN法存在的不足之处[29]。
2.6支持向量机
SVM以统计学习理论和结构风险最小原理为基础,根据有限的样本信息在模型的复杂性和学习能力之间寻求最佳折中,以期获得最好的推广能力。对于小样本、非线性问题,SVM具有较强的稳定性能及泛化能力,在大多数情况下能够得到优于PLS等传统方法的建模效果。对于分类问题,SVM对训练集和测试集都达到了100%的分类正确率。SVM 也有许多问题需要解决或改进,如核函数及相关参数的选择、训练速度的提高、多类问题及回归问题等[17]。
3 结 语
传统的建模方法仅根据样本集分子自身信息来构建预测模型,能够不同程度地反映抗菌肽分子结构性质和活性之间的关系,但都存在一个共同的问题:无论是2D-QSAR还是3D-QSAR,都没有体现靶细胞因素在建模中的作用,即数据异质性对建模效果的影响[35]。具体表现为:1)同一组多肽针对不同的靶细胞,建模效果不同[13,27];2)在不考虑作用对象差异的情况下,模型拟合性和预测力均下降(R2<0.9,Q2<0.8)[15-16,21,29,31]。
Mehla等[36]首次从表征细胞膜流动性和带电性的角度,利用ANN法建立了细胞膜膜脂组成与抗菌肽活性的QSAR模型,为基于病原菌细胞膜组成进行特异性抗菌肽的设计提供了思路,但该模型没有体现多肽因素的影响。多肽活性是肽、膜相互作用的结果,因此构建兼顾多肽因素和靶细胞膜因素的QSAR模型更能反映出肽、膜相互作用的真实情况,也是今后抗菌肽QSAR研究的发展方向。随着计算技术的发展,以及相互作用机制的深入研究,抗菌肽QSAR将从2D-QSAR、3D-QSAR研究逐渐向可模拟全部作用分子的4D-QSAR发展,甚至可模拟相互作用过程的5D-QSAR发展,从而对多肽药物的开发发挥积极作用。
[1] BAHAR A A, REN D. Antimicrobial peptides[J]. Pharmaceuticals,2013, 6(12): 1543-1575.
[2] RIEDL S, ZWEYTICK D, LOHNER K. Membrane-active host defense peptides: challenges and perspectives for the development of novel anticancer drugs[J]. Chemistry and Physics of Lipids, 2011,164(8): 766-781.
[3] GASPAR D, VEIGA A S, CASTANHO M A R B. From antimicrobial to anticancer peptides. A review[J]. Frontiers in Microbiology, 2013, 4: 294.
[4] FOX J L. Antimicrobial peptides stage a comeback[J]. Nature Biotechnology, 2013, 31(5): 379-382.
[5] MENG S, XU H, WANG F. Research advances of antimicrobial peptides and applications in food industry and agriculture[J]. Current Protein and Peptide Science, 2010, 11(4): 264-273.
[6] WU Dongdong, GAO Yanfeng, QI Yuanming, et al. Peptide-based cancer therapy: opportunity and challenge[J]. Cancer Letters, 2014,351(1): 13-22.
[7] FJELL C D, HISS J A, HANCOCK R E W, et al. Designing antimicrobial peptides: form follows function[J]. Nature Reviews Drug Discovery, 2012, 11(1): 37-51.
[8] JENSSEN H. Descriptors for antimicrobial peptides[J]. Expert Opinion on Drug Discovery, 2011, 6(2): 171-184.
[9] HELLBERG S, SJÖSTRÖM M, SKAGERBERG B, et al. Peptide quantitative structure-activity relationships, a multivariate approach[J]. Journal of Medicinal Chemistry, 1987, 30(7): 1126-1135.
[10] COLLANTES E R, DUNN III W J. Amino acid side chain descriptors for quantitative structure-activity relationship studies of peptide analogs[J]. Journal of Medicinal Chemistry, 1995, 38(14): 2705-2713.
[11] 许航, 田菲菲, 吴坚. 系统比较氨基酸描述子在多肽定量构效关系中的应用[J]. 生物学杂志, 2014, 31(1): 87-93.
[12] SANDBERG M, ERIKSSON L, JONSSON J, et al. New chemical descriptors relevant for the design of biologically active peptides. A multivariate characterization of 87 amino acids[J]. Journal of Medicinal Chemistry, 1998, 41(14): 2481-2491.
[13] LEJON T, STIBERG T, STRØM M B, et al. Prediction of antibiotic activity and synthesis of new pentadecapeptides based on lactoferricins[J]. Journal of Peptide Science, 2004, 10(6): 329-335.
[14] SHU Mao, HUO Danqun, MEI Hu, et al. New descriptors of amino acids and its applications to peptide quantitative structure-activity relationship[J]. Chinese Journal of Structural Chemistry, 2008, 27(11):1375-1383.
[15] SHU Mao, JIANG Yongjun, YANG Li, et al. Application of ‘HESH' descriptors for the structure-activity relationships of antimicrobial peptides[J]. Protein and Peptide Letters, 2009, 16(2): 143-149.
[16] 舒茂, 路亚阔, 张云茹, 等. 基于序列信息的阳离子抗菌肽设计[J].计算机与应用化学, 2012, 29(8): 921-926.
[17] 周鹏, 曾晖, 李波, 等. 支持向量机分类和回归用于肽的QSAR研究[J].化学通报, 2006, 69(5): 342-346.
[18] JENSSEN H Å, FJELL C D, CHERKASOV A, et al. QSAR modeling and computer-aided design of antimicrobial peptides[J]. Journal of Peptide Science, 2008, 14(1): 110-114.
[19] TIAN F, ZHOU P, LI Z. T-scale as a novel vector of topological descriptors for amino acids and its application in QSARs of peptides[J]. Journal of Molecular Structure, 2007, 830(1): 106-115.
[20] 梅虎, 周原, 孙立力, 等. 一种新的氨基酸描述子及其在肽QSAR中的应用[J]. 物理化学学报, 2004, 20(8): 821-825.
[21] SHU Mao, YU Rui, ZHANG Yunru, et al. Predicting the activity of antimicrobial peptides with amino acid topological information[J]. Medicinal Chemistry, 2013, 9(1): 32-44.
[22] YANG Li, SHU Mao, MA Kaiwang, et al. ST-scale as a novel amino acid descriptor and its application in QSAM of peptides and analogues[J]. Amino acids, 2010, 38(3): 805-816.
[23] TONG Jianbo, LIU Shuling, ZHOU Peng, et al. A novel descriptor of amino acids and its application in peptide QSAR[J]. Journal of Theoretical Biology, 2008, 253(1): 90-97.
[24] TONG Jianbo, CHE Ting, LIU Shuling, et al. SVEEVA descriptor application to peptide QSAR[J]. Archiv der Pharmazie, 2011, 344(11):719-725.
[25] PATEL S, STOTT I P, BHAKOO M, et al. Patenting computerdesigned peptides[J]. Journal of Computer-Aided Molecular Design,1998, 12(6): 543-556.
[26] SÁNCHEZ-GÓMEZ S, JAPELJ B, JERALA R, et al. Structural features governing the activity of lactoferricin-derived peptides that act in synergy with antibiotics against Pseudomonas aeruginosa in vitro and in vivo[J]. Antimicrobial Agents and Chemotherapy, 2011, 55(1):218-228.
[27] BHONSLE J B, VENUGOPAL D, HUDDLER D P, et al. Application of 3D-QSAR for identification of descriptors defining bioactivity of antimicrobial peptides[J]. Journal of Medicinal Chemistry, 2007,50(26): 6545-6553.
[28] MACCARI G, DI LUCA M, NIFOSI R, et al. Antimicrobial peptides design by evolutionary multiobjective optimization[J]. PLoS Computational Biology, 2013, 9(9): e1003212. doi: 10.1371/journal. pcbi.1003212.
[29] CHERKASOV A, JANKOVIC B. Application of ‘inductive' QSAR descriptors for quantification of antibacterial activity of cationic polypeptides[J]. Molecules, 2004, 9(12): 1034-1052.
[30] WU Shufen, QI Wei, SU Rongxin, et al. CoMFA and CoMSIA analysis of ACE-inhibitory, antimicrobial and bitter-tasting peptides[J]. European Journal of Medicinal Chemistry, 2014, 84: 100-106.
[31] TORRENT M, ANDREU D, NOGUES V M, et al. Connecting peptide physicochemical and antimicrobial properties by a rational prediction model[J]. PLoS ONE, 2011, 6(2): e16968. doi: 10.1371/journal. pone.0016968.
[32] 苏淅娜, 管骁, 刘静. 基于不同建模方法的ACE抑制肽QSAR比较研究[J]. 分析测试学报, 2013, 32(5): 604-608.
[33] WANG Yuanqiang, DING Yuan, WEN Haixia, et al. QSAR modeling and design of cationic antimicrobial peptides based on structural properties of amino acids[J]. Combinatorial Chemistry & High Throughput Screening, 2012, 15(4): 347-353.
[34] FJELL C D, JENSSEN H, CHEUNG W A, et al. Optimization of antibacterial peptides by genetic algorithms and cheminformatics[J]. Chemical Biology & Drug Design, 2011, 77(1): 48-56.
[35] CHERKASOV A, MURATOV E N, FOURCHES D, et al. QSAR modeling: where have you been? Where are you going to?[J]. Journal of Medicinal Chemistry, 2014, 57(12): 4977-5010.
[36] MEHLA J, SOOD S K. Connecting membrane fluidity and surface charge to pore-forming antimicrobial peptides resistance by an ANN-based predictive model[J]. Applied Microbiology and Biotechnology,2013, 97(10): 4377-4384.
Progress in Quantitative Structure-Activity Relationships Research of Antimicrobial Peptides
LU Yongzhong, KANG Daole
(College of Chemical Engineering, Qingdao University of Science and Technology, Qingdao 266042, China)
Antimicrobial peptides have potential applications in many fields, such as medicine, food, agriculture,and aquiculture. Quantitative structure-activity relationships (QSAR) research will be helpful for the optimization of antimicrobial peptides. Herein, the recent progress in molecular description and modeling of antimicrobial peptides in comparison to oligo peptides is reviewed. Meanwhile, the existing problems and the future trends are also discussed.
antimicrobial peptide; quantitative structure-activity relationship; descriptor; amino acid; mathematical model
Q74
A
1002-6630(2015)15-0299-06
10.7506/spkx1002-6630-201515055
2014-09-18
卢永忠(1968—),男,副教授,博士,研究方向为生物技术。E-mail:luyz@yahoo.com
