植物乳杆菌在不同盐质量浓度下生长至对数期中期全蛋白SDS-PAGE分析
2015-10-29乌日娜王茜茜宋雪飞薛雅婷唐筱扬武俊瑞
乌日娜,徐 鑫,王茜茜,宋雪飞,薛雅婷,唐筱扬,武俊瑞,*
(1.沈阳农业大学食品学院,辽宁 沈阳 110866;2. 江南大学食品学院,食品科学与技术国家重点实验室,江苏 无锡 214122)
植物乳杆菌在不同盐质量浓度下生长至对数期中期全蛋白SDS-PAGE分析
乌日娜1,2,徐鑫1,王茜茜1,宋雪飞1,薛雅婷1,唐筱扬1,武俊瑞1,*
(1.沈阳农业大学食品学院,辽宁 沈阳110866;2. 江南大学食品学院,食品科学与技术国家重点实验室,江苏 无锡214122)
以耐盐植物乳杆菌FS5-5为研究对象,采用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳技术构建了菌株在NaCl质量浓度分别为0、3、6、9 g/100 mL的培养基中生长至对数期中期的全蛋白表达图谱。通过比较分析,选取了6 个差异蛋白质条带,并采用液相色谱-质谱/质谱联用对差异蛋白质条带进行质谱分析。结果表明:1号样品条带鉴定得到6 种蛋白;2号样品条带鉴定得到11 种蛋白;3号样品条带鉴定得到9 种蛋白;4号样品条带鉴定得到4 种蛋白;5号样品条带鉴定得到15 种蛋白;6号样品条带鉴定得到15 种蛋白。去除相同的蛋白,共有45 种蛋白得到鉴定。这些蛋白大致可以分为4 类:与蛋白质合成有关的蛋白25 种;与代谢相关的蛋白10 种;与核苷酸合成有关的蛋白8 种;未知功能蛋白2 种。可能由于这些蛋白的表达发生变化,才导致菌体中蛋白质合成、能量代谢、DNA复制能够正常进行,最终使植物乳杆菌FS5-5能够更好地在盐环境下生存下去。
植物乳杆菌FS5-5;盐胁迫;十二烷基硫酸钠-聚丙烯酰胺凝胶电泳;差异蛋白
1994年,Marc Wikins等首次提出蛋白质组这一概念,用来描述在特定时期或特定状态下,一种生物体的某一组织或某一细胞的基因组所表达的全部蛋白质[1]。研究细胞内全部蛋白质的存在及其活动方式的学科即为蛋白质组学(proteomics)[2]。随着人们认识到人类基因组计划不能为治疗疾病提供理想的切入点后,蛋白质组学技术越来越受到人们的重视。
植物乳杆菌是乳酸菌的一种,常见于奶油、肉类及许多蔬菜发酵制品中。最适生长温度为30~35 ℃,兼性厌氧,最适pH 6.5左右。菌体呈短杆状,有时成对或成链状,不产芽孢[3]。在食品工业生产中,常常会利用植物乳杆菌作为发酵剂来生产发酵型食品。在发酵型食品的生产过程中,经常会遇到外界盐质量浓度较高的环境。在这种环境中,菌株为了生长繁殖会激发自身的应激反应机制来应对来自外界盐环境的胁迫[4]。对植物乳杆菌盐胁迫条件下蛋白质表达情况进行分析,有助于人们了解其盐胁迫条件下蛋白质的表达差异,进而为阐明植物乳杆菌盐胁迫应激机制提供理论基础。
目前,研究者的研究目标主要集中在乳酸菌的酸胁迫、碱胁迫、胆盐胁迫过程中蛋白质的表达变化,而关于乳酸菌在盐胁迫条件下蛋白质变化研究甚少。研究表明,乳杆菌在不同外界环境条件下,蛋白质的表达有所不同。Wu Rina等[5]研究了益生菌Lactobacillus casei Zhang在酸胁迫下的蛋白质组,发现在pH 2.5和pH 6.4时,有33 个蛋白位点发生改变。Johanna等[6]研究了酸胁迫对Lactobacillus rhamnosus GG蛋白质表达和磷酸化的影响,结果表明参与中枢细胞通路的蛋白质被磷酸化,并且其中糖酵解酶被磷酸化最为广泛。Lee等[7]研究了Lactobacillus johnsonii PF01在胆盐胁迫下蛋白质表达情况,发现大约215 个蛋白质发生改变,这其中有94 种蛋白质含量降低,121 种蛋白质含量增加。Belfiore等[8]对Lactobacillus sakei CRL1756在NaCl环境下的基础适应性进行研究发现,在盐环境下菌体自身会生成应激蛋白Hsp20、ClpB、Chaperone GrpE、ClpL ATPase protein、Chaperone DnaK,同时蛋白质Fba、Pgk、Gpm5、Tpi的表达量降低。Zhao Shanshan等[9]研究结果表明Glycine betaine(GB)可以调节碳水化合物的运输和代谢、保护核糖体的结构,对Lactobacillus plantarum ST-III应对盐胁迫起到了非常重要的作用。当用NaCl作为外界胁迫因素时,细胞外的渗透压会增大,Li Chun等[10]发现GB具有调节细胞内外的渗透压的作用。Leontiev等[11]发现在盐胁迫条件下,菌体Transport ATPase的表达量会增加,可以逆向运输将进入细胞内部的Na+运输到细胞外,从而使乳酸菌能够更好地生存下去。
本实验以耐盐植物乳杆菌FS5-5为研究对象,采用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gelelectrophoresis,SDS-PAGE)技术构建该菌株在NaCl质量浓度分别为0、3、6、9 g/100 mL的MRS培养基中生长至对数期中期的蛋白质图谱,并对图谱进行比较分析,找出差异蛋白,以期为揭示植物乳杆菌FS5-5耐盐分子机制提供理论依据。
1 材料与方法
1.1菌株、培养基与试剂
菌种:3 株耐盐性较好的植物乳杆菌,由沈阳农业大学食品微生物实验室提供。
MRS培养基:葡萄糖20.0 g/L、MnSO4·4H2O 0.25 g/L、牛肉浸膏8 g/L、MgSO4·7H2O 0.58 g/L、蛋白胨8 g/L、吐温-80 1 g/L、酵母粉4 g/L、柠檬酸钠2 g/L、三水乙酸钠5 g/L、K2HPO42 g/L,121 ℃高压灭菌20 min。
丙烯酸胺、Tris、SDS、四甲基乙二胺(N,N,N',N'-tetramethylethylenediamine,TEMED)、N,N-甲叉双丙烯酞胺、三氯乙酸美国Sigma公司;尿素、硫脲、二硫苏糖醇(dithiothreitol,DTT)、3-((3-胆固醇氨丙基)二甲基氨基)-1-丙磺酸(3-((3-cholamidopropyl)dimethylammonio) propanesulfonate,CHAPS) 美国Bio-Rad公司;过硫酸铵、苯甲基磺酰氟(phenylmethanesulfonyl fluoride,PMSF)、3-(N-吗啉)丙磺酸 (3-(N-morpholino) propanesulfonic acid,MOPS)、2-(N-吗啉)乙磺酸(2-(N-morpholino)ethanesulfonic acid,MES)北京鼎国生物技术有限公司。
1.2仪器与设备
LDZX-50KBS型立式压力蒸汽灭菌锅上海申安医疗器械厂;NP-9080型生化培养箱、恒温水浴锅上海精宏试验仪器有限公司;VS-1型涡旋仪上海泸西分析仪器厂有限公司;DW-86L486型超低温保存箱青岛海尔特种电器有限公司;Bio-Rad Mini、PROTEAN II XL Cell垂直电泳系统、Image Lab图像分析系统、电泳仪美国Bio-Rad公司;低温冷冻离心机德国Eppendorf公司。
1.3方法
1.3.1耐盐植物乳杆菌的培养
将采用真空冷冻干燥法冻干保存的3 株植物乳杆菌菌种[12],接种于MRS液体培养基中,37 ℃培养24 h,活化3 代。经革兰氏染色镜检确定为纯菌后,取第3代MRS液体培养物,12 000×g离心10 min,弃掉上清液,在沉淀中加入5 mL灭菌生理盐水,12 000×g、10 min、4 ℃离心洗涤菌体,重复离心洗涤菌体2 次。最后加入5 mL灭菌生理盐水,用涡旋仪振荡混匀,即为供试菌液。
1.3.2耐盐性菌株挑选
吸取混匀后的3 代菌液100 .L分别加入5 mL NaCl质量浓度分别为4、6、8、10、14、16、20 g/100 mL的MRS液体培养基中,37 ℃培养24 h。培养液用涡旋仪混匀后,以空白MRS液体培养基调零,在600 nm波长处测OD值,挑选出一株耐盐性最好的菌株。
1.3.3耐盐菌株生长曲线的建立
对3 株植物乳杆菌中耐盐性较好的菌株FS5-5,参照GB 4789.2—2010《食品安全国家标准 食品微生物学检验 菌落总数测定》,在菌株生长过程中采用倾注法进行菌落计数。构建菌株在NaCl质量浓度分别为0、3、6、9 g/100 mL的MRS培养基中的生长曲线。
1.4蛋白质样品的提取与制备
1.4.1菌体的培养与收集
参照Lorena等[13]收集菌株在含有NaCl质量浓度分别为0、3、6、9 g/100 mL的MRS培养基中生长至对数期中期的菌体。为保证培养基pH值的稳定,向其中添加MOPS和MES,以减小pH值对菌体中蛋白的影响。
1.4.2植物乳杆菌全蛋白的提取
将收集到的菌体,利用研钵采用液氮研磨法对菌体进行研磨破碎。将研好的菌体三氯乙酸(trichloroacetic acid,TCA)/丙酮法制取全蛋白样品[14]。
1.4.3蛋白质的裂解
分别取一定量的菌体蛋白质干粉,加入400 .L的裂解液,进行超声助溶10 min。之后放入冰盒中摇床振荡2 h,然后4 ℃、20 000×g离心1 h,弃沉淀,在上清中加入DNase I (1 U/50 .L)和RNase A(25 .g/.L),于4 ℃冰箱中消化过夜以去除其中的核酸。最后将蛋白质裂解液分装,于-20 ℃短期保存[15]。
1.4.4Bradford微量蛋白质测定法
配制10 mg/mL牛血清白蛋白(bovine albumin,BSA)储存液,稀释至1 mg/mL母液。按照Bradford微量蛋白质测定法制作标准曲线,测定蛋白质的质量浓度[16]。
1.4.5SDS-PAGE方法鉴定蛋白
利用SDS-PAGE方法对在不同盐质量浓度下生长的植物乳杆菌蛋白质样品进行电泳实验。灌制分离胶12%,浓缩胶5%[17]。梯度胶为预制的4%~20%的梯度胶。电泳条件:80 V,10 min;120 V,2 h。
1.4.6染色与脱色
电泳完毕,取下凝胶用去离子水清洗凝胶表面残留的电泳液等。然后用提前加热至95 ℃的考马斯亮蓝染色液,将凝胶染色20 min。染色完毕,用去离子水清洗凝胶表面,去除多余的染色液,最后用脱色液将凝胶脱色。直至出现清晰的条带为止,期间可以更换几次脱色液。
1.4.7图像分析
利用Image Lab软件对所得的SDS-PAGE图谱进行分析,找出差异条带。
1.4.8蛋白质质谱鉴定
从胶上选取差异蛋白条带,切下后放入干净的1.5 mL离心管中,脱色后经胰蛋白酶消化,并利用AB Sciex 4000 QTRAP.液相色谱-质谱/质谱(liquid chromatograph-mass spectrometer/mass spectrometer,LC-MS/MS)对肽段样品进行质谱分析。
1.4.9生物信息学分析
通过软件Mascot 2.3.01(Matrix Science),以NCBInr数据库中革兰氏阳性菌已知数据为基础,进行蛋白质序列数据库检索,获取蛋白质信息。查询条件设定为:物种来源:乳杆菌属;表观等电点(isoelectric point,PI)与表观相对分子质量(relative molecular mass,Mr)的误差范围设定为:无限制;一级质谱精度(peptide mass tolerance):±50×10-6,二级质谱精度(fragment mass tolerance):±0.25 u;固定修饰:半胱氨酸碘乙酰胺化(carbamidomethyl);可变修饰:蛋氨酸氧化(methionine oxidation)[18]。当检索结果蛋白分数超过19时,可靠性达到显著水平(P<0.05)。
2 结果与分析
2.1耐盐植物乳杆菌的筛选
对已鉴定的3 株耐盐性较好的乳酸菌进行更细致的分离,测定其在不同盐质量浓度下生长24 h后的OD600nm值,确定一株耐盐性较好的植物乳杆菌,结果见表1。

表1 3 株植物乳杆菌在不同盐质量浓度下的生长情况(OODD60000 nnmm)Table 1 Growth status of 3Lactobacillus plantaarruumm strains undeerr different NaCl concentrations (OODD60000 nnmm)
由表1可知,当NaCl质量浓度达到14 g/100 mL时,3 株植物乳杆菌基本上不再生长,所以最高的生长质量浓度在12 g/100 mL左右。当NaCl质量浓度为12 g/100 mL时,DL3-1的OD600nm值为0.103;DL4-5的OD600nm值为0.097;FS5-5的OD600nm值为0.137。所以确定3 株植物乳杆菌中,FS5-5的耐盐性最好。
2.2FS5-5在不同盐质量浓度下的生长曲线

图1 植物乳杆菌FS5-5在不同盐质量浓度下的生长曲线Fig.1 Growth curves of Lactobacillus plantarum FS5-5 in different salt concentrations
对菌株FS5-5采用菌落计数的方法建立其生长曲线。将活化3 代的植物乳杆菌FS5-5接种到不同盐质量浓度的MRS培养基中。在菌株的生长过程中,每间隔2 h,参照国标GB 4789.2—2010采用倾注法测定其菌落数,每个质量浓度做两个平行。根据测得结果,以培养时间为横坐标,菌落数为纵坐标,构建菌株FS5-5在NaCl质量浓度为0、3、6、9 g/100 mL的MRS培养基中的生长曲线,结果见图1。在0 g/100 mL NaCl的MRS培养基中,植物乳杆菌FS5-5的对数期中期为生长6 h。在3 g/100 mL NaCl的MRS培养基中,植物乳杆菌FS5-5的对数期中期为生长6 h。在6 g/100 mL NaCl的MRS培养基中,植物乳杆菌FS5-5的对数期中期为生长10 h。在9 g/100 mL NaCl的MRS培养基中,植物乳杆菌FS5-5的对数期中期为生长21 h。
2.3样品中蛋白质含量的测定结果
采用最基本的方法——Bradford法测定蛋白质的含量。首先制作标准曲线,根据BSA标准溶液质量浓度为横坐标(x,mg/mL),595 nm波长处的OD值为纵坐标(y),可得到回归方程:y=6.898 2x+0.037 3(R2=0.999 5),表明在该蛋白质量浓度范围内线性关系良好。根据标准曲线测得样品在0、3、6、9 g/100 mL NaCl条件下的蛋白质含量分别为13.51、15.67、7.36、16.95 mg/mL。提取的蛋白质的含量均大于4 mg/mL,能够满足SDS-PAGE的需要。
2.4FS5-5不同盐质量浓度下蛋白质表达的SDS-PAGE分析
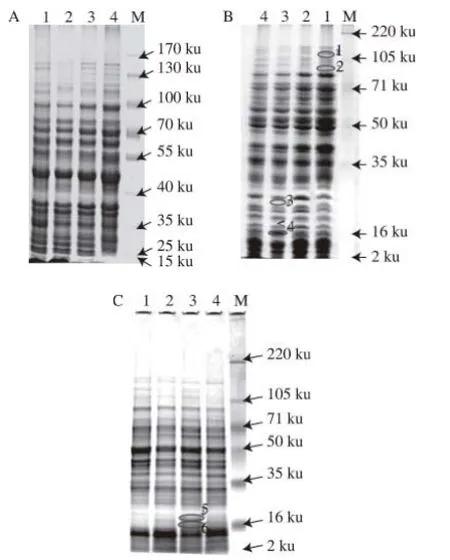
图2 不同盐质量浓度下蛋白质表达的SDS-PAGEE图谱Fig.2 SDS-PAGE electrophoresis of protein expression profiles under different salt concentrations
由图2A可知,由于蛋白条带较多,分离效果不明显,很多条带没有分离开,尤其是15~40 ku之间。由图2B可知,由于Bio-Rad 18.5 cm×20 cm胶较长,蛋白分离效果要明显好于Bio-Rad Mini 聚丙烯酰胺凝胶,条带也相对清晰。由图2C可知,4%~20%梯度胶对蛋白质的分离较为清晰,在6 g/100 mL对数期条带中,在16 ku处有2 个条带表达明显。但是由于梯度胶较短而样品中蛋白质种类较多,导致有很多蛋白紧密相连,不易区分开来。同时可以发现,植物乳杆菌FS5-5在不同盐质量浓度环境下,蛋白质表达差异很小。
2.5图像分析结果
由于Bio-Rad 18.5 cm×20 cm分离效果相对较好,所以利用Image Lab软件对其所得的SDS-PAGE图谱进行分析,结果见图3。

图3 不同SDS-PAGE泳道的图谱分析Fig.3 Analysis of different SDS-PAGE lanes
对图3A~3D的图谱结果进行分析整理合并,可以得到不同泳道蛋白质条带对应分子质量,结果如表2所示。

表2 不同泳道蛋白质条带对应分子质量Table 2 Molecular weights of different protein bands
通过对Image Lab软件自动分析的结果(图3和表2)可以看出,植物乳杆菌在3 g/100 mL NaCl条件下,全蛋白的表达与0 g/100 mL NaCl条件下相比基本上没有差异;在6 g/100 mL NaCl条件下,1号泳道中的4号和7号条带是缺失的,3号泳道中增加了一条分子质量大约为14.3 ku的条带,同时在分子质量大约为6.6 ku时蛋白表达量明显增加;在9 g/100 mL NaCl条件下,4号泳道中的4号和7号条带也是缺失的,增加了一条分子质量大约为14.3 ku的条带。所以将1号泳道中的4号蛋白条带定义为1号样品,6号条带定义为2号样品,3号泳道中的24号条带定义为3号样品,31号条带定义为4号样品,对梯度胶中两条差异比较明显的条带定义为5号、6号样品。
2.6蛋白质质谱鉴定及生物信息学检索
将1、2、3、4、5、6号样品条带切下后放入干净的1.5 mL离心管中,脱色后经胰蛋白酶消化,并利用LC-MS/MS对肽段样品进行分析。通过软件Mascot 2.3.01(Matrix Science),以NCBInr数据库中革兰氏阳性菌已知数据为基础,进行蛋白质序列数据库检索,获取蛋白质信息。

表3 与蛋白质合成有关的蛋白Table 3 Proteins associated with protein synthesis

表4 与代谢相关的蛋白Table 4 Proteins associated with metabolism

表5 与DNA合成有关的蛋白Table 5 Proteins associated with DNA synthesis
在检测的6 个条带中,差异蛋白条带均为蛋白混合物。1号样品条带鉴定得到6 种蛋白;2号样品条带鉴定得到11 种蛋白;3号样品条带鉴定得到9 种蛋白;4号样品条带鉴定得到4 种蛋白;5号样品条带鉴定得到15 种蛋白;6号样品条带鉴定得到15 种蛋白。去除相同的蛋白,共有45 种蛋白得到鉴定。这些蛋白大致可以分为4 类:与蛋白质合成有关的蛋白25 种(表3);与代谢相关的蛋白10 种(表4);与核苷酸合成有关的蛋白8 种(表5);未知功能蛋白2 种,分别为xaa蛋白的二肽基肽(Xaa-Pro dipeptidylpeptidase)和非规范嘌呤NTP焦磷酸酶(non-canonical purine NTP pyrophosphatase),如表6所示。

表6 未知功能蛋白Table 6 Proteins with unknown functions
3 讨 论
盐胁迫反应是一个相当复杂的生理过程,当植物乳杆菌FS5-5在一定盐质量浓度的外界环境中生长时,为了抵御外界盐质量浓度环境的影响,其本身蛋白质的表达会发生一系列变化,从而使自己能够更好地在盐质量浓度环境下生长[19]。对于本实验中的鉴定结果发现,变化最多的蛋白大致可以分为三大类:与对蛋白质合成有关的蛋白;与代谢有关的蛋白;与核苷酸合成有关的蛋白。
3.1 与蛋白质合成有关的蛋白
蛋白质的生物合成过程可以分为起始、延伸和终止3 个阶段,每一阶段都会有各自相关的蛋白质因子发挥作用[20-21]。通过对植物乳杆菌FS5-5的SDS-PAGE差异条带的鉴定得到翻译起始因子2、翻译起始因子3、延伸因子Tu、延伸因子4、延伸因子G、核糖体成熟因子RimM。由此可见,当菌株应对外界压力时,在蛋白质的合成过程中从起始因子到最后的成熟因子都发生了一系列的变化,从而导致蛋白质的表达发生差异。同时,表达差异较多的还有核糖体蛋白,包括30S核糖体蛋白、50S核糖体蛋白等。
3.2 与代谢有关的蛋白
从目前报道的相关结果来看,糖代谢相关蛋白的变化,对乳酸菌应对外界应激具有极其重要的作用。糖代谢反应主要为糖酵解途径,是指细胞通过消耗葡萄糖生成丙酮酸同时释放出一定能量的过程。当乳酸菌面临外界不利环境胁迫时,乳酸菌消耗的能量就会增加,从而会使糖酵解途径中相关的酶发生变化[22]。3-磷酸甘油醛脱氢酶(glyceraldehyde-3-phosphate dehydrogenase)是糖酵解途径中的关键酶。在细胞的糖代谢过程中,3-磷酸甘油醛脱氢酶催化3-磷酸甘油醛脱氢、加磷酸,其辅酶为烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide,NAD+),反应脱下的氢交给NAD+成为NADH+H+;反应时释放的能量储存在所生成的1,3-二磷酸甘油酸1位的羧酸与磷酸构成的混合酸酐内,此高能磷酸基团可将能量转移给ADP形成ATP,从而使能量增加[23],有利于菌株在盐胁迫条件下更好地存活下来。
3.3与核苷酸合成有关的蛋白
DNA的正确复制是保证乳酸菌存活、增殖的必备条件,也是蛋白质氨基酸序列合成的依据。本实验中鉴定得到了8 种与核苷酸相关的蛋白,当乳酸菌受到外界胁迫时,基因序列的复制会受到影响,此时一些相关的与DNA相关的酶发生作用,以确保DNA的正确复制。本实验中鉴定得到了DNA错配修复蛋白Muts。DNA错配修复蛋白是通过修复由于DNA复制时的错误而使基因组保持稳定的一种蛋白。在盐胁迫条件下鉴定出该种蛋白,说明外界环境已经影响到了该菌株DNA的正常复制[24-25]。所以与核苷酸合成相关的蛋白有效地保护了乳酸菌的DNA的正确复制,从而使得该菌株能够耐受盐性环境的胁迫而存活、增殖。
4 结 论
本实验以植物乳杆菌FS5-5为研究对象,采用SDS-PAGE技术构建该菌株在NaCl质量浓度为0、3、6、9 g/100 mL的MRS培养基中,生长至对数期中期蛋白质一维图谱,并对图谱进行比较分析,找出差异蛋白条带,并利用LC-MS/MS质谱对肽段样品进行质谱分析。
共鉴定了6 个蛋白质条带,均为蛋白混合物。1号样品条带鉴定得到6 种蛋白;2号样品条带鉴定得到11 种蛋白;3号样品条带鉴定得到9 种蛋白;4号样品条带鉴定得到4 种蛋白;5号样品条带鉴定得到15 种蛋白;6号样品条带鉴定得到15 种蛋白。去除相同的蛋白,共有45 种蛋白得到鉴定。这些蛋白大致可以分为4 类:与蛋白质合成有关的蛋白25 种;与代谢相关的蛋白10 种;与核苷酸合成有关的蛋白8 种;未知功能蛋白2 种。也许正是由于这些蛋白的表达发生变化,才导致菌体中蛋白质的合成、能量代谢、DNA的复制能够正常的进行,最终使植物乳杆菌FS5-5能够更好地在盐环境下生存。
[1]BOLOTIN A, WINCKER P, MAUGER S, et al. The complete genome sequence of the lactic acid bacterium Lactococcus lactis ssp. lactis IL1403[J]. Genome Research, 2001, 11(5): 731-739.
[2]乌日娜, 孙志宏, 张文羿, 等. 蛋白质组学在乳酸菌应激反应机制研究中的应用[J]. 食品科学, 2013, 34(1): 324-327.
[3]da SILVA SABO S, VITOLO M, JOS. MANUEL DOMÍNGUEZ GONZ.LEZ J M D, et al. Overview of Lactobacillus plantarumas a promising bacteriocin producer among lactic acid bacteria[J]. Food Research International, 2014, 64(10): 527-536.
[4]VERONICA F, ANDREA B, JORGE R, et al. Resistance of functional Lactobacillus plantarum strains against food stress conditions[J]. Food Microbiology, 2015, 48(6): 63-71.
[5]WU Rina, WU Junrui, YUE Xiqing, et al. Proteomic analysis of responses of a new probiotic bacterium Lactobacillus casei-Zhang to low acid stress[J]. International Journal of Food Microbiology, 2011,147(3): 181-187.
[6]JOHANNA K, KATI L, KERTTU K, et al. Effect of acid stress on protein expression and phosphorylation in Lactobacillus rhamnosus GG[J]. Journal of Proteomics, 2012, 75(4): 1357-1374.
[7]LEE J Y, PAJARILLO E A B, KIM M J. Proteomic and transcriptional analysis of Lactobacillus johnsonii PF01 during bile salt exposure by iTRAQ shotgun proteomics and quantitative RT-PCR[J]. Journal of Proteome Research, 2013, 12(1): 432-443.
[8]BELFIORE C, FADDA S, RAYA R, et al. Molecular basis of the adaption of the anchovy isolate Lactobacillus sakei CRL1756 to salted environments through a proteomic approach[J]. Food Research International, 2013, 54(1): 1334-1341.
[9]ZHAO Shanshan, ZHANG Qiuxiang, HAO Guangfei, et al. The protective role of glycine betaine in Lactobacillus plantarum ST-III against salt stress[J]. Food Control, 2014, 44: 208-213.
[10] LI Chun, LIU Libo, SUN Di, et al. Response of osmotic adjustment of Lactobacillus bulgaricus to NaCl stress[J]. Journal of Northeast Agricultural University, 2012, 9(4): 66-74.
[11] LEONTIEV L, SELIVERSTOV A, LYUBETSKY V. Algorithm for a large-scale search for T-Box transcription regulation in bacteria[J]. Molecular Microbiology, 2005, 39(6): 946-948.
[12] 徐鑫, 王茜茜, 乌日娜, 等. 传统农家大酱中耐盐性乳酸菌的分离与鉴定[J]. 食品与发酵工业, 2014, 40(11): 33-40.
[13] LORENA R, YOHANN C, BORJA S, et al. The cell-envelope proteome of Bifi dobacterium longum in an in vitro bile environment[J]. Microbiology, 2009, 155(3): 957-967.
[14] 乌日娜. 益生菌Lactobacillus casei Zhang蛋白质组学研究[D]. 呼和浩特: 内蒙古农业大学, 2009.
[15] 何庆华, 吴永宁, 印遇龙. 乳酸菌差异蛋白组学研究进展[J]. 食品与发酵工业, 2007, 33(8): 113-116.
[16] 王延华, 形如新, 游潮. 蛋白质理论与技术[M]. 2版. 北京: 科学出版社, 2009: 225-230.
[17] WU Rina, XU Xin, MENG Lingshuai, et al. Identification of salt stress responsive protein in Lactobacillus parac asei LN-1 using SDSPAGE[J]. IERI Procedia, 2014, 8(1): 60-65.
[18]黄桂东. Lactobacillus brevis NCL912的耐酸特性及其酸胁迫下差异表达蛋白的研究[D]. 南昌: 南昌大学, 2011.
[19] 周方方, 吴正钧, 艾连中, 等. 蛋白组学技术在乳酸菌环境胁迫应激研究中的应用[J]. 食品与发酵工业, 2012, 38(8): 101-106.
[20] de LEEUW F, ZHANG T, WAUQUIER C, et al. The cold-inducible RNA-binding protein migrates from the nucleus to cytoplasmic stress granules by a methylation-dependent mechanism and acts as a translational repressor[J]. Experimental Cell Research, 2007, 313(20):4130-4140.
[21] MITHU M, DANIEL M, SERGEI M, et al. Residues required for phosphorylation of translation initiation factor IF2 under diverse stress conditions are divergent between yeast and human[J]. The International Journal of Biochemistry & Cell Biology, 2015, 59(2):135-141.
[22] KISTRUP M, JACOBSEN S, HAMMER K, et al. Induction of heat shock proteins DnaK, GroEL and GroES by salt stress in Lactococcus lactis[J]. Applied and Environmental Microbiology, 1997, 63(5): 1826-1837.
[23] MICHAEL A, SIROVE R. Structural analysis of glyceraldehyde-3-phosphate dehydrogenase functional diversity[J]. The International Journal of Biochemistry & Cell Biology, 2014, 57(1): 20-26.
[24] BI Lijun, ZHANG Xianen, ZHOU Yafeng. Observation of the interaction between MutS and MutL mismatch repair proteins by fusion protein syste ms[J]. Progress in Biochemistry and Biophysics,2005, 32(12): 1178-1183.
[25] 钟天映, 毕利军, 张先恩. 错配修复蛋白MutS的新功能位点[J]. 中国科学: 生命科学, 2011, 41(2): 168-172.
SDS-PAGE Analysis of Total Proteins in Lactobacillus plantarum in the MiddleLogarithmic Growth at Different Salt Concentrations
WU Rina1,2, XU Xin1, WANG Qianqian1, SONG Xuefei1, XUE Yating1, TANG Xiaoyang1, WU Junrui1,*
(1. College of Food Science, Shenyang Agricultural University, Shenyang110866, China; 2. State Key Laboratory of Food Science and Technology, College of Food Science, Jiangnan University, Wuxi214122, China)
In this study, the total protein profiles of Lactobacillus plantarum FS5-5 with salt tolerance grown in media with NaCl concentrations of 0, 3, 6, and 9 g/100 mL to the middle logarithmic phase were established and compared by sodium dodecyl sulfate-polyacrylamide gel electrophoresis (SDS-PAGE), and six different protein bands were selected for analysis by liquid chromatography-tandem mass spectrometry (AB Sciex 4000 QTRAP.LC/MS/MS). Results showed that 6, 11, 9, 4, 15 and 15 proteins were contained in bands 1, 2, 3, 4, 5 and 6, respectively. Totally 45 proteins were identified. These proteins could be divided into 4 groups including 25 proteins involved in protein synthesis, 10 proteins associated with metabolism, 8 proteins associated with nucleotide synthesis, and 2 proteins with unknown functions. The differential expression of these proteins could lead to bacterial protein synthesis, energy metabolism and DNA replication, and eventually enable Lactobacillus plantarum FS5-5 to better survive in salt stress environment.
Lactobacillus plantarum FS5-5; salt stress; sodium dodecyl sulfate-polyacrylamide gel electrophoresis(SDS-PAGE); different proteins
Q939.117
A
1002-6630(2015)23-0155-07
10.7506/spkx1002-6630-201523029
2015-01-29
国家自然科学基金面上项目(31000805;31471713);辽宁省农业领域青年科技创新人才培养资助计划项目(2014048);辽宁省高等学校优秀人才支持计划项目(LR2015059);江苏省博士后科研资助计划项目(1402071C);沈阳农业大学“天柱山英才支持计划”项目
乌日娜(1979—),女,副教授,博士,研究方向为生物技术。E-mail:wrn6956@163.com
武俊瑞(1977—),男,副教授,博士,研究方向为生物技术。E-mail:junruiwu@126.com
