实时荧光聚合酶链式反应法检测食品中狗源性成分
2015-10-21高晓丽杨昕霆薛晨玉赵琳娜侯彩云中国农业大学食品科学与营养工程学院北京00083北京市食品安全监控中心北京0004
高晓丽,杨昕霆,薛晨玉,王 丹,赵琳娜,侯彩云,*(.中国农业大学食品科学与营养工程学院,北京00083;.北京市食品安全监控中心,北京0004)
实时荧光聚合酶链式反应法检测食品中狗源性成分
高晓丽1,杨昕霆2,+,薛晨玉2,王丹2,赵琳娜2,侯彩云1,*
(1.中国农业大学食品科学与营养工程学院,北京100083;2.北京市食品安全监控中心,北京100041)
根据狗线粒体NADH氧化还原酶亚基2基因(ND2)中的序列设计了狗特异性引物和Taqman探针,建立了食品中狗源性成分的实时荧光聚合酶链式(Real-time PCR)检测方法。实验表明,该方法可以很好地区分狗与其他常见的14个物种,引物、Taqman探针特异性良好,检测灵敏度可达100fg,适用于食品中狗源性成分的快速鉴定。
Real-time PCR,线粒体ND2基因,狗源性成分
近年来,食品中肉制品的安全问题日益突出,如国内的“假牛、羊肉事件”、“假鸭血事件”,国外的“马肉风波”、“假鳕鱼事件”。食品中的掺杂作假现象,不仅严重侵害消费者的权益,引发对食品安全的信任危机,一些疫情的发生与传播也往往起因于不安全的动物食品[1-2]。我国针对饲料、明胶、化妆品中含有的猪、牛、羊、马、驴、兔、狗等动物源性成分制定了相关标准[3],但关于食品中动物源性成分的检测标准并不完善。因此,建立准确、快速的食品中动物源性成分筛查方法至关重要。
国内外学者主要基于PCR方法对动物源性成分的鉴定进行了研究,大量报道了猪、牛、羊、鸡、火鸡、鸭、鹅、马、驴等常见物种的普通PCR[4-10]和荧光PCR[11-18]研究,但针对狗源性成分鉴定的研究报道还不多。Tobe等[19]基于线粒体cytb基因建立了18个欧洲常见物种(狗、猫、牛、马、驴、狐狸、山羊等)的多重PCR检测方法,能够鉴别混合肉中各物种340fg的模板DNA。Martin等[20]建立了食品和饲料中狗、猫、鼠源性成分鉴定的普通PCR方法,检测限为0.1%。我国国标中只制定了动物源性饲料中狗源性成分的普通PCR定性检测方法,检出限为0.1%[21]。
在食品中动物源性成分的鉴定方面,与普通PCR和多重PCR法相比,实时荧光PCR法具有特异性强、灵敏度高、重现性好、有效降低污染、可实时监测等优点,是目前应用较普遍的方法之一[22]。本研究设计了狗线粒体ND2基因特异性引物和Taqman探针,建立了食品中狗源性成分的实时荧光PCR检测方法。该方法可为保障食品安全、制定相应标准等提供参考。
1 材料与方法
1.1材料与仪器
猪肉、牛肉、山羊肉、绵羊肉、鸡肉、鸭肉、鹅肉、鸽子肉、鹌鹑肉、兔肉等动物材料购自北京市农贸市场或超市;骆驼肉、马肉、驴肉、狗肉、猫肉等动物材料为实验室自存;20个牛羊肉串样品购自北京市农贸市场;Tris-平衡酚、氯仿、无水乙醇、异丙醇、乙酸钠均为国产分析纯;CTAB提取缓冲液(20g/L CTAB,1.4mol/L NaCl,0.02mol/L Na2EDTA,0.1mol/L Tris-HCl,pH=8.0)、TE缓冲溶液(10mmol/L Tris-HCl,pH=8.0,1mmol/L EDTA)由本实验室配制;Taqman实时荧光PCR预混合液(Premix Ex Taq)购自宝生物工程(大连)有限公司;狗ND2基因序列特异的上下游引物、Taqman探针由北京赛百盛基因技术有限公司合成。
核酸蛋白定量分析仪(分光光度计)德国Eppendorf公司;核酸自动提取仪QIA Cube美国QIA Gene公司;3K18冷冻离心机德国Sigma公司;MyiQ单色实时定量PCR仪美国伯乐公司;超低温冰箱(-40℃)Biomedical freezer日本SANYO公司;Mili-Q纯水器美国Milipore公司。
1.2动物组织基因组DNA的提取[23]
用灭菌手术剪分别剪取适量的猪肉、牛肉、山羊肉、绵羊肉、鸡肉、鸭肉、鹅肉、鸽子肉、鹌鹑肉、兔肉、骆驼肉、马肉、驴肉、狗肉、猫肉的肌肉组织,剪碎置于研钵中(注意防止交叉污染);加入石英砂,用液氮充分研磨至粉末状,迅速取2g左右于2.0mL离心管中,立即加入1mL CTAB,混匀;置于核酸自动提取仪中加热振荡,消化30min;离心取600μL上清液于新的2.0mL离心管中,加入等体积的氯仿和Tris-平衡酚各300μL(1∶1,V/V),用力振荡5min,使其充分反应,12000r/min室温离心10min;取上清,加入600μL氯仿,同上,12000r/min室温离心10min;取上清,加10%体积3mol/L乙酸钠和2倍体积无水乙醇,轻缓颠倒数次,可见白色絮状DNA,12000r/min室温离心10min,弃上清液;加入1mL 70%乙醇洗涤沉淀2次,12000r/min室温离心10min,弃乙醇;待DNA沉淀干燥后,将DNA溶于50~100μL无菌水中,用核酸蛋白定量分析仪测定DNA浓度,置于-20℃保存备用。
1.3引物与Taqman探针的设计
根据NCBI基因数据库已注释的、不同品系的狗(Canis lupus familiaris)线粒体序列(Accession Number:NC_002008,GI:17737322)和猪、牛、山羊、绵羊、鸡、鸭、鹅、鸽子、鹌鹑、兔、骆驼、马、驴、猫等物种的线粒体序列,分析生物进化树的规律,使用DNAMAN软件比对以上所有基因序列。选择种内保守、种间差异较大的基因片段,根据引物和Taqman探针的设计原则,应用Primer Express软件,设计引物和Taqman探针,将设计的引物和探针序列在NCBI的Blast中搜索、比对,最后选取既可以保证种内保守,又能保证种间特异的狗源性成分引物和Taqman探针。
1.4引物和Taqman探针特异性的验证
以狗肉基因组DNA为阳性对照,猪、牛、山羊、绵羊、鸭、鸡等14个物种基因组DNA为阴性对照,空白对照中以水代替DNA模板。所有DNA模板的质量浓度均稀释至100ng/μL。实时荧光PCR反应体系为20μL,具体包括:Premix buffer(2×)10μL、上游引物(10μmol/L)0.5μL、下游引物(10μmol/L)0.5μL、Taqman探针(10μmol/L)1μL、DNA模板1μL、无菌水将体系补充至总体积20μL。实时荧光PCR扩增条件为:95℃预变性30s;95℃变性5s;60℃延伸30s,记录荧光值,40个循环。
在实时荧光PCR中,Ct值是指每个反应管内的荧光信号达到设定的阈值时所经历的循环数。实时荧光PCR反应的前15个循环的荧光信号作为荧光本底信号,荧光域值的缺损设置是3~15个循环的荧光信号的标准偏差的10倍,即threshold=10XSD cycle 6~15。因此,以Ct值小于35为阳性,Ct值大于35为阴性。特异性验证实验重复三遍。
1.5灵敏度和扩增效率检测
在检测该方法的灵敏度实验中,将狗肉基因组DNA溶液分别稀释为102、101、100、10-1、10-2、10-3、10-4、10-5ng/μL,每个浓度进行3次重复实验,设置空白对照。根据各梯度的扩增曲线Ct值,建立Ct值-lg[DNA]的标准曲线。依据扩增效率计算公式Efficiency(%)=(-1+10[-1/slope])×100,得到PCR反应扩增效率。以Ct值为判断标准,确定检出的最低浓度,作为该反应体系的检测灵敏度。
1.6市售肉制品中狗源性成分的检测
在北京农贸市场中随机取样20个牛、羊肉串,检验是否有掺杂掺假狗肉的情况。提取样本基因组DNA,按照实时荧光反应体系,设置阳性对照、阴性对照和空白对照,进行实时荧光PCR反应。
2 结果与分析
2.1阳性物种样本的鉴定
以CO1基因的DNA条形码[24-25]扩增狗肉基因组DNA,并对扩增产物进行测序。将测序结果在NCBI中Blast搜索,结果如图1所示。序列相似性为99%,从而确定狗物种样本的真实性。
2.2引物和Taqman探针的特异性检测结果
在特异性实验中,用狗ND2基因特异性引物和Taqman探针体系分别扩增猪肉、牛肉、山羊肉、绵羊肉、鸭肉、鸡肉、鹅肉、鸽子肉、鸵鸟肉、鹌鹑肉、兔肉、马肉、驴肉、猫肉的基因组DNA模板。结果表明,非目标物种基因组DNA的扩增曲线的Ct值均大于35,说明狗引物和Taqman探针特异性良好,如图2所示。
2.3灵敏度、扩增效率检测结果
以10倍为稀释梯度,狗基因组DNA分别稀释为102、101、100、10-1、10-2、10-3、10-4、10-5ng/μL。在10-5ng/μL浓度下,未检测到目标物种。在102~10-4ng/μL浓度范围内,建立Ct值-lg[DNA]的标准曲线,如图3所示。标准曲线的R2为0.9957,线性相关度良好。PCR反应扩增效率为108%。DNA浓度为10-4ng/μL时,Ct值为34.6。该引物、Taqman探针的实时荧光反应体系可检出10-4ng的模板DNA量。因此,灵敏度为100fg。
2.4市售样本的检测结果
在对市售的20个牛、羊肉串检测实验中,设置阳性对照、阴性对照和空白对照。检测样本、阴性对照、空白对照中并未检出狗源性成分,阳性对照扩增曲线完好。说明,狗引物、Taqman探针具有很强的实际应用价值。
3 结论与讨论
线粒体DNA是高等动物唯一的核外遗传物质,是检测中比较理想的靶基因,而且细胞中均含有大量的线粒体。目前,国内外学者关于线粒体的测序及其他研究已经非常透彻,在NCBI基因数据库中有大量已发表并做注释的线粒体序列,便于针对不同的物种进行引物和探针的设计。目前,国内外学者主要以线粒体中cytb、D-loop、12S rRNA、16S rRNA、ND1、ND2等基因作为目标基因位点,本研究通过筛查狗线粒体中的不同基因,并将设计的引物和探针序列在NCBI的Blast中搜索、比对,最终在狗线粒体ND2基因位点上,发现既可以保证种内保守,又可保证种间特异的引物和探针序列。因此,选取了狗线粒体ND2基因序列片段设计了引物和Taqman探针。

图1 DNA条形码扩增产物在NCBI中Blast搜索比对结果Fig.1 Sequence alignment of sequence amplification product
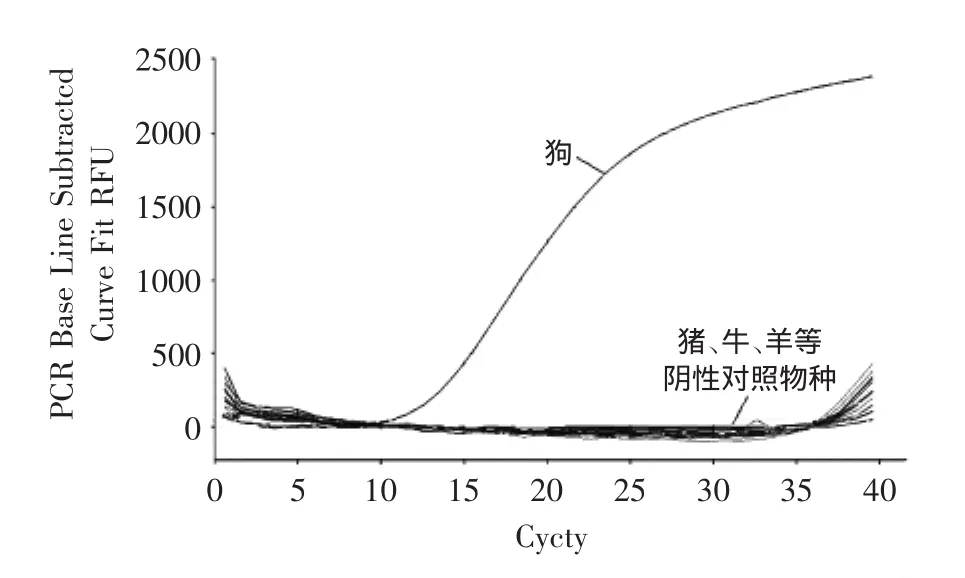
图2 狗源性引物探针实时荧光PCR的特异性检测结果图Fig.2 Specific detection of real-time PCR for dog
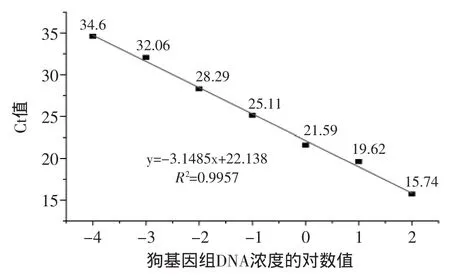
图3 Ct值-lg[DNA]的标准曲线Fig.3 Standard curve showing ct values in relation to the concentration of initial target gene copies
在实际样本的检测中,通过阳性对照、阴性对照和空白对照实验,检测样本中并未检出狗源性成分。需要注意点是,由于该方法的灵敏度较高,根据动物源性成分检出量,不能判断是意外污染还是蓄意添加。因此,需要结合其他方法进一步实验,以便更好的判断掺杂作假的程度。此外,该Taqman探针的荧光PCR反应体系是否适用于肉骨粉、明胶、化妆品中的狗源性成分的检测,还有待于进一步验证。
总之,狗ND2基因特异性引物、Taqman探针的荧光PCR反应体系特异性好,灵敏度高,稳定性强,实用性强,适用于食品中狗源性成分的检测,可为食品安全检测、打击掺假掺杂等违法行为提供技术参考。
[1]卜登攀,王加启,贺云霞,等.动物源性饲料检测技术研究进展[J].中国畜牧兽医,2008,35(2):54-59.
[2]陈文炳,邵碧英,廖宪彪,等.加工食品中若干动物成分的PCR检测技术应用研究[J].食品科学,2005,26(8):338-342.
[3]李家鹏,乔晓玲,田寒友,等.食品和饲料中动物源性成分检测技术研究进展[J].食品科学,2011,32(9):340-347.
[4]张慧霞,张利平,吴建平,等.鹌鹑源性成分PCR检测方法的建立[J].中国兽医科学,2008,38(4):346-349.
[5]陈颖,吴亚军,徐宝梁,等.食品及饲料中马属动物源性成分的PCR检测研究[J].中国生物工程杂志,2004,24(5):78-83.
[6]Colombo F,Viacava R,Giaretti M.Differentiation of the species ostrich(Struthio camelus)and emu(Dromaius novaehollandiae)by polymerase chain reaction using an ostrich-specific primer pair[J].Meat Science,2000,56(1):15-17.
[7]Wan Q H,Fang S G.Application of species-specific polymerase chain reaction in the forensic identification of tiger species[J].Forensic Science International,2003,131(1):75-78.
[8]Chen Y,Wu Y J,Xu B L,et al.Species-specific polymerase chain reaction amplification of camel(Camelus)DNA extracts[J]. Journal of Aoac International,2005,88(5):1394-1398.
[9]Haunshi S,Basumatary R,Girish P S,et al.Identification of chicken,duck,pigeon and pig meat by species-specific markers of mitochondrial origin[J].Meat Science,2009,83(3):454-459.
[10]Rodriguez M A,Garcia T,González I,et al.PCR identification of beef,sheep,goat,and pork in raw and heat-treated meat mixtures[J].Journal of Food Protection®,2004,67(1):172-177.
[11]Ahmad A I,Ghasemi J B.New FRET primers for quantitativereal-time PCR[J].Analytical and Bioanalytical Chemistry,2007,387(8):2737-2743.
[12]Jonker K M,Tilburg J,Hägele G H,et al.Species identification in meat products using real-time PCR[J].Food Additives and Contaminants,2008,25(5):527-533.
[13]Laube I,Zagon J,Broll H.Quantitative determination of commercially relevant species in foods by real-time PCR[J]. International Journal of Food Science&Technology,2007,42(3):336-341.
[14]Köppel R,Zimmerli F,Breitenmoser A.Heptaplex real-time PCR for the identification and quantification of DNA from beef,pork,chicken,turkey,horse meat,sheep(mutton)and goat[J]. European Food Research and Technology,2009,230(1):125-133.
[15]Hanna S E,Connor C J,Wang H H.Real-time Polymerase ChainReactionfortheFoodMicrobiologist:Technologies,Applications,and Limitations[J].Journal of Food Science,2005,70(3):R49-R53.
[16]Sawyer J,Wood C,Shanahan D,et al.Real-time PCR for quantitative meat species testing[J].Food Control,2003,14(8):579-583.
[17]Kesmen Z,Gulluce A,Sahin F,et al.Identification of meat species by TaqMan-based real-time PCR assay[J].Meat Science,2009,82(4):444-449.
[18]Chisholm J,Sánchez A,Brown J,et al.The development of species-specific real-time PCR assays for the detection of pheasant and quail in food[J].Food Analytical Methods,2008,1(3):190-194.
[19]Tobe S S,Linacre A M T.A multiplex assay to identify 18 European mammal species from mixtures using the mitochondrial cytochrome b gene[J].Electrophoresis,2008,29(2):340-347.
[20]Martín I,García T,Fajardo V,et al.Technical Note:Detection of cat,dog,and rat or mouse tissues in food and animal feed using species-specific polymerase chain reaction[J].Journal of Animal Science,2007,85(10):2734-2739.
[21]中国人民共和国山东出入境检验检疫局,中国检验检疫科学研究院,中华人民共和国辽宁出入境检验检疫局,等.GB/T 21105-2007动物源性饲料中狗源性成分定性检测方法PCR方法[S].2007.
[22]Fajardo V,González I,Rojas M,et al.A review of current PCR-based methodologies for the authentication of meats from game animal species[J].Trends in Food Science&Technology,2010,21(8):408-421.
[23]Sambrook J,萨姆布鲁克,Russell D W,et al.分子克隆实验指南[M].北京:科学出版社,2002.
[24]Hebert P D N,Cywinska A,Ball S L.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society of London.Series B:Biological Sciences,2003,270(1512):313-321.
[25]Hebert P D N,Ratnasingham S,de Waard J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J].Proceedings of the Royal Society of London.Series B:Biological Sciences,2003,270(S1):96-99.
Detection for dog-derived components in foods by real-time polymerase chain reaction
GAO Xiao-li1,YANG Xin-ting2,+,XUE Chen-yu2,WANG Dan2,ZHAO Lin-na2,HOU Cai-yun1,*
(1.School of Food Science and Nutritional Engineering,China Agricultural University,Beijing 100083,China;2.Beijing Municipal Center for Food Safety Monitoring,Beijing 100041,China)
A rapid and highly species-specific real-time polymerase chain reaction(PCR)assay had been developed for the detection of dog-derived components in meat and meat mixtures.Primers and Taqman probe were designed on the mitochondrial ND2,and the performance of the method was tested.Results showed that no cross-reaction was observed between the dog and non-target species,and this method revealed a high sensitivity and could detect 100fg of dog template DNA.In conclusion,this real-time PCR assay could be a rapid and straightforward method for the accurate identification of dog-derived components in food.
Real-time PCR;mitochondrial ND2 gene;dog-origin ingredient
TS201.6
A
1002-0306(2015)02-0061-04
10.13386/j.issn1002-0306.2015.02.004
2014-04-09+为并列第一作者
高晓丽(1990-),女,硕士研究生,研究方向:农产品加工与贮藏。杨昕霆(1986-),男,硕士研究生,研究方向:动物源性成分检测。
侯彩云(1963-),女,博士研究生,教授,研究方向:食品科学与加工技术。
北京市科技计划课题(Z131100005613005)。
