WRKY转录因子的基因组水平研究现状
2015-09-14颜君郭兴启曹学成
颜君 郭兴启 曹学成
WRKY转录因子家族是植物中最大的转录调控因子家族之一,能够调控植物的生长和发育进程及生物和非生物胁迫。植物基因组测序的大规模完成,为在基因组水平全面研究WRKY转录因子家族提供了可能。2003年,72个拟南芥AtWRKY从测序基因组数据中鉴定出,并且发现49个参与了病菌防御过程。之后,实时定量PCR(qRT-PCR)、微阵列芯片(Microarray)、RNA测序(RNA-seq)等技术应用到大规模分析植物WRKY家族的研究中。尤其是2013年后,全基因组鉴定植物WRKY基因家族的研究报道日益增多。本文综述了近年来全基因组鉴定植物WRKY基因家族和其功能的研究成果,旨在为研究人员提供一定的参考。
1 WRKY转录因子的发现、功能及起源
1.1 WRKY转录因子的发现
1994年,Ishiguro[1]首次从甘薯中克隆出一个WRKY转录因子并命名为SPF1,之后人们相继克隆出WRKY家族的其他成员并研究了它们的特性和功能,主要集中在拟南芥、烟草、大豆和辣椒中[2]。
WRKY基因家族的成员都含有WRKY结构域,其蛋白质序列N端含有高度保守的氨基酸序列WRKYGQK和CX4-5CXnHXH/C(X为任意氨基酸),属于锌指转录因子,通过结合靶基因启动子区域的W-box[(T)TGACC(A/T)]核苷酸序列而调控基因表达[3]。根据WRKY的结构和特点,可分为3个组(图1),其中组Ⅰ含有两个WRKY结构域,而组Ⅱ和组Ⅲ都只含有一个WRKY结构域[4]。

图1 拟南芥全长WRKY蛋白的结构图和家族分类图[4,5]
1.2 WRKY转录因子的功能
WRKY转录因子家族是植物中最大的转录调控因子家族之一,是很多信号网络不可缺少的一部分,能调控多种植物进程:疾病防御、非生物胁迫应答、营养不足、衰老、种子和毛状体发育、胚胎发生、多种发育方面及植物激素控制的生物进程;WRKYs可以以同源二聚体或异源二聚体的形式,充当转录的激活子或抑制子[6]。WRKY调控涉及到的信号通路,包括脱落酸(Abscisic acid,ABA)、水杨酸(salicylic acid,SA)、茉莉酸(Jasmonic acid,JA)/乙烯(Ethylene,ET)、丝裂原活化蛋白激酶(Mitogen activated protein kinase kinase,MAPKK)、14-3-3 蛋白、钙调蛋白(calmodulin)、组蛋白去乙酰化酶(Histone deacetylases)、阻力蛋白(Resistance proteins)等[6,7]。
1.3 WRKY转录因子的起源和进化
早在 2004年,Ulker[8]就提出组Ⅰ WRKY 基因或许代表着WRKY基因的祖先状态。2005年,Zhang等[9]研究了当时公布的基因组数据(包括拟南芥、水稻、绿藻Chlamydomonas reinhardtii等),提出了一个进化模型——WRKY基因起源于原生生物,在绿藻等植物中经过多次基因复制和歧化事件,导致在开花植物中形成一个大的基因家族,例如在水稻中有 100个成员(图 2)。2005年,Wu等[10]研究了水稻和拟南芥中WRKY基因的起源,发现水稻组Ⅲ 的基因通过复制和剧烈的扩增产生;在原生生物(Giardia lamblia,Dictyostelium discoideum)和绿藻(Chlamydomonas reinhardtii)中存在WRKY-like的类似祖先的基因。
2 拟南芥和水稻全基因组的WRKY转录因子研究
WRKY转录因子发现后,其生物学功能的研究主要依赖于经典分子生物学实验。随着基因组测序技术的发展,特别是植物基因组测序的完成,为在基因组水平全面研究WRKY转录因子家族提供了可能。2000年模式植物拟南芥基因组测序完成[11],2002年水稻基因组测序完成[12],故全基因组水平WRKY转录因子家族的研究首先开始于这两种植物。
2.1 拟南芥全基因组的WRKY研究
2000年,Eulgem[4]通过分析基因组序列,鉴定出61个拟南芥(Arabidopsis thaliana)WRKY基因(AtWRKY1 to AtWRKY61)。2003年,Dong 等[13]又发现一些新的WRKY基因(共鉴定出72个);Northern blotting印记法揭示出这72个AtWRKY中有49个有差别地调节了抗病进程,或被SA处理后的反应进程。2006年,Tosti等[14]用实时反转录PCR(Real-time reverse transcriptase PCR)和微阵列基因芯片(Gene chip microarray)的方法发现,WRKY介导了O3压力诱导的RLKs(Receptor-like kinases)信号通路,这一点和病原体诱导的转录反应信号通路相似。2011年,Wang等[15]分析了拟南芥WRKY基因家族的进化,建立了系统进化树,发现其分为五个亚组(Ⅰa、Ⅰb、Ⅱa、Ⅱb和Ⅲ);检查了进化动力,发现净化选择(purifying selection)和功能歧化(Functional divergence)在本家族进化中存在。
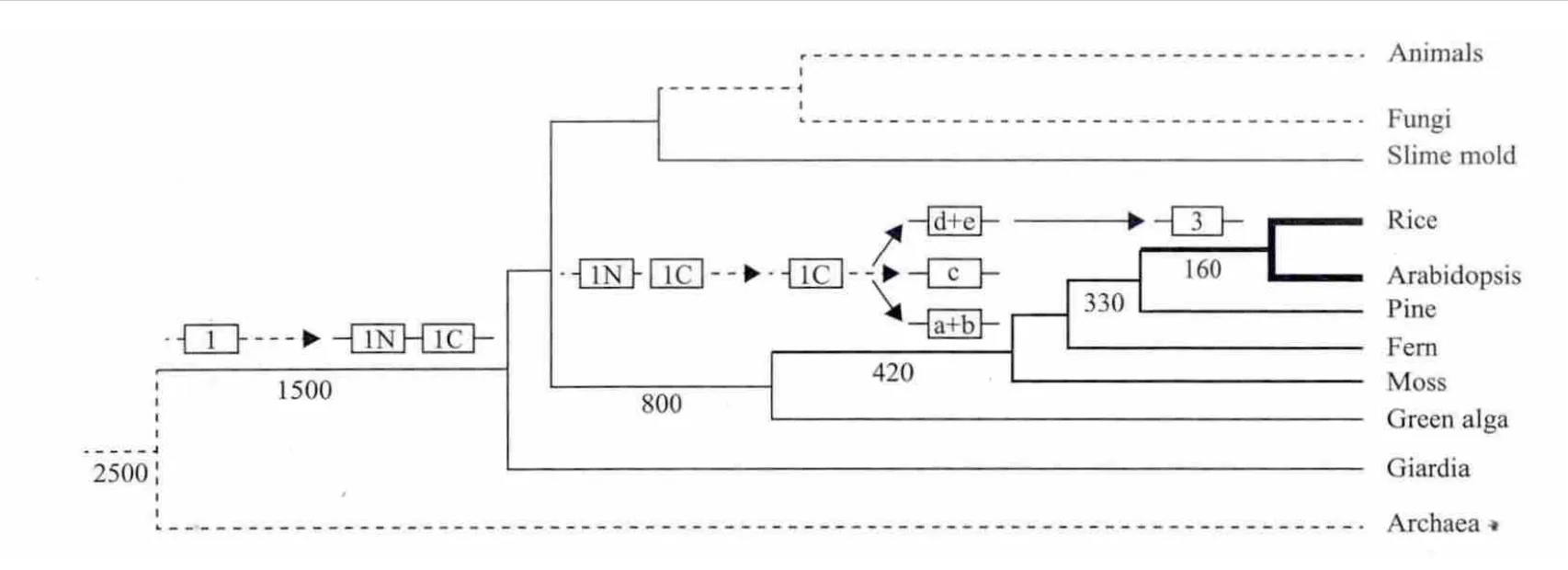
图2 WRKY基因家族的起源和复制模式图[9]
2.2 水稻全基因组的WRKY研究
2005年,Xie等[16]预测出 81个水稻(Oryza sativa)OsWRKY基因,进化分析支持假说“WRKY基因经过了一次WRKY结构域的复制形成祖先状态,后又经过丢失一个WRKY结构域的过程形成了组Ⅱ和组Ⅲ”;染色体分布分析发现26%的OsWRKY位于1号染色体;Northern blot研究糊粉细胞的WRKY基因的表达模式,发现OsWRKY-24、-51、-71、 和 -72被 ABA所 诱 导。2006年,Ryu等[17]用半定量 RT-PCR和 Northern blot分析证明一部分的WRKY涉及到了应答水稻病原体感染的防卫相关基因的转录激活进程:SA处理后,叶片的OsWRKY-45、-62表达量增高;JA处理后,OsWRKY-10、-82和-85增高;同时两者处理,OsWRKY-30和-83增高。2008年Ramamoorthy等[18]预测出103个OsWRKY基因,77.7%位于水稻复制区域,45.6%是零碎复制(Fragmentally duplicated),而35%(包含在45.6%中)是串联复制(Tandem duplicate);全面转录谱分析,发现正常生长组有65个OsWRKY基因在转录丰度和基因表达模式有差异,而在非生物胁迫(冷、干旱、高盐)压力和植物激素处理下54个在转录丰度表现出很大的不同。
2009年,Berri等[19]发现 104个 OsWRKY基因中的24个成员差异性地调节至少一种检测的生长压力,存在9个OsWRKY基因簇(由系统进化相关基因和不相关基因组成)显著地共表达;用基因微阵列芯片和泊松相关系数分析方法发现AtWRKYs从属于两个主要的共调控网络(COR-A,COR-B)和两个小点的网络(COR-C,COR-D)。2014年,Nuruzzaman等[20]分析了水稻近等基因系在可控制情况下、激素处理、干旱状况下的全转录组概况,发现存在差异基因活化和组织特异性基因活化。
3 其他植物全基因组WRKY转录因子研究进展
随着植物基因组的大面积测序,其他植物全基因组的WRKY转录因子研究也在如火如荼地进行。此类研究策略一般如图3所示,主要分为生物信息学分析和表达模式研究两部分。
3.1 鉴定和进化方面的分析
全基因组WRKY转录因子研究的基础是通过BLAST或HMM扫描基因组,鉴定出本植物基因组WRKY基因家族成员的数量和序列等(表1);然后基于保守序列构建系统进化树,从而对WRKY家族进行分类等其他生物信息学分析。大部分植物的WRKY基因家族的分类是相似的,可以分为3个组和若干亚组[Ⅰ、Ⅱ(Ⅱa-Ⅱe)、Ⅲ]。
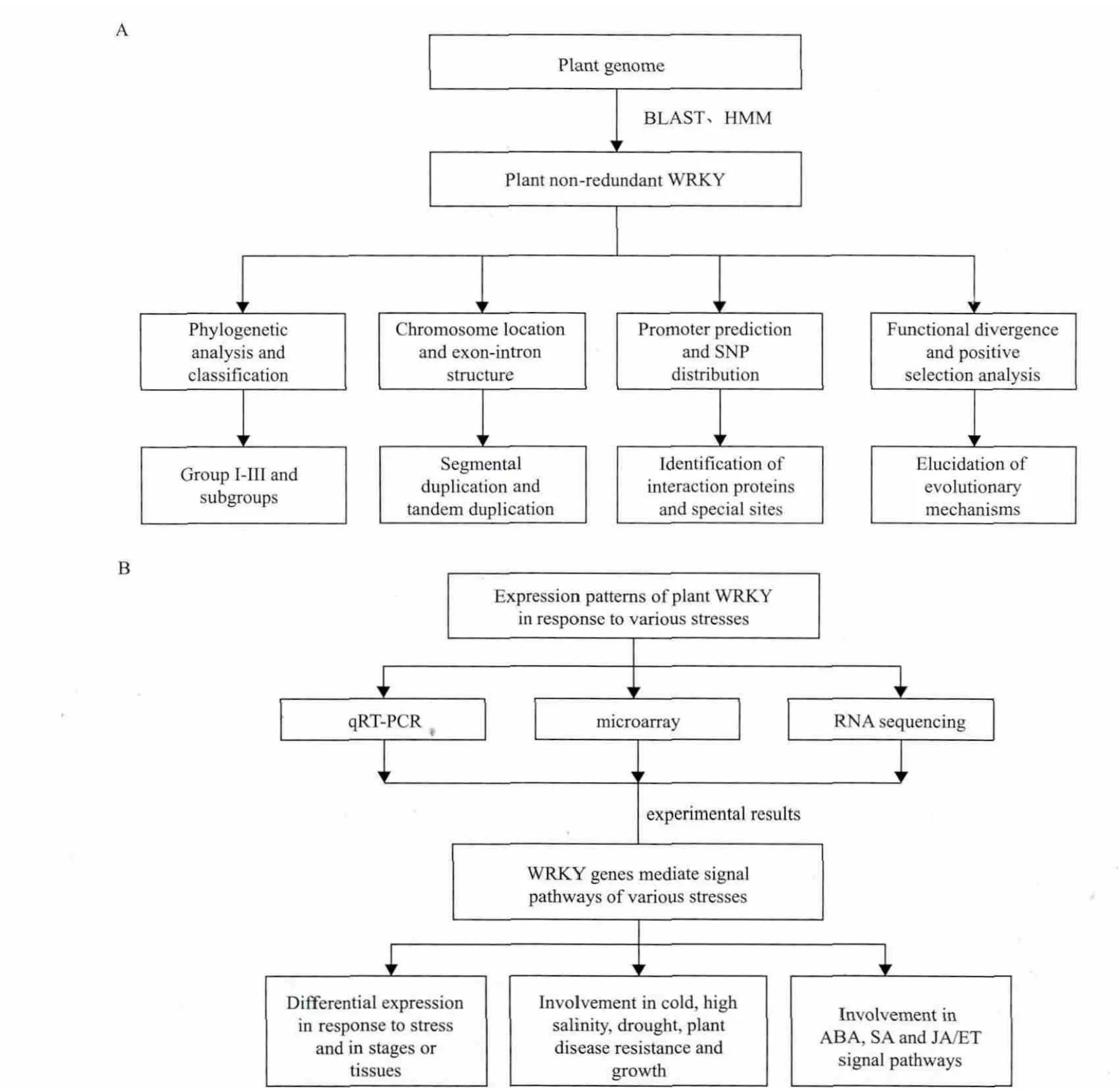
图3 全基因组鉴定和分析WRKY基因家族的一般研究策略
除了家族分类,其他生物信息学分析也是基于研究植物WRKY的全基因组鉴定信息,但是各文献的研究侧重点各不相同,现总结如下。(1)染色体位置分析:有研究发现其植物的WRKY在有些染色体位置上富集形成了物种特异性的基因簇,来源于片段复制(Segmental duplication)或者串联复制(Tandem duplication)[23,27,38-40,45,46]。 巴 豆 与水稻、拟南芥、蓖麻的WRKY基因进化分析比较发现JcWRKY基因没有发生基因复制事件[36]。大部分的二穗短柄草BdWRKY集群基因对(Clustered gene pairs)有着高度相似的WRKY结构域,暗示它们或许具有功能冗余[28]。棉花物理图谱比对发现GrWRKY和GaWRKY的染色体位置并不相互对应,暗示其二倍体化时发生了剧烈的染色体重排[38]。(2)进化动力分析 :玉米[23]、大豆[46]、棉花[38]进化中存在功能歧化现象。中国白菜[32]和棉花[38]经历了进化选择。大豆不同的亚组具有不同的进化速率,组Ⅱe和组Ⅲ检测到很强的正选择压力[46]。(3)启动子分析:欧洲大叶杨很多涉及压力和防御应答的顺式作用元件分布在PtWRKY基因的上游[5]。(4)单核苷酸多态性(Single nucleotide polymorphism,SNP)分析:许多棉花(GrWRKY和GaWRKY)SNP位点不均等的分布在外显子和内含子区域[38]。(5)构建数据库:构建了一个包含二穗短柄草BrWRKY和其他植物WRKY等功能的数据库网站(http://www.igece.org/WRKY/BrachyWRKY/BrachyWRKYIndex.html)[27]。(6)功能注释和标签表达分析:揭示小麦TaWRKY的主要功能是对刺激物的应答和对病菌侵扰的防御,分子机制主要是结合DNA;在空白对照植物和由叶锈病菌(Puccinia triticina)接种过的小麦近等基因系之间表达有差异[26]。通过同源分析发现有些菊花CmWRKY与拟南芥AtWRKY功能相同,有些不一样[53]。(7)亚细胞定位预测:这些假定的小白菜BcWRKY 蛋白富集于细胞核区域,BcWRKY-25和-40的实验更加证实了这一预测[30]。(8)地质年代分析:通过系统进化分析7个WRKY基因,鉴定出山葵属(Syagrus)是椰子属(Cocos)的一个姐妹群,两者分歧时间发生在3 500万年前,建立发生在3 700万年前的渐新世(Oligocene)的东部巴西地区[54]。
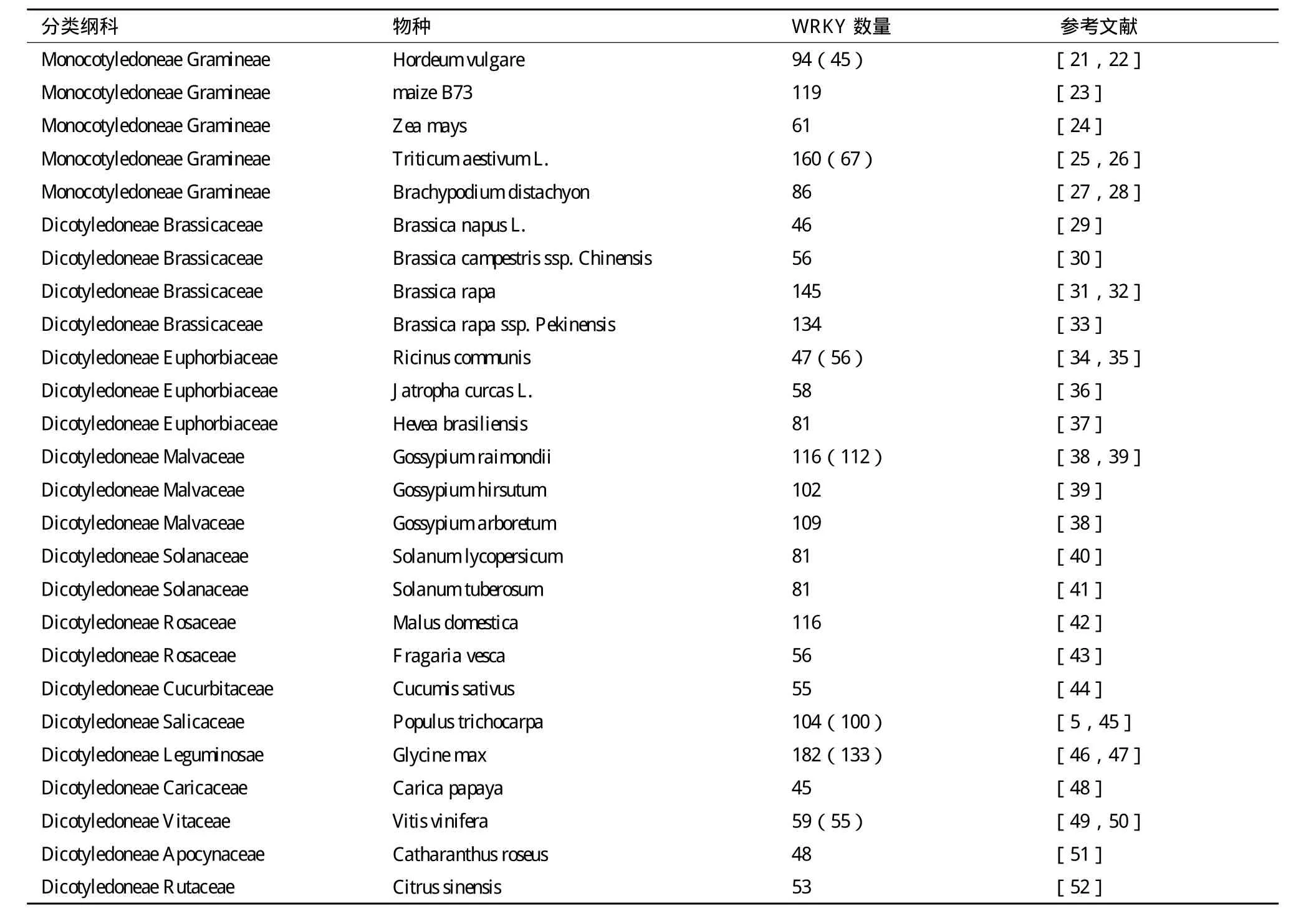
表1 植物中WRKY的鉴定
还有一些鉴定方面的研究。(1)亚细胞定位:通过绿色荧光蛋白(GFP)观察了4个欧洲油菜BnWRKY蛋白的细胞亚定位[29]。GFP观察了大麦HvWRKY1和2的核定位,功能实验证明两者是病菌侵染(BghA6,Blumeria graminis f. sp. hordei Speer isolate A6)诱导基因 HvGER4c 的抑制子[22]。(2)蛋白相互作用:在酵母实验中观察到橡胶树HbWRKY-3、-14、-55蛋白结合HbSRPP(橡胶小粒子蛋白,Small rubber particle protein)启动子并激活其表达,说明HbWRKY可能涉及到自然橡胶的生物合成的转录调控[37]。
3.2 表达模式研究
WRKY转录因子家族作为重要的基因家族参与应答环境信号、调节植物生长发育过程等。基于全基因组鉴定研究植物WRKY家族成员的信息,通过qRT-PCR、微阵列基因芯片、转录组测序等高通量手段全面研究本植物WRKY家族成员在不同试剂处理、不同环境压力下的基因表达模式,为揭示其具体作用机制提供了一定的线索。
3.2.1 qRT-PCR 主要用qRT-PCR的方法验证了各植物WRKY家族的差异表达、参与了生物胁迫和非生物胁迫、涉及到了JA等各信号通路。欧洲油菜13个BnWRKY基因会对病原真菌和激素处理(ABA、JA、SA、ET、细胞分裂素的一种或多种)反应应答[29]。黄瓜在正常生长条件下48个CsWRKY在转录丰度和基因表达模式上表现出明显的差别,在非生物胁迫(冷、干旱、高盐)压力下23个表达有差异性;压力诱导的CsWRKY 表达模式和拟南芥同系物的表达模式很相似(除了组ⅢCsWRKYs)[44]。欧洲大叶杨PtWRKY亚组III在各种压力(冷、旱、高盐、水杨酸)下表达,PtWRKY76在 0.1mmol/L的SA 或25% PEG-6000(聚乙二醇,polyethylene glycol)处理下可以被剧烈地诱导出来[45]。番茄在不同的发育进程和应答各种的生物胁迫和非生物胁迫过程中,SlWRKY基因表现出不同的时空表达,18个选择好的SlWRKY基因应答干旱高盐压力和病菌感染压力[40]。蓖麻正常发育情况下,RcWRKY基因无论在转录丰度水平还是在基因表达模式水平上都表现出差异性[34]。巴豆在正常发育情况下根、茎、叶、种子4种组织的表达数据揭示,47个JcWRKY基因至少可应答一种非生物胁迫压力(干旱、盐碱、磷酸饥饿、氮饥饿)[36]。中国白菜叶片BrWRKY基因应答生物胁迫和非生物胁迫压力时的表达模式有差异和相似性[32]。番木瓜TF807.3、72.14在低温下上调,TF807.3、43.76、12.199、12.62涉及到了干旱压力,TF9.35、18.51、72.14、12.199涉及到了受伤反应,TF12.199、807.3、21.156、18.51被病原体诱导,TF72.14、43.76被 ABA诱导[48]。二穗短柄草BdWRKY基因的表达快速地被各种压力和植物激素所调节[28]。橡胶树组织特异性表达模式检测结果显示74个HbWRKYs至少在一种检测组织中表达,而其他7个HbWRKY则在所有检测组织中表达量较低,说明HbWRKYs参加细胞内的各种进程;在嫩枝植株加入JA和ET试剂处理后观察选择20个HbWRKY基因(其中13个在乳胶中富含)的表达情况,发现其中17个HbWRKY至少应答一种试剂,说明HbWRKY被JA和ET信号通路调控[37]。15个菊花的CmWRKY基因应答各种植物激素处理、生物胁迫、非生物胁迫(qRT-PCR)[53]。检测了大豆被P. pachyrhizi感染后8个GmWRKY基因的表达,发现与敏感型植株相比,抗性型植株的这些基因表达的更早更强[47]。qRT-PCR检测61个欧洲大叶杨PtWRKY基因的组织特异性表达,发现只有46个表达在各种组织中(根、茎、叶等),且不同组织中的表达模式大不相同,18个PtWRKY基因被一种或多种压力处理时表达且转录丰度相对高;过量表达SA诱导的基因PtrWRKY89,加速PR蛋白的表达且改善转基因杨树对病菌的抗性,说明PtrWRKY89是杨树中SA依赖的防御信号通路的调节子[5]。检验小白菜叶片,共有22个BcWRKYs在应答至少一种压力状况下(脱落酸、冷、高盐、热、高渗)差异性地表达,共表达分析结果显示压力诱导的BcWRKYs共调控多个非生物胁迫压力应答信号通路;BcWRKY-33、-40、-53和-70 作为核心调节子,在压力诱导的BcWRKYs 共调控网络中起主要作用[30]。
有些研究用了不止qRT-PCR一种手段。用微阵列芯片Br135K(针对所有BrWRKY基因设计)和RT-PCR(以根茎叶等特异组织为模板)分析,发现中国白菜BrWRKY的表达有一定的组织特异性,大白菜亚种Chiifu和Kenshin对冷、干旱、高盐、病菌侵染的BrWRKY应答表达模式不相同[31]。葡萄的7种组织(幼叶、成熟叶、叶卷须、茎尖、根、幼果、成熟果实)中检测VvWRKYs表达,发现大部分至少在两种组织中表达;36种VvWRKYs在冷压力下表达水平改变;结合各种数据分析(基因芯片、qRT-PCR、全局转录组)鉴定出15种VvWRKYs应答冷压力,其中3个VvWRKY基因可以被ABA所诱导[50]。棉花的微阵列芯片、表达模式数据和qRT-PCR结果显示,GhWRKY可能调节纤维、花药、组织(根、茎、叶、胚胎)的发育,且涉及到了压力应答;表达模式分析结果显示大部分的组Ⅱ和组ⅢGhWRKY在各种压力下高度表达;而代表着祖先状态的组Ⅰ在压力下的表达量低,似乎对各种压力都不敏感[39]。2014年,Wang等[55]用 qRT-PCR和RNA测序方法分析了双单倍体(di-haploid)杨树(Populus simonii×P. nigra)的51个WRKY基因(共119个)在应答高盐压力的基因表达模式,发现有13个PthWRKY基因在高盐处理后在根茎叶组织中表达但它们的表达模式很不相同,根据表达量可分为3组:组Ⅰ(PthWRKY-28、-45、-105)表达量低,组Ⅱ(PthWRKY-56、-88、-116)中等,组Ⅲ(PthW RKY-41、-44、-51、-61、-62、-75、-106)高 ;组Ⅱ和组Ⅲ的“诱导扩增-恢复”的动态表达模式或许说明它们在调控高盐应答上作用很重要。
3.2.2 微阵列基因芯片 芯片检测到大麦一些特殊的表达模式;在单子叶植物(大麦)和双子叶植物(拟南芥)之间的WRKY基因有保守保留的功能[21]。玉米WRKY基因或许参与了其生长和发育的调控,涉及到了干旱压力和病菌感染的应答[23]。胡杨对长时程干旱压力的反应,发现在转录丰度上改变的基因包括一些转录因子家族,如WRKY、AP2/EREPB、bZIP等[56]。 一 大 批 葡 萄VvWRKYs应答各种压力而激活[50]。6个锈病相关的小麦微阵列芯片数据分析后支持功能注释结论,即TaWRKY应答抗病反应;两者分析结果的主要代表是 TaWRKY-10、-15、-17、-56[26]。 用 superSAGE、微分离创伤后的RNA测序、微阵列芯片检验到75个大豆GmWRKY基因涉及大豆锈菌(Phakopsora pachyrhizi)感染应答的功能[47]。
3.2.3 转录组测序 中国白菜的根、茎、叶等组织的BrWRKY有组织特异性表达和差异性表达模式[32]。大豆正常生长状况下的127个GmWRKY基因在RNA转录丰度和基因表达模式水平上都具有显著的差异性[46]。棉花WRKY基因参与到了不同棉花亚种特殊纤维的发育;在二倍体棉花和四倍体棉花的纤维发育过程中,复杂的WRKY基因表达模式也被观察到,如G. hirsutum中差异性的等位基因Dt和At的基因表达及可变剪切事件[38]。利用公布的RNA-Seq数据在转录组水平分析了小麦TaWRKYs基因对干旱应答的情况,在耐旱品种Sivas 111/33和易旱品种Atay 85植株的根和叶片组织中检测了TaWRKY16/16-A、TaWRKY-17、-19-C,-24、-59、-61、-82的 相 对表达水平发现,Sivas 111/33根组织中的以上所有基因的量都上调了,其中小麦应答干旱的基因TaWRKY-16、-24、-59、-61、-82 是 首 次 发 现[25]。2014年,Huang等[57]RNA测序了成熟前和成熟后中国梨(Pyrus ussuriensis)的转录组发现了3 729个差异表达的基因,含有PuWRKYs。高通量转录组分析鉴定出61个欧洲大叶杨PtWRKY基因应答生物胁迫和非生物胁迫压力[5]。
4 问题与展望
目前WRKY转录因子的研究还主要集中在单个基因的克隆、表达和功能方面,它们的具体作用机制还不完全清楚。随着测序技术的发展和各植物基因组测序信息的公布,使得大规模全基因组水平鉴定WRKY基因家族以及研究它们在各种压力应答下的基因表达模式成为可能。这样的整体性研究会为单个WRKY基因作用机制研究提供一定的预测。文献分析结果显示,目前全基因组鉴定WRKY基因家族,主要局限在WRKY的生物信息学分析和表达模式的初步研究,较少有涉及其信号调控网络精细机制。完善各种环境压力应激条件下WRKY的表达模式研究,利用包括二代测序mRNA-seq和蛋白质组学技术等,研究其基因表达变化,同时结合ChIP-seq技术,研究全基因组水平WRKY的结合靶基因,构建WRKY转录因子调控网络,分析不同应激条件下WRKY通过哪些靶基因和通路起到抵抗应答作用等,将是未来的研究热点。
[1]Ishiguro S, Nakamura K. Characterization of a cDNA encoding a novel DNA-binding protein, SPF1, that recognizes SP8 sequences in the 5' upstream regions of genes coding for sporamin and betaamylase from sweet potato[J]. Mol Gen Genet, 1994, 244(6):563-571.
[2]Eulgem T, Somssich IE. Networks of WRKY transcription factors in defense signaling[J]. Curr Opin Plant Biol, 2007, 10(4):366-371.
[3]余迪求, 陈利钢, 张利平. 转录调控因子WRKY 超级家族[J].云南植物研究, 2006, 28(1):69-77.
[4]Eulgem T, Rushton PJ, et al. The WRKY superfamily of plant transcription factors[J]. Trends Plant Sci, 2000, 5(5):199-206.
[5]Jiang Y, Duan Y, Yin J, et al. Genome-wide identification and characterization of the Populus WRKY transcription factor family and analysis of their expression in response to biotic and abiotic stresses[J]. J Exp Bot, 2014, 65(22):6629-6644.
[6]Bakshi M, Oelmuller R. WRKY transcription factors:Jack of many trades in plants[J]. Plant Signal Behav, 2014, 9:e27700.
[7]Rushton PJ, Somssich IE, Ringler P, et al. WRKY transcription factors[J]. Trends Plant Sci, 2010, 15(5):247-258.
[8]Ulker B, Somssich IE. WRKY transcription factors:from DNA binding towards biological function[J]. Curr Opin Plant Biol,2004, 7(5):491-498.
[9]Zhang Y, Wang L. The WRKY transcription factor superfamily:its origin in eukaryotes and expansion in plants[J]. BMC Evol Biol,2005, 5:1.
[10]Wu KL, Guo ZJ, Wang HH, et al. The WRKY family of transcription factors in rice and Arabidopsis and their origins[J].DNA Res, 2005, 12(1):9-26.
[11]Analysis of the genome sequence of the flowering plant Arabidopsis thaliana[J]. Nature, 2000, 408(6814):796-815.
[12]Yu J, Hu S, et al. A draft sequence of the rice genome(Oryza sativa L. ssp. indica)[J]. Science, 2002, 296(5565):79-92.
[13]Dong J, Chen C, Chen Z. Expression profiles of the Arabidopsis WRKY gene superfamily during plant defense response[J].Plant Mol Biol, 2003, 51(1):21-37.
[14]Tosti N, Pasqualini S, Borgogni A, et al. Gene expression profiles of O3-treated Arabidopsis plants[J]. Plant Cell Environ, 2006, 29(9):1686-1702.
[15]Wang Q, Wang M, Zhang X, et al. WRKY gene family evolution in Arabidopsis thaliana[J]. Genetica, 2011, 139(8):973-983.
[16]Xie Z, Zhang ZL, Zou X, et al. Annotations and functional analyses of the rice WRKY gene superfamily reveal positive and negative regulators of abscisic acid signaling in aleurone cells[J]. Plant Physiol, 2005, 137(1):176-189.
[17]Ryu HS, Han M, Lee SK, et al. A comprehensive expression analysis of the WRKY gene superfamily in rice plants during defense response[J]. Plant Cell Rep, 2006, 25(8):836-847.
[18]Ramamoorthy R, Jiang SY, Kumar N, et al. A comprehensive transcriptional profiling of the WRKY gene family in rice under various abiotic and phytohormone treatments[J]. Plant Cell Physiol, 2008, 49(6):865-879.
[19]Berri S, Abbruscato P, Faivre-Rampant O, et al. Characterization of WRKY co-regulatory networks in rice and Arabidopsis[J]. BMC Plant Biol, 2009, 9:120.
[20]Nuruzzaman M, Sharoni AM, Satoh K, et al. Comparative transcriptome profiles of the WRKY gene family under control,hormone-treated, and drought conditions in near-isogenic rice lines reveal differential, tissue specific gene activation[J]. J Plant Physiol, 2014, 171(1):2-13.
[21]Mangelsen E, Kilian J, Berendzen KW, et al. Phylogenetic and comparative gene expression analysis of barley(Hordeum vulgare)WRKY transcription factor family reveals putatively retained functions between monocots and dicots[J]. BMC Genomics,2008, 9:194.
[22]Liu D, Leib K, Zhao P, et al. Phylogenetic analysis of barley WRKY proteins and characterization of HvWRKY1 and -2 as repressors of the pathogen-inducible gene HvGER4c[J]. Mol Genet Genomics, 2014, 289(6):1331-1345.
[23]Wei KF, Chen J, Chen YF, et al. Molecular phylogenetic and expression analysis of the complete WRKY transcription factor family in maize[J]. DNA Res, 2012, 19(2):153-164.
[24]李修善. 玉米WRKY基因聚类分析[J]. 曲阜师范大学学报,2012, 38(3):93-97.
[25]Okay S, Derelli E,Unver T. Transcriptome-wide identification of bread wheat WRKY transcription factors in response to drought stress[J]. Mol Genet Genomics, 2014, 289(5):765-781.
[26]Satapathy L, Singh D, Ranjan P, et al. Transcriptome-wide analysis of WRKY transcription factors in wheat and their leaf rust responsive expression profiling[J]. Mol Genet Genomics, 2014,289(6):1289-1306.
[27]Tripathi P, Rabara RC, Langum TJ, et al. The WRKY transcription factor family in Brachypodium distachyon[J]. BMC Genomics,2012, 13:270.
[28]Wen F, Zhu H, Li P, et al. Genome-wide evolutionary characterization and expression analyses of WRKY family genes in Brachypodium distachyon[J]. DNA Res, 2014, 21(3):327-339.
[29]Yang B, Jiang Y, Rahman MH, et al. Identification and expression analysis of WRKY transcription factor genes in canola(Brassica napus L.)in response to fungal pathogens and hormone treatments[J]. BMC Plant Biol, 2009, 9:68.
[30]Tang J, Wang F, Wang Z, et al. Characterization and coexpression analysis of WRKY orthologs involved in responses to multiple abiotic stresses in Pak-choi(Brassica campestris ssp.chinensis)[J]. BMC Plant Biol, 2013, 13:188.
[31]Kayum MA, Jung HJ, Park JI, et al. Identification and expression analysis of WRKY family genes under biotic and abiotic stresses in Brassica rapa[J]. Mol Genet Genomics, 2014.
[32]Tang J, Wang F, Hou X, et al. Genome-wide fractionation and identification of WRKY transcription factors in chinese cabbage(Brassica rapa ssp. pekinensis)reveals collinearity and their expression patterns under abiotic and biotic stresses[J]. Plant Molecular Biology Reporter, 2014, 32(4):781-795.
[33]蒋明, 苗立祥, 张豫超, 等. 大白菜WRKY转录因子的全基因组鉴定与分析[J]. 浙江农业学报, 2013, 25(5):980-986.
[34]Li HL, Zhang LB, Guo D, et al. Identification and expression profiles of the WRKY transcription factor family in Ricinus communis[J].Gene, 2012, 503(2):248-253.
[35]邹智. 蓖麻WRKY转录因子的全基因组鉴定及其进化分析[J].中国油料作物学报, 2013, 35(1):36-42.
[36]Xiong W, Xu X, Zhang L, et al. Genome-wide analysis of the WRKY gene family in physic nut(Jatropha curcas L.)[J].Gene, 2013, 524(2):124-132.
[37]Li HL, Guo D, Yang ZP, et al. Genome-wide identification and characterization of WRKY gene family in Hevea brasiliensis[J].Genomics, 2014, 104(1):14-23.
[38]Ding M, Chen J, Jiang Y, et al. Genome-wide investigation and transcriptome analysis of the WRKY gene family in Gossypium[J].Mol Genet Genomics, 2015, 290(1):151-171.
[39]Dou L, Zhang X, Pang C, et al. Genome-wide analysis of the WRKY gene family in cotton[J]. Mol Genet Genomics, 2014, 289(6):1103-1121.
[40]Huang S, Gao Y, Liu J, et al. Genome-wide analysis of WRKY transcription factors in Solanum lycopersicum[J]. Mol Genet Genomics, 2012, 287(6):495-513.
[41]黄胜雄, 刘永胜. 土豆WRKY转录因子家族的生物信息学分析[J]. 应用与环境生物学报, 2013, 19(2):205-214.
[42]许瑞瑞, 张世忠, 曹慧, 等. 苹果WRKY转录因子家族基因生物信息学分析[J]. 园艺学报, 2012, 39(10):2049-2060.
[43]苗立祥, 张豫超, 等. 森林草莓全基因组WRKY转录因子基因的鉴定与分析[J]. 核农学报, 2012, 26(8):1124-1131.
[44]Ling J, Jiang W, et al. Genome-wide analysis of WRKY gene family in Cucumis sativus[J]. BMC Genomics, 2011, 12:471.
[45]He H, Dong Q, Shao Y, et al. Genome-wide survey and characterization of the WRKY gene family in Populus trichocarpa[J]. Plant Cell Rep, 2012, 31(7):1199-1217.
[46]Yin G, Xu H, Xiao S, et al. The large soybean(Glycine max)WRKY TF family expanded by segmental duplication events and subsequent divergent selection among subgroups[J]. BMC Plant Biol, 2013, 13:148.
[47]Bencke-Malato M, Cabreira C, Wiebke-Strohm B, et al. Genomewide annotation of the soybean WRKY family and functional characterization of genes involved in response to Phakopsora pachyrhizi infection[J]. BMC Plant Biol, 2014, 14(1):236.
[48]Pan LJ, Jiang L. Identification and expression of the WRKY transcription factors of Carica papaya in response to abiotic and biotic stresses[J]. Mol Biol Rep, 2014, 41(3):1215-1225.
[49]李成慧, 蔡斌. 葡萄 WRKY 转录因子家族全基因组分析[J].经济林研究, 2013, 31(4):127-131.
[50]Wang L, Zhu W, Fang L, et al. Genome-wide identification of WRKY family genes and their response to cold stress in Vitis vinifera[J]. BMC Plant Biol, 2014, 14:103.
[51]Schluttenhofer C, Pattanaik S, Patra B, et al. Analyses of Catharanthus roseus and Arabidopsis thaliana WRKY transcription factors reveal involvement in jasmonate signaling[J]. BMC Genomics, 2014, 15:502.
[52]李学鑫, 白志川, 岳欢, 等. 甜橙WRKY转录因子序列的生物信息学分析[J]. 北方园艺, 2013, 2013(1):119-123.
[53]Song A, Li P, Jiang J, et al. Phylogenetic and transcription analysis of chrysanthemum WRKY transcription factors[J]. Int J Mol Sci,2014, 15(8):14442-14455.
[54]Meerow AW, Noblick L, Borrone JW, et al. Phylogenetic analysis of seven WRKY genes across the palm subtribe Attaleinae(Arecaceae)[corrected]identifies Syagrus as sister group of the coconut[J]. PLoS One, 2009, 4(10):e7353.
[55]Wang S, Wang J, Yao W, et al. Expression patterns of WRKY genes in di-haploid Populus simonii× P. nigra in response to salinity stress revealed by quantitative real-time PCR and RNA sequencing[J]. Plant Cell Rep, 2014, 33(10):1687-1696.
[56]Yan DH, Fenning T, Tang S, et al. Genome-wide transcriptional response of Populus euphratica to long-term drought stress[J].Plant Sci, 2012, 195:24-35.
[57]Huang G, Li T, Li X, et al. Comparative transcriptome analysis of climacteric fruit of chinese pear(Pyrus ussuriensis)reveals new insights into fruit ripening[J]. PLoS One, 2014, 9(9):e107562.
