DNA甲基化定量测量国际比对CCQM P94.2
2015-07-19高运华陈鸿飞盛灵慧武利庆王晶
高运华,陈鸿飞,盛灵慧,武利庆,王晶
(1.中国计量科学研究院,北京100029;2.南京市计量监督检测院,江苏南京210037)
DNA甲基化定量测量国际比对CCQM P94.2
高运华1,陈鸿飞2,盛灵慧1,武利庆1,王晶1
(1.中国计量科学研究院,北京100029;2.南京市计量监督检测院,江苏南京210037)
建立基因组DNA基质中CDKN2A基因甲基化含量的液相色谱同位素稀释质谱法和Sanger序列测定法两种定量方法,应用于国际物质量咨询委员会(consultative committee for amount of substance,CCQM)开展的DNA甲基化定量测量国际比对(CCQM P94.2)研究。考察DNA甲基化效率、酶解条件、色谱分离条件等对测定结果的影响,评定测量结果的不确定度。液相色谱同位素稀释质谱定量两个比对未知样的结果分别为未知样1:64.5%±6.1%;未知样2:46.7%±8.0%。Sanger序列测定法测定两个比对未知样的结果分别为未知样1:48.9%±1.0%;未知样2:48.9%±1.1%。与主导实验室标准值未知样1:51%±5.9%,未知样2:46.9%±4.9%十分符合,为我国DNA甲基化定量测量标准物质的研制奠定坚实的基础。
DNA甲基化;同位素稀释质谱;Sanger测序;国际比对
0 引言
DNA甲基化能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达[1-3]。DNA甲基化定量信息将有利于更好地解释这些生物现象,并提升DNA甲基化相关疾病诊断的正确性。DNA甲基化定量分析的方法现已有多种,除了传统的免疫、酶催化和化学方法外,高效毛细管电泳、高效液相色谱、液质联用等现代仪器分析方法也取得显著进展[4-6]。然而,没有一种测量参考系统可以评价不同测量方法和不同测量仪器之间结果的一致性[7-8]。亚硫酸氢盐介导的方法是最方便而被广泛应用的技术,亚硫酸氢盐将没有甲基化的胞嘧啶核苷酸转化为尿嘧啶核苷酸,再通过PCR反应转化为胸腺嘧啶核苷酸,而甲基化的胞嘧啶核苷酸保持不变,PCR反应转化后仍为胞嘧啶核苷酸,通过测定胸腺嘧啶核苷酸和胞嘧啶核苷酸的含量变化实现甲基化的定量分析。但是,现在还没有一个能够充分评估其转化反应及后续分析过程(包括PCR反应)偏差的方法,不能确保DNA甲基化分析结果的可比性、有效性[9-10]。因此,急需建立DNA甲基化测量标准方法,研制DNA甲基化方法和仪器评价相关的标准物质。为了建立一个DNA甲基化标准测量系统,国际物质量咨询委员会生物工作组(CCQM BAWG)开展了DNA甲基化定量测量国际比对研究,该比对代号为CCQM P94.2,由韩国标准科学研究院(KRISS)主导,7个国家计量院参加,分别采用序列测定技术、熔点法、液相色谱-同位素稀释质谱法、限制性内切酶法等对样品进行了定量比对分析。中国计量科学研究院实验室采用了液相色谱同位素稀释质谱和序列测定技术两种方法参加了此次比对活动,取得了较好的结果。
1 实验部分
1.1 仪器与试剂
液相色谱(Agilent 1290,Agilent公司);质谱仪(Qtrap 5500,AB Sciex公司);PCR扩增仪(PTC0200,Bio-Rad公司);琼脂糖凝胶电泳仪(Power Pac系列,Bio-Rad公司);紫外分光光度计(Beckman DU800,Beckman公司);DNA序列分析仪(ABI 3500)。
DNA甲基化试剂盒(EZ DNA methylation kit D5002,Zymo),蛇毒磷酸二酯酶I(Phosphodiesterase I,Sigma)和DNA酶I(DNase I,Sigma),腺嘌呤脱氧核苷一磷酸(dAMP)标准物质(GBW09805),胞嘧啶脱氧核苷一磷酸(dCMP)标准物质(GBW09806),鸟嘌呤脱氧核苷一磷酸(dGMP)标准物质(GBW09807),胸腺嘧啶脱氧核苷一磷酸(TMP)标准物质(GBW09808)标准物质,均来自中国计量科学研究院;dAMP-15N(Silantes公司),dCMP-13C15N(Silantes公司),dGMP-13C15N(Isotec公司),TMP-13C15N(剑桥同位素实验室)。乙酸铵(色谱纯,阿拉丁公司);乙腈(色谱纯,Merck公司);实验室用水来自Milli-Q超纯水系统。比对样品来自韩国标准科学研究院。
1.2 样品亚硫酸盐甲基化修饰
比对样品共两个,其中比对样品1(U1)由比对主导实验室利用亚硫酸盐处理并纯化,直接作为模板进行PCR扩增。未知样2(U2)由参加实验室进行亚硫酸盐处理。首先取14μL DNA样品,加入5μL M-Dilution缓冲溶液,再加入31μL超纯水,混匀,37℃温浴15min。然后加入100μL CT转化试剂,混匀后,50℃暗处温浴12~16h。然后将样品置于冰上10min。利用试剂盒中的纯化试剂将修饰后的样品进行纯化,纯化后的样本用于后续的PCR扩增。
1.3 比对样品的PCR扩增和纯化
PCR反应体系为:模板DNA 1μL;引物F4:GA GGGGAGTTAGGAATAAAATAA 1μL;R1:ACCACC ATCTTCCCACCCTCAA,1μL;AmpliTaq Gold PCR master mix 25μL,水22μL。热反应条件为:第1步,95℃,5min,1个循环;第2步,先94℃,30 s,然后58℃30s,最后72℃,30s,30个循环;第3步,72℃,7min,1个循环。反应后的样品用QIA quick PCR纯化试剂盒进行纯化,用琼脂糖凝胶电泳进行产物检验。
1.4 DNA甲基化定量分析
1.4.1 DNA甲基化液相色谱同位素稀释质谱(HPLCIDMS)定量分析
1)HPLC-IDMS分析条件优化。利用核苷酸标准物质、消解后的样本对液相色谱和质谱分析条件进行了系统优化,使4种核苷酸dAMP,dGMP,dCMP,TMP之间及其他杂质成分基线分离,然后利用优化后的液相色谱同位素稀释质谱定量测量条件,对酶解后的比对样品进行了定量分析。
2)比对样品的酶消解条件优化及消解。系统优化了Phosphodiesterase I酶用量和酶消解时间,具体的优化方案见表1。

表1 酶反应条件优化方案
反应结束后,置于65℃水浴中反应20min灭活,置于4℃的冰箱中保存待测。校准样品和未知样品在相同的条件下进行酶解处理。
1.4.2 比对样品甲基化含量的序列测定分析
将纯化后的PCR产物,通过DNA连接酶插入DNA质粒载体中,然后将转化的质粒DNA导入大肠杆菌中,随机挑取200个单菌落,进行DNA序列分析,通过统计计算得到其甲基化含量。
2 结果与讨论
2.1 PCR扩增产物的琼脂糖凝胶电泳分析
比对样品1、2,100%甲基化样本M100和0%甲基化样本M0,通过PCR扩增后产物琼脂糖凝胶电游分析可知,DNA分子量标准全部分开,条带清晰,扩增产物处于200bp片段长度标准处,表明PCR扩增产物约200bp。另外上样孔处干净,证明样品中蛋白质成分含量极低,用琼脂糖凝胶电泳方法不能检出,图中下部没有明显的亮色,证明扩增产物中RNA成分去除干净。
2.2 HPLC-IDMS分析条件优化
优化后的液相色谱分析条件为:色谱柱:Waters ACQUITY UPLC HSS T3;流动相:(A相)5%乙腈-(B相)醋酸铵(pH=3.6);梯度洗脱程序为:0~6min,100%B;6~8min,100%B降为98%B;8~12min,98%B降为60%B;12~20min,维持60%B;20~21min,60%B升为100%B;21~30min维持100%B。柱温:25℃,进样量:10μL。优化后的质谱条件为:正离子扫描;多反应检测模式(MRM);离子源温度(TEM)650.00℃;离子源喷雾电压5500.00V,雾化器(GS1)50.00L/h,辅助加热器(GS2)40.00L/h;气帘气(CUR)30.00kPa。检测离子对见表2。
利用优化后的色谱质谱分析条件对酶解后的比对样品进行定量分析,分析结果见图1。4种脱氧核苷酸在优化的色谱质谱分析条件下达到基线分离,互不干扰,样品浓度合适,峰高和峰面积满足定量分析的要求。
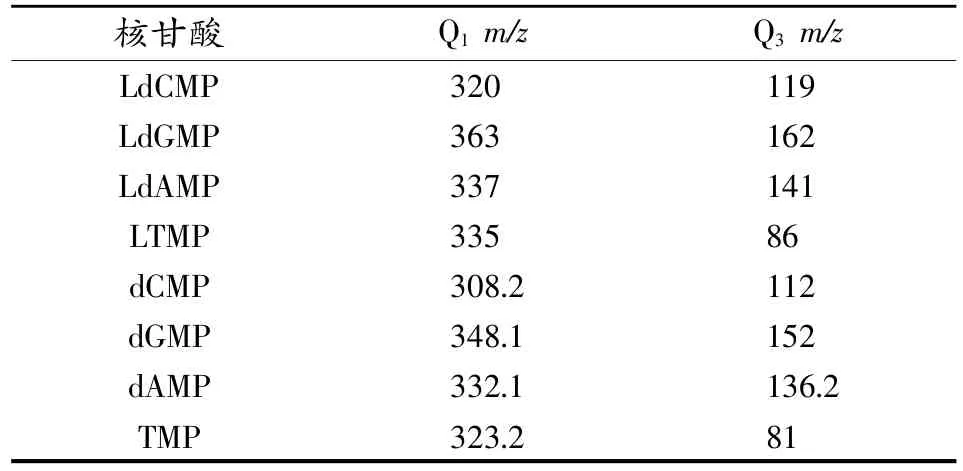
表2 检测离子对

图1 脱氧核苷酸液相色谱同位素稀释质谱分析
2.3 酶反应条件的优化
酶反应条件主要从Phosphodiesterase I用量和水解时间两方面进行优化。利用液相色谱-同位素稀释质谱定量分析各反应条件下的核苷酸含量,统计分析结果见图2。
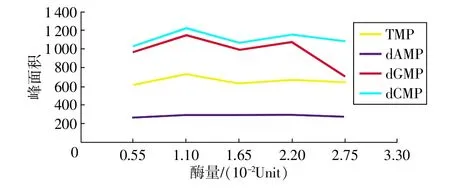
图2 Phosphodiesterase I酶用量优化
分析图2可知,随着Phosphodiesterase I量的增加,各核苷酸的量初期增加,达到1.1×10-2Unit时含量达到了最高,随后产物的量趋于平衡并稍微减少,表明酶量在1.1×10-2Unit时较为合适,超过1.1×10-2Unit时产物趋于平衡或下降,原因可能是酶中含有杂质,促使产物进一步降解所致。
酶水解时间的优化结果见图3,酶反应时间在60min时产物量基本达到了平衡,最后确定了酶的优化时间为60min。
图3 比对样品酶水解条件优化
因此,优化后的酶解条件为:酶解时间为1 h,DNase I为2Unit,反应温度37℃,Phosphodiesterase I为1.1×10-2Unit,样品量为400ng。
2.4 酶消解产物的HPLC-IDMS分析及不确定度评定
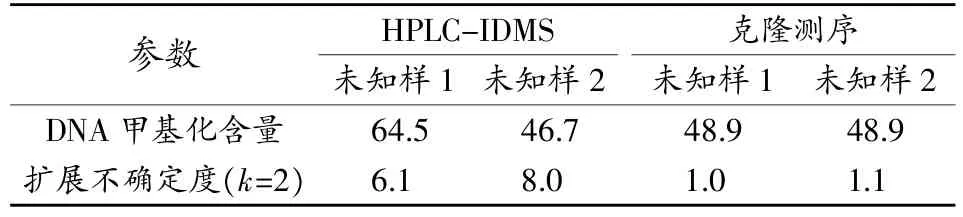
表3 CCQM P94.2比对样品HPLC-IDMS定量结果
利用优化后的液相色谱同位素稀释质谱分离分析条件,对经过酶解并定容后的比对样品进行了定量分析,结果见表3。测量结果的不确定度评定主要从方法的精度、样品和标准溶液的配制称量、酶消解程度、DNA甲基化效率等方面进行了系统考察,结果见表4。

表4 DNA甲基化HPLC-IDMS定量结果各项相对标准不确定度及合成标准不确定度
2.5 DNA甲基化克隆测序结果及不确定度分析
亚硫酸盐处理,然后克隆测序测定DNA甲基化含量是DNA甲基化定量测定的金标准;但这种方法操作较为复杂,且定量结果的准确度受选取的单克隆数量的限制。为尽量提高克隆测序的准确度,比对过程中每个样品随机选取了200个克隆进行测序,克隆的分辨率提升到0.5%。通过统计计算得到其甲基化含量,评定了其测量结果的不确定度,结果见表4。
2.6 比对结果等效图
CCQM P94.2国际比对结果见图4,图中实红线为主导实验室设定参考值,两侧的红虚线为设定参考值的不确定度。C-seq为中国计量科学研究院克隆测序结果,C-dNMP为中国计量科学研究院HPLC-IDMS测量结果。可以看出,中国计量科学研究院克隆测序的两个比对样品的结果均与标准值十分吻合。HPLC-IDMS测量结果均取得了等效度,未知样2测量结果与标准值十分吻合。综合分析表4和图4可知,克隆测序和HPLC-IDMS测量dNMP含量两种方法均可较好地完成DNA甲基化定量分析。HPLC-IDMS测量结果可以通过dNMP标准物质实现到国际基本单位的溯源,溯源途径清晰。而克隆测序结果的溯源途径还不十分清晰,相对应的计量标准物质缺乏,急需加强这方面的科技研究。
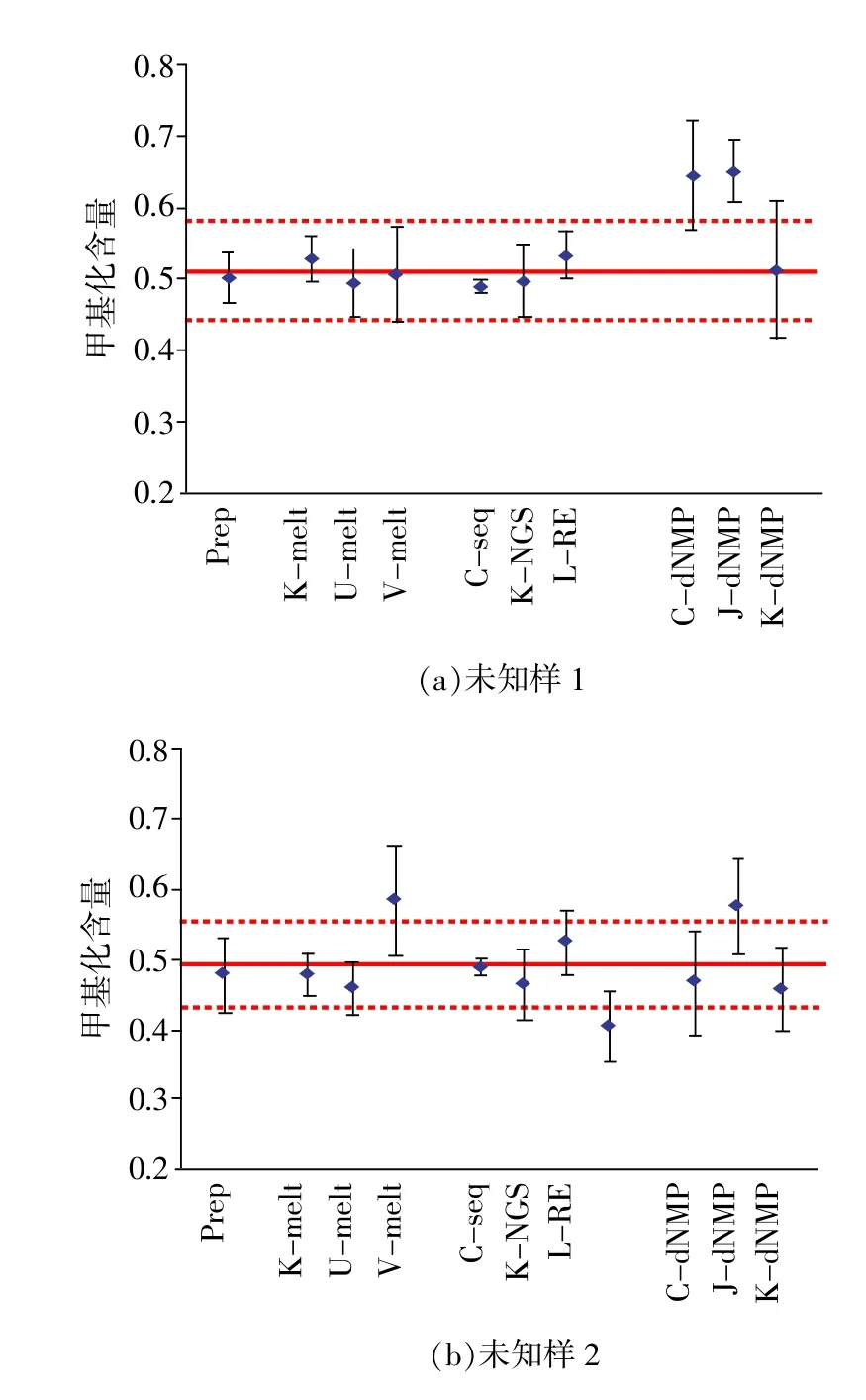
图4 CCQM P94.2国际比对结果
3 结束语
通过HPLC-IDMS和序列测定两种方法对比对样品进行了DNA甲基化含量定量测量,优化了酶消解条件、液相色谱同位素稀释质谱分析条件,选取了足够量的克隆进行序列测定分析,充分考虑各可能影响测定准确度的因素,评定了测量结果的不确定度,取得了较好的比对结果。比对结果证明了文中建立的方法准确可靠,可为后续的DNA甲基化计量标准物质的研制及临床诊断中DNA甲基化的定量分析提供借鉴和参考。
[1]Shi H,Maier S,Nimmrich I,et al.Oligonucleotidebased microarray for DNA methylation analysis:Principles and applications[J].Journal of Cellular Biochemistry,2003,88(1):138-143.
[2]邓大君.DNA甲基化和去甲基化的研究现状及思考[J].遗传,2014,36(5):403-410.
[3]张敏,权力,张霆.DNA甲基化水平定量分析方法研究进展[J].国际遗传学杂志,2010,33(1):29-31.
[4]Volkers N.DNA methylation:what is its role in carcinogenesis[J].Journal of the National Cancer Institute,2000,92(10):789-790.
[5]Dhingra T,Mittal K,Sarma G S.Analytical techniques for DNA methylation[J].Current Pharmaceutical Analysis,2014,10(1):71-85.
[6]彭思远,张洁,田美平,等.液相色谱-串联质谱法测定生物样本全基因组DNA甲基化[J].分析化学,2012,40(8):1201-1206.
[7]国振,符惠,李秀琴,等.橙汁中苯甲酸含量测定的国际比对APMP.QM-P23[J].色谱,2013,31(12):1194-1200.
[8]张慧敏,陶功华,杨建平,等.毛细管胶束电动色谱测定基因组DNA甲基化水平[J].中华预防医学杂志,2010,44(6):539-541.
[9]Akman K,Haaf T,Vijg J,et al.Genome-wide quantitative analysis of DNA methylation from bisulfite sequencing data[J].Bioinformatics,2014,30(13):1933-1934.
[10]Longley B J,Spiegelman V S,Wood G S.Quantitative gene analysis of methylation and expression(Q-GAME)in fresh or fixed cells and tissues[J].Experimental Dermatology,2014,23(5):304-309.
International com parison CCQM P94.2:quantitative analysis of DNA methylation
GAO Yunhua1,CHEN Hongfei2,SHENG Linghui1,WU Liqing1,WANG Jing1
(1.National Institute of Metrology,Beijing 100029,China;2.Nanjing Institute of Measurement and Technology,Nanjing 210037,China)
Two quantitative methods namely liquid chromatography-isotope dilution mass spectrometry(HPLC-IDMS)and Sanger sequencing method(SS)have been established to analyze and determine the CDKN2A gene methylation in the matrix of genomic DNA.The two methods,applied to the international comparison(CCQM P94.2)on quantitative measurement of DNA methylation carried out by the Consultative Committee for the Amount of Substance(CCQM),has investigated the influence of the DNA methylation efficiency,enzymolysis conditions and chromatographic separation conditions upon the measurement results and evaluated the uncertainty in measurement results.The DNA methylation of two unknown comparison samples measured were respectively U1:64.5%±6.1%and U2:46.7%±8.0%by HPLC-IDMS and U1:respectively 48.9%±1.0%and U2:48.9%±1.1%by the SS method.These figures are very close to the reference values from the leading laboratory,i.e.U1:51%±5.9%and U2:46.9%±4.9%.The established HPLC-IDMS and SS method have laid a solid foundation for China to quantitatively measure the DNA methylation in reference materials.
DNA methylation;isotope dilution mass spectrometry;Sanger sequencing;international comparison
A文章编号:1674-5124(2015)06-0047-05
10.11857/j.issn.1674-5124.2015.06.011
2014-11-03;
2015-01-17
国家质检行业公益专项项目资助(201310008)中央级公益性科研院所基本科研业务费项目资助(AKY1311)
高运华(1972-),男,山东定陶县人,副研究员,主要从事核酸计量及相关的法制计量工作。
