山药种质资源的ISSR 分析及初级核心种质库的构建
2015-07-05刘向宇霍秀文周翼虎赵智宏邰丽华
刘向宇,霍秀文*,杨 明,王 东,周翼虎,赵智宏,邰丽华
(1 内蒙古农业大学 农学院,内蒙古野生蔬菜种质资源与创新重点实验室,呼和浩特010019;2 内蒙古师范大学 生命科学与技术学院,呼和浩特010022)
山药(Dioscorea opposita Thunb.)是百合目(Liliales)薯 蓣 科(Dioscoreaceae)薯 蓣 属(Dioscorea)植物,为一年或多年生缠绕性藤本植物[1-3]。山药能形成肥大的地下块茎,其肉质细腻,风味鲜美,可供人们食用,同时山药还是重要的中药成分之一,具有增强人体免疫能力、延缓衰老、抑制肿瘤、抗氧化活性等作用[4]。但是由于山药在悠长的历史中各地区相互引种交流,造成市场上山药出现品种混杂、同名异物和同物异名的现象,严重制约了山药产业的发展。因此,收集优质高产的山药种质资源,并快速、准确地筛选优质种质,培育高产优质、高抗的新品种或为解决问题的关键。
植物遗传多样性资源是生物遗传的基础。近年来随着遗传资源的急剧增加,种质资源的保存利用成为急需解决的问题。Frankel[5-6]和Brown[7]提出以最小的资源份数最大限度地代表该物种的遗传多样性,即核心种质的概念,在一定程度上解决了这一问题。近年来国内外关于核心种质的研究较多,但多是基于表型性状。表型性状包括植物的农艺性状和有效成分含量等,而这些性状多为数量性状,易受环境条件及栽培措施的影响。因此,完全按照其作为核心种质构建的方法是不够准确的。分子标记的方法与表型数据相比,不受环境、采样部位的影响,并且稳定性相对较高,能从根本上反映植物群体以及个体之间的关系[8]。近年来采用分子标记技术研究植物种质遗传多样性并构建核心种质在很多农作物以及经济作物中得到了广泛的应用,邱丽娟等[9]、白成科 等[10]、周 延 清 等[11]、吴 慧 芳 等[12]和 李 慧 峰等[13]分别对大豆、山茱萸、地黄、天麻和甘薯的研究都得到了可靠的结果。本实验运用ISSR 分子标记技术对来自中国不同产地的35份山药种质资源进行遗传多样性分析,并利用聚类分析结果采用最小遗传距离逐步抽样法(LDSS)构建山药的初级核心种质库,为今后进一步构建山药核心种质奠定基础,为山药种质资源的保护利用提供理论依据。
1 材料和方法
1.1 材 料
供试35份材料(表1)种植于内蒙古农业大学科技园区山药种质资源圃。参考钱韦等[14]的取样方法,于2013年8月采集了种质资源圃生长旺盛期的山药幼嫩叶片,将采集的叶片放入有标记的塑封袋中,立即放入冰盒,带回实验室置于-80 ℃冰箱中冷冻备用。
1.2 PCR 反应
采用试剂盒(天根生化有限公司)提取35份材料的基因组DNA,用1%琼脂糖凝胶电泳,紫外分光 光 度 计 检 测DNA 纯 度 和 浓 度(ng/μL),将OD260/OD280在1.7~1.9 之间的DNA 稀释,-20℃保存备用。
参考 周 延 清 等[15]、胡 建 斌 等[16]、黄 玉 仙[17]的ISSR 引物,选用了32 条引物以山药基因组DNA为模板进行PCR 反应。筛选出电泳条带清晰、多态性高、重复性好的12条引物(表2),分别对35份样品基因组DNA 进行PCR 扩增。PCR 反应体系为25μL[18]:模 板DNA 5 μL(约50 ng/μL),1.0 μmol/L 引物2μL,2×Taq PCR Mix(0.1 U/μL Taq Polymerase、500 μmol/L dNTP each、20 mmol/L Tris-HCl pH 8.3、100 mmol/L KCl、3 mmol/L MgCl2)12.5μL,ddH2O 5.5μL。PCR 扩增程序为:94℃预变性5min,94℃变性30s,53℃退火45s,72 ℃延伸90s,45个循环,最后72 ℃延伸10min,4 ℃保存以备电泳,扩增产物用1.5%琼脂糖凝胶于90 V 电压下电泳90 min 后,用GAS700IB凝胶成像系统拍照分析。
1.3 数据分析
利用0、1矩阵的统计方式构建ISSR 数据库。采用POPGENE 32软件分析扩增产物的多态位点数、多态位点比率、Shannon信息指数(I)等遗传多样性指数。采用NTsys2.10e[19]聚类分析软件UPGMA 法进行聚类分析,构建聚类图。
1.4 初级核心种质库构建及其评价
用NTsys2.10e软件对种质资源聚类分析后,在聚类图中依据不同的遗传相似系数进行最小距离逐步抽样法(LDSS)构建核心种质[20]。具体方法为:通过观察35份山药种质的UPGMA 聚类图,将遗传相似系数较大的种质组合随机删除1份,再将剩余的种质再次聚类分析;再次删除遗传相似系数较大的成对种质中的1份种质。以此类推,直到筛选出能够达到要求的核心种质。第1次抽样时,选出一定份数种质,运用NTsys2.10e软件对其进行聚类分析;第2次抽样在第1次抽样聚类分析的基础上进行,以此类推。每次抽样后,均采用POPGENE32软件分别对构建的核心种质的ISSR-PCR扩增数据进行核心种质的遗传多样性分析,进而评价初级核心种质的代表性。
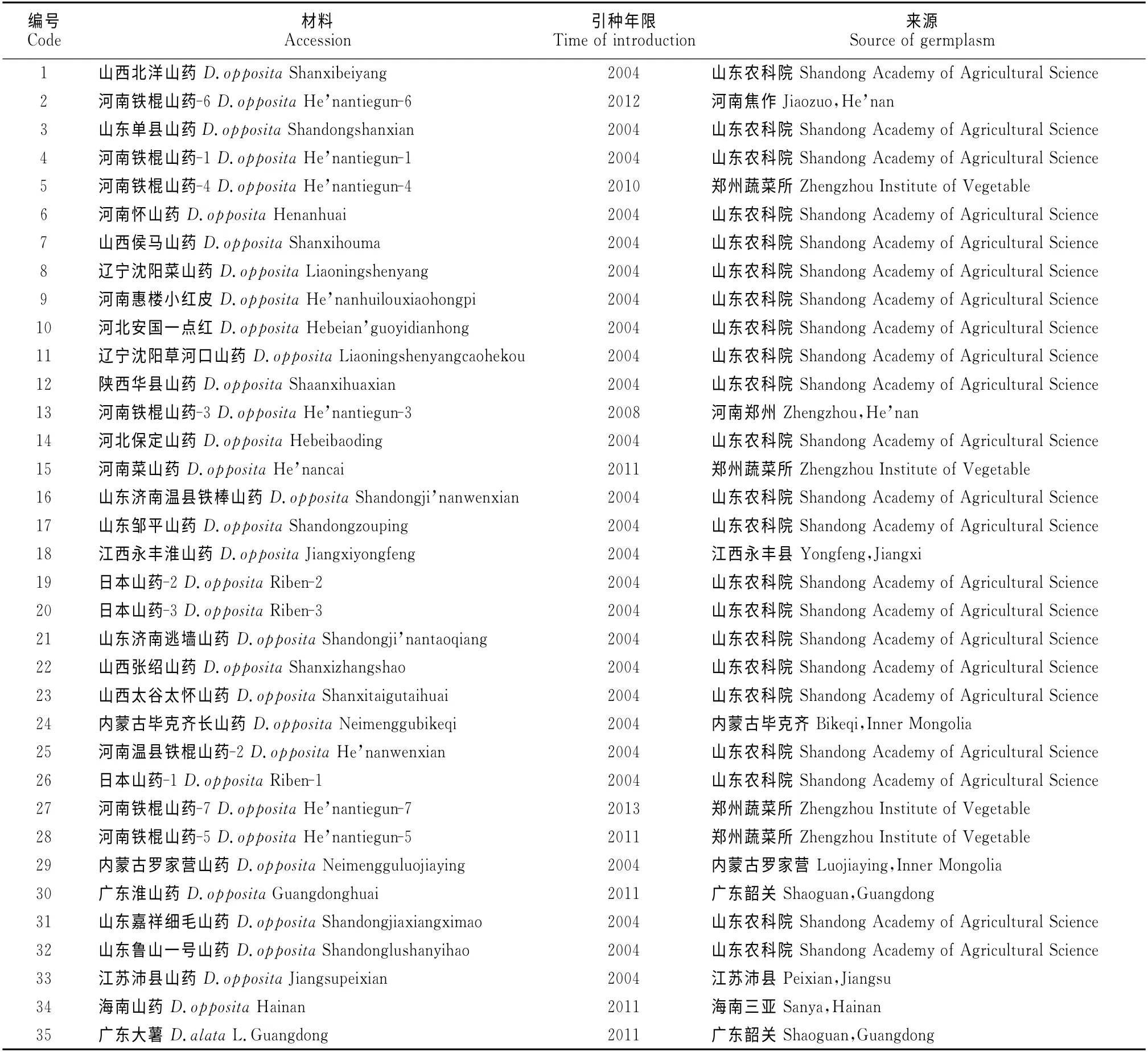
表1 供试山药材料及来源Table 1 Accessions of yam and source of germplasm
2 结果与分析
2.1 ISSR 标记的多态性分析
从32条ISSR 引物中共筛选出12 条多态性好、条带清晰的引物,分别对35 份山药材料进行PCR 扩增,均得到条带清晰、多态性高、重复性好的图片,图1为其中的引物ISSR-4对35份山药材料进行PCR 扩增的电泳图。由图1可以看出每个样品体系中均得到了清晰、丰富的条带,可进行下一步的多样性分析。
12条引物(表2)对35份山药材料共扩增出142个位点,每条引物最多能得到14条清晰条带(ISSR-10、ISSR-11),最少有9条(ISSR-4、ISSR-20)。除ISSR-2、3、4、25外,每条引物扩增出的多态性条带比率均达100%。在这142条带中,有138条重复性好、清晰的多态性条带,多态性条带比率高达97.18%,表明本研究所收集的山药种质资源在分子水平上具有丰富多态性,种质具有很好的代表性。
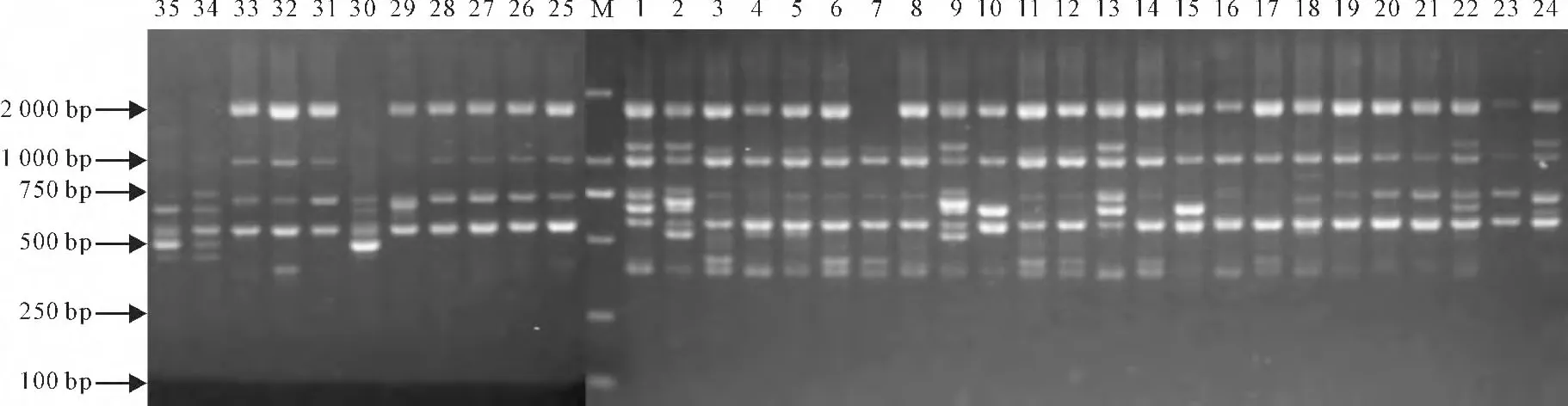
图1 引物ISSR-4对35个山药材料的PCR 扩增产物琼脂糖凝胶电泳图谱M.DL2000;1~35.编号同表1;下同Fig.1 Agarose gel electrophoresis of PCR products from primer ISSR-4in 35yam accessions M.DL2000;1-35.Same as Table 1and as below
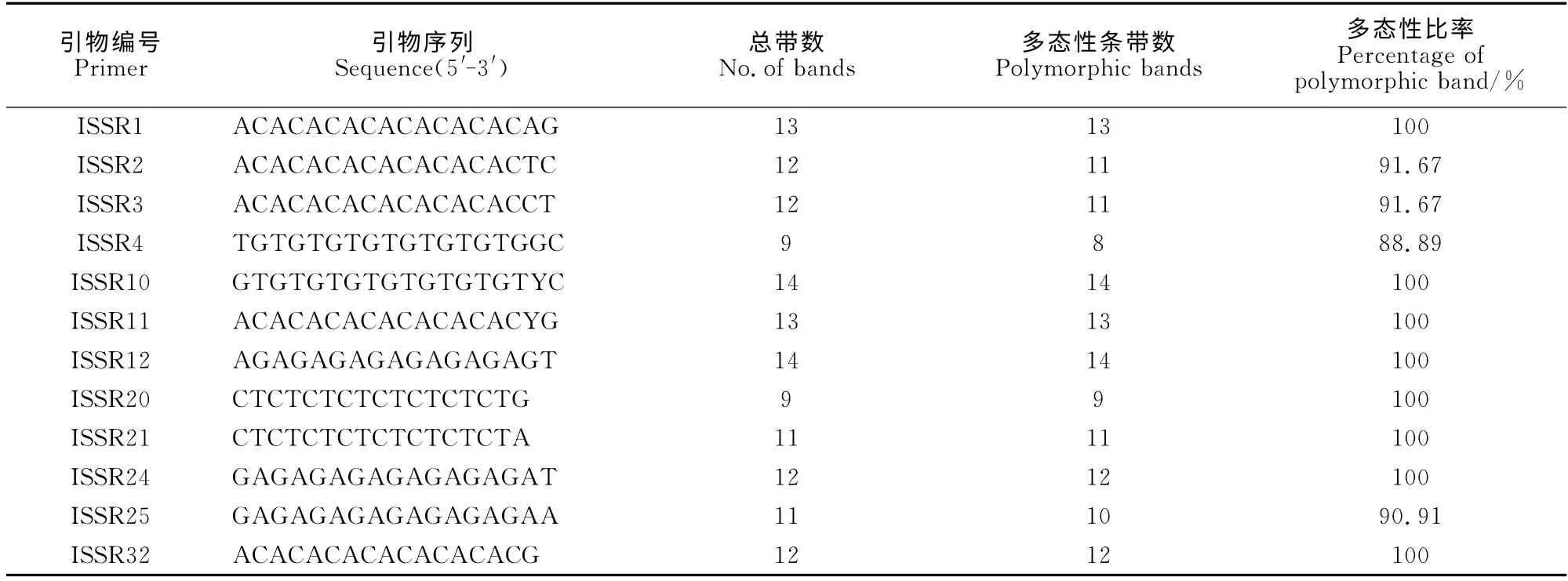
表2 供试ISSR 引物及其PCR 扩增的多态性Table 2 ISSR primers and polymorphism of PCR amplification
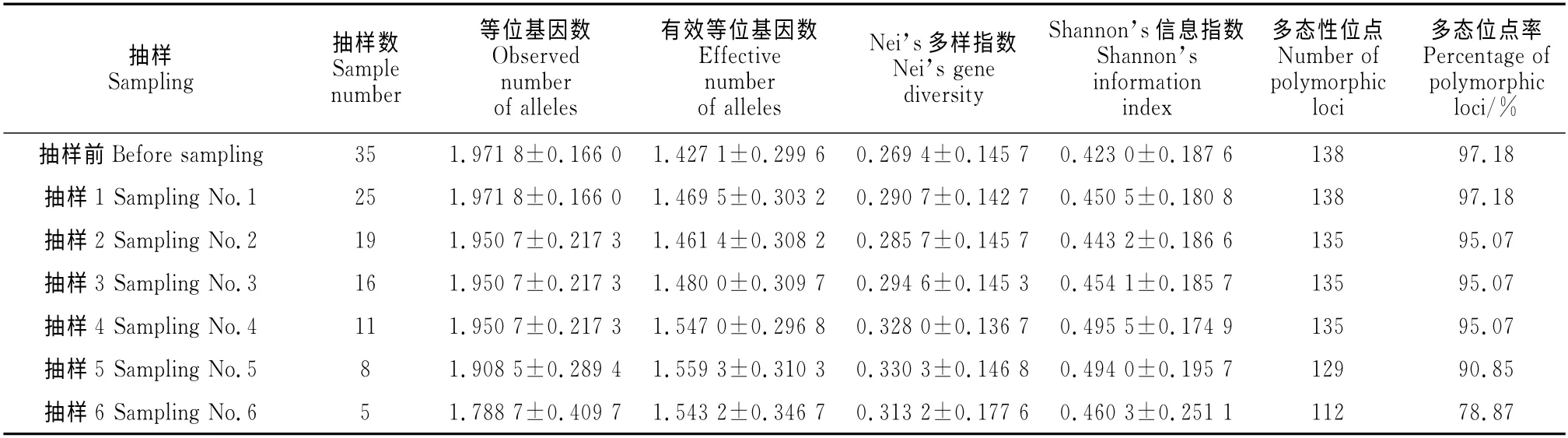
表3 最小距离逐步抽样法构建山药核心种质的遗传多样性Table 3 Genetic diversity index of core collection of yam by LDSS
2.2 山药种质资源遗传多样性分析
由POPGENE32软件分析结果(表3)可知,35个样品的扩增条带平均Shannon’s信息指数(I)为0.423 0,平均Nei’s基因多样性指数(h)为0.269 4,等位基因数目(Na)为1.971 8,每个位点平均有效等位基因数(Ne)为1.427 1。35份山药材料的遗传一致性为0.464 8~0.971 8 之间,遗传距离在0.028 6~0.693 1之间,其中河南铁棍山药-4(5号)与广东大薯(35 号)之间的遗传距离最大(0.693 1),而山西侯马山药(7号)与辽宁沈阳山药(8号)之间的遗传一致性最高(0.971 8)。
2.3 聚类分析
采用NTsys2.10e软件对35 份山药种质材料进行聚类分析(图2)。由图2可以看出,种质间的遗传相似系数介于0.54~0.97之间,在遗传相似系数Gs=0.628时,35份山药种质聚为两大类,为广东淮山药(30号)、海南山药(34号)与广东大薯(35号)聚为一类,其它种质聚为一大类。在遗传相似系数Gs=0.66处,35份山药种质被聚为四大类,其中广东淮山药(30号)单独聚为一类。河北安国一点红(10号)与河南菜山药(15号)聚为一类;海南山药(34号)与广东大薯(35号)聚为一类,其它种质聚为一大类,其中河南惠楼小红皮(9号)、内蒙古罗家营山药(29号)和内蒙古毕克齐长山药(24号)较为特殊在这一大类中分别单独聚类。
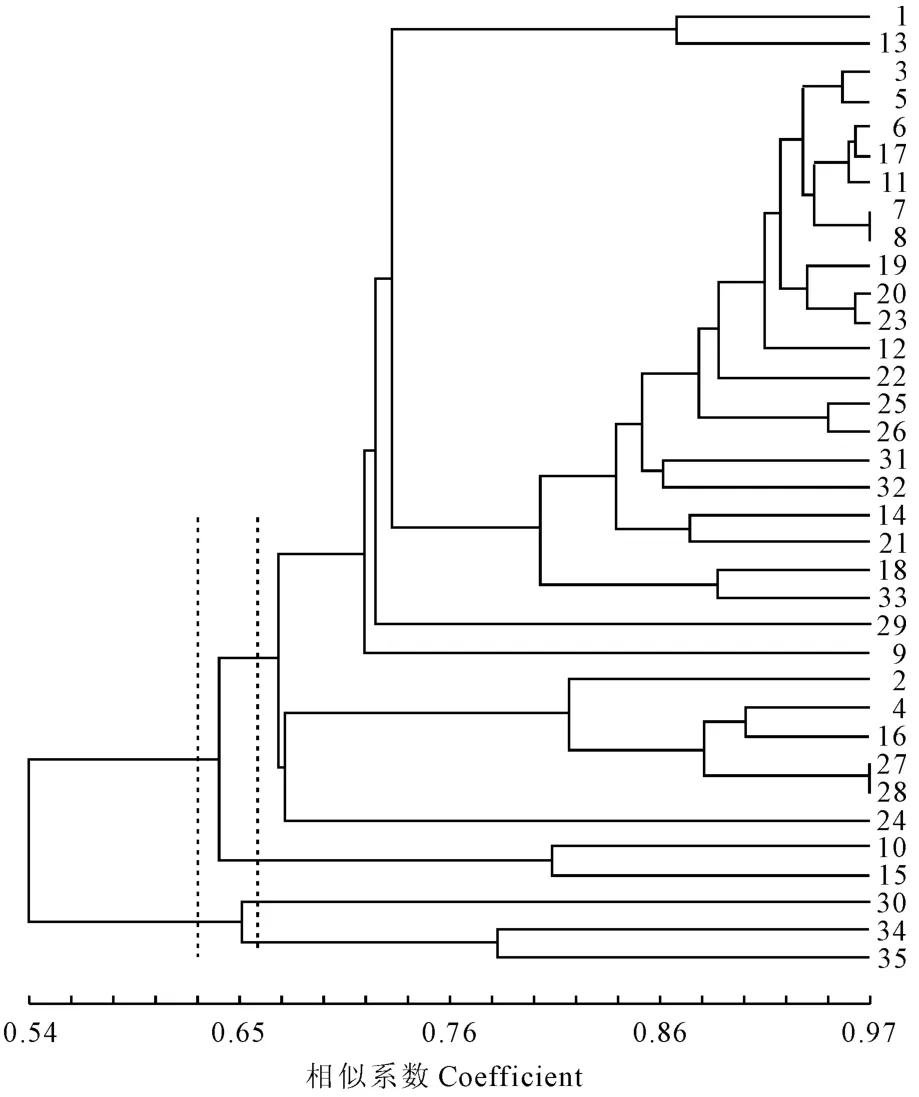
图2 35份山药种质资源的UPGMA 聚类图Fig.2 Dendrogram of 35yam accessions based on UPGMA
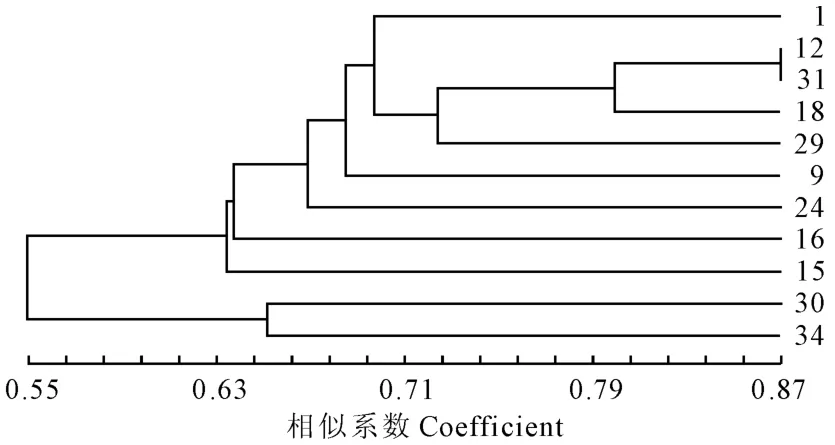
图3 抽样4构建山药核心种质库的ISSR 分子标记聚类图1、9、12、15、16、18、24、29、30、31、34.初级核心种质库的11个山药样品Fig.3 Dendrogram of core collection of yam No.4sampling by ISSR based on UPGMA 1,9,12,15,16,18,24,29,30,31and 34are 11samples of primary core collection of yam
2.4 山药核心种质库的构建及数据分析
对35份山药种质的聚类结果进行最小距离逐步抽样法(LDSS)构建核心种质。共抽样6次,每次抽样构建山药核心种质的遗传多样性如表3。
由表3可以看出,随着抽样种质数目的减少,遗传多样性参数总体上变化较小。有效等位基因数目变化较小,且抽样4的有效等位基因数目最接近抽样前的值。Nei’s基因多样性指数(h)和Shannon’s信息指数(I)均有略增,而分别在抽样5和抽样4中达到最高值。多态性位点百分率呈现下降趋势,在抽样5、6(尤其是抽样6)下降较明显。抽样4所构建的种质库的抽样数是抽样前的31%。综合上述分析,取抽样4为初级核心种质(图3)。
3 讨 论
山药在中国已有2 500多年的栽培历史,许多地区都有栽培,且地区与地区之间相互引种交流较频繁,再加上山药易受周围环境影响,导致部分物种外部形态特征相近、变异范围重叠及珠芽、花、果实、种子等器官很难在同一栽培地区全部形成,因此很难从形态学上鉴定相关物种是否为相同种或类群[17]。
本实验通过12 条ISSR 引物,对所收集的35份山药材料进行PCR 扩增,共扩增出142条清晰的条带,多态性位点比率为97.18%,表明山药种质资源在分子水平上具有丰富的多态性。周延清等[15]用7条ISSR 引物对28份山药品种扩增出的多态性条带比率为83.01%,Shannon’s指数为0.319 1;雷伏贵等[21]采用24 条引物对94份山药材料共扩增出356条ISSR 谱带,其多态性比率为96.6%;华树妹等[22]对34份山药资源进行RAPD 分析,24条引物共扩增出182条谱带,其多态性比例为88.5%。用不同方法对不同山药材料的研究均表明了山药DNA 分子水平上的高度多态性。
本实验35 份山药材料的有效等位基因数(1.427 1)、Nei’s 基 因 多 样 性 指 数(0.269 4)和Shannon’s指数(0.423 0)均表明了35份山药材料具有丰富的遗传多样性。35份山药材料的遗传距离也表明样品间存在遗传差异,如河南铁棍山药-4(5号)与广东大薯(35号)在DNA 分子水平存在很大的差异,而山西侯马山药(7号)与辽宁沈阳菜山药(8号)之间亲缘关系较近。杨明[18]研究表明,形态上河南铁棍山药-4叶片呈浅裂三角形,块茎龙头细长且表皮呈淡黄色;广东大薯叶片呈箭形,块茎无龙头且表皮呈棕红色,它们之间差异很大。而山西侯马山药在地上部和地下部与辽宁沈阳菜山药之间都较相近,本实验结果与形态分类基本相符。
本实验聚类分析中将35份山药种质分为两大类,根据形态特征等来区分,其中广东淮山药(30号)、海南山药(34号)与广东大薯(35号)可归为田薯,其它山药种质可归为普通山药。周延清等[15]用ISSR 聚类将28 个山药品种划分为4 组;雷伏贵等[21]也用聚类分析将94份山药资源分为薯蓣群、参薯群、山薯群、褐苞薯蓣群等4类群,并证明在分子水平上山药是以类群为单位进行分类的。华树妹等[22]通过RAPD 技术将34份资源分为普通山药、田薯、扁山药和福建大薯4类,且当遗传相似系数为0.68时,可将普通山药分为长山药和棒山药2个亚类。与上述研究结果相比,本实验所分类群相对简单,这可能与本实验材料引种区域的局限性有关,但本实验结果与上述研究结果还是相近的。广东淮山药(30号)在所在类群中单独聚为一类,说明这一份种质的基因组与其所携带的遗传信息与其它种质之间的差异较大,并与它们之间的亲缘关系较远。辽宁沈阳菜山药(8号)与山东邹平山药(17号)虽然是在地理距离上相距较远,生态环境不同,却在分子水平上聚为同一类,遗传距离仅为0.050 6,表明这2个来自不同地区的种质资源之间在分子水平上亲缘关系较近,推测可能是由于山药在不同地区之间引种所引起的。山西侯马山药(7号)、山西张绍山药(14号)、河北保定山药(22号)、山东嘉祥细毛山药(31号)这4个山药品种聚为一类,这与陈占勇等[23]对10份山药材料进行花粉形态聚类结果基本一致。
本实验所选的7份河南铁棍山药资源聚在一个亚群,其中河南铁棍-1(4 号)、河南铁棍-5(28 号)、河南铁棍-6(2号)、河南铁棍-7(27号)这4 份河南铁棍山药聚在一起,且它们之间遗传距离0.210 9~0.028 6也印证了这一点。河南铁棍-4(5 号)与河南温县铁棍山药-2(25 号)之间的遗传距离较小为0.127 5,说明两者之间亲缘关系较近。而河南铁棍-3(13号)与其它河南铁棍山药之间的遗传距离均达到了0.3以上,且与河南铁棍-5(28号)之间遗传距离达到0.501 5。这些河南铁棍山药虽来自于河南省,但由于长期以来各地区相互引种,可能造成品种混杂现象,这也证实了当地一些种植户的说法。有关进一步的解释有待今后深入研究。
核心种质的概念是由Frankel在1984 年提出来的,发展至今已有近30年了,核心种质的研究在这30年中已经取得了很大的进展,目前已有很多人开始利用分子标记技术对核心种质库中的种质代表性和遗传多样性进行评价,并且这种方法已成为一种有效和应用广泛的方法[24-25]。
徐海明等[26-27]研究表明,完全随机取样法和分层聚类取样法是构建核心种质的两种重要的取样方法。多数研究者采用后者或把二者结合起来的方法,以保证核心种质的代表性。关于总体取样比例,Brown指出大多数植物资源核心种质取样比例为5%~10%[7],也有学者认为遗传冗余度在0.2~0.9之间时取样比例在20%~30%才合适[28]。李自超等[29]认为各物种适宜的核心种质规模不应简单化而定,对应具体的物种,应按照该物种的遗传多样性来定。本实验对35份山药种质资源进行最小距离逐步抽样法构建核心种质,并对各抽样进行遗传多样性分析,结果表明随着抽取种质数目的减少,各种质库遗传多样性参数变化较小,但多态性位点百分率呈现下降趋势。综合上述分析,确定抽样4为初级核心种质。抽样4构建的种质库的抽样数是抽样前的31%,Nei’s基因多样性指数和Shannon’s信息指数分别为0.328 0和0.495 5,而且多态位点比率和等位基因数均达到抽样前的90%以上,符合核心种质资源代表初始种质资源遗传多样性达到70%~80%的要求[30],表明抽样4所构建的种质库具有较高的代表性。从抽样4的聚类结果中看,其中的山西北洋山药(1号)、河南惠楼小红皮(9号)、河南菜山药(15 号)、山东济南温县铁棒山药(16号)、内蒙古毕克齐长山药(24号)、内蒙古罗家营山药(29号)、广东淮山药(30号)种质分别单独聚类,为较特殊种质,可以在今后的种质资源保存、评价及核心种质库构建的过程中作为重要种质。
[1] 吴国芳,冯志坚,马炜梁,等.植物学(第2版下册)[M].北京:高等教育出版社,1992:352-353.
[2] 裴 鉴.中国植物志(第16卷,第1分册)[M].北京:科学出版社,1985:54-120.
[3] 中国农业科学院蔬菜花卉研究所.中国蔬菜品种志(下卷)[M].北京:中国农业科技出版社,2001:1 054-1 070.
[4] 国家药典委员会.中华人民共和国药典 第1增补本 M.北京 中国医药科技出版社2010.
[5] FRANKEL O H.Genetic manipulation:Impact on man and society[M]//ARBER W,ILLMENSEE R,PEACOCK W J.Genetic perspectives of germplasm conservation.London:Cambridge University Press,1989:161-170.
[6] FRANKEL O H,BROWN A H D.Crop genetic resources:Conservation &Evolution[M]//HOLDEN J H W,WILLIAMS J T,et al.Current plant genetic resources today-A critical appraisal.London:George Allen & Urwin L t d,1984:249-257.
[7] BROWN A H D.Core collections:A practical approach to genetic resources management[J].Genome,1989,31:818-824.
[8] 周延清.DNA 分子标记技术在植物研究中的应用[M].北京:化学工业出版社,2005:9-47.
[9] QIU L J(邱丽娟),LI Y H(李英慧),GUAN R X(关荣霞),et al.Establishment,representative testing and research progress of soybean core collection and mini core collection[J].Acta Agronomica Sinica(作物学报),2009,35(4):571-579(in Chinese).
[10] BAI CH K(白成科),YU J R(俞君如),YU F(于 凤),et al.Genetic diversity and construction of primary core germplasm in Cornus officinalis by ISSR marker[J].Acta Bot.Boreal.-Occident.Sin.(西北植物学报),2009,29(12):2 401-2 407(in Chinese).
[11] ZHOU Y Q(周延清),JING J ZH(景建洲),JIA J F(贾敬芬),et al.ISSR identification of genetic diversity of Rehmannia glutinosain Huai zone[J].Chinese Traditional and Herbal Drugs(中草药),2005,36(2):257-261(in Chinese).
[12] WU H F(吴慧芳),LI Z ZH(李作洲),HUANG H W(黄宏文).Genetic differentiation among natural populations of Gastrodia elata(Orchidaceae)in Hubei and germplasm assessment of the cultivated populations[J].Biodiversity Science(生物多样性),2006,14(4):315-326(in Chinese).
[13] LI H F(李慧峰),LU S Q(卢森权),LI Y Q(李彦青),et al.Preliminary study on constructing core collection of sweet potato germplasm in Guangxi[J].Guangxi Agricultural Sciences(广西农业科学),2010,41(7):732-735(in Chinese).
[14] QIAN W(钱 韦),GE S(葛 颂),HONG D Y(洪德元).Assessment of genetic variation of Oryza granulate detected by RAPDs and ISSRs[J].Acta Botanica Sinica(植物学报),2000,42(7):741-750(in Chinese).
[15] ZHOU Y Q(周延清),JING J ZH(景建洲),LI ZH Y(李振勇),et al.Genetic diversity of yam(Dioscorea opposita Thunb.)detected by ISSR markers[J].Acta Biologiae Experimentalis Sinica(实验生物学报),2005,38(4):324-329(in Chinese).
[16] HU J B(胡建斌),LI J(李 静),LI Q(李 琼),et al.Establishment and optimization of ISSR-PCR amplification Dioscorea zingiberensis C.H.Wright[J].Journal of Huazhong Agricultural University(华中农业大学学报),2008,27(1):105-109(in Chinese).
[17] 黄玉仙.山药种质资源研究[D].福州:福建农林大学,2012.
[18] 杨 明.山药种质资源遗传多样性的研究[D].呼和浩特:内蒙古农业大学,2012.
[19] ROHL F F J.NTSYSpc:Numerical taxonomy and multivariate analysis system,Version2.1[CP].New York:Exeter Software,2000.
[20] HU J(胡 晋),XU H M(徐海明),ZHU J(朱 军).Constructing core collection of crop germplasm by multiple clusters based on genotypic values[J].Journal of Biomathematics(生物数学学报),2000,15(1):103-109(in Chinese).
[21] LEI F G(雷伏贵),HUA SH M(华树妹),TU Q CH(涂前程),et al.Genetic relationship of Dioscorea polystachya Turcz.resources based on ISSR markers[J].Fujian Journal of Agricultural Sciences(福建农业学报),2013,28(1):27-32(in Chinese).
[22] HUA SH M(华树妹),TU Q CH(涂前程),LEI F G(雷伏贵).Genetic diversity of Dioscorea polystachya Turcz.revealed by RAPD markers[J].Journal of Plant Genetic Resources(植物遗传资源学报),2009,10(2):195-200(in Chinese).
[23] CHEN ZH Y(陈占勇),HUO X W(霍秀文),YIN CH(尹 春),et al.Study of classification of different Chinese yam(Dioscorea opposita Thunb.)by pollen morphology[J].Acta Agriculturae Boreali-Sinica(华北农学报),2011,26(5):141-145(in Chinese).
[24] FUKUNAGA K,HILL J,VIGOUROUX Y,et al.Genetic diversity and population structure of teosinte[J].Genetics Society of America,2005,169(4):2 241-2 254.
[25] ESCRIBANO P,VIRULE M A,HORMAZA J I.Comparison of different methods to construct a core germplasm collection in woody perennial species with simple sequence repeat markers.A case study in cherimoya(Annona cherimola,Annonaceae),an underutilized subtropical fruit tree species[J].Annals of Applied Biology,2008,153(1):25-32.
[26] XU H M(徐海明),HU J(胡 晋),ZHU J(朱 军).An efficient method of sampling core collection from crop germplasm[J].Acta Agronomica Sinica(作物学报),2000,26(2):157-162(in Chinese).
[27] XU H M(徐海明),HU J(胡 晋),et al.Methods of constructing core collection of crop germplasm by comparing different genetic distances,cluster methods and sampling strategies[J].Acta Agronomica Sinica(作物学报),2004,30(9):932-936(in Chinese).
[28] SHASHIDHARA G,HEMA M V,KOSHY B,et al.Assessment of genetic diversity and identification of core collection in sandalwood germplasm using RAPDs[J].The Journal of Horticultural Science &Biotechnology,2003,78(4):528-536.
[29] LI Z CH(李自超),ZHANG H L(张洪亮),SUN CH Q(孙传 清),et al.Status and prospects of core collection in plant germplasm resource[J].Journal of China Agricultural University(中国农业大学学报),1999,4(5):51-62(in Chinese).
[30] GU X F,YANG A F,MENG H,et al.In vitro induction of tetraploid plants from diploid Zizyphus jujuba Mill.cv.Zhanhua[J].Plant Cell Reports,2005,24(11):671-676.
