花生中巨大芽孢杆菌对黄曲霉毒素合成相关基因的抑制
2015-07-05孔青迟晨单世华李琦玉
孔青, 迟晨, 单世华, 李琦玉
(1.中国海洋大学食品科学与工程学院,山东 青岛266003;2.山东省花生研究所,山东 青岛 266100)
花生中巨大芽孢杆菌对黄曲霉毒素合成相关基因的抑制
孔青1*, 迟晨1, 单世华2, 李琦玉1
(1.中国海洋大学食品科学与工程学院,山东 青岛266003;2.山东省花生研究所,山东 青岛 266100)
分别在UF 715133-1和Jinhua 1012花生仁上接种黄曲霉和巨大芽孢杆菌,高压液相色谱法测定花生中黄曲霉毒素含量,借助生物芯片和实时定量聚合酶链式反应(quantitative real-time polymerase chain reaction, qRT-PCR)测定巨大芽孢杆菌对黄曲霉毒素生物合成途径基因表达的影响,并用基因本体(gene ontology, GO)功能分类和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)通路方法对基因进行分类分析。在实验组Jinhua 1012 和UF 715133-1中,黄曲霉毒素B1的含量降低了85%以上,一些编码蛋白质酶的基因显著下调,如短链脱氢酶、非核糖体多肽合成酶等。结果表明,从海洋中分离出的巨大芽孢杆菌可显著抑制黄曲霉毒素在花生上的合成,黄曲霉中多种基因的表达受到抑制,但这些基因在黄曲霉毒素生物合成中的作用并未阐明,其作用机制还有待进一步深入研究。
巨大芽孢杆菌; 黄曲霉毒素; 抑制; 基因表达; 花生
花生和玉米是最容易受黄曲霉毒素污染的农产品[4]。为了减少人类接触黄曲霉毒素的可能,已经采取多种措施来控制黄曲霉的生长、抑制黄曲霉毒素的形成[5]。物理方法和化学降解的方法由于处理不完全、成本比较高等原因不被大众接受[6]。而采用拮抗微生物或天然提取物的生物方法是一种高效率、低危险的去除毒素方法[7]。Brown等[8]、Cotty等[9]测试了在玉米,棉籽、花生等作物中,培养不产毒素黄曲霉菌株来抑制产毒菌株的生长。木霉属真菌也可作为某些植物病原真菌的生物拮抗剂,包括生物防治黄曲霉[10]。已有研究表明,一些细菌可以降解黄曲霉毒素,如黄杆菌属NRRL b-184能在受污染的作物中去除黄曲霉毒素[11];从加州杏仁样品和玉米土壤分离出的杆菌、假单胞菌、伯克氏菌株、青枯菌,可以完全抑制黄曲霉生长和黄曲霉毒素合成[12-13]。放线菌也可以降解黄曲霉毒素,从分枝杆菌提取的2种F420H2-依赖性还原酶,可以催化降解黄曲霉毒素[14]。
前期研究[15]表明,海洋巨大芽孢杆菌可以抑制黄曲霉的生长和黄曲霉毒素B1的生物合成,还需要进一步的研究巨大芽孢杆菌抑制黄曲霉毒素生物合成的机制。生物芯片技术已经成为检查成千上万个基因表达的好方法,这种方法已经广泛应用于不同温度下,黄曲霉毒素的生物合成中的转录规律[16-17]。这些研究发现,在不同的温度条件下,黄曲霉不同基因的表达水平是不一样的。但是,生物拮抗剂对花生上黄曲霉全基因组中各基因表达的影响还未见报道。在本研究中,我们首次利用基因芯片的方法,研究巨大芽孢杆菌在花生中对黄曲霉毒素生物合成途径的抑制作用。
1 材料与方法
1.1 实验材料
1.1.1 菌种与培养基 海洋巨大芽孢杆菌分离自黄海东部,利用形态、生理生化、16S rRNA的方法对其菌种鉴定[18]。巨大芽孢杆菌培养在牛肉膏蛋白胨肉汤培养基(beef extract and peptone broth,BEPB)(牛肉膏3 g,蛋白胨10 g,氯化钠5 g,蒸馏水1 L)中,200 r/min在37 ℃培养24 h后,8 000 r/min离心45 s收集菌体,无菌蒸馏水洗2次去掉残留培养基。细菌用平板菌落计数法对水样中的细菌计数,然后用无菌蒸馏水稀释到所需的含量。
病原菌黄曲霉NRRL3357保存在马铃薯葡萄糖琼脂培养基(potato dextrose agar,PDA)(200 g土豆煮后的滤液,葡萄糖20 g,琼脂20 g,蒸馏水1 L)。用0.1% Tween-80溶液洗涤黄曲霉PDA培养基,过滤制成孢子悬浮液。孢子用血球计数器计数,然后用0.1% Tween-80溶液稀释。
1.1.2 花生仁 花生品种UF 715133-1和Jinhua 1012由山东省花生研究所提供。UF 715133-1是一种抗黄曲霉侵染的花生品种,而Jinhua 1012是易被黄曲霉侵染的花生品种。处理前,花生仁用自来水清洗,然后用0.1%次氯酸钠消毒表面1 min,打孔前用自来水清洗和风干。
粗颗粒的冻融循环试验不仅与试验时的含水率有关,还与颗粒的级配有关。袁俊平等[21]认为粗粒土冻融变形量受其颗粒粒径大小的影响,如图5所示。颗粒粒径大小影响水分充满孔隙的多少和水分的迁移通道,以及影响冻融过程中颗粒排列和孔隙分布。试样冻胀变形量随颗粒粒径增大而逐渐减小; 而冻胀融沉后试样总变形率随限制粒径呈现先增大再减小趋势。此外,冻融循环还可以使土的颗粒级配发生变化[4]。
1.1.3 主要试剂和仪器 冷冻离心机3-18R(美国TOMOS公司);人工气候箱(QHX-300BS-Ⅲ,上海新苗医疗器械有限公司);黄曲霉毒素B1免疫亲和柱(AflaT-est,美国VICAM公司);安捷伦1100系列高效液相色谱仪(美国Agilent公司);Zorbax C18色谱柱(150 mm×4.6 mm,5 μm,美国Agilent公司);黄曲霉全基因组“8×15 K”基因芯片(美国Agilent公司);RNeasy mini kit(美国Invitrogen公司);荧光染料Cy3-dCTP(美国GE Healthcare公司);基因芯片扫描仪G2505C(美国Agilent公司);SYBR PremixExTaqkit(大连宝生物工程有限公司);ABI实时PCR系统(美国Applied Biosystems公司)。
1.2 实验方法
1.2.1 在花生上接种海洋巨大芽孢杆菌和黄曲霉
花生仁使用无菌打孔器打孔(直径6 mm,约3 mm深),然后分别在已打孔的花生仁上接种20 μL的巨大芽孢杆菌(108CFU/mL)。接种细菌2 h后,将10 μL的黄曲霉孢子悬浮液(106个/mL)接种到每个孔中。花生仁置于人工气候箱,在28 ℃高湿度(85%)培养7 d。每处理重复3次,每个测试20粒花生仁。在第7天收集花生仁上的菌丝,立即在液氮下冷冻,磨成粉,储存在-80℃。
1.2.2 黄曲霉毒素的提取和HPLC分析 从10 g花生提取黄曲霉毒素,用免疫亲和柱纯化,三氟乙酸衍生,使用安捷伦1100系列高效液相色谱仪测定,荧光检测器激发波长360 nm、发射波长440 nm,色谱柱为Zorbax C18(150 mm×4.6 mm,5 μm)。流动相为V(甲醇)∶V(水)∶V(乙腈)=50∶40∶10,流速为0.8 mL/min,检测时间为15 min。
1.2.3 基因芯片 黄曲霉全基因组芯片(8×15 K)上至少用1个60-mer寡核苷酸探针为每个黄曲霉基因测试,寡核苷酸探针和基因芯片由美国安捷伦公司设计并制造。
1.2.4 RNA分离和基因芯片分析 每个样本用总RNA抽提试剂盒提取总RNA,并根据说明用试剂盒(RNeasy mini Kit)进一步纯化。总RNA模板通过分光光度法进行量化,并用1.0%甲醛变性琼脂糖凝胶电泳对其检测。
cDNA逆转录于5 μg的RNA,然后根据制造商的说明加上荧光染料(Cy3-dCTP)。在80 μL杂交溶液中进行杂交,杂交液含3×SSC(氯化钠柠檬酸钠缓冲液,saline sodium citrate),0.2%十二烷基硫酸钠(sodium dodecyl sulfate,SDS)、5×Denhardt的溶液和25%甲酰胺,然后在65 ℃过夜培养。在杂交后,切片用洗涤溶液清洗(0.2% SDS,分别加2×SSC和2×SSC),在37 ℃放置5 min。芯片用软件扫描(Agilent G2505C Scanner),图像用GeneSpring软件(Agilent Technologies Co., Ltd.)进行分析。用Cluster 3.0软件进行平均信号强度分析。利用BlastX来搜索同源物和基因本体(gene ontology,GO)功能分类[19],GO使用BGI-WEGO网络服务(http://wego.genomics.org.cn)进行分类。log2比率(log2ratio)用来衡量相对表达水平的变化,如果log2>2或log2<-2,认为基因表达有差异;如果log2比率>5或<-5,则认为基因表达差异显著。
1.2.5 途径分析 由于对黄曲霉合成途径的信息了解有限,所有基因的注释使用酿酒酵母(Saccharomycescerevisiae)作为参考。使用京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路方法(http://www.genome.jp/kegg/)对基因进行分类和分析。
1.2.6 定量RT-PCR 使用实时定量聚合酶链式反应(quantitative real-time polymerase chain reaction,qRT-PCR)确认基因芯片数据。序列的引物合成于上海生工生物工程有限公司(表1),β-微管蛋白(β-tubulin)基因作为管家基因。使用生物染料荧光定量试剂盒(SYBR PremixExTaqkit)完成qRT-PCR,每个反应制备液25 μL,包含互补DNA 2 μL,SYBR预混料Taq试剂12.5 μL,10 mmol/L引物0.5 μL(上游与下游)。根据说明来进行PCR反应:在95 ℃ 预变性2 min,94 ℃变性10 s,60 ℃退火40 s,60 ℃延伸40 s,反应运行了40个周期。qRT-PCR重复3次。
1.2.7 统计分析 用统计软件SAS 8.0对所有的数据进行统计,显著水平设置在P=0.05。基因芯片结果已提交到美国NCBI的GEO数据库(登录号:GSE51469)。
2 结果与分析
2.1 在花生Jinhua 1012和UF 715133-1中黄曲霉基因表达的差异
在黄曲霉基因组已知的13 487个基因中,Jinhua 1012实验组上的黄曲霉有817个基因表达有差异,453个基因表达水平上调,而364基因表达水平下调;其中13个基因表达显著上调,而7个基因表达显著下调。在Jinhua 1012对照组和实验组中黄曲霉毒素B1的质量比分别为(1 683.60±75.27) μg/kg和(206.15±19.18) μg/kg(表2)。
类似的结果在UF 715133-1中出现。用海洋巨大芽孢杆菌处理后,黄曲霉中201个基因表达有差异,其中125个基因表达上调,而76个基因表达下调;其中2个基因表达显著上调,1个基因表达显著下调。实验组和对照组黄曲霉毒素B1质量比分别为(149.45±12.87) μg/kg和(1 040.79±41.62) μg/kg(表2)。经GO分析,大多数表达差异的基因参与运输(36.2%)和代谢过程(28.6%)(图1)。
2.2 京都基因与基因组百科全书(KEGG)注释和分析
KEGG途径分析Jinhua 1与Jinhua 2、UF 1与UF 2表明,大部分表达差异的基因参与次生代谢物(占30%以上)生物合成和代谢途径(占80%以上)(图2)。

表2 不同方法处理后黄曲霉毒素B1的质量比

图1 基因本体分析表达差异基因参与的生物过程Fig.1 Gene ontology analysis of the differentially expressed genes in biological process
2.3 qRT-PCR分析
laeA、veA、aflR、aflS和rolA的表达由qRT-PCR进行验证(图3)。相比Jinhua 1012对照组(Jinhua 2),实验组Jinhua 1012(Jinhua 1)的基因rolA、laeA、veA、aflR和aflS表达未达到显著差异水平(P>0.05)。相似的结果在花生UF 715133-1中出现,与对照组UF 715133-1(UF 2)比较,实验组UF 715133-1(UF 1)中基因aflR表达接近差异显著,而基因rolA、laeA、veA和aflS的表达差异不显著(P>0.05)。
3 讨论
Jinhua 1012和UF 715133-1上的研究结果表明,经海洋巨大芽孢杆菌处理,黄曲霉毒素生物合成基因簇中各基因表达差异不明显(表3)。相应地,测定实验组Jinhua 1012和UF 715133-1的黄曲霉毒素B1质量比,黄曲霉毒素B1的质量比降低了85%以上(表2)。花生并不是海洋巨大芽孢杆菌生长的最优培养基,而实验条件(28℃,相对湿度85%)是黄曲霉毒素生物合成的最优条件,这是花生中巨大芽孢杆菌对黄曲霉毒素生物合成基因簇中基因表达抑制作用较低的主要原因。因此我们推断,黄曲霉毒素的生物合成不仅受到毒素生物合成基因簇中基因的调控,还受到其他一些基因的调控。
相比UF 715133-1对照组,Jinhua 1012对照组更多的基因表达是上调的,在Jinhua 1012对照组产生黄曲霉毒素B1比UF 715133-1对照组多(表2)。此外,Jinhua 1012经海洋巨大芽孢杆菌处理,一些黄曲霉素生物合成途径的基因表达上调;UF 715133-1经海洋巨大芽孢杆菌处理,黄曲霉毒素生物合成途径没有基因表达上调(表3)。相应地,黄曲霉毒素B1质量比在Jinhua 1012显著高于UF 715133-1(表2)。

图2 京都基因与基因组百科全书分析表达差异基因参与的生物过程Fig.2 Kyoto encyclopedia of genes and genomes (KEGG) analysis of the differentially expressed genes in biological process
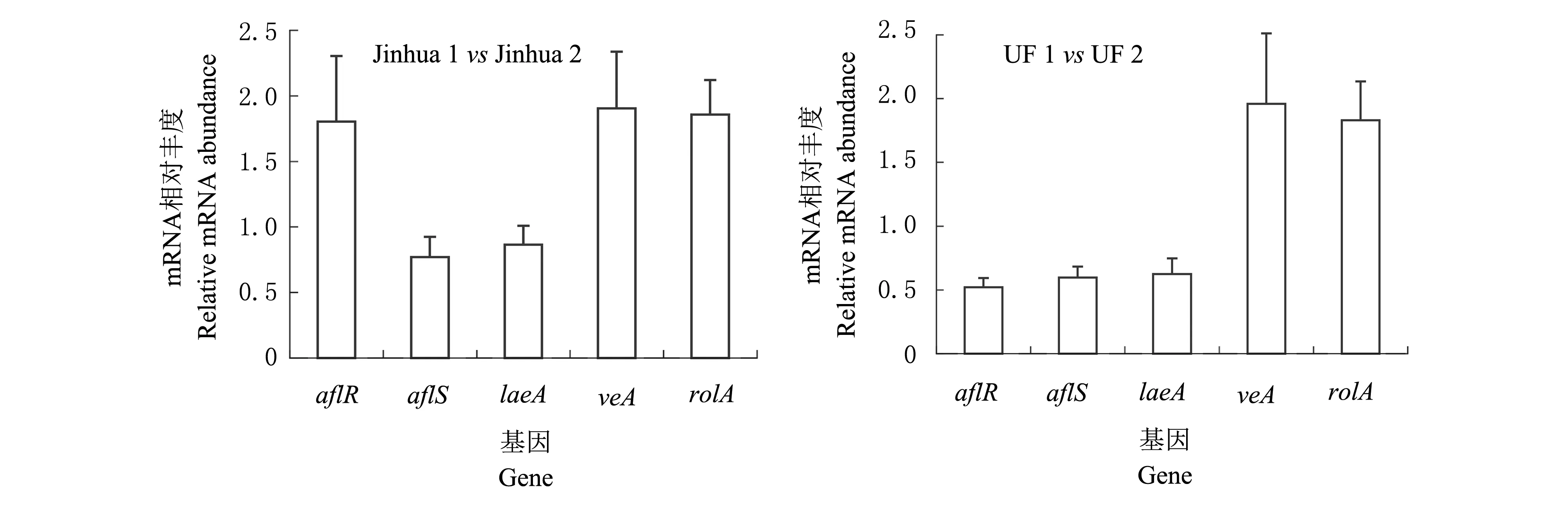
图3 qRT-PCR 证明aflR,aflS,laeA,veA和rolA的基因表达Fig.3 qRT-PCR analysis of the expression of aflR, aflS, laeA, veA and rolA
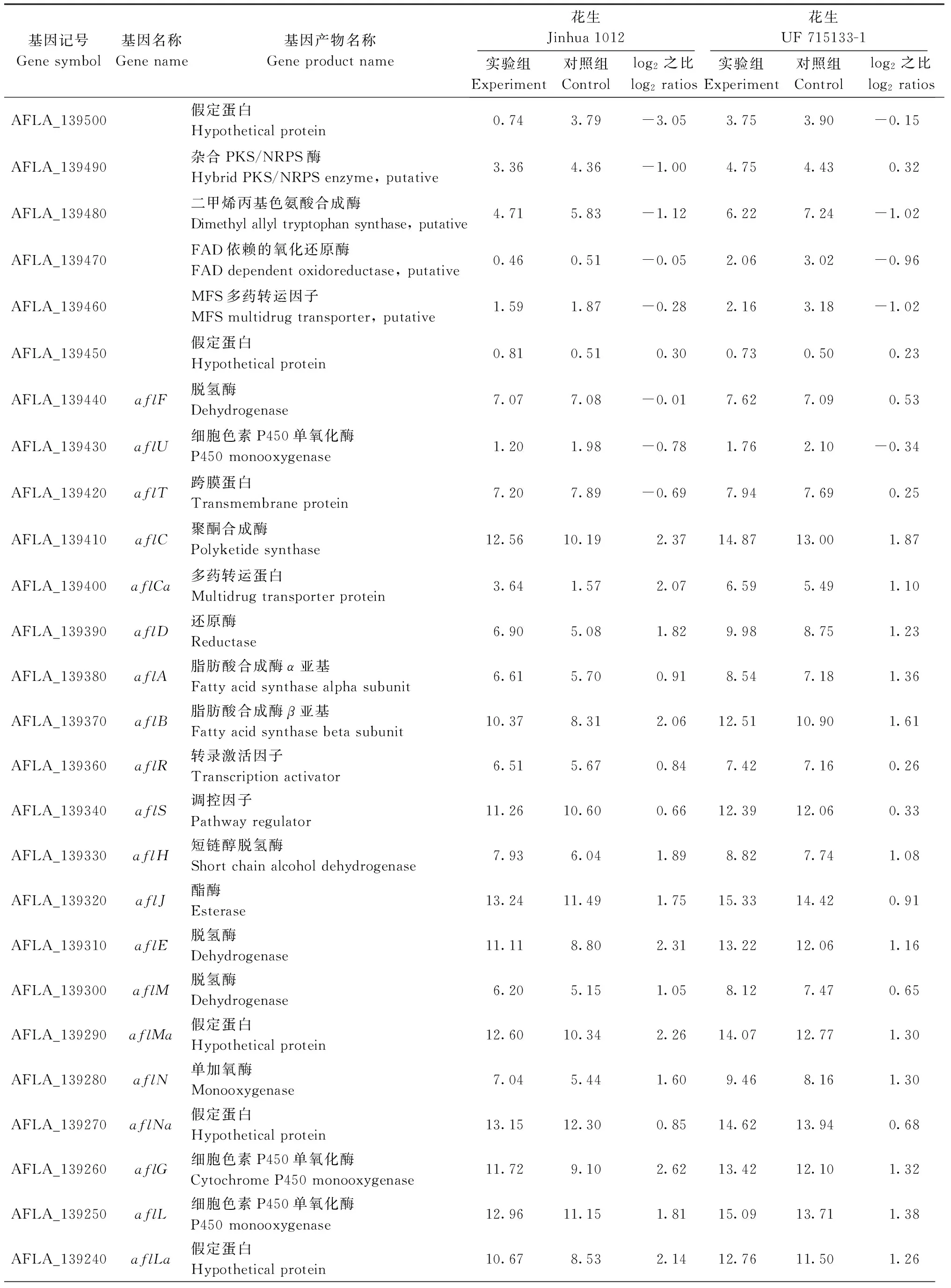
表3 巨大芽孢杆菌影响黄曲霉毒素生物合成基因和laeA与veA的表达

续表3 巨大芽孢杆菌影响黄曲霉毒素生物合成基因和laeA与veA的表达
这个结果与UF 715133-1是抗黄曲霉品种而Jinhua 1012是易感黄曲霉品种相吻合。
基因芯片数据显示海洋巨大芽孢杆菌也可抑制某些参与环匹阿尼酸(cyclopiazonic acid,CPA)生物合成的基因表达(表4)。CPA的产生总是与霉菌毒素的产生特别是黄曲霉毒素的产生有关[20]。我们推断海洋巨大芽孢杆菌也可抑制环匹阿尼酸的产生。
除了黄曲霉毒素生物合成基因簇的基因(表3),此项研究还发现了一些有效控制黄曲霉毒素生物合成的目标基因,如非核糖体多肽合成酶(NRPSs)、短链脱氢酶和翻译延伸因子(EF-1)。非核糖体肽合成酶具有多种对微生物生存、生长繁殖等所必需的生理功能,比如可作为调节生长、繁殖和分化的信号分子等[21];短链脱氢酶是一类NADPH依赖型氧化还原酶家族,具有相似的序列模型及催化机制,短链脱氢酶在脂质、碳水化合物、氨基酸、辅酶和异物的代谢中具有关键性作用;翻译延伸因子是一个主要的翻译因子,它不仅仅是翻译必需的蛋白质,而且是一个重要的多功能蛋白质,参与许多重要的细胞过程和疾病,包括信号传导、翻译控制、凋亡、细胞骨架组成等[22]。
此外,AFLA_063980,AFLA_059490,AFLA_073880,AFLA_116480,AFLA_000910,AFLA_117420,AFLA_042140,AFLA_064240,AFLA_114330,AFLA_039240,AFLA_082260,AFLA_075300,AFLA_136890,AFLA_075950,AFLA_107060,AFLA_104430,AFLA_040740,AFLA_110190,AFLA_035900,AFLA_008770,AFLA_110170等基因通过海洋巨大芽孢杆菌处理,在UF 715133-1实验组和Jinhua 1012实验组都显著下调(表5),可以推断这些基因直接或间接影响黄曲霉毒素的生物合成(表2)。目前,这些基因的作用是未知的,进一步的研究将增加我们对控制黄曲霉毒素生物合成的理解。

表4 巨大芽孢杆菌影响环匹阿尼酸生物合成基因的表达

表5 海洋巨大芽孢杆菌显著抑制黄曲霉中表达差异的基因(前20个基因)
[1] Yu J J, Fedorova N D, Montalbano B G,etal. Tight control of mycotoxin biosynthesis gene expression inAspergillusflavusby temperature as revealed by RNA-Seq.FEMSMicrobiologyLetters, 2011,322(2):145-149.
[2] Calvo A M, Wilson R A, Bok J W,etal. Relationship between secondary metabolism and fungal development.MicrobiologyandMolecularBiologyReviews, 2002,66(3):447-459.
[3] Commission Regulation (EU) No 165/2010. Amending Regulation (EC) No 1881/2006 setting maximum levels for certain contaminants in foodstuffs as regards aflatoxins.OfficialJournalofEuropeanUnion, 2010,53:8-12.
[4] Amaike S, Keller N P.Aspergillusflavus.AnnualReviewofPhytopathology, 2011,49:107-133.
[5] Chang P K, Horn B W, Dorner J W. Sequence breakpoints in the aflatoxin biosynthesis gene cluster and flanking regions in non-aflatoxigenicAspergillusflavusisolates.FungalGeneticsandBiology, 2005,42(11):914-923
[6] Sindhu S, Chempakam B, Leela N K,etal. Chemoprevention by essential oil of turmeric leaves (CurcumalongaL.) on the growth ofAspergillusflavusand aflatoxin production.FoodandChemicalToxicology, 2011,49:1188-1192.
[7] Reddy K R N, Reddy C S, Muralidharan K. Potential of botanicals and biocontrol agents on growth and aflatoxin production byAspergillusflavusinfecting rice grains.FoodControl, 2009,20:173-178.
[8] Brown R L, Cotty P J, Cleveland T E. Reduction in aflatoxin content of maize by atoxigenic strains ofAspergillusflavus.JournalofFoodProtection, 1991,54:623-626.
[9] Cotty P J. Influence of field application of an atoxigenic strain ofAspergillusflavuson the populations ofA.flavusinfecting cotton bolls and on the aflatoxin content of cottonseed.Phytopathology, 1994,84:1270-1277.
[10] Papavizas G C.TrichodermaandGlicladium: Biology, ecology and potential for biocontrol.AnnualReviewofPhytopathology, 1985,23:23-54.
[11] Ciegler A, Lillehoj E B, Peterson R E,etal. Microbial detoxification of aflatoxin.AppliedMicrobiology, 1966,14:934-939.
[12] Nesci A V, Bluma R V, Etcheverry M G.Invitroselection of maize rhizobacteria to study potential biological control ofAspergillussection Flavi and aflatoxin production.EuropeanJournalofPlantPathology, 2005,113:159-171.
[13] Palumbo J D, Baker J L, Mahoney N E. Isolation of bacterial antagonists ofAspergillusflavusfrom almonds.MicrobialEcology, 2006,52:45-52.
[14] Taylor M C, Jackson C J, Tattersall D B,etal. Identification and characterization of two families of F420H2-dependent reductases fromMycobacteriathat catalyse aflatoxin degradation.MolecularMicrobiology, 2010,78:561-575.
[15] Kong Q, Shan S H, Liu Q Z,etal. Biocontrol ofAspergillusflavuson peanut kernels by use of a strain of marineBacillusmegaterium.InternationalJournalofFoodMicrobiology, 2010,139:31-35.
[16] O’Brian G R, Georgianna D R, Wilkinson J R,etal. The effect of elevated temperature on gene transcription and aflatoxin biosynthesis.Mycologia, 2007,99(2):232-239.
[17] Georgianna D R, Fedorova N D, Burroughs J L,etal. Beyond aflatoxin: Four distinct expression patterns and functional roles associated withAspergillusflavussecondary metabolism gene clusters.MolecularPlantPathology, 2010,11(2):213-226.
[18] Olofsson T C, Ahrné S, Molin G. Composition of the bacterial population of refrigerated beef, identified with direct 16S rRNA gene analysis and pure culture technique.InternationalJournalofFoodMicrobiology, 2007,118:233-240.
[19] Ashburner M, Ball C A, Blake J A,etal. Gene ontology: tool for the unification of biology.NatureGenetics, 2000,25(1):25-29.
[20] Horn B W, Dorner J W. Regional differences in production of aflatoxin B1and cyclopiazonic acid by soil isolates ofAspergillusflavusalong a transect within the United States.AppliedandEnvironmentalMicrobiology, 1999,65:1444-1449.
[21] Grunewald J, Marahiel M A. Chemoenzymatic and template-directed synthesis of bioactive macrocyclic peptides.MicrobiologyandMolecularBiologyReviews, 2006,70(1):121-146.
[22] 周冰,曹诚,刘传暄.翻译延伸因子1A的研究进展.生物技术通讯,2007,18(2):281-284. Zhou B, Cao C, Liu C X. Advances in research on translation elongation factor 1 alpha.LettersinBiotechnology, 2007,18(2):281-284. (in Chinese with English abstract)
Inhibitory effect ofBacillusmegateriumon aflatoxin biosynthetic pathway genes expression ofAspergillusflavusin peanuts.
Journal of Zhejiang University (Agric. & Life Sci.), 2015,41(5):567-576
Kong Qing1*, Chi Chen1, Shan Shihua2, Li Qiyu1
(1.SchoolofFoodScienceandEngineering,OceanUniversityofChina,Qingdao266003,Shandong,China; 2.ShandongPeanutInstitute,Qingdao266100,Shandong,China)
Previous studies in our laboratory demonstrated that a marineBacillusmegateriumcould inhibit the growth ofAspergillusflavusand turn off the biosynthesis of aflatoxin B1. In order to investigate the mechanisms of inhibition byB.megateriumonA.flavusgrowth and aflatoxin production in peanuts and to evaluate the possible application ofB.megateriumas a biocontrol agent, genes expression analysis using wholeA.flavusgenome gene chip whenA.flavuswas co-cultured withB.megateriumin peanuts were performed.
The aim of this research was to study the inhibitory effect ofB.megateriumon aflatoxin biosynthetic pathway genes expression inA.flavus. The effects ofB.megateriumon aflatoxin biosynthesis and genes expression inA.flavuswere tested in two types of peanut varieties, UF 715133-1 and Jinhua 1012 by the aid of high performance liquid chromatography (HPLC), gene chip and quantitative real-time polymerase chain reaction (qRT-PCR). UF 715133-1 is a kind of peanut that could resist toA.flavusinvasion, while Jinhua 1012 is susceptible to the infection ofA.flavus.
The results showed that though more than 85% of aflatoxin B1was inhibited by the treatment ofB.megateriumin UF 715133-1 and Jinhua 1012, no important aflatoxin biosynthetic pathway gene was differentially expressed. Peanuts unsuitable for the growth ofB.megateriumwas the main reason forB.megateriumunthoroughly inhibit the biosynthesis of aflatoxin like in minimal medium (MM) or potato dextrose broth (PDB). Furthermore, the experimental condition (28 ℃, relative humidity 85%), which is the optimal condition for aflatoxin biosynthesis, wasn’t suitable for the growth ofB.megateriumneither. So we inferred thatB.megateriuminhibited aflatoxin biosynthesis by inhibiting the growth ofA.flavusin peanuts, and the secondary metabolites inB.megateriumaccounted for the inhibition. Furthermore, the results showed some coding protease genes were highly downregulated, such as short-chain dehydrogenase, and non-ribosomal peptide synthetase, which may relate to aflatoxin biosynthesis.
In conclusion, this strain of marineB.megateriumisolated from the Yellow Sea of East China could significantly inhibit the aflatoxin biosynthesis in peanuts through inhibiting genes expression inA.flavus. While the role of most highly downregulated genes in aflatoxin biosynthesis hasn’t been clarified, the mechanism remains to be further studied.
Bacillusmegaterium; aflatoxin; inhibitory; genes expression; peanut
国家自然科学基金(31471657;31000823)。
2015-03-06;接受日期(Accepted):2015-06-17;网络出版日期(Published online):2015-09-18
S 565.2; Q 939.92
A
*通信作者(Corresponding author):孔青(http://orcid.org/0000-0002-5147-7832),Tel:+86-532-82031851;E-mail:kongqing@ouc.edu.cn
URL:http://www.cnki.net/kcms/detail/33.1247.s.20150918.1800.016.html
