无籽刺梨杂交起源:来自分子数据的证据
2015-06-24邓亨宁高信芬李先源周洪英
邓亨宁, 高信芬,①, 李先源, 周洪英
(1. 中国科学院成都生物研究所: a. 中国科学院山地生态恢复与生物资源利用重点实验室, b. 生态恢复与生物多样性保育四川省重点实验室, 四川 成都 610041; 2. 西南大学园艺园林学院 重庆市花卉工程技术研究中心, 重庆 400716; 3. 贵州省植物园, 贵州 贵阳 550004)
无籽刺梨杂交起源:来自分子数据的证据
邓亨宁1a,1b, 高信芬1a,1b,①, 李先源2, 周洪英3
(1. 中国科学院成都生物研究所: a. 中国科学院山地生态恢复与生物资源利用重点实验室, b. 生态恢复与生物多样性保育四川省重点实验室, 四川 成都 610041; 2. 西南大学园艺园林学院 重庆市花卉工程技术研究中心, 重庆 400716; 3. 贵州省植物园, 贵州 贵阳 550004)
为追溯无籽刺梨(Rosa×sterilisS. D. Shi)的物种起源,以该种及同域分布的蔷薇属(RosaLinn.)14种、2变种和2变型为实验材料,并以小叶金露梅(PotentillaparvifoliaFisch.)为外类群,选取5个叶绿体基因片段(psbA-trnH、trnL-trnF、trnK-matK、psbI-psbK和rpoC1)及2个核基因片段(ITS和GAPDH)进行扩增和测序,并据此利用贝叶斯分析(BI)和最大似然法(ML)进行系统发育重建。结果显示:5个叶绿体基因片段联合组成1个全长3 365 bp的数据集,在据此构建的BI系统树中,无籽刺梨与长尖叶蔷薇(R.longicuspisBertol.)聚在一起,后检验率(PP)为0.96、靴带支持率(LB)为81%。43条ITS序列组成1个全长617 bp的数据集,在据此构建的BI系统树中,无籽刺梨的4个单倍型与缫丝花(R.roxburghiiTratt.)、白花刺梨(R.roxburghiif.candidaS. D. Shi)和无刺刺梨(R.roxburghiif.inermisS. D. Shi)构成分支a,PP值为1.00、LB值为98%;无籽刺梨的另外3个单倍型与软条七蔷薇(R.henryiBouleng.)及长尖叶蔷薇的2个单倍型共同构成分支b,PP值为0.93、LB值为85%。63条GAPDH基因序列组成1个全长883 bp的数据集,在据此构建的BI系统树中,无籽刺梨单倍型H2与长尖叶蔷薇的2个单倍型聚为分支A,PP值为1.00、LB值为99%;无籽刺梨单倍型H1和H3与缫丝花、白花刺梨和无刺刺梨聚为分支B,PP值为1.00、LB值为100%;无籽刺梨单倍型H4单独聚为一支。综合分析结果表明:无籽刺梨起源于长尖叶蔷薇与缫丝花的天然杂交,长尖叶蔷薇和缫丝花分别为其母本和父本。
无籽刺梨; 物种形成; 核基因; 叶绿体基因; 杂交亲本; 系统发育树
无籽刺梨(Rosa×sterilisS. D. Shi)又名无子刺梨、搭钩刺梨或金刺梨,为蔷薇科(Rosaceae)蔷薇属(RosaLinn.)攀缘灌木,是贵州省特有种,仅集中分布于黔西南的兴仁和安顺地区,1985年时圣德将其作为新分类群予以发表[1]。无籽刺梨果实富含VC及多种有益成分[2],在医药及保健方面具有一定的开发潜力,经济价值较高,受到学术界及相关企业的高度关注,贵州省已率先进行了相关的产品开发,目前已初具规模。无籽刺梨因其瘦果疏被刺,故名为“刺梨”;个别瘦果偶见少量种子,且种子可育,但平均发芽率仅为10.8%[3]。
无籽刺梨与常见的同属种类缫丝花(RosaroxburghiiTratt.)近缘,其与后者的主要形态差异为花瓣白色、复伞房花序和无籽等。对于无籽刺梨的物种起源方式,学术界一直存在争议。从形态解剖学特征看,无籽刺梨可能是自然杂交种[4]。然而,形态学性状以及AFLP和RAPD标记分析结果显示,无籽刺梨与贵州缫丝花(RosakweichowensisT. T. Yu et T. C. Ku)的遗传距离较近,推测无籽刺梨很可能是贵州缫丝花的高度雄性不育变异[5-6]。通过对物种形态特征的研究以及物种分布地点的考察,推测无籽刺梨很可能是蔷薇属内形成的天然杂交种,并且缫丝花为无籽刺梨的亲本之一。
相比于其他基因分型分析方法(genotyping method),如同工酶、AFLPs、SSR等,系统发育分析法具有自身特有的优势[7],并且已成功用于其他类群的物种起源研究[7-11]。为了探究无籽刺梨的物种起源方式,作者选择与无籽刺梨同域分布的蔷薇属植物14种、2个变种和2个变型以及外类群小叶金露梅(PotentillaparvifoliaFisch.),利用5个叶绿体基因片段(psbA-trnH、trnL-trnF、trnK-matK、psbI-psbK和rpoC1)和2个核基因片段(ITS和GAPDH),采用系统发育分析方法追溯无籽刺梨的物种起源。
1 材料和方法
1.1 材料
供试材料包括16种、2个变种和2个变型,其中蔷薇属植物15种、2个变种和2个变型,外类群1种(为小叶金露梅),凭证标本均保存于中国科学院成都生物研究所植物标本馆(CDBI),各种类的凭证标本及采集地信息见表1。在野外取各种类的新鲜嫩叶,置于硅胶中干燥,然后于-20 ℃保存,用于植物总DNA的提取。
1.2 方法
1.2.1 总DNA提取 称取经硅胶干燥后的叶片20~30 mg,用TIAGEN植物基因组DNA提取试剂盒(北京全式金生物技术有限公司) 提取总DNA,提取方法参见试剂盒说明书。
1.2.2 基因片段筛选、PCR及测序 根据前期的筛选结果,对5个叶绿体基因片段(psbA-trnH、trnL-trnF、trnK-matK、psbI-psbK和rpoC1)和2个核基因片段(ITS和GAPDH)进行扩增,扩增片段及其引物信息见表2。扩增反应体系总体积均为25.0 μL,包括双蒸水15.7 μL、25 mmol·L-1MgCl2溶液1.5 μL、Taq酶缓冲液2.5 μL、2.5 mmol·L-1dNTPs溶液2.0 μL、5 U·μL-1TaqDNA聚合酶〔天根生化科技(北京)有限公司〕 0.3 μL、10 pmol·mL-1正向引物和反向引物各1.0 μL以及植物全基因组DNA 1.0 μL(约50~100 ng)。
表1 供试无籽刺梨、蔷薇属其他种类及外类群的凭证标本和采集地信息
Table 1 Information of voucher specimen and location ofRosa×sterilisS. D. Shi, other species inRosaLinn. and outgroup tested

种类Species采集人和标本号CollectorandNo.ofspecimen采集地Location小叶组Sect.Microphyllae 缫丝花Rosaroxburghii邓亨宁D116 DENGHengningD116贵州贵阳GuiyangofGuizhou 白花刺梨Rosaroxburghiif.candida高信芬,等12903 GAOXinfen,etal12903重庆南川NanchuanofChongqing 无刺刺梨Rosaroxburghiif.inermis邓亨宁D120 DENGHengningD120四川汶川WenchuanofSichuan 无籽刺梨Rosa×sterilis周洪英,等D111 ZHOUHongying,etalD111贵州兴仁XingrenofGuizhou芹叶组Sect.Pimpinellifoliae 毛叶蔷薇Rosamairei朱章明8 ZHUZhangming8云南腾冲TengchongofYunnan 绢毛蔷薇Rosasericea高信芬,等12818 GAOXinfen,etal12818云南威信WeixinofYunnan木香组Sect.Banksianae 木香花Rosabanksiae高信芬,等12816 GAOXinfen,etal12816云南威信WeixinofYunnan 小果蔷薇Rosacymosa高信芬,等12746 GAOXinfen,etal12746四川江安Jiang’anofSichuan合柱组Sect.Synstylae 绣球蔷薇Rosaglomerata朱章明213 ZHUZhangming213四川峨边EbianofSichuan 卵果蔷薇Rosahelenae高信芬,等11131 GAOXinfen,etal11131云南永胜YongshengofYunnan 软条七蔷薇Rosahenryi朱章明647 ZHUZhangming647湖南芷江ZhijiangofHu’nan 长尖叶蔷薇Rosalongicuspis高信芬,等11031 GAOXinfen,etal11031云南鹤庆HeqingofYunnan 野蔷薇Rosamultiflora朱章明229 ZHUZhangming229贵州贵阳GuiyangofGuizhou 粉团蔷薇Rosamultifloravar.cathayensis高信芬,等10547 GAOXinfen,etal10547四川北川BeichuanofSichuan 悬钩子蔷薇Rosarubus朱章明227 ZHUZhangming227贵州贵阳GuiyangofGuizhou月季组Sect.Chinenses 单瓣月季花Rosachinensisvar.spontanea高信芬,等12963 GAOXinfen,etal12963重庆南川NanchuanofChongqing 亮叶月季Rosalucidissima高信芬16331 GAOXinfen16331贵州贵阳GuiyangofGuizhou金樱子组Sect.Laevigatae 金樱子Rosalaevigata朱章明,等460 ZHUZhangming,etal460广西环江HuanjiangofGuangxi蔷薇组Sect.Rosa 赫章蔷薇Rosahezhangensis邓亨宁D115 DENGHengningD115贵州赫章HezhangofGuizhou小叶金露梅1)Potentillaparvifolia1)高信芬,等12085 GAOXinfen,etal12085四川雅江YajiangofSichuan
1)外类群Outgroup.
表2 用于无籽刺梨、蔷薇属其他种类及外类群基因片段扩增的引物信息
Table 2 Information of primers used for gene fragment amplification ofRosa×sterilisS. D. Shi, other species inRosaLinn. and outgroup

引物类型Primertype引物Primer引物序列(5'→3')Primersequence(5'→3')设计者DesignerpsbA-trnHforwardpsbAFGTTATGCATGAACGTAATGCTCSANG,etalpsbA-trnHreversetrnHRCGCGCATGGTGGATTCACAAATCSANG,etalpsbI-psbKforwardpsbKITTAGCCTTTGTTTGGCAAGKIMKim-JoongpsbI-psbKreversepsbIAGAGTTTGAGAGTAAGCATKIMKim-JoongtrnK-matKforwardtrnKF2GCAACATGACTTCCTATACCCZHANGYutrnK-matKreversetrnK2RAACTAGTCGCATGGAGTAGPOTTER,etaltrnL-trnFforwardcCGAAATCGGTAGACGCTAGGTABERLET,etaltrnL-trnFreversefATTTGAACTGGTGACACGAGTABERLET,etalrpoC1forwardrpoC1FTATGAAACCAGAATGGATGGRoyalBotanicGardensrpoC1reverserpoC5RCAAGAAGCATATCTTGASTYGGRoyalBotanicGardensITSforwardITS-4TCCTCCGCTTATTGATATGCWHITE,etalITSreverseITS-AGGAAGGAGAAGTCGTAACAAGGWHITE,etalGAPDHforwardGAP×7FGATAGATTTGGAATTGTTGAGGSTRAND,etalGAPDHreverseGAP×11RGACATTGAATGAGATAAACCJOLY,etal
基本扩增程序为:95 ℃预变性5 min;95 ℃变性45 s、51 ℃~55 ℃退火30~45 s、72 ℃延伸30~90 s,34个循环;最后于72 ℃延伸10 min。其中,ITS片段为55 ℃退火45 s、72 ℃延伸60 s;GAPDH基因片段为51 ℃退火45 s、72 ℃延伸60 s;psbA-trnH基因片段为52 ℃退火30 s、72 ℃延伸45 s;psbI-psbK基因片段为53 ℃退火30 s、72 ℃延伸30 s;rpoC1基因片段为53 ℃退火45 s、72 ℃延伸60 s;trnL-trnF基因片段为54 ℃退火45 s,72 ℃延伸60 s;trnK-matK基因片段为53 ℃退火45 s,72 ℃延伸90 s。用质量体积分数1.0%琼脂糖凝胶电泳检测PCR产物,送上海英骏生物技术有限公司进行纯化和测序。
1.2.3 目的基因片段测序及克隆 对2个核基因片段ITS和GAPDH直接测序,无法直接测序的片段则进行克隆测序;用E.Z.N.A.®Gel Extraction Kit试剂盒(美国OMEGA Bio-Tek公司)进行PCR产物纯化;用pEASY®-T1 Cloning Kit试剂盒〔天根生化科技(北京)有限公司〕进行目的基因片段克隆,具体操作步骤参照操作指南(略有改动)。每个样品挑选10~30个阳性克隆,经培养后进行PCR鉴定;将符合要求的菌液送上海英骏生物技术有限公司进行测序。
1.2.4 序列拼接及比对 采用Sequencher 4.1软件拼接正向序列和反向序列,采用BioEdit version 5.0.6软件进行多序列比对并加以人工调整,去除模糊及比对不整齐的区域,最终得到用于后续分析的数据集。
1.2.5 系统发育分析 分别采用MrBayes V3.0中的贝叶斯分析(Bayesian inference,BI)和The CIPRES Science Gateway V. 3.3 (RAxML-HPC Blackbox)中的最大似然法(Maximum likelihood method,ML)进行系统发育分析。
2 结果和分析
利用BI法和ML法重建无籽刺梨的系统发育关系,所得到系统发育树拓扑结构一致,仅部分节点获得的支持率不同,因而,仅选用BI系统发育树用于结果分析;并且,将采用BI法和ML法分别得到的具有显著性的后检验率(PP)和靴带支持率(LB)标注在BI系统发育树上。
2.1 基于叶绿体基因片段的系统发育重建
利用5个叶绿体基因片段联合建树分析,经比对调整后,得到1个全长3 365 bp的数据集。系统发育重建结果(图1)显示:蔷薇属植物聚于分支Ⅰ和Ⅱ内。在分支Ⅱ里,无籽刺梨与长尖叶蔷薇(RosalongicuspisBertol.)聚在一起,且支持率较高,PP值为0.96、LB值为81%。

分支上和分支下的数据分别为后检验率(PP)和靴带支持率(LB) Datums above and below the branches indicate posterior probability (PP) and bootstrap percentage (LB), respectively.
图1 依据5个cpDNA 基因序列的扩增结果联合构建的无籽刺梨与蔷薇属部分种类的贝叶斯分析(BI)系统发育树
Fig. 1 Bayesian inference (BI) phylogenetic tree ofRosa×sterilisS. D. Shi and some species inRosaLinn. combined constructed by amplification result of five cpDNA gene sequences
2.2 基于ITS基因片段的系统发育重建
从供试的19种蔷薇属植物和小叶金露梅中总共获得43条ITS序列,经比对调整后,得到1个全长617 bp的数据集。系统发育重建结果(图2)显示:无籽刺梨所有单倍型分别位于2个分支上。其中,无籽刺梨单倍型H4、H5、H6和H7与缫丝花、白花刺梨(Rosaroxburghiif.candidaS. D. Shi)和无刺刺梨(R.roxburghiif.inermisS. D. Shi)构成分支a,该节点PP值为1.00、LB值为98%;无籽刺梨单倍型H1、H2和H3与软条七蔷薇(R.henryiBouleng.)以及长尖叶蔷薇单倍型H1和H2共同构成分支b,该节点PP值为0.93、LB值为85%。

分支上和分支下的数据分别为后检验率(PP)和靴带支持率(LB) Datums above and below the branches indicate posterior probability (PP) and bootstrap percentage (LB), respectively. H: 单倍型 Haplotype.
图2 依据ITS片段序列的扩增结果构建的无籽刺梨与蔷薇属部分种类的贝叶斯分析(BI)系统发育树
Fig. 2 Bayesian inference (BI) phylogenetic tree ofRosa×sterilisS. D. Shi and some species inRosaLinn. constructed by amplification result of ITS fragment sequence
2.3 基于GAPDH基因片段的系统发育重建
从供试的19种蔷薇属植物和小叶金露梅中共获得63条GAPDH基因序列,经比对调整后,得到1个全长883 bp的数据集。系统发育重建结果(图3)显示:无籽刺梨单倍型H2与H1和H3分别聚于分支A与分支B内,无籽刺梨单倍型H4单独聚为一支。无籽刺梨单倍型H2与长尖叶蔷薇单倍型H1和H2聚成分支A,该节点PP值为1.00、LB值为99%;无籽刺梨单倍型H1和H3与缫丝花、白花刺梨、无刺刺梨聚成分支B,该节点PP值为1.00、LB值为100%。观察和比对结果显示:无籽刺梨单倍型H4的碱基构成由缫丝花与长尖叶蔷薇单倍型H1整合而成。据此推测,由于无籽刺梨单倍型H4特殊的碱基构成,导致其系统位置异于其他单倍型。
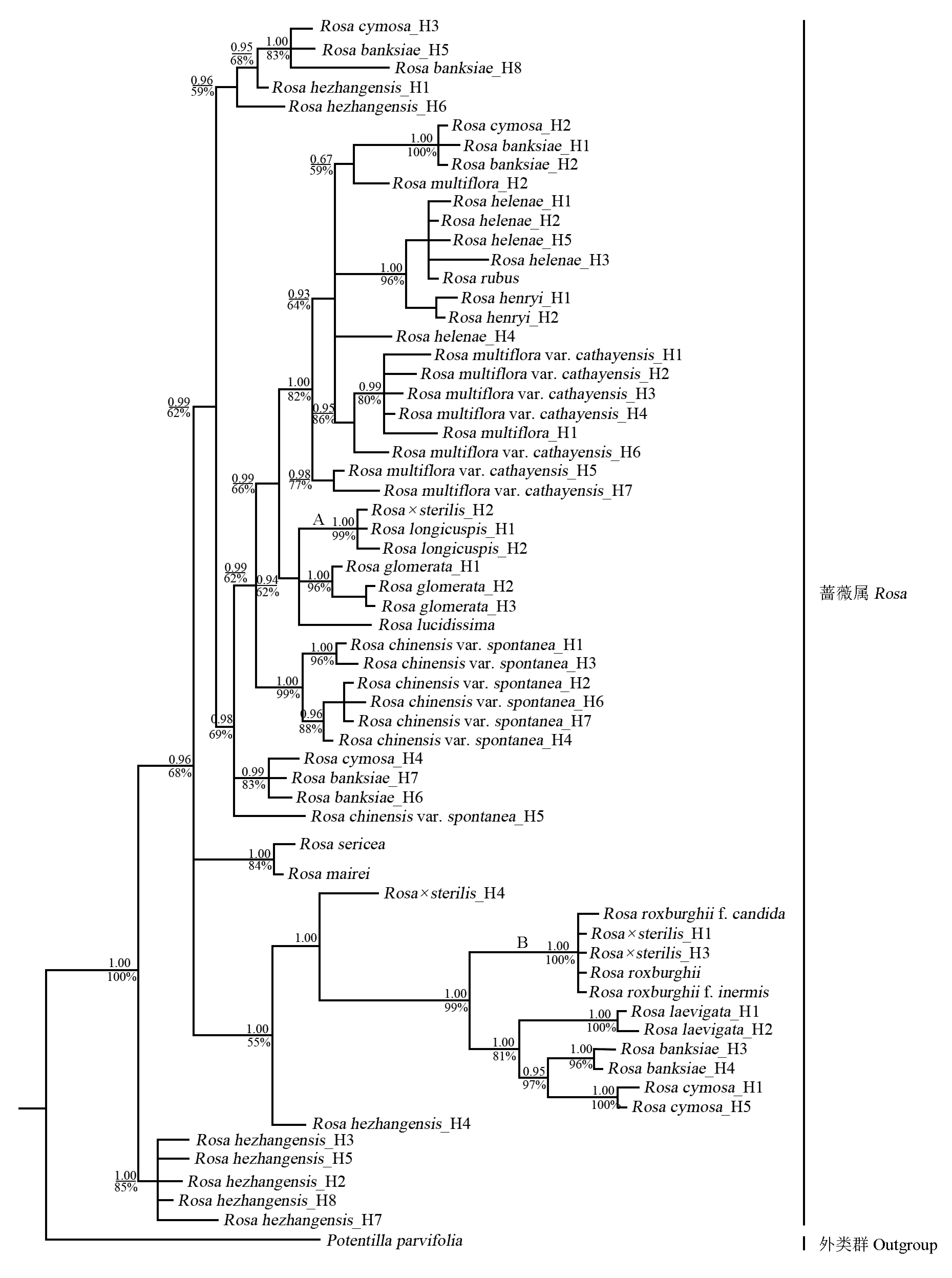
分支上和分支下的数据分别为后检验率值(PP)和靴带支持率值(LB) Datums above and below the branches indicate posterior probability values (PP) and bootstrap percentage values (LB), respectively. H: 单倍型 Haplotype.
图3 依据GAPDH基因序列的扩增结果构建的无籽刺梨与蔷薇属部分种类的贝叶斯分析(BI)系统发育树
Fig. 3 Bayesian inference (BI) phylogenetic tree ofRosa×sterilisS. D. Shi and some species inRosaLinn. constructed by amplification result ofGAPDHgene sequence
基于叶绿体基因片段psbA-trnH、trnL-trnF、trnK-matK、psbI-psbK和rpoC1以及核基因片段ITS和GAPDH的系统发育重建分析结果显示:无籽刺梨与同属的长尖叶蔷薇和缫丝花为近缘种,并且在系统发育树上得到了可信的支持。叶绿体基因片段的系统发育重建分析中,无籽刺梨与长尖叶蔷薇互为姐妹关系;在核基因片段的系统发育重建分析中,无籽刺梨的相应基因单倍型均分别与长尖叶蔷薇和缫丝花聚在一起。由于蔷薇科植物的叶绿体基因系母系遗传,因而,综合分析认为无籽刺梨起源于长尖叶蔷薇与缫丝花的天然杂交,长尖叶蔷薇为母本、缫丝花为父本。
3 讨 论
对杂交物种的准确鉴定,传统的方法是找寻物种的中间或过渡性状,尤其是形态上的中间性状,并通过杂交实验验证其亲本物种。但是在杂交物种的形成过程中也存在相应的亲本性状丢失的现象,误导判定工作,因而,即使得到形态学和细胞遗传学研究支持,仍需其他证据验证杂交物种的亲本来源[12]。
在本研究中,选取了5个叶绿体基因片段psbA-trnH、trnL-trnF、trnK-matK、psbI-psbK和rpoC1,以及2个核基因片段ITS和GAPDH,并利用系统发育分析方法探讨了无籽刺梨的物种形成方式。基于5个叶绿体基因联合构建的系统发育树及基于2个核基因片段分别构建的系统发育树均得出无籽刺梨系杂交起源的结论,其亲本为长尖叶蔷薇和缫丝花;并且在杂交成种过程中,长尖叶蔷薇及缫丝花分别扮演母本及父本的角色。长尖叶蔷薇为常绿高大攀缘灌木,广泛分布于中国云南、四川和贵州海拔400~2 700 m的山区(也见于印度北部),花期5月至7月[13]375;缫丝花为开展灌木,中国大多数地方均有分布(也见于日本),在中国西南地区分布较多,生长在海拔500~1 400 m的林缘、向阳山坡、灌丛和溪边等处,花期5月至7月[13]381。长尖叶蔷薇和缫丝花的花期一致,生境与分布海拔大部分重叠,为二者的基因交流奠定了基础条件;无籽刺梨为狭域分布于贵州省的1个天然杂交种,迄今为止仅发现在贵州省西南部的兴仁和安顺地区有分布。因此,推测在长尖叶蔷薇及缫丝花的进化历史中,贵州地区扮演了杂交带(hybrid zone)的角色,在这个杂交带内,长尖叶蔷薇和缫丝花进行频繁的基因交流,最终形成天然杂交种——无籽刺梨。形态特征比较结果也显示无籽刺梨的主要形态均与长尖叶蔷薇和缫丝花相应性状一致。
杂交不亲和及杂交后代高度不育是月季花(RosachinensisJacq.)远缘育种面临的重要问题[14],同样的问题在无籽刺梨中也存在。在无籽刺梨果实的发育过程中,最开始能形成胚珠,但伴随果实的发育胚珠逐渐枯萎[4],最终蔷薇果内仅能形成极少的种子[15-16]。染色体数目及花粉形态观察结果显示:无籽刺梨与缫丝花同为二倍体植物(2n=2x=14),其不育机制属于花粉败育型[15]。对于无籽刺梨少籽的现象,推测其原因为无籽刺梨在物种形成过程中经历了基因组冲击(genomic shock)。在自然杂交过程中,来自2个不同种的基因组同时进入1个核内,新形成的基因组发生了一系列的变化,其变化可能体现在DNA水平[17]、染色体水平[18]以及基因调控表达调节[19-21]等方面,最终引起控制无籽刺梨花粉正常发育的相关基因的表达受到一定程度抑制,从而造成无籽刺梨少籽现象。然而,对于无籽刺梨杂交成种后因花粉败育造成的少籽现象,目前鲜有研究报道,因此亟待开展更深入的研究来揭示控制这一现象的根本机制。
文晓鹏等[5]认为无籽刺梨与贵州缫丝花近缘。贵州缫丝花是1981年由俞德浚和谷粹之发表的贵州特有种,对于贵州缫丝花的采集信息,目前仅有1份模式标本(1936年采于贵阳清镇)和1份普通标本(1975年采于贵阳花溪区农学院),均为花期标本,未见果期标本。作者于2013年和2014年多次前往贵州缫丝花的标本采集地及其周边地区搜寻调查,均未发现该种的踪迹。当年用于实验研究的贵州缫丝花并未保存凭证标本、影像图片、分子材料或DNA样品等相关材料,因而,对于贵州缫丝花现今是否存在依然存疑。基于上述情况,无籽刺梨与贵州缫丝花之间的物种关系还有待进一步研究。
致谢: 野外调查过程中得到贵州省科学技术厅陈训老师、贵州生物研究所陈翔老师、贵州植物园金平老师及贵州师范大学向刚老师等的协助,在此一并致谢!
[1] 时圣德. 贵州蔷薇属新分类群[J]. 贵州科学, 1985(1): 8-9.
[2] 沈昱翔, 彭 珊, 熊 果. 安顺市金刺梨的成分差异探究[J]. 安顺学院学报, 2013, 15(4): 120-123.
[3] 陈 睿, 吴洪娥, 周 艳, 等. 7种蔷薇属植物种子的发芽试验研究[J]. 种子, 2013, 32(3): 72-74.
[4] 季祥彪, 李淑久. 贵州4种刺梨的比较形态解剖学研究[J]. 山地农业生物学报, 1998, 1(1): 28-33.
[5] 文晓鹏, 庞晓明, 邓秀新. 刺梨及部分近缘种形态学性状及RAPD标记分析[J]. 园艺学报, 2003, 30(2): 204-206.
[6] WEN X P, PANG X M, DENG X X. Characterization of genetic relationships ofRosaroxburghiiTratt and its relatives using morphological traits, RAPD and AFLP markers[J]. Journal of Horticultural Science and Biotechnology, 2004, 79: 189-196.
[7] JOLY S, STARR J R, LEWIS W H, et al. Polyploid and hybrid evolution in roses east of the Rocky Mountains[J]. American Journal of Botany, 2006, 93: 412-425.
[8] GRUNDT H H, POPP M, BROCHMANN C, et al. Polyploid origins in a circumpolar complex inDraba(Brassicaceae) inferred from cloned nuclear DNA sequences and fingerprints[J]. Molecular Phylogenetics and Evolution, 2004, 32: 695-710.
[9] MENG J, FOUGRE-DANEZAN M, ZHANG L B, et al. Untang-ling the hybrid origin of the Chinese tea roses: evidence from DNA sequences of single-copy nuclear and chloroplast genes[J]. Plant Systematics and Evolution, 2011, 297: 157-170.
[10] BRASSAC J, JAKOB S S, BLATTNER F R. Progenitor-derivative relationships ofHordeumpolyploids(Poaceae, Triticeae) inferred from sequences ofTOPO6, a nuclear low-copy gene region[J]. PLoS One, 2012, 7: e33808.
[11] KELLY L J, LEITCH A R, CLARKSON J J, et al. Reconstructing the complex evolutionary origin of wild allopolyploid tobaccos (Nicotianasectionsuaveolentes)[J]. International Journal of Organic Evolution, 2013, 67: 80-94.
[12] SOLTIS P S,DOYLE J J,SOLTIS DE.Molecular data and polyploidy evolution in plant[M]∥SOLTIS P S, SOLTIS D E, DOYLE J J. Molecular Systematic of Plant. New York: Chapman and Hall, 1992: 177-201.
[13] WU Z Y, RAVEN P H, HONG D Y. Flora of China: Vol. 9[M]. Beijing: Science Press, 2003.
[14] 冯 慧, 常卫民, 从日晨. 远缘杂交在现代月季育种中的作用[J]. 广东农业科学, 2009(12): 72-74.
[16] 邓朝义, 方仕能, 黄 勇. 贵州特有种子植物无子刺梨形态特征研究及分类学订正[J]. 种子, 2009, 28(9): 62, 68.
[17] WENDEL J F, SCHNABEL A, SEELANAN T. Bidirectional inter-locus concerted evolution following allopolyploid speciation in cotton (Gossypium)[J]. Proceedings of the National Academy of Sciences of the United States of America, 1995, 92: 280-284.
[18] LEITCH I J,BENNET M D.Polyploidyinangiosperms[J]. Trends in Plant Science, 1997, 2: 470-476.
[19] MITTELSTEN SCHEID O, JAKOVLEVA L, AFSAR K, et al. A change in ploidy can modify epigenetic silencing[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93: 7114-7119.
[20] GALITSKI T, SALDANHA A J, STYLES C A, et al. Ploidy regu-lation of gene expression[J]. Science, 1999, 285: 251-254.
[21] COMAI L, TYAGI A P, WINTER K, et al. Phenotypic instability and rapid gene silencing in newly formedArabidopsisallotetraploids[J]. The Plant Cell, 2000, 12: 1551-1568.
(责任编辑: 张明霞)
Molecular evidence for hybridization origin ofRosa×sterilis(Rosaceae)
DENG Hengning1a,1b, GAO Xinfen1a,1b,①, LI Xianyuan2, ZHOU Hongying3
(1. Chengdu Institute of Biology, Chinese Academy of Sciences: a. Key Laboratory of Mountain Ecological Restoration and Bioresource Utilization of Chinese Academy of Sciences, b. Key Laboratory of Ecological Restoration and Biodiversity Conservation of Sichuan Province, Chengdu 610041, China; 2. Chongqing Engineering Research Center for Floriculture, College of Horticulture and Landscape Architecture, Southwest University, Chongqing 400716, China; 3. Guizhou Botanical Garden, Guiyang 550004, China),
J.PlantResour. &Environ., 2015, 24(4): 10-17
In order to trace back to origin ofRosa×sterilisS. D. Shi, takingR.×sterilisand fourteen species, two varieties and two forms ofRosaLinn. in its same domain distribution as materials and takingPotentillaparvifoliaFisch. as outgroup, five chloroplast gene fragments (includingpsbA-trnH,trnL-trnF,trnK-matK,psbI-psbKandrpoC1) and two nuclear gene fragments (including ITS andGAPDH) were amplified and sequenced, on this basis, phylogenetic reconstruction was performed by Bayesian inference (BI) and Maximum likelihood method (ML). The results show that a dataset with whole length of 3 365 bp is constructed by combining five chloroplast gene fragments, and in BI phylogenetic tree hereby constructed,R.×sterilisandR.longicuspisBertol. are clustered together, with posterior probability (PP) of 0.96 and bootstrap percentage (LB) of 81%. A dataset with whole length of 617 bp is constructed by 43 ITS sequences,and inBIphylogenetic tree hereby constructed,four haplotypes ofR.×sterilis,R.roxburghiiTratt.,R.roxburghiif.candidaS. D. Shi andR.roxburghiif.inermisS. D. Shi are constituted together in branch a, with PP value of 1.00 and LB value of 98%, and other three haplotypes ofR.×sterilis,R.henryiBouleng. and two haplotypes ofR.longicuspisare constituted together in branch b, with PP value of 0.93 and LB value of 85%. A dataset with whole length of 883 bp is constructed by 63GAPDHgene sequences, and in BI phylogenetic tree hereby constructed,R.×sterilishaplotype H2 and two haplotypes ofR.longicuspisare clustered in branch A, with PP value of 1.00 and LB value of 99%,R.×sterilishaplotype H1 and H3,R.roxburghii,R.roxburghiif.candidaandR.roxburghiif.inermisare clustered in branch B, with PP value of 1.00 and LB value of 100%, andR.×sterilishaplotype H4 is clustered alone in a branch. The comprehensive analyzed result shows thatR.×sterilisoriginates in natural hybrid ofR.longicuspisandR.roxburghii, andR.longicuspisandR.roxburghiiare its maternal parent and paternal parent, respectively.
Rosa×sterilisS. D. Shi; speciation; nuclear gene; chloroplast gene; hybrid parent; phylogenetic tree
2015-03-24
国家自然科学基金资助项目(31070173; 31110103911); 中国科学院知识创新项目(KSCX2-EW-J-22; KSCX2-EW-Z-1); 贵州省农业科技攻关计划项目(黔科合NY字[2010]3030)
邓亨宁(1988—),男,四川资阳人,硕士研究生,主要从事植物资源利用与系统发育方面的研究。
①通信作者 E-mail: xfgao@cib.ac.cn
Q74; Q949.751.8
A
1674-7895(2015)04-0010-08
10.3969/j.issn.1674-7895.2015.04.02
