猪附红细胞体ORF5 蛋白的空间结构与B细胞抗原表位预测
2015-06-24王丽君巴彩凤
吕 晴,单 思,王丽君,巴彩凤
猪附红细胞体ORF5 蛋白的空间结构与B细胞抗原表位预测
吕 晴1,单 思2,王丽君1,巴彩凤3
目的 预测猪附红细胞体(Mycoplasmasuis,M.suis)ORF5 基因编码蛋白的二级结构及其B细胞抗原表位。方法 联合应用生物信息学软件DNAstar与在线分析软件IEDB的B Cell Epitope Prediction Tools预测M.suisORF5 蛋白的二级结构及其亲水性区域、柔韧性区域、抗原指数、表面可及性等结构特征,经综合分析后预测其B细胞优势抗原表位。结果 经分析可见,M.suisORF5 蛋白具有较为规则的二级结构,潜在的优势B 细胞抗原表位为GGVDGGRD、GMRLPEDSR、EGHPDLESAR氨基酸区段。结论 预测出M.suisORF5 蛋白二级结构和B细胞抗原表位,为M.suis免疫原性研究提供新手段,为病原微生物免疫原性研究提供新思路。
猪附红细胞体;ORF5;二级结构;B 细胞抗原表位
猪附红细胞体主要寄生于哺乳动物(包括人类)的红细胞导致出血性疾病或凝血功能障碍而引起贫血性传染病[1]。目前,世界已有三十多个国家报道了猪附红细胞体病的感染,而我国是感染最为严重的国家之一,但我国对其研究较晚,直至1972年才发现和报道猪附红细胞体病[2]。至今,猪附红细胞体的体外培养体系尚未建立,以至于对其生物学特性与发病机制了解十分有限[3],而关于猪附红细胞体基因组学信息与蛋白表达研究亦较少。Hoelzle 等提出M.suis的AJ504999DNA序列,长度为1 805 bp,含有11个ORF(Open reading frame),编码11种假设蛋白,但并未对其进行深入研究。多处文献报道,蛋白的二级结构及其亲水性、柔韧性、抗原性和表面可及性等结构特征决定与影响着B细胞抗原表位分布[4-5]。
本实验运用生物信息学方法对M.suisORF5蛋白二级结构与生物学特性进行分析预测,将二级结构与亲水性、柔韧性、抗原指数和表面可及性等参数相结合预测B细胞抗原表位,为M.suis免疫原性研究提供新手段,为病原微生物免疫原性研究提供新思路。
1 材料与方法
1.1 查找AJ504999基因序列及其ORF资料 登陆PubMed网站选择Nucleotide模块,输入GenBank序列号AJ504999,获得猪附红细胞体的基因序列及M.suisORF5的资料。
1.2 蛋白二级结构预测 启动DNAStar软件程序中的Editseq模块,将M.suisORF5中的氨基酸序列保存为pro格式,应用Protean模块分析和预测M.suisORF5蛋白的二级结构及其生物学特性。应用Gamier-Robson法和Chou-Fasman法分别分析预测M.suisORF5蛋白的二级结构,应用Kyte-Doolittle法预测其亲水性区域,Karplus-Schulz法预测其柔韧性区段,Jameson-Wolf法预测其抗原指数,Emini法预测表面可及性。登陆IEDB的B Cell Epitope Prediction Tools,应用Kolaskar-tongaonkar法预测平均抗原指数。
1.3 抗原表位的预测 综合考虑空间结构、亲水性、柔韧性、抗原指数和表面可及性等参数,进行分析和预测M.suisORF5编码蛋白的潜在优势B细胞抗原表位。
2 结 果
2.1M.suis的AJ504999-ORF5氨基酸序列 MCPNFRGGVDGGRDGFRLGILRTRFQGCLASNLCPGCPCCLTAGWRCEEMRLPEDSRPPCTLQGLSEYPLMLGFRTSVLSYCSGNFRWTRRRVLRGRGSGEGHPDLESARSGTWTQRGWARTSM。其含有124个氨基酸,相对分子量为13 836.92,等电点为9.02。
2.2M.suisORF5蛋白质二级结构预测 Protean模块运用Gamier-Robson法和Chou-Fasma法预测M.suis ORF5蛋白的二级结构发现,两者预测的结果显示,β-折叠和β-转角区域的重合率相对较高。Gamier-Robson法预测不存在α-螺旋,存在较多的β-折叠和β-转角,偶见单个氨基酸出现,其β-折叠最大的一个区段是60-80氨基酸,β-转角最大区段是27-36,无规则卷曲分布区段较分散,而Chou-Fasman法预测α-螺旋分布45-52、69-72区域。β-折叠分布区段均较长,最大区段是70-81,β-转角区域较分散,最大区段是30-37。Gamier-Robson法预测的无规则卷曲分布区段分散。见图1、表1。

A:α-螺旋;B:β-折叠;T:β-转角区域;C:无规则卷曲区域

表1 Gamier-Robson 法与Chou-Fasman 法预ORF5蛋白的二级结构
2.3M.suis ORF5蛋白结构特征
2.3.1 亲水性分析 Protean模块运用Kyte-Doolittle 法分析ORF5蛋白的亲水性发现,其亲水区段分布较多,主要分布于2-16,23-28,45-67,83-124氨基酸,见图2。

图2 Kyte-Doolittle 法分析ORF5蛋白的亲水性
2.3.2 柔韧性分析 Protean模块运用Karplus-Schulz法预测蛋白骨架区的柔韧性,柔韧性区段主要分布于5-16,23-25,53-60,64-66,76-77,84,90-92,94-119,121氨基酸,见图3。
2.3.3 抗原指数分析 Protean模块运用Jameson-Wolf方案预测的抗原指数可见,ORF5蛋白抗原指数较高的区段较多,主要位于7-15,50-60,93-113,118-121,见图4。在线分析软件IEDB运用Kolaskar-Tongaonkar 法预测ORF5蛋白抗原表位指数,平均抗原指数为1.015,见图5。

图3 Karplus-Schulz 法预测ORF5蛋白的柔韧性区段

图4 ORF5编码的蛋白的抗原指数分析

图5 ORF5结构蛋白的抗原表位指数分析
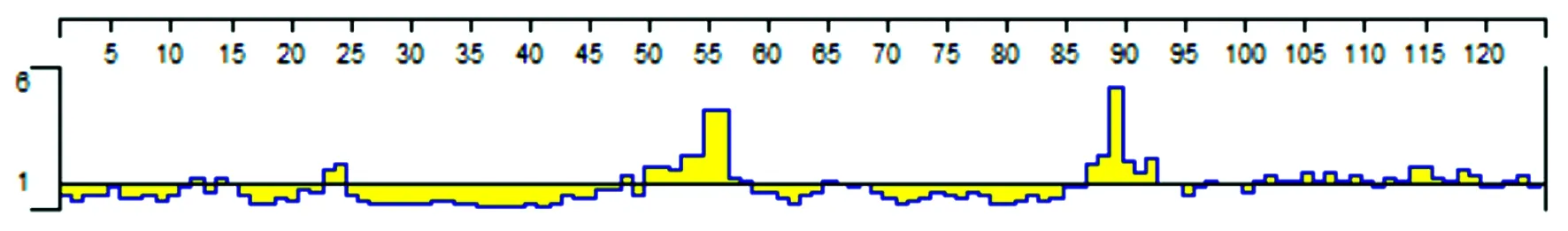
图6 ORF5蛋白的表面可及性分析
2.3.4 表面可及性分析 Protean模块运用Emini法预测表面可及性可见,其氨基酸位于蛋白质分子表面可能性较大的区段位于1-2,12,22,27-32,44-45,57-61,故这些很有可能位于蛋白质的表面的区段可能成为优势表位,为构建表位疫苗提供靶点。见图6。
2.4 B细胞抗原表位预测 结合ORF5蛋白的二级结构及结构特征等参数分析,若表面可及性指数≥1,亲水性指数≥0,抗原指数≥1.015,表面可及性指数≥1,且其内部或附近又具有柔韧性结构,区段氨基酸个数≥7,则这一区段成为抗原表位的可能性较大,经过筛选有3个区段可作为B细胞优势抗原表位,见表2。

表2 ORF5 编码蛋白B 细胞抗原表位的氨基酸序列
3 讨 论
B细胞抗原表位是由肽链上顺序连续的氨基酸组成,对其深入研究有益于推进免疫识别基础理论和实际应用,对疾病的诊断、定点改造蛋白分子降低蛋白药物免疫原性,设计无毒副作用的人工疫苗以及免疫干预治疗等有重要意义[6]。学者们指出,蛋白的空间结构决定了蛋白的功能与B细胞抗原表位的分布。在蛋白二级结构中,β-转角结构为凸出结构,多位于蛋白抗原表面,利于与抗体嵌合,较可能成为抗原表位,而α-螺旋、β-折叠结构规则稳定不易变形,较难嵌和抗体,成为B细胞抗原表位的几率极小。换言之,B细胞抗原表位多位于蛋白外侧,故而位于蛋白的外侧亲水氨基酸残基区段与表面可及性指数高的区段作为抗原表位的几率较高[7]。另外,柔韧性与抗原指数等结构特征也决定了B细胞抗原表位的分布。柔韧性区段具有较强的可塑性,易于抗体嵌入。抗原指数高的区段免疫原性强,成为抗原表位的几率大。迄今,对于B细胞抗原表位的预测,学者们提出了各自不同的分析方法,目前多采用综合分析法,权重选择蛋白的空间结构、亲水性、柔韧性、表面可及性、抗原指数等结构参数进行预测[8-11]。由于软件分析存在一定的局限性,故将多种方法和多参数分析的结果进行综合,这样可提高预测的准确性和可信性[12]。
目前,M.suis蛋白的结构还未成功解析, 其相应的二级结构也未知。本研究联合采用生物信息学软件DNAstar与IDEB的B Cell Epitope Prediction Tools综合分析M.suisORF5编码的蛋白,研究结果显示,M.suisORF5编码的蛋白的氨基端β-转角区段和无规则卷曲分布区段较多,相应区域相的亲水性、柔韧性、抗原指数和表面可及性等参数也较高,故而预测此区段为潜在优势B细胞抗原表位所在区段。该预测结果为M.suis免疫原性研究提供新手段,为病原微生物免疫原性研究提供新思路。
[1]Sokoli A, Groebel K, Hoelzle K, et al.Mycoplasmasuisinfection results endothelial cell damage and activation: new insight into the cell tropism and pathogenicity of hemotrophic mycoplasma[J]. Vet Res, 2013, 44: 6. DOI: 10.1186/1297-9716-44-6
[2]Wu YM. Diagnosis and treatment of Eperythrozoonosis in one case of piglet[J]. Fujian J Anim Husband Vet Med, 2013, 35(5): 67-68. (in Chinese) 吴奕渺.一例仔猪附红细胞体病的诊治[J] .福建畜牧兽医, 2013, 35(5): 67-68.
[3]Hoelzle LE, Felder KM, Hoelzle K. Porcine eperythrozoonosis: from Eperythrozoon suis toMycoplasmasuis[J]. Tierarztl Prax Ausg G Grosstiere Nutztiere, 2011, 39(4): 215-220.
[4]Ou Q, Zhu SL, Zhang LF. Prediction of the secondary structure and B-cell epitopes of EBV LMP2[J]. J Wenzhou Med Coll, 2007, 37(2): 114-118. (in Chinese) 欧琴, 朱珊丽, 张丽芳. EB病毒潜伏膜蛋白2的二级结构分析和B细胞表位预测[J] . 温州医学院学报, 2007, 37(2):114-118.
[5]Shan S, Ba CF. Prediction of secondary structure and B-cell antigenic epitopes ofMycoplasmasuisORF2 protein[J]. Chin J Biologicals, 2013, 26(2): 218-211. (in Chinese) 单思, 巴彩凤. 猪附红细胞体ORF2蛋白的二级结构与B细胞抗原表位预测[J] . 中国生物制品学杂志, 2013, 26(2): 218-211.(in Chinese)
[6]Wu J, Wu WT. Research progress on the method of B cell protein antigen epitope[J]. Pharmaceutical Biotechnol, 2000, 7(4): 239-242. (in Chinese) 吴敬, 吴梧桐. B细胞蛋白质抗原表位的研究方法进展[J]. 药物生物技术, 2000, 7(4): 239-242.
[7]Li H, Hu P. Prediction of the B cell epitopes of hemagglutinin and neuraminidase of human infected avian influenza A H7N9 virus[J]. Chin J Cell Mol Immunol, 2013, 29(12): 1319-1321.
[8] Song J, He QF. Bioinformatics analysis of the structure and linear B-cell epitopes of aquaporin-3 fromSchistosomajaponicum[J]. Asian Pac J Trop Med, 2012, 5(2): 107-109. DOI: 10.1016/S1995-7645(12)60005-4
[9]Kyte J, Doolittle RF. A simple method for displaying the hydropathic charactor of a protein[J]. J Mol Biol, 1982, 157(1): 105-132.
[10]Xue X, Zhu S, Li W, et al. Identification and characterization of novel B-cell epitopes within EBV latent membrane protein 2 (LMP2)[J]. Viral Immunol, 2011, 24(3): 227-236.
[11]Sweredoski MJ, Baldi P. A novel system for predicting continuous B-cell epitopes[J]. Protein Eng Des Sel, 2009, 22(3): 113-120. DOI: 10.1093/protein/gzn075
[12]Liu LN, Pan Q, Zhu JM. Prediction of B cell epitope for protective antigen RfeA against Streptococcussuisserotype2[J]. J Chengdu Med Coll, 2011, 6(2): 133-135. (in Chinese) 刘丽娜, 潘渠, 朱军民, 等. 2-型猪链球菌保护性抗原RfeA的B细胞表位预测[J]. 成都医学院学报, 2011, 6(2):133-135.
Prediction of the secondary structure and B-cell epitopes ofMycoplasmasuisORF5 protein
LYU Qing1,SHAN Si2,WANG Li-jun1,BA Cai-feng3
(1.DepartmentofPediatrics,LiaoheOilfieldGeneralHospital,Panjin121000,China; 2.ResearchCenterforDifferentionandDevelopmentofTCMBasicTheory,JiangxiUniversityofTraditionalChineseMedicine,Nanchang330004,China; 3.CenterofLaboratoryAnimals,LiaoningMedicalUniversity,Jinzhou121000,China)
The objective of the study is to predict the spatial structure and B-cell epitopes ofMycoplasmasuisORF5 protein. The secondary structure, hydrophilicity, flexible region, antigenic index and surface probability were analyzed and predicted by the Protean module in DNAStar software and B Cell Epitope Prediction Tools of IDEB, then B-cell epitopes were predicted by aggregate analysis. Results showed that the secondary structure ofMycoplasmasuisORF5 protein was relatively regular, in which the potential B cell antigenic epitopes were located at GGVDGGRD, GMRLPEDSR, and EGHPDLESAR. The methods of prediction of the secondary structure and B-cell epitopes ofMycoplasmasuisORF5 protein may provide a new method for the study ofM.suisimmunogenicity, and provides a new idea for the study on immunogenicity of pathogenic microorganisms.
Mycoplasmasuis; ORF5; Secondary structure; B-cell epitopes
Ba Cai-feng,Email:bacaifeng@163.com
10.3969/cjz.j.issn.1002-2694.2015.03.014
辽宁省科技基金资助项目(No.2006408002-6)
巴彩凤,Email:bacaifeng@163.com
1.辽河油田总医院,盘锦 124010; 2.江西中医药大学,中医基础理论分化发展研究中心,南昌 330004; 3辽宁医学院,实验动物中心,锦州 121000
R518.1;R392.12
A
1002-2694(2015)03-0251-04
Supported by the Liaoning province Science and Technololgy Fund Project(No.2006408002-6)
2014-04-02;
2014-10-28
