一株水产益生菌的鉴定及其净水研究
2015-06-06姜文联
王 军,姜文联
(广东科贸职业学院动物科技系, 510640)
一株水产益生菌的鉴定及其净水研究
王 军,姜文联
(广东科贸职业学院动物科技系, 510640)
鉴定1株分离自刺参养殖水体的益生菌DR1,进行了常规生理生化及16S rRNA基因序列分析,将DR1鉴定为马氏副球菌。进一步研究了该益生菌水质净化能力,结果表明:该菌株能有效降低水体化学耗氧量(COD),减少氨氮、亚硝酸盐等有害物质含量。
益生菌;16S rRNA基因;生物学鉴定;净水研究
随着我国水产养殖业的飞速发展,大规模、集约化的养殖模式逐渐取代了粗放、半粗放、半精养的养殖模式。集约化养殖过程中天然饵料已经不能完全满足养殖生物生长的需要,必须补充投喂人工配合饲料,以保证养殖生物的快速生长。不科学的投喂和管理模式使得饲料的利用率低下,没有被养殖生物摄食的残饵以及粪便等有机物沉入养殖池底,从而造成水体中营养物质和有机物大量积累,在水体溶氧不足的情况下经不完全氧化转化为对养殖生物有毒有害的氨氮及亚硝酸盐等物质,进而引起养殖生物中毒或死亡。
水产益生菌通过氨化、氧化、硝化、反硝化和固氮等作用[1],将养殖过程中产生的残饵、粪便以及水体中其他的有害物质等迅速地分解为二氧化碳、硝酸盐、磷酸盐、硫酸盐等无毒无害的营养物质来达到净化水质的目的,在此过程中水体的溶解氧也得到了提高,以此保证养殖水环境的洁净,从而促进养殖生物的健康生长。由于其绿色环保、不产生抗性、无毒副作用、无残留污染、无记忆性、作用范围广等优点目前已广泛应用于水产养殖。
从刺参养殖池塘水体中分离筛选出1株海洋益生菌DR1。然后采用常规的生理生化方法和细菌16S rRNA基因序列测定,进行了该菌株的生物学分类鉴定,并在实验室条件下研究了该菌株对养殖污水中COD、氨氮及亚硝酸盐的降解能力。
1 材料与方法
1.1 待测菌种及培养基
菌株DR1分离自刺参养殖池塘水体中。选用2216E海水培养基:酵母膏 1g,蛋白胨 5g,FePO40.1g,陈海水 1 000mL,pH=7.6~7.8,琼脂 2%。
1.2 形态学鉴定
将两菌株分别接种于2216E固体培养基上,28℃恒温培养24h 后,观察平板上的菌落形态及表面特征,常规的检验还包括革兰氏染色、芽孢染色、菌体运动性观察以及形态观察,具体方法和步骤参照《常见细菌系统鉴定手册》和《微生物学实验技术》[2-3]。
1.3 生理生化指标测定
测定菌株DR1的生理生化特性,具体鉴定内容及方法参照武心华《刺参池塘有机物降解菌固定化及其对水质净化作用研究》[4]进行。
1.4 待测菌株的16S rRNA基因序列鉴定和分析
1.4.1 PCR模板制备
将细菌接种在平板上,28℃培养过夜。取单一菌落悬浮于50 L无菌蒸馏水中,于100℃水浴加热5min,离心,取上清作为PCR模板DNA。
1.4.2 16S rRNA基因的PCR扩增与测序
以细菌16S rDNA扩增常用引物27F(5'-A G A G T T T G A T CC T G G C T CA G -3')和1492R(5'-A C G G C T A C C T T G T T A C G A C T -3')为PCR引物。 采用50μL的反应体系,扩增模板DNA,并交由相关测试公司(北京三博远志生物技术有限责任公司)进行序列测定。
1.4.3 16S rRNA基因的序列分析及数据处理
将菌株 DR1的 16S rRNA基 因序列与从Genbank数据库中获得的细菌的16S rRNA基因序列进行比对,采用ClustalX、BioEdit和MEGA4.1等软件对菌株DR1进行系统进化分析,用Neighbor-Joning法构建系统发育树,1000次RePlications,计算Bootstrap值评估树的置信度,对DR1进行系统发育树的构建。
1.5 水质净化研究
将经过2216E液体培养基培养至对数期的DR1培养液离心收集菌体,用无菌生理盐水洗涤2次后重悬作为水质净化研究的接种菌液,并用血球计数板计数菌浓度。本实验按施用终浓度1.0×l06cfu/mL接种于盛有3L养殖污水的三角瓶中,以不加菌的处理作为对照组,每个处理设5个重复,每天定时取样测定水体中COD、氨氮及亚硝酸盐含量,连续监测一周。水体中COD、氨氮、亚硝酸盐测定方法参照照雷衍之《养殖水环境化学实验》[5]进行。
2 结果
2.1 菌株的形态学鉴定
荧光显微镜下DR1菌体呈球状,无鞭毛,无芽孢,2216E平板上培养48~72h,肉眼观察菌落呈橘黄色、圆形、隆起、不透明、边缘整齐、表面较湿润光滑。
2.2 生理生化指标测定结果
菌株DR1生理生化鉴定结果见表1。

表1 菌株生理生化特性
可以看出,该菌株为低温种,在水温超过25℃,菌体生长明显受到抑制,最适生长温度10~18℃。DR1对盐度的适应范围较广,在0~4%的水体中均能正常生长。
2.3 16S rRNA 基因序列分析和系统发育树的构建
将菌株DR1的16S rRNA基因序列在GenBank数据库中进行比对,发现与该菌株序列同源性最高的绝大多数为副球菌属的细菌,选取相关菌株的16S rRNA序列用Neighbour-Joining method构建系统发育树(图1)。如图1所示,菌株DR1与副球菌属的Paracoccus marcusii(EF491972)序列相似性达99%以上,结合形态特征、生理生化特性,将菌株DR1初步鉴定为马氏副球菌。

图1 DR1的16S rDNA 基因序列聚类分析结果
2.4 DR1净水结果
菌株DR1对水体中COD、氨氮及亚硝酸盐的降解效果见图2。
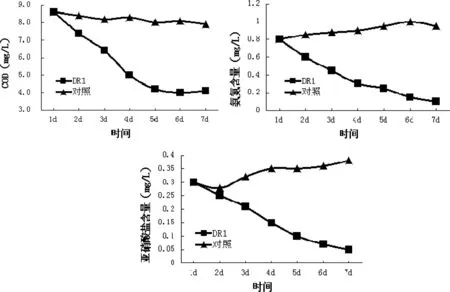
图2 DR1对水体COD、氨氮及亚硝酸盐的降解
由图2可以看出,与对照组相比,施加DR1可以明显降低养殖废水中的化学耗氧量(COD),间接提高水体中溶解氧供养殖生物呼吸作用。同时,添加DR1的处理组,氨氮1周内由0.9mg/L迅速降低到0.15mg/L,达到安全浓度;而水体中的亚硝酸盐也由0.3mg/L降低到0.05mg/L,而对照组2个指标在此过程中均有所上升。
3 讨论
根据常见细菌的鉴定方法,菌株DR1属于革兰氏阴性球状菌;菌株好氧,无运动能力;接触酶、氧化酶阳性;不产芽孢;不能利用柠檬酸盐,硝酸盐还原阳性。具有上述显著特征的代表是副球菌属菌株。
16S rRNA作为一种重要的核酸水平鉴定工具,其显著特点就是能及时鉴定出生长缓慢或生化惰性、细菌培养阴性以及非常规的菌株[6]。近年来,大量细菌的16S rRNA序列已被检测并被NCBI等国际基因数据库收录,成为目前细菌鉴定和分类的主要参照系统,并得到NASA、FDA、TIGR、NIAID等多个国际权威机构的认可[7-8]。从16S rRNA基因序列相似性和系统发育树上看,菌株DR1与Paracoeeus marcusii聚为一群,具有最高的相似性。从形态、生理、生化、系统发育学、16S rRNA基因序列同源性等方面分析,菌株DR1可鉴定为马氏副球菌。
水中氨态氮含量增加会抑制鱼体内的氮排泄,损害养殖生物鳃等器官组织,消耗水中溶解氧;亚硝态氮毒性较强,是养殖水域中诱发暴发性疾病的重要因素,因此去除水体中过量的氨氮、亚硝态氮对于改善水质、提高养殖生物成活率和生长率、保护环境等具有重要意义。水体有益微生物能通过降低水体中有机物含量,以及一系列的消化、反硝化作用,降低水体中氨氮、亚硝酸盐的含量,提高水体溶氧,是进行养殖生态防治重要手段。本文在实验室条件下,研究了DR1对养殖污水的净化能力,结果显示,在小水体中,DR1对水体化学耗氧量(COD)、氨氮、亚硝酸盐均有较好的降解能力,可以作为商业微生态制剂开发材料。
[1] 冯俊荣,陈营,付学军,等. 微生态制剂对养殖水体水质条件的影响[J].海洋湖沼通报,2005,(4):104-108.
[2] 东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001:349-370.
[3] 林连祥, 路福平. 微生物学实验技术[M]. 北京: 中国轻工业出版社, 2006:25-31.
[4] 武心华. 刺参池塘有机物降解菌固定化及其对水质净化作用研究[D]. 青岛:中国海洋大学, 2011.1-76.
[5] 雷衍之. 养殖水环境化学实验[M]. 北京:中国农业出版社,2006.
[6] 刘朝军. 16SrDNA 序列测定在细菌鉴定中的应用[J]. 军医进修学院学报, 2011,32(7):774-777.
[7] Hugenholtz P, Huber T. Chimeric 16SrDNA sequences of diverse origin are accumulating in the public databases[J]. International journal of systematic and evolutionary microbiology, 2003, 53(1):289-293.
[8] Fuller N, Marie D, Partensky F, et al. Clade-specific 16S ribosomal DNA oligonucleotides reveal the predominance of a single marine Synechococcus clade throughout a stratified water column in the Red Sea[J]. Applied and environmental microbiology, 2003,69(5): 2430-2443.
