肠道微生态与炎症性肠病
2015-06-01何丽娟张军东安毛毛姜远英第二军医大学药学院新药研究中心上海004同济大学附属第十人民医院上海0007同济大学医学院上海0009
何丽娟,张军东,安毛毛,姜远英(.第二军医大学药学院新药研究中心,上海004;.同济大学附属第十人民医院,上海0007;.同济大学医学院,上海0009)
・综述・
肠道微生态与炎症性肠病
何丽娟1,张军东2,安毛毛3,姜远英1(1.第二军医大学药学院新药研究中心,上海200433;2.同济大学附属第十人民医院,上海200072;3.同济大学医学院,上海200092)
炎症性肠病(inflammatory bowel disease,IBD)是一组病因未明、发病机制亦不明确的慢性肠道炎症性疾病,主要包括克罗恩病(Crohn's disease,CD)和溃疡性结肠炎(ulcerative colitis,UC)。近几十年的研究结果认为,其发病是环境、易感基因和肠道微生态3个要素相互作用的结果,且这些要素使IBD成为一种适合研究宿主与肠道微生物相互作用的高优先平台。最近,肠道菌群的图谱分析将IBD的发病机制与菌群各组成部分特征的改变相联系,进一步支持“肠道微生物和宿主相互作用的改变能形成IBD”这一观点。该文回顾性分析有关IBD患者体内微生物的研究文献,综述肠道微生态失衡对IBD的多方面影响,以动物模型和临床验证资料阐述不同的治疗方法改善肠道微生态变化的最新进展。
炎症性肠病;肠道微生态;相互作用
在北美和欧洲,IBD曾是一种极为罕见的疾病,直至20世纪中期发病率开始大幅上升。如今,欧洲UC最高发病率为24.3/10万人・年,北美CD最高发病率为20.2/10万人・年;而亚洲、非洲等IBD发病率偏低的地区在前20年中其发病率也在逐年上升[1],这种变化可能是由于生活方式及饮食的改变所导致,因此,环境因素也是造成IBD的重要原因。其中,肠道菌群正获得越来越多的关注,特别是它对人类健康等多方面的影响。笔者主要就肠道微生态和IBD的相互影响进行讨论。
1 肠道微生态失衡对IBD的影响
肠道菌群是人体最大的微生物菌落,在整个胃肠道中密集程度有所不同,在结肠内数量最多,达到1011~1012个菌/g肠容物[2]。这个菌落对于宿主来说有着一系列功能,包括分解人体内由酶消化后难以被利用的物质,刺激免疫系统,抑制有害微生物的增长等。最近研究发现,IBD宿主肠道微生物群在生态学和功能学上均有改变,因此,IBD也成为研究肠道微生物结构和功能的一个理想模型[3]。
近十年中,DNA测序技术和计算机分析方法的飞速发展,为研究IBD患者体内微生物群的特征创造了条件。多项研究发现,在IBD的初始阶段,微生物群结构发生失衡,然后出现典型的特征表型,包括生物多样性的减少、一些厚壁菌门代表性的种群降低和一些变形菌属的增加等[4]。如表1所列,肠杆菌科,尤其是有黏附-侵袭能力的大肠杆菌、梭状芽孢杆菌(艰难梭菌),以及变形菌门在IBD患者体内均增加;而厚壁菌门、拟杆菌门以及梭菌群IV均减少[5]。这些变化都有可能影响IBD患者的炎症特征。
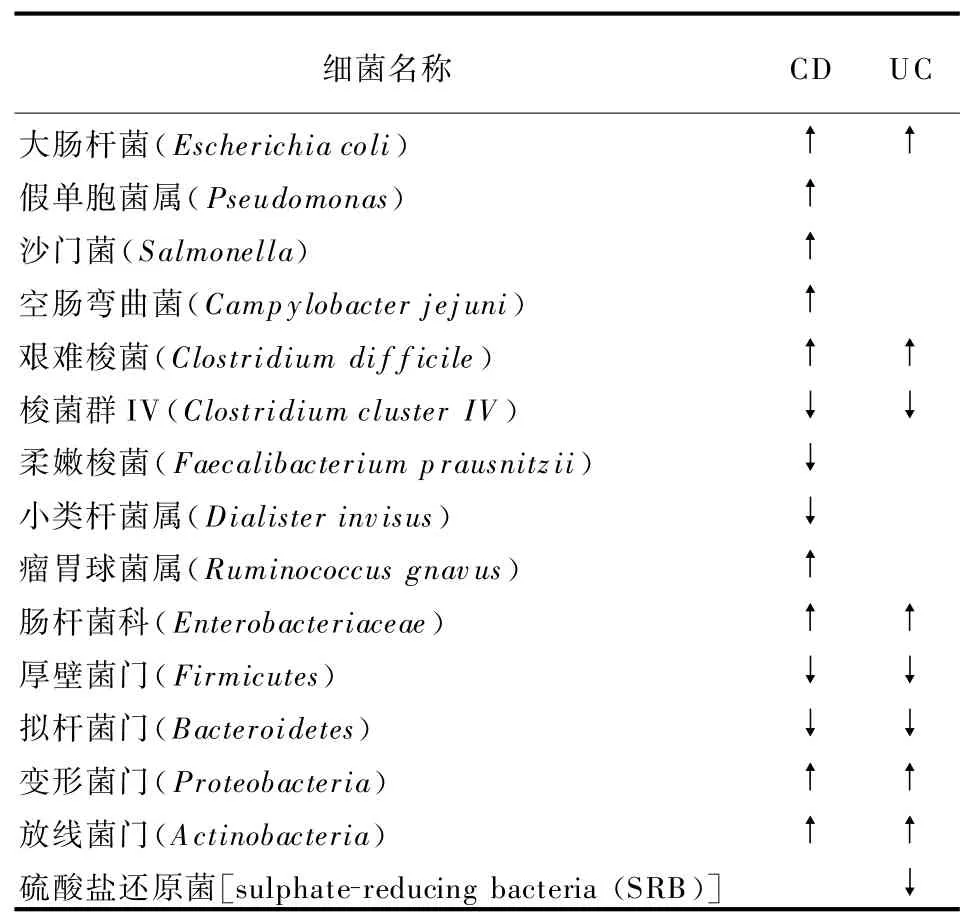
表1 IBD患者肠道菌群分类变化
1.1肠道有害菌群富集对IBD的影响 健康人体内,由于黏液层的存在以及诸如防卫素、抗菌肽和溶菌酶等抗菌因子的生成,细菌一般都附着在黏液中,不会直接进入上皮组织。但与健康人群相比,UC患者结肠黏液层中会附着数量更多的细菌[6]。Boudeau等[7]从CD患者的回肠中分离出具有黏附-侵袭能力的大肠杆菌致病菌(adherent invasive E.coli,AIEC),这种菌也黏附于UC患者肠黏膜上,但目前在UC患者中还没有发现它的侵袭作用。AIEC在CD患者肠黏膜活检标本中比健康受试者更为密集,在回肠样本中尤为显著。AIEC侵入上皮细胞和巨噬细胞内能够大量繁殖,在体外可以诱导出肉芽,同时也有实验结果显示其参与了CD患者体内息肉的形成[8]。最近,有文献详细地阐述了这种菌株的潜在致癌功能[9]。
第二类具有黏附和侵袭作用的菌种是梭状杆菌。研究发现UC患者与对照组(健康受试者)相比,前者结肠黏膜上的具核梭杆菌相对丰度较高,可作为胃肠道疾病的生物指标来判断IBD患者病情的轻重[10]。Ohkusa等[11]将临床上UC患者体内分离的梭杆菌菌株通过人工培养得其上清液,再经过直肠灌肠诱导小鼠结肠黏膜受损,这表明此类菌可能是UC的发病机制之一。临床上IBD患者的病情严重程度与其体内梭杆菌菌株的侵袭能力呈正相关[12],表明侵袭性梭杆菌可能影响IBD的病理变化。最近研究发现,在结直肠癌中梭杆菌大量富集在癌症部位而其邻近正常组织较少,患者粪便中的梭杆菌也比健康人群多,患者体内分离的梭杆菌在小鼠模型中能够生成炎症微环境,促进肿瘤发生[13]。由于IBD是导致结直肠癌的高风险因素之一,因此梭杆菌可能代表了这些疾病之间的某种潜在联系。
1.2肠道共生菌群减少对IBD患者的影响 研究表明肠道中某些微生物能够对IBD具有保护作用。例如,Kitajima等[14]用葡聚糖硫酸钠(DSS)造成的结肠炎模型中,无菌小鼠模型出血及死亡情况比常规饲养的小鼠模型要严重许多。Kamada等[15]认为肠道共生菌群保护宿主的机制之一是肠道菌群的抗移植性。即肠道共生菌群在宿主体内占据主要生态位,通过竞争排除病原菌和其他致病菌在体内的定植。因此,肠道内微生态平衡被破坏后,随着生物多样性的减少,势必造成一系列问题。
肠道内有部分种属的细菌,如大多数的双歧杆菌、乳酸杆菌、肠球菌类等益生菌能够通过一系列机制保护宿主避免炎症,包括下调炎症因子和刺激抑炎因子IL-10,调节黏膜屏障功能和通透性等[16]。近期发现,柔嫩梭菌也是一种具有抗炎性能的微生物。在CD患者的回肠活检样本中,随着大肠杆菌数量的增加,柔嫩梭菌群数量相对减少,而这往往导致CD患者术后复发危险性的增加。相反,UC复发后该菌群的恢复则有利于维持UC患者的临床缓解[17]。因此,柔嫩梭菌也被认为是IBD的潜在治疗菌种之一。
1.3真菌等非细菌类微生物对IBD的影响 肠道菌群中其他非细菌类成员,如真菌、病毒、古细菌和噬菌体等,也可能在胃肠道疾病中起重要作用。然而,近年来绝大多数对肠道微生物群的研究都是基于16S rRNA测序的方法,因此在很大程度上忽略了这些群体。例如,CD易感基因ATG16L1突变的小鼠感染了诺瓦克病毒后表现出和CD类似的症状,因此该研究认为诺瓦克病毒和突变的CD易感基因ATG16L1相互作用可引发小鼠肠道病变[18]。近年来多项研究探讨真菌和IBD之间的关系,包括在UC和CD中真菌多样性的总体增加等[19]。Iliyan等[20]对肠道共生真菌和C型凝集素受体dectin-1的相互作用进行研究,发现dectin-1缺失小鼠比同窝野生型小鼠对DSS造成的结肠炎更为明显,但如果口服氟康唑将肠道真菌抑制后,这两种小鼠对DSS造成的结肠炎则无显著性差异。因此,从真菌刺激肠道固有免疫方面来寻找新的药物治疗方法,为IBD的临床治疗提供了新的思路。预计未来的数年内真菌和IBD之间的关系将会有更深入的报道。
此外,IBD患者体内肠道菌群的功能改变比构成改变更为严重。Morgan等[21]通过对231个IBD患者和健康对照组的活检样本、粪便样本进行16S RNA检测和代谢功能的分析,发现仅2%的肠道菌群属发生改变,却有12%的代谢通路发生变化。因此,该研究进一步验证了微生态失衡对IBD影响之大。
2 微生态平衡缓解IBD
IBD可以通过药物治疗好转,也可以自行缓解,但都会反复发作。缓解和发作的机制也都不明确,有待深入研究。从肠道共生菌群方面着手,其针对潜在的病原体具有直接的调节功能(如抑制病毒有关的基因表达)。此外,肠道菌群对调节肠免疫系统也起着一定的作用。如拟杆菌和丁酸梭菌都能诱导调节性T细胞的高表达,减轻肠道炎症,乳酸菌和双歧杆菌等还可以通过调节NF-κB核通路来减轻黏膜炎症[22]。
肠道微生态的平衡意味着在一个健康的肠道环境下,IBD患者可以通过服用益生元或者活的益生菌,补充体内缺少的一部分有益菌,使症状得到缓解。
益生元,是存在于发酵膳食纤维中的某些成分,它能够促进肠道益生菌的定植。益生菌生成短链脂肪酸(SCFA),它能抑制有害菌的繁殖,维持肠道内菌群的平衡。SCFA包括乙酸、丙酸、丁酸等,其优先于葡萄糖被肠黏膜细胞吸收,是肠道上皮细胞的初级能源。最近有研究证明,SCFA能刺激结肠调节性T细胞的增加,进而控制炎症反应[23]。但在IBD患者体内,由于肠道微生态失衡导致益生菌减少,SCFA产量相对降低。研究证明瘤胃菌科,尤其是产丁酸菌属柔嫩梭菌群,在CD患者回肠中明显降低。其他产SCFA的菌,如考拉杆菌群和罗氏菌群,两者在CD患者体内有所减少;而明串珠菌群在UC患者体内有所减少[22]。同时由于炎症和组织破坏处有未被肠道吸收的营养物质,细菌可以直接吸收使用,因而虽然它们生产营养因子的能力降低了,但从环境中摄取的较多。同时有害菌群大量吸收营养快速增殖,进而使炎症更加难以缓解。因此一旦菌群恢复之前的结构和功能,患者体内微生态即趋向平衡,IBD患者症状就能得到缓解。
3 IBD治疗对微生态失衡的影响
部分研究认为,抗生素的滥用使得肠道内大肠杆菌大幅增加以及代谢紊乱。然而,一些较新的荟萃分析则显示,抗生素的使用能够缓解IBD和预防其复发,但此研究需进一步做对照试验[24]。因此,为更好地了解肠道菌群失衡的后果,以及在药物产生的疗效中微生物所起的作用,需要在微生态环境和功能构成变化水平上进行瞬态分析[25]。迄今为止,已有部分研究揭示了健康人群短暂服用抗生素造成肠道菌群紊乱和恢复后的水平。如果健康人群重复使用一种抗生素,将会导致肠道菌群结构长时间内累积持续性变化。菌群种类复杂性的损伤必然会扰乱微生物内稳态,导致微生态失调和炎症危险因素大大增加[26]。而抗生素引起肠道感染概率增加的另一个机制是过薄的黏液层导致肠道屏障功能的损伤[27]。
为防止抗生素扰乱肠道微生态的菌群多样性,人们开始向肠道内植入部分有益的菌群。这种方法在过去几年中得到了普及,但仍存在很多问题需要关注,如待植入肠道的菌群复杂性和组成结构很重要,菌种的选择和安全性也需要重复验证。由于菌种和疾病亚型的不同,益生菌在IBD患者肠道内的定植结果变化很大。一个典型的临床试验——对艰难梭菌反复感染的治疗,使得“粪便微生物移植能够高效补充肠道菌群”的观点获得广泛关注[28]。与之相关的一些研究表明,从健康者提供的粪便中精选出的某些菌群已经能够代替他们的粪便,使得患者病情恢复[29]。因此,治疗艰难梭菌重复感染的高成功率使“粪便微生物移植”作为一种新兴的治疗手段,在临床用于治疗某些胃肠道疾病和代谢紊乱的疾病,包括治疗IBD[28]。但至今为止,有关IBD患者用该法治疗的结果报道很少,关于IBD病情缓解的成功率也大不相同,因此目前仍需设计更加严格精确的随机对照试验[30]。微生物组成和炎症的相互关系,以及抗生素和益生菌的使用对未来研究起着重要的指导作用。Morgan等[31]对肠道菌群的生物信息学分析和统计工具获得的数据能够对正常人的各项指标作出早期诊断,判断其是否有潜在的肠道疾病。
4 结语
迄今为止,已有多项关于微生物多层次多因素影响IBD发病的研究,包括从基因、免疫反应、微生物功能活性等各方面进行解释。然而,由于人体肠道与微生物系统相互作用的复杂性,还没有找到真正能解释微生物与宿主相互作用机制的关键证据,因此我们需要对IBD患者体内微生物组成有系统性的了解。近期科技的发展使得检测技术更加成熟,计算机生物信息学的技术进步也能用来进行宿主与微生物群预测等相关性研究。这些科学技术的发展使未来肠道微生物研究的重点从广泛的功能特征转向识别特定的效应分子,包括微生物结构组件,细胞内或者分泌出的代谢物和蛋白质,以及其他由于宿主和微生物相互作用产生的信号组分等。这样能深入探讨宿主和微生物相互作用的机制,并发展出新的诊断和治疗方法,为这种疾病提供更多的治疗途径。
[1] M olodecky NA,Soon IS,Rabi DM,etal.Increasing incidence and prevalence of the inflammatory bow el diseases w ith time,based on systematic review[J].Gastroenterology,2012,142(1):46-54.
[2] Dave M,Higgins PD,M iddha S,et al.The human gutm icrobiome:current know ledge,challenges,and future directions[J].Transl Res,2012,160(4):246-257.
[3] Huttenhow er C,Kostic AD,Xavier RJ.Inflammatory bowel disease as a model for translating them icrobiome[J].Immunity,2014,40(6):843-854.
[4] M organ XC,Segata N,Huttenhower C.Biodiversity and functional genom ics in the human m icrobiome[J].Trends Genet,2013,29(1):51-58.
[5] Scaldaferri F,Gerardi V,Lopetuso LR,et al.Gut m icrobial flora,p rebiotics,and p robiotics in IBD:their current usage and utility[J].Biomed Res Int,2013,2013:435268.
[6] Hooper LV,Littman DR,M acpherson AJ.Interactions between the m icrobiota and the immune system[J].Science,2012,336(6086):1268-1273.
[7] Boudeau J,Glasser AL,M asseret E,et al.Invasive ability of an Escherichia coli strain isolated from the ileal mucosa of a patient with Crohn's disease[J].Infect Immun,1999,67(9):4499-4509.
[8] Rolhion N,Darfeuille-M ichaud A.Adherent-invasive Escherichia coli in inflammatory bowel disease[J].Inflamm Bow el Dis,2007,13(10):1277-1283.
[9] A llen-Vercoe E,Jobin C.Fusobacterium and Enterobacteriaceae:important players for CRC?[J].Immunol Lett,2014,162(2PA):54-61.
[10] A llen-Vercoe E,Strauss J,Chadee K.Fusobacterium nucleatum:an emerging gut pathogen?[J].Gut M icrob,2011,2(5):294-298.
[11] Ohkusa T,Okayasu I,Ogihara T,et al.Induction of experimental ulcerative colitis by Fusobacterium varium isolated from colonic mucosa of patients w ith ulcerative colitis[J].Gut,2003,52(1):79-83.
[12] Strauss J,Kap lan GG,Beck PL,et al.Invasive potential of gut mucosa-derived Fusobacterium nucleatum positively correlates w ith IBD status of the host[J].Inflamm Bow el Dis,2011,17(9):1971-1978.
[13] Kostic AD,Chun E,Robertson L,et al.Fusobacterium nucleatum potentiates intestinal tumorigenesis and modulates the tumor-immunem icroenvironment[J].Cell Host M icrob,2013,14(2):207-215.
[14] Kitajima S,M orimoto M,Sagara E,etal.Dex tran sodium sulfate-induced colitis in germ-free IQI/Jic m ice[J].Exp Anim,2001,50(5):387-395.
[15] Kamada N,Chen G,Núñez G.A com plex m icroworld in the gut:harnessing pathogen-commensal relations[J].Nat Med,2012,18(8):1190-1191.
[16] Llopis M,Antolin M,Carol M,et al.Lactobacillus casei dow nregulates commensals′inflammatory signals in Crohn's diseasemucosa[J].Inflamm Bow el Dis,2009,15(2):275-283.
[17] Varela E,Manichanh C,Gallart M,et al.Colonisation by Faecalibacterium p rausnitzii and maintenance of clinical rem ission in patients w ith ulcerative colitis[J].A liment Pharmacol Ther,2013,38(2):151-161.
[18] Cadwell K,Patel KK,M aloney NS,etal.Virus-plus-suscep tibility gene interaction determ ines Crohn's disease gene A tg16L1 phenotypes in intestine[J].Cell,2010,141(7):1135-1145.
[19] T rojanowska D,Zwolinska-W cislo M,Tokarczyk M,et al.The role of Cand ida in inflammatory bow el disease.Estimation of transm ission of C.albicans fungi in gastrointestinal tract based on genetic affinity between strains[J].Med Sci M onit,2010,16(10):CR451-457.
[20] Iliev ID,Funari VA,Tay lor KD,et al.Interactions between commensal fungiand the C-type lectin receptor Dectin-1 influence colitis[J].Science,2012,336(6086):1314-1317.
[21] M organ XC,Tick le TL,Sokol H,etal.Dysfunction of the intestinalm icrobiome in inflammatory bowel disease and treatment[J].Genome Biol,2012,13(9):R79.
[22] A tarashi K,Tanoue T,Oshima K,et al.Treg induction by a rationally selected m ixture of Clostridia strains from the human m icrobiota[J].Nature,2013,500(7461):232-236.
[23] Duncan SH,Hold GL,Barcenilla A,etal.Roseburia intestinalis sp.nov.,a novel saccharolytic,butyrate-producing bacterium from human faeces[J].Int JSyst Evol M icrobiol,2002,52(Pt 5):1615-1620.
[24] Khan KJ,U llman TA,Ford A,et al.Antibiotic therapy in inflammatory bow el disease:a systematic review and Meta-analysis[J].Am JGastroenterol,2011,106(4):661-673.
[25] Lemon KP,A rmitage GC,Relman DA,et al.M icrobiota-targeted therapies:an ecological perspective[J].Sci Transl M ed,2012,4(137):137rv5.[26] Gevers D,Kugathasan S,Denson LA,et al.The treatmentnaivem icrobiome in new-onset Crohn's disease[J].Cell Host M icrob,2014,15(3):382-392.
[27] W lodarska M,W illing B,Keeney KM,et al.Antibiotic treatment alters the colonic mucus layer and p redisposes the host to exacerbated Citrobacter rodentium induced colitis[J].Infect Immun,2011,79(4):1536-1545.
[28] Damman CJ,M iller SI,Suraw icz CM,et al.The m icrobiome and inflammatory bowel disease:is there a therapeutic role for fecal m icrobiota transplantation?[J].Am J Gastroenterol,2012,107(10):1452-1459.
[29] Shahinas D,Silverman M,Sittler T,et al.Toward an understanding of changes in diversity associated w ith fecalm icrobiome transplantation based on 16S rRNA gene deep sequencing[J].MBio,2012,3(5):e00338-12.
[30] Anderson JL,Edney RJ,W helan K.Systematic review:faecal m icrobiota transplantation in themanagement of inflammatory bow el disease[J].A liment Pharmacol Ther,2012,36(6):503-516.
[31] M organ XC,Huttenhower C.M eta'om ic analy tic techniques for studying the intestinalm icrobiome[J].Gastroenterology,2014,146(6):1437-1448.
Gutm icrobiota and inflammatory bowel disease
HE Lijuan1,ZHANG Jundong2,AN Maomao3,JIANG Yuanying1(1.New Drug Research and Development Center,School of Pharmacy,Second M ilitary Medical University,Shanghai200433,China;2.Shanghai Tenth People's Hospital,School of Medicine,Tongji University,Shanghai200072,China;3.School of Medicine,Tongji University,Shanghai200092,China)
Inflammatory bowel disease is a chronic inflammation of the intestinal tract,which comprises two primary forms of Crohn's disease(CD)and ulcerative colitis(UC).Decades of studies have revealed thatenvironmental factors,susceptibility genes,and gutmicrobiota are considered as themajor determinants for the induction of IBD.The combination of factors hasmade IBD as an appropriateand a high-priority platform for studying host-microbiome interactions.More recently,profiling studies of the intestinalm icrobe have associated pathogenesis of IBD w ith characteristic alterations in the composition of the intestinalmicrobiota,reinforcing the viewpoint that IBD results from the altered interplays between the host and intestinalmicrobe.the studies of the gut flora in IBD were reviewed andthemultiple effects of intestinalmicrobiota-dysfunction on the IBD were described.The progress of intestinalm icrobiota alterations w ith different therapeuticmethods in animalmodels and clinic trials were provided.
inflammatory bowel disease;intestinalmicrobe;interaction
R574;R975
A
1006-0111(2015)05-0385-05
10.3969/j.issn.1006-0111.2015.05.001
2014-11-13
2015-03-17
[本文编辑] 顾文华
何丽娟,硕士研究生.E-mail:advb_788@163.com
姜远英,教授,博士生导师.研究方向:抗感染药物药理研究.Tel:(021)81871357;E-mail:13761571578@163.com
