同源不同辐射抗拒NPC细胞间总甲基化水平及基因甲基化表达差异的探讨*
2015-05-16罗海丹王冬辉林伟达苏箔金赵存友杨惠玲中山大学中山医学院中山医学院病理生理学教研室广东广州50080南方医科大学基础医学院医学遗传学教研室广东广州5055
罗海丹,王冬辉,林伟达,苏箔金,赵存友,杨惠玲△(中山大学中山医学院,中山医学院病理生理学教研室,广东广州50080;南方医科大学基础医学院医学遗传学教研室,广东广州5055)
同源不同辐射抗拒NPC细胞间总甲基化水平及基因甲基化表达差异的探讨*
罗海丹1,王冬辉2,林伟达1,苏箔金2,赵存友3,杨惠玲2△
(中山大学1中山医学院,2中山医学院病理生理学教研室,广东广州510080;3南方医科大学基础医学院医学遗传学教研室,广东广州510515)
[摘要]目的:比较同源不同辐射抗拒能力的鼻咽癌细胞CNE2R/CNE2间总甲基化水平及其区域的差异。方法:采用赵存友博士改良的基于甲基化敏感酶全基因组甲基化法检测CNE2R/CNE2细胞全基因组甲基化水平;同时采用MeDIP-Seq测序法对CNE2R/CNE2细胞进行测序,分析比较DNA甲基化区域(主要包括6个基因功能元件即CDS、intron、downstream2k、upstream2k、3’UTR及5’UTR)的差异。结果: CNE2R细胞全基因组甲基化水平比CNE2细胞低约30%;在CNE2R和CNE2细胞中,虽然分布在6个基因功能元件上的峰值个数和峰值在各基因元件上的覆盖度均无明显差异,但2种细胞的6个基因功能元件上基因甲基化存在差异。结论:同源不同辐射抗拒能力的鼻咽癌细胞间总甲基化水平及基因甲基化存在差异,推测DNA甲基化的差异可能与鼻咽癌辐射抗拒相关。
[关键词]辐射抗拒;甲基化;鼻咽癌;基因元件
目前放疗仍为鼻咽癌( nasopharyngeal carcinoma,NPC)的首选疗法,但是放疗引发的辐射抗拒严重制约NPC病人5年生存率的提高[1],而NPC辐射抗拒机制至今尚未阐明。近年表观遗传调控特别是DNA甲基化因其作用位点广泛且调控结果可逆等特性而备受关注,已有研究报道DNA甲基化与肿瘤的放化疗抗拒相关,如Chen等[2]定量MSP检测发现头颈部肿瘤病人组织NEFL启动子CpG岛的超甲基化与顺铂治疗耐受正相关,而影响病人存活时间; Luzhna等[3]报道MCF-7/DOX细胞辐射抗拒与全基因组显著低甲基化相关。另外有研究提示NPC是一种高频率基因CpG岛甲基化的肿瘤[4-5],至于NPC辐射抗拒时细胞DNA甲基化变化如何值得深入探讨。本研究拟采用Zhao等[6]改良的基于甲基化敏感酶全基因组甲基化法和MeDIP-Seq测序法检测和比较NPC辐射抗拒细胞CNE2R和NPC辐射敏感细胞CNE2[7]的全基因组甲基化水平及甲基化区域(主要为6个基因功能元件即CDS、intron、downstream2k、upstream2k、3’UTR及5’UTR)的差异,为阐明NPC辐射抗拒机制、确立预测和逆转NPC辐射抗拒新标记物和靶点提供实验依据。
材料和方法
1实验材料
人低分化鼻咽癌细胞株CNE-2由中山大学肿瘤防治中心院提供,CNE2R细胞是本实验室在CNE2细胞基础上采用爬坡式放疗诱导生成的对放疗抵抗的细胞株。
Paired-End DNA Sample Prep kit购自Illumina; agnetic甲基化DNA免疫沉淀反应设备购自Diagenod; Covaris sonication system超声波反应器为Covaris产品; PCR Thermal Cycler基因扩增仪为ABI产品; NanoDrop 1000分光光度计为Thermo产品; Agilent 2100生物分析仪为Agilent产品;恒温均匀器和离心机为Eppendorf产品; Qubit核酸/蛋白定量系统为Invitrogen产品。
2方法
2.1细胞培养胰酶消化对数生长期的CNE-2R和CNE-2细胞,用含10%灭活胎牛血清、1×105U/L青霉素、1×105U/L链霉素的RPMI-1640培养液稀释细胞为2×108cells/L的细胞悬液,接种于10 mL的培养皿,每皿2×106个,并置于37℃、5% CO2和饱和湿度的培养箱中培养。
2.2CNE2R/CNE2细胞基因组DNA的制备和提取
培养48 h后弃上清,用PBS洗2次,每皿加1 mL
TRIzol使细胞裂解,转移至微量离心管中,加入200 μL PBS,吹打重悬。随后清除RNA,加入4 μL 100 g/L RNase A,旋涡混匀后室温放置2 min;再加入20 μL蛋白酶K,旋涡混匀;加入200 μL样品裂解液B,旋涡混匀后70℃孵育10 min。加入200 μL无水乙醇,旋涡混匀后将所得混合物加入到DNA纯化柱内,6 000×g离心1 min。弃废液收集管内液体,加入500 μL洗涤液I,6 000×g离心1 min。弃废液收集管内液体。加入600 μL洗涤液II,18 000×g离心1 min。弃废液,收集管内液体。进行以上离心步骤前需将纯化柱置于废液收集管上,弃废液后回收废液收集管。18 000×g离心1 min,以去除残留的乙醇。将DNA纯化柱置于一洁净的1.5 mL离心管上,加入50~200 μL洗脱液。室温放置1~3 min。18 000×g离心1 min,所得液体即为纯化得到的总DNA。洗脱液需要直接加至纯化柱管内柱面中央,使液体被纯化柱吸收。对提取的DNA采用1%的琼脂糖凝胶电泳进行检测。
2.3检测CNE2R/CNE2细胞的全基因组甲基化水平-FPDM鉴定对甲基化敏感的Hpa II核酸内切酶和对甲基化不敏感的Msp I核酸内切酶是同切酶,具有相同的靶序列CCGG,在酶切产生的5’端生成1 个CpG的突出序列,可作为互补链扩增的模板。分别取50~500 ng CNE2R/CNE2细胞DNA样本置于50 μL的混合溶液中进行酶切反应。混合溶液中包含1×107U/g Hpa II或Msp I DNA、1×buffer( 33 mmol/L三羟基氨甲烷醋酸盐、10 mmol/L醋酸镁、66 mmol/L乙酸钾和0. 5 mmol/L二硫苏糖醇)和0. 01% BSA,37℃保温12 h( CNE2R/CNE2细胞基因组DNA)。将酶切产生的DNA与DNA聚合酶及TAMRA-dCTP混合,在每条链上将添加1个TAMRA-dCMP,即每个酶切位点增添2个TAMRA-dCTP。取10 μL酶切的DNA置于1个含15 μL增链反应溶液的384孔的培养皿中。溶液包含1×PCR buffer,0. 75 U Taq DNA聚合酶( TaKaRa),0.56 nmol荧光TAMRA-dCTP。在57.8℃恒温箱中静置1 h。其中,Msp I核酸内切酶能识别甲基化的Cm5CGG序列而Hpa II核酸内切酶无法识别,因此,当该CCGG酶切位点中的CpG二核苷酸被甲基化时,Cm5CGG序列仅能被Msp I核酸内切酶识别并进行切割。使用Wallac VictorTM2V 1420多标记分析仪( PerkinElmer)通过FP对每孔中与DNA结合的TAMRA-dCMP进行测量,对扩增的DNA无需提纯。对空白FP阅读框进行校正,Hpa II和Msp I处理的样品的FP阅读框产生的甲基化的范围:甲基化( %) = ( 1-Hpa II/Msp I)×100%,其中Hpa II/Msp I代表Hpa II诱导的TAMRA-dCMP结合物与Msp I诱导的TAMRA-dCMP结合物校正后的空白FP阅读框的比值。
2.4MeDIP-Seq测序采用Diagenod公司的agnetic甲基化DNA免疫沉淀反应设备对基因组DNA进行甲基化处理,用Covaris sonication system超声波反应器将基因组DNA超声打断至100~500 bp片段,DNA片段末端修复、3’端加A碱基,连接测序接头,双链DNA变性成单链DNA。采用5-mC抗体对基因组进行沉淀富集,QPCR对抗体富集结果进行验证。PCR扩增后跑胶,切胶纯化,进行DNA片段大小选择,切取220~320 bp的片段回收,并用Agilent 2100 Analyzer定量,合格的文库行上机测序。将测序结果与参考基因组比对,比对上唯一位置的序列用于后续标准信息分析及个性化分析。
3统计学处理
统计MeDIP-Seq数据富集区域( Peak)在不同基因功能元件上的分布和差异,应用华大基因工程自制软件分别统计Peak在upstream2k、5’UTR、CDS、Intron、3’UTR、downstream2k等6个基因功能元件上的个数分布和覆盖度分布。同时比较peak在不同基因功能元件上分布的差异。
结果
1CNE2R/CNE2细胞的全基因组甲基化水平
通过对提取的DNA采用1%的琼脂糖凝胶电泳进行检测获得电泳条带,结果表明条带单一,纯度较高,可用于进一步实验。随后采用Zhao等[6]改良的新方法对CNE2R/CNE2细胞的全基因组甲基化水平进行测定,获得CNE2R/CNE2细胞的全基因组甲基化水平如图1所示。结果显示CNE2R细胞的全基因组甲基化水平与CNE2细胞相比大约下降30%,2者存在显著差异。

Figure 1.The genome-wide methylation levels of DNA in the CNE-2 cells and CNE-2R cells.*P<0. 05 vs CNE-2.图1 CNE-2和CNE-2R细胞DNA全基因组甲基化水平
26个基因功能元件上的峰值( peak)个数及覆盖度比较
计算CNE-2R和CNE-2细胞分布在upstream2k、5’UTR、CDS、intron、3’UTR、downstream2k等6个基因功能元件上的peak相关基因个数及覆盖度和差异见图2、3。结果表明CNE2R/CNE2两细胞株的基因组在upstream2k、5’UTR、CDS、intron、3’UTR、downstream2k等6个基因功能元件上的peak个数及覆盖度无明显差异。
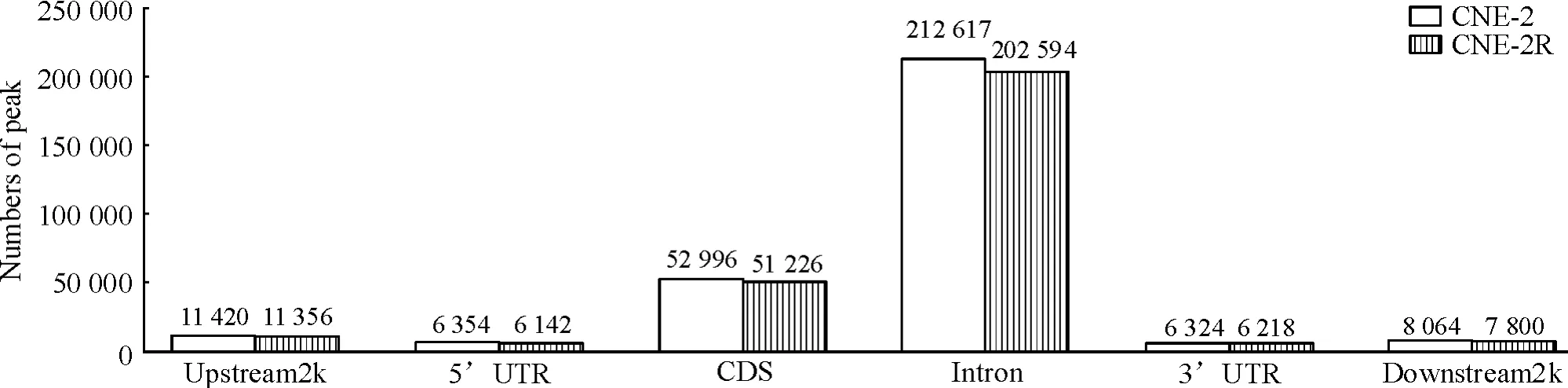
Figure 2.The numbers of peak in each gene function element of the DNA isolated from CNE-2 cells and CNE-2R cells.图2 分布在CNE-2和CNE-2R细胞各个元件上的peak个数
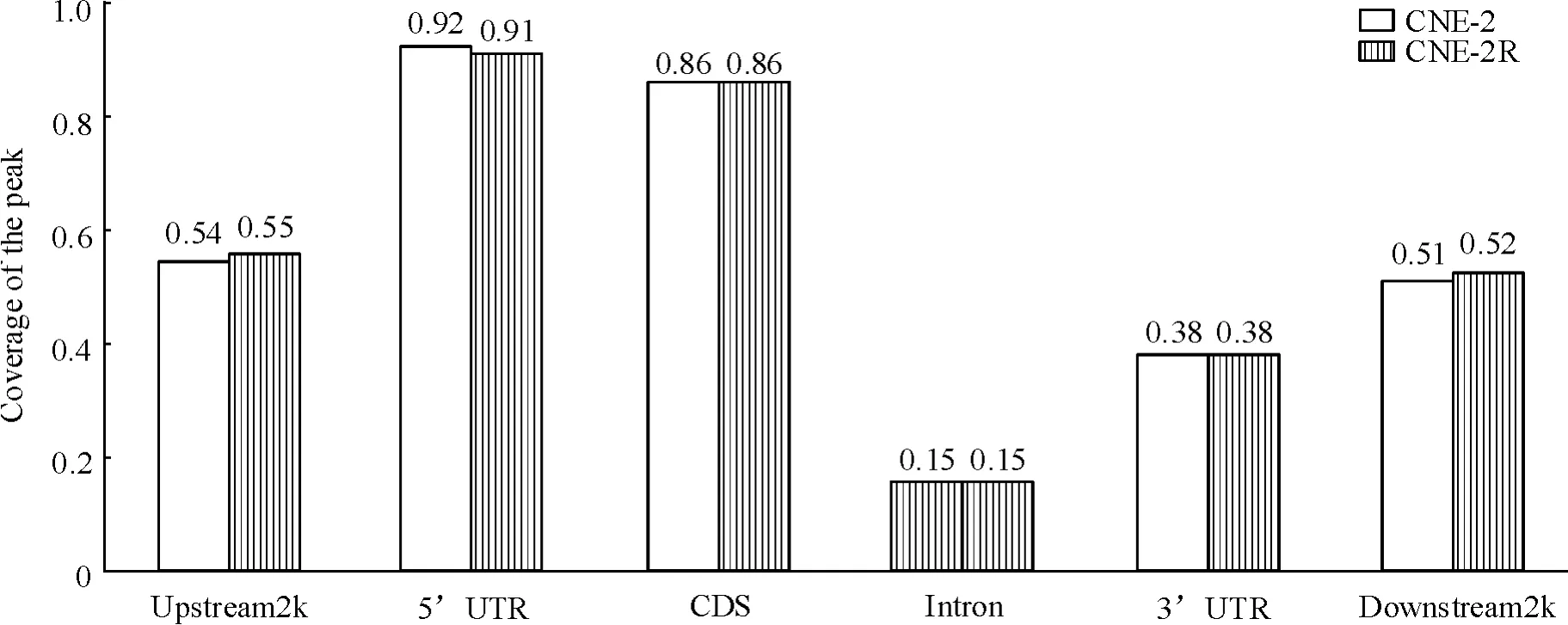
Figure 3.The coverage of the peak in each gene function element of the DNA isolated from CNE-2 cells and CNE-2R cells.图3 Peak在CNE-2和CNE-2R细胞中各基因元件上的覆盖度
3分析CNE-2R和CNE-2细胞间的peak相关差异基因
将2个样本的peak区域合并,统计合并后的区域中每个样品归一化后的reads个数并做χ2检验。根据区域内reads数的差异,我们将差异区域分为上升和下降2种趋势差异区域,如同一peak区域内CNE-2R的reads数高于CNE-2的基因即为up,反之为down。筛选条件为P<0. 01,2个样本在相同基因元件内均覆盖,且覆盖度的差异在2倍以上。结果表明CNE-2R和CNE-2细胞在6个基因功能元件上基因甲基化表达存在差异。CNE-2R和CNE-2细胞各基因功能元件上甲基化水平改变的基因个数存在差异,而差异基因个数差异不明显,见图4。
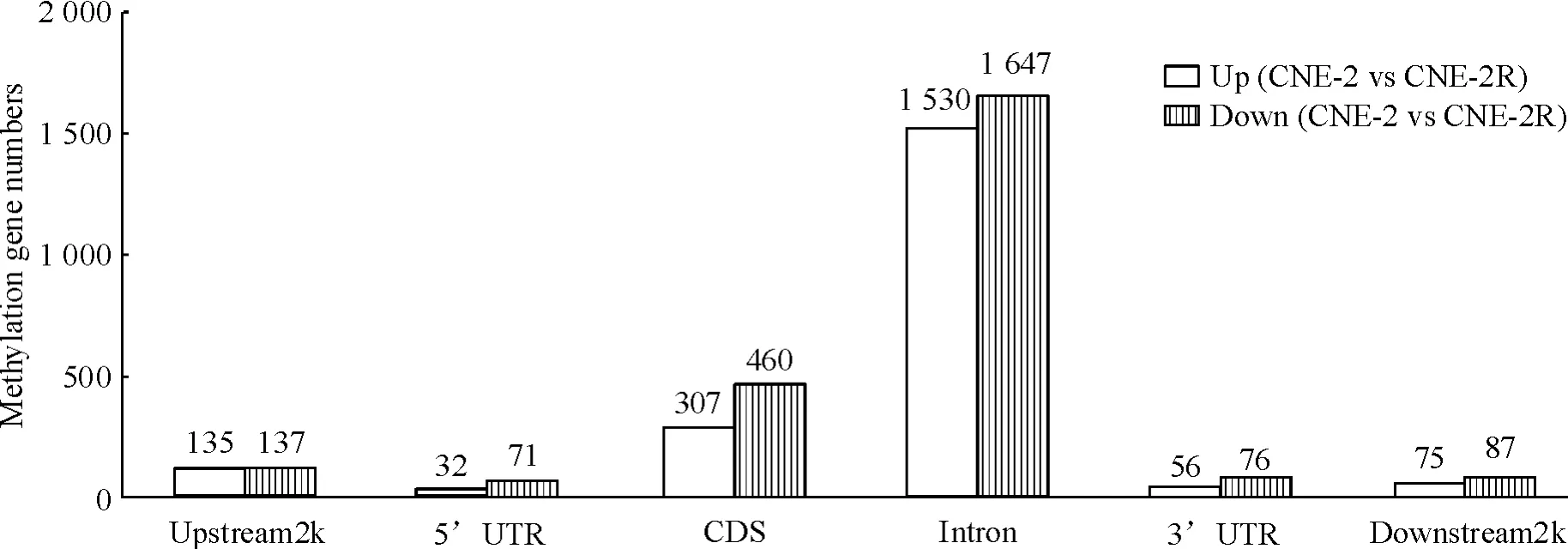
Figure 4.The numbers of genes altered on methylation in each gene function element in the CNE-2 cells and CNE-2R cells.图4 CNE-2和CNE-2R细胞各基因功能元件上甲基化水平改变的基因个数
讨论
目前制约鼻咽癌生存率的主要是辐射抗拒,故阐明NPC辐射抗拒机制并逆转辐射抗拒尤为迫切。表观遗传学包括DNA甲基化、microRNA、组蛋白修饰、染色质重塑及非编码RNA调控等,近年来表观遗传学尤其是DNA甲基化与肿瘤的相关研究备受关注。已有研究表明DNA甲基化与肿瘤的放化疗抗拒相关,如Zhou等[8]证实恶性胶质瘤中SOCS1超甲基化致其表达减少,通过ERK-MAPK信号通路介导放疗抵抗;在口腔鳞状细胞癌中放疗可提高RASSF1A/RASSF2A甲基化水平,激活Ras相关通路而诱导放疗抵抗[9];张晓伟[10]通过实验表明DNA甲基化水平与紫杉醇耐药形成密切相关,DNA异常甲基化后的耐药相关基因表达改变导致肿瘤耐药的形成。但尚未见全基因组甲基化水平异常与肿瘤辐射抗拒的相关报道。
本研究采用Zhao等[6]改良的基于甲基化敏感酶全基因组甲基化法对CNE-2R与CNE-2细胞全基因组甲基化水平进行检测和比较,该法步骤较为简便,且可直接在多孔培养皿中对亚微克剂量的DNA进行测量,更适用于生物和临床实验中高通量、多样本的测定需要。检测结果显示CNE2R细胞全基因组甲基化水平与CNE2细胞的相比大约低30%,表明辐射抗拒差异的细胞间全基因甲基化水平存在差异,推测该差异可能与细胞辐射抗拒差异相关。
同时,我们采用MeDIP-Seq测序法对CNE2R/ CNE2细胞进行测序,验证CNE2R/CNE2细胞的全基因组甲基化水平及差异,并分析比较DNA甲基化区域的差异,其中主要6个基因功能元件包括CDS、intron、downstream2k、upstream2k、3’UTR及5’UTR。结果表明CNE2R/CNE2两细胞株基因组在upstream2k、5’UTR、CDS、intron、3’UTR、downstream2k 等6个基因功能元件上的peak个数及覆盖度无明显差异,与CNE2R/CNE2细胞的全基因组甲基化水平存在的差异不相符。这可能与本方法的局限性有关,MeDIP-Seq测序法测序时主要针对以上6个基因功能元件,显示结果仅代表6个基因功能元件的甲基化差异,而非全基因组内所有基因甲基化的差异。如何克服这一局限性,定位到具体甲基化基因的差异也是我们今后研究的重点。另外,MeDIP-Seq测序法测得6个基因功能元件上的peak个数及覆盖度为各个功能元件的整体水平,通过分析CNE-2R和CNE-2细胞间的peak相关差异基因和差异,结果表明6个基因功能元件上基因甲基化表达存在上调和下调差异,但上调和下调基因的个数大致接近,因此可能在各个功能元件表现为无差异。
本研究各项结果表明同源不同辐射抗拒NPC细胞间总甲基化水平及基因甲基化表达存在差异,推测DNA甲基化的差异可能与鼻咽癌辐射抗拒相关。然而,DNA甲基化与NPC辐射抗拒之间是否存在相关关系,特别是寻找某基因的甲基化水平与NPC辐射抗拒相关的作用位点及相关通路仍需要深入探讨。
[参考文献]
[1]Qin HD,Shugart YY,Bei JX,et al.Comprehensive pathway-based association study of DNA repair gene variants and the risk of nasopharyngeal carcinoma[J].Cancer Res,2011,71( 8) : 3000-3008.
[2]Chen B,Chen J,House MG,et al.Role of neurofilament light polypeptide in head and neck cancer chemoresistance [J].Mol Cancer Res,2012,10( 3) : 305-315.
[3]Luzhna L,Kovalchuk O.Modulation of DNA methylation levels sensitizes doxorubicin-resistant breast adenocarcinoma cells to radiation-induced apoptosis[J].Biochem Biophys Res Commun,2010,392( 2) : 113-117.
[4]He D,Zeng Q,Ren G,et al.Protocadherin8 is a functional tumor suppressor frequently inactivated by promoter methylation in nasopharyngeal carcinoma[J].Eur J Cancer Prev,2012,21( 6) : 569-575.
[5]Tao Q,Chan AT.Nasopharyngeal carcinoma: molecular pathogenesis and therapeutic developments[J].Expert Rev Mol Med,2007,9( 12) : 1-24.
[6]Zhao C,Xue H.A simple method for high-throughput quantification of genome-wide DNA methylation by fluorescence polarization[J].Epigenetics,2012,7 ( 4) : 335-339.
[7]曲昌菊,王旭丹,杨惠玲,等.相同遗传背景不同辐射抗拒鼻咽癌细胞miRNA差异表达的研究[J].中国病理生理杂志,2009,25( 3) : 484-488.
[8]Zhou H,Miki R,Eeva M,et al.Reciprocal regulation of SOCS 1 and SOCS3 enhances resistance to ionizing radiation in glioblastoma multiforme[J].Clin Cancer Res,2007,13( 8) : 2344-2353.
[9]Huang KH,Huang SF,Chen IH,et al.Methylation of RASSF1A,RASSF2A,and HIN-1 is associated with poor outcome after radiotherapy,but not surgery,in oral squamous cell carcinoma[J].Clin Cancer Res,2009,15 ( 12) : 4174-4180.
[10]张晓伟.DNA甲基化在鼻咽癌紫杉醇耐药形成及逆转耐药机制中的初步研究[D].湖南:中南大学,2012.
Differences of genome-wide methylation level and methylated genes between nasopharyngeal carcinoma cells in same genetic background but different radiation resistance
LUO Hai-dan1,WANG Dong-hui2,LIN Wei-da1,SU Bo-jin2,ZHAO Cun-you3,YANG Hui-ling2
(1Zhongshan School of Medicine,2Department of Pathophysiology,Zhongshan School of Medicine,Sun Yat-sen University,Guangzhou 510080,China;3Department of Medical Genetics,School of Basic Medical Sciences,Southern Medical University,Guangzhou 510515,China.E-mail: yanghl@ mail.sysu.edu.cn)
[ABSTRACT]AIM: To compare the differences of the genome-wide methylation levels and methylated regions between nasopharyngeal carcinoma ( NPC) cells in the same genetic background but different radiation resistance ( CNE-2 cells and CNE-2R cells).METHODS: Using the method which was developed by Doctor Zhao Cun-you,based on using methyl-sensitive restriction enzyme to measure the genome-wide methylation levels.In addition,MeDIP-Seq was used to analyze the methylated regions in 6 gene functional elements,including the upstream 2k sequence,5’UTR,coding sequence,intron,3’UTR and downstream 2k sequence,between CNE-2 cells and CNE-2R cells.RESULTS: The genomewide methylation level was approximately 30% lower in CNE-2R cells than that in CNE-2 cells.No obvious difference on the amount of genes and the coverage of the peak in the 6 gene functional elements was observed.However,the methylation pattern of plentiful genes had altered in the gene function elements.CONCLUSION: The genome-wide methylation levels and methylated regions between NPC cells in the same genetic background but different radiation resistance were quite different,indicating that the DNA methylation may be associated with NPC radioresistance.
[KEY WORDS]Radiation resistant; Methylation; Nasopharyngeal carcinoma; Gene element
通讯作者△Tel: 020-87332268; E-mail: yanghl@ mail.sysu.edu.cn
*[基金项目]国家自然科学基金资助项目( No.81071837; No.81372410) ;广东省自然科学基金重点项目( No.92510089; No.01000005) ;国家大学生创新训练计划项目( No.201310558074; No.201210558076) ;广东省大学生创新训练项目( No.1055813171)
[收稿日期]2014-12-15[修回日期]2015-02-04
[文章编号]1000-4718( 2015)07-1300-05
[中图分类号]R363. 1+21
[文献标志码]A
doi:10.3969/j.issn.1000-4718.2015.07.026
