酵母剪接体高分辨率三维结构的解析
2015-05-12段艳芳
段艳芳(本刊记者)
酵母剪接体高分辨率三维结构的解析
段艳芳(本刊记者)
2015年8月21日,清华大学生命科学学院施一公教授带领的研究团队在美国《科学》杂志上同时发表了两篇论文——《3.6 Å的酵母剪接体结构》(Structure of a yeast spliceosome at 3.6-Angstrom resolution)和《前体信使RNA剪接的结构基础》(Structural basis of pre-mRNA splicing),介绍了通过单颗粒冷冻电子显微技术(冷冻电镜)解析的酵母剪接体(spliceosome)在3.6 Å分辨率的三维结构,并在此结构基础上进行详细分析,阐述了剪接体对前体信使RNA (pre-mRNA)执行剪接的基本工作机理。该项工作将分子生物学的“中心法则”在分子机理的研究上大幅度向前推进,是我国科学家在生命科学领域做出的重大原创性突破。
首次获得剪接体近原子分辨率的高清结构图
剪接是转录后修饰的重要环节,是指在真核生物RNA中将非编码的内含子(intron)序列去除,将编码的外显子(exon)序列连接起来的过程。真核生物编码蛋白质的前体信使RNA剪接过程需要剪接体的参与。剪接体是一个动态的、复杂的RNA-蛋白质大分子复合物,沉降系数大约为60S,由U1、U2、U4、U5、U6等5个核内小核糖核蛋白(snRNP)和多种其他蛋白质所组成。每个snRNP包含一条核内小RNA(snRNA)和数个或十多个蛋白质。更为重要的是,伴随着不同snRNP的结合与解离,剪接体行使拼接功能是一个复杂的、动态的拼接过程。首先,U1结合至内含子的5′拼接点形成复合物E,然后U2结合至分支点(branch site,BS)形成复合物A,接下来U4/U6. U5三聚snRNP装配上去形成复合物B。伴随着U1、U4的陆续解离,剪接体最终成为处于激活状态的复合物Bact和具有催化活性的复合物B*。U6/U2催化酯转移反应,5′拼接点断开,内含子形成套索结构,标志着第一阶段催化反应的完成,形成包含有U2、U5、U6和内含子套索的复合物C。接下来完成第二阶段的酯转移反应,内含子3′位点断开,外显子连接在一起(图1)。
施一公团队解析的是包含有U2和U5 snRNP,NTC,NTC相关蛋白,U6 snRNA,以及一个RNA内含子套索的剪接体三维结构。该复合物包括源于37个蛋白质的10 574个氨基酸和4条RNA分子,总分子量高达130万Da。从酵母剪接体高分辨率的三维结构中可以看出,剪接体的外形轮廓十分不对称,各个蛋白相互缠绕,形成了分子量和体积都很巨大的复合物(图2)。U5 snRNP起到了中心支架的作用,U6和U2 RNA相互缠绕形成催化中心。剪接体本质上是蛋白质指引的核酶,控制关键RNA的位置和距离,从而控制剪接反应。
这是首次在近原子分辨率上看到了剪接体的细节,而且解析对象是在真核生物体内真正发挥作用的完整的剪接体,因此意义重大。论文发表后,施一公表示这项研究成果的意义很可能超过了自己过去25年科研生涯中所有研究成果的总和。
自1977年Richard J. Roberts和Phillip A. Sharp分别发现真核生物断裂基因,并因此荣获1993年诺贝尔生理学或医学奖以来,对RNA剪接的研究不断深入,剪接体的结构解析是分子生物学里最热门的研究之一。国际上多家实验室也进行着类似的研究,如剑桥大学Kiyoshi Nagai团队、德国的Reinhard Lührmann团队、美国马萨诸塞大学医学院的Melissa Moore团队、哈佛大学的Robin Reed团队等,竞争非常激烈。例如,此前已有多个课题组利用冷冻电镜获得了一系列剪接体复合物三维结构,只是分辨率在20~50 Å,无法看清细节。2015年6月24日,剑桥大学分子生物学实验室的日裔学者Kiyoshi Nagai博士的课题组在《自然》杂志上发表文章,解析了酿酒酵母的U4/U6.U5三聚snRNP在5.9 Å的分辨率的三维结构,一度引起轰动。Nagai的最新工作被称为近原子尺度的结构研究。施一公团队不但将分辨率提高至3.6 Å,可以将绝大部分氨基酸看得清楚楚楚,而且解析对象是完整的剪接体,因而是一项重大突破。该成果阐释清楚了剪接体对前体mRNA执行剪接的基本工作原理,不仅初步解答了这一基础生命科学领域长期以来备受关注的核心问题,也为进一步揭示与剪接体相关疾病的发病机理提供了结构基础和理论指导。

图1 剪接体行使pre-mRNA剪接功能是个复杂的、动态的过程[3]。施一公团队解析了完成第一步催化反应,包含U2、U5、U6 snRNP,内含子套索结构和其他蛋白质的剪接体的三维结构
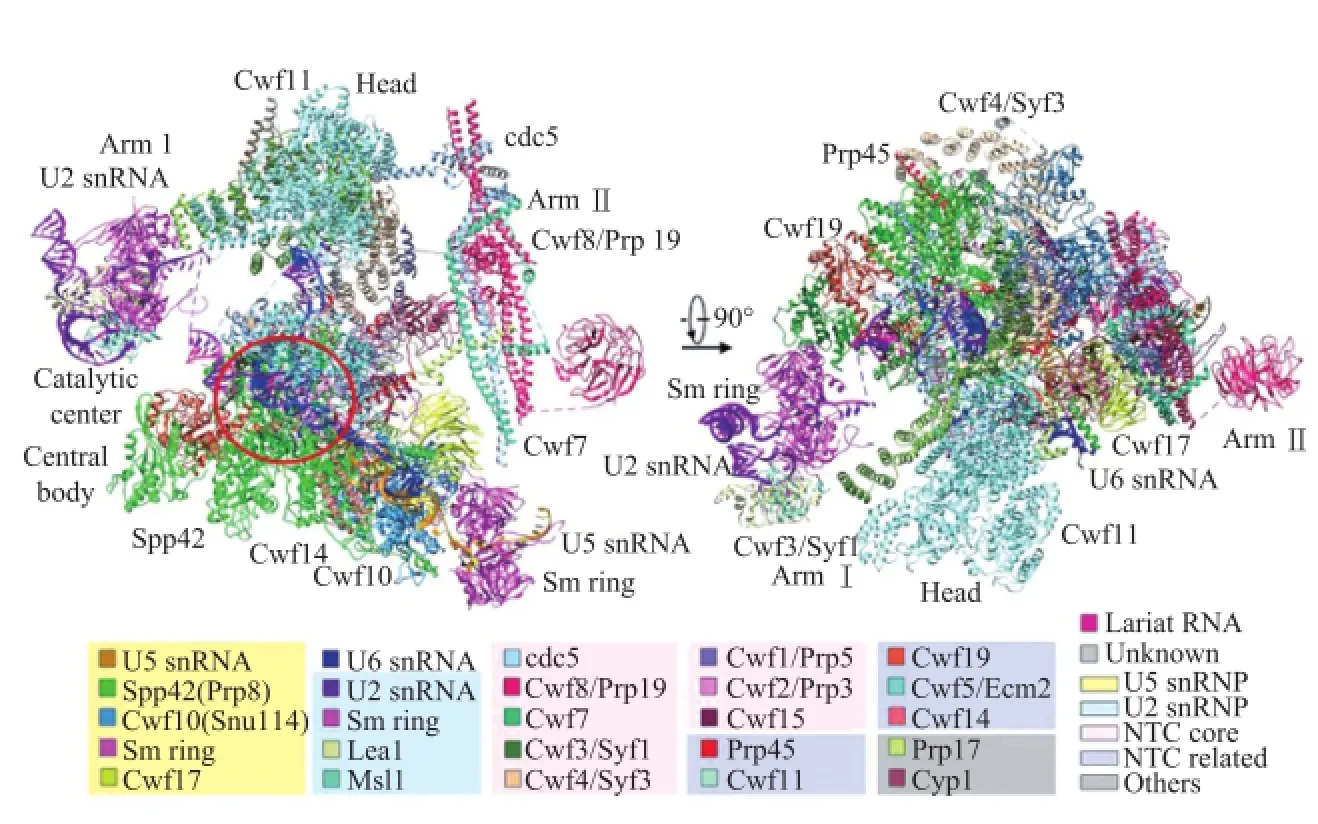
图2 施一公团队解析的酵母剪接体在3.6 Å分辨率的三维结构图[1]
对RNA剪接研究具有重要的推动作用
RNA剪接对真核生物基因表达调控具有重要的意义。不编码的内含子序列通过剪接被精确而高效地去除,才能被翻译成具有正常功能的蛋白质,而且越是高等的生物,内含子越多,剪接也越复杂。更为重要的是,真核生物中还普遍存在着选择性剪接,即一个基因的前体mRNA可以形成多种mRNA产物,从而形成不同的蛋白质,发挥不同的生物学功能。选择性剪接显著提高了基因组的复杂性,是基因表达调控的重要方式。例如,昆虫的性别决定即由选择性剪接级联反应调控。剪接是一个至关重要的生物学机制——至少15%的人类疾病是由于剪接错误所致。例如,脊髓性肌萎缩(一种影响运动神经的常染色体隐性遗传病)和色素性视网膜炎(一种会导致失明的常见疾病),均与RNA剪接不能正常进行密切相关。
通过间接实验,科学家对剪接体如何发挥作用已经有了比较深入的了解。这一次是实实在在地“看到”剪接体的各个部分,不同snRNP如何结合在一起并行使功能的,把RNA剪接相关研究向前推进了一大步。近原子分辨率剪接体三维结构的获得,可以把大部分生化数据连在一起,能够很好地解释过去的数据,也可以预测将来的实验结果,对剪接体功能方面的研究也具有重要的推动作用。
据清华大学官网,施一公本人表示“还要继续推进这一项基础研究工作,并且下一步的工作重点是把不同剪接体相互间不同的地方看清楚,从而阐述内含子被去除、外显子被连接在一起的分子机制。”
成果从哪里来
2009年施一公带领杭婧、万蕊雪博士和闫创业博士后进入剪接体领域,开始致力于剪接体及其相关复合物的结构生物学研究。他们在2014年初首次报道了剪接体复合物中重要蛋白质Lsm蛋白七聚体以及其RNA结合状态下的晶体结构。这一成果被《自然》杂志刊发后,课题组继续聚焦于极富挑战性的攻坚战——完整剪接体的结构生物学研究,终于在2015年5月份取得重大突破——捕获了真核细胞剪接体复合物的高分辨率空间三维结构。
剪接体是结构生物学领域公认的难题,是细胞内最后一个待解析结构的超大复合体。一个年轻的团队在短短几年实现重大突破,原因何在呢?应该说机会是留给有准备的人的,这是多种因素共同作用的结果,下面介绍其中几个比较重要的因素。
首先,技术突破给科研带来创新机会。如果没有冷冻电镜技术的革新,就完全不可能得到剪接体近原子水平分辨率的结构。一直以来,X射线晶体衍射、核磁共振方法和冷冻电镜技术是研究生物大分子晶体结构的主要方法。核磁共振方法只能解析小分子量的蛋白质和核酸结构。X射线晶体衍射的分辨率较高,但对样品浓度要求较高,需要筛选合适的晶体生长条件,形成晶体后方能分析其结构。对于剪接体这样动态的、复杂的细胞机器,获得其晶体几乎是不可能的。冷冻电镜技术逐渐发展成熟,将结构生物学的研究对象由纳米级扩增至百纳米甚至微米级,尤其是最近几年冷冻电镜技术取得革命性的突破,数据采集系统和图像处理技术都有了很大发展,冷冻电镜的分辨率大大提高,已经接近X射线晶体衍射的分辨率。2014年中国科学院生物物理所李国红课题组解析出30 nm染色质纤维左手双螺旋结构,也是利用冷冻电镜技术做出的重要成果(李国红研究员曾为我刊撰文《30 nm染色质纤维高级结构的研究进展》介绍了该成果[4])。因此,冷冻电镜技术的突破和清华大学冷冻电镜平台的建设为解析剪接体这一巨大、动态、复杂的核酸蛋白复合体的结构奠定了基础。
其次,实验材料的正确选择功不可没。酵母易于培养和操作、增值快,便于大量表达目的蛋白,是分子和细胞生物学中最常用的实验材料,也是研究真核生物生命活动的首选。剑桥大学Kiyoshi Nagai博士课题组解析的U4/U6.U5三聚snRNP三维结构,所选用的实验材料是酿酒酵母(Saccharomyces cerevisiae),而施一公团队选用的是粟酒裂殖酵母(Schizosaccharomyces pombe)。中国科学院植物生理与生态研究所徐永镇研究员介绍说:“虽然两种酵母的基因数差别不大,但内含子数目差异非常大,酿酒酵母的内含子数为377个,粟酒裂殖酵母则含有约6 000个内含子。更为重要的是,在酿酒酵母中,剪接是个动态的过程,U4/U6.U5三聚snRNP相对较多;而粟酒裂殖酵母因为存在某种突变,U2、U5和U6复合物天然地大量富集。施一公团队选择粟酒裂殖酵母体现了其智慧,也是能获得剪接体高分辨率三维结构的重要因素。”
另外,年轻的团队为此课题付出了艰辛的努力。施一公团队掌握了极为成熟的样品处理方法,能够让蛋白质性质稳定,从而适宜在电镜下观察,这是该课题组能够陆续取得科技成果的重要原因。比如,此次解析剪接体的三维结构,万蕊雪和杭婧对样品百般驯化,让它们适合电镜观察;闫创业巧妙地革新了计算软件,可以让所有重要的颗粒都被再挑选出来。坚持不懈的努力和细节处的创新同样闪着光辉,值得学习。
致谢 感谢中国科学院植物生理与生态研究所徐永镇研究员对本文的帮助。
(2015年11月20日收稿)
[1] YAN C Y, HANG J, WAN R X, et al. Structure of a yeast spliceosome at 3.6-angstrom resolution [J]. Science, 2015, 349(6253): 1182-1191.
[2] HANG J, WAN R X, YAN C Y, et al. Structural basis of pre-mRNA splicing [J]. Science, 2015, 349(6253): 1191-1198.
[3] WILL C L, LÜHRMANN R. Spliceosome Structure and Function [J]. Cold Spring Harb Perspect Biol, 2011, 3: a003707.
[4] 董立平, 陈萍, 李国红. 30 nm染色质纤维高级结构的研究进展 [J]. 自然杂志, 36(4): 274-279.
(编辑:沈美芳)
Structure of a yeast spliceosome at high resolution
DUAN Yanfang
10.3969/j.issn.0253-9608.2015.06.011
†通信作者,E-mail:yfduan@shu.edu.cn
