降解组测序技术在玉米雌穗发育相关miRNA靶基因中的应用
2015-05-10赵晓锋吕盼晴付忠军许蒙蒙李慧敏杨慧丽
赵晓锋,吕盼晴,张 珂,2,付忠军,3,许蒙蒙,彭 倩,李慧敏,杨慧丽,丁 冬
(1.河南农业大学农学院,河南 郑州450002;2.石河子大学,新疆石河子832000;3.重庆市农业科学院,重庆401329)
MicroRNAs(miRNAs)为21~23个核苷酸(nt)片段的、非编码的、相对分子质量较小的RNA,在转录后水平基因表达调控中起关键作用,包括生物代谢[1]、激素反应[2]、表观遗传控制的转座因子[3]、生物胁迫[4]和非生物胁迫[5]等多个方面.植物miRNAs介导的基因调控涉及植物发育多个过程,如器官分化[6]、叶片生长[7]、性别决定[8]、雌 雄 不 育[9]、果 核 形 成[10]、子 粒 灌 浆[11]等.MiRNAs主要通过切割靶mRNA或抑制靶mRNA的翻译实现转录后水平调控[12].通过芯片技术和高通量测序可以快速高效地检测并鉴定miRNA,而对于miRNA剪切的靶基因鉴定常使用低通量的5’-RACE技术.降解组测序技术(degradome Sequencing)利用植物miRNA在调控靶基因的表达时通常与靶mRNA进行几乎完全的配对的特点,结合高通量测序技术、生物信息学分析和RACE验证,能够快速高效鉴定miRNA的靶基因.降解组测序技术已应用于拟南芥(Arabidopsis thaliana)[13]、小立碗藓(Physcomitrella patens)[14]、水稻(Oryza sativa)[15]、大豆(Glycine max)[16]、葡萄(Vitis vinifera)[17]、黄瓜(Cucumis sativus)[18]、马铃薯(Solannum tuberosum)[19]、海 岛 棉 (Gossypium barbadense)[20]、泡 桐 (Paulownia australis)[21]及 玉 米(Zea mays)[22]等多种植物miRNA靶基因的检测。本研究采用降解组测序的方法,分别对昌7-2,郑58等2个玉米自交系的雌穗发育5个关键时期取样,并混样进行测序分析,以鉴定出与玉米雌穗发育相关miRNAs的靶基因,为进一步研究miRNAs在玉米雌穗发育过程中的调控作用奠定基础。
1 材料与方法
1.1 试验材料
试验材料选用2013年4月种植在河南农业大学郑州科教园区(平均气温14.3℃,年均降雨量为640.9 mm)的昌7-2,郑58等2个玉米自交系材料,每个自交系选生长锥未伸长期,生长锥伸长期,小穗分化期,小花分化期,性器官形成期等5个时期的雌穗进行取样,每个时期取样量为30个,取样后放在-80℃冰箱中保存。
1.2 雌穗组织总RNA提取
用液氮将样品在研钵中研磨至细碎,转移到用DEPC处理过的 1.5 mL Eppendorf管中,以 100 mg∶1 mL(样品:TRIzol)的量向管中加入TRIzol溶液,充分摇匀后12 000 r·min-1离心10 min;收集上清液到新的离心管中,加入1/5体积氯仿充分混匀,室温放置 5 min 后,12 000 r·min-1离心 10 min,收集上清液;再加等体积的异丙醇,放在-20℃冰箱过夜。第2天12 000 r·min-1离心10 min后,倒掉废液,轻微离心,用移液枪吸除管底残液,自然干燥10 min;RNA沉淀溶于50 μL DEPC水中,溶解完全后对RNA进行质量检测。
1.3 文库构建与测序
对检测后的RNA进行降解组测序文库的构建:
1)miRNA靶基因mRNA片段的分选,将3’接头(adapter)与靶基因mRNA片段的3’端退火互补,获得含有3’接头-mRNA连接产物的连接混合物;
2)将步骤1所获的连接混合物中的3’接头-mRNA连接产物连上5’接头反转录获得两端均带有接头的靶基因mRNA片段;
3)miRNA靶基因cDNA的扩增:以两端均带有接头的靶基因mRNA片段为模板,进行PCR扩增,凝胶电泳分离PCR产物,切取预期大小分子并回收,得到靶基因mRNA片段的降解组测序文库,进行高通量测序。
1.4 数据分析
高通量测序得到的数据中去除接头序列后得到目的序列,应用CleaveLand分析软件将目的序列与玉米的genome进行比对,记录匹配上的序列舍弃未比对上的序列,通过匹配可以得到被切割mRNA的完整序列信息。计算机分析结果可以直观地显示在mRNA序列的某个位点会出现一个波峰,而该处正是候选的miRNA剪切位点。为了进一步确定miRNA的靶基因,又基于植物miRNA的切割常常发生在互补位点的第10或11位核苷酸上,因此,取波峰上游和下游各15 nt核苷酸序列产生1个30 nt的序列(t-signature),将该序列反向互补并与玉米miRNA数据库去匹配,符合要求的匹配则为确定了的miRNA靶基因。
1.5 Real-time PCR 验证
选取昌7-2,郑58雌穗在小穗分化期和小花分化期的花序分生组织(inflorescence meristems,IM)分化为成对小穗分生组织(spikelet pair meristems,SPM)、成对小穗分生组织分化为小穗分生组织(spikelet meristems,SM),小花分生组织(Floral meristems,FM)3 个阶段,对 miR159,miR160,miR167,miR169,miR172a,miR172e,miR390 等 7个miRNAs与其靶基因进行了Real-time PCR验证。Real-time PCR反应选用 RR820A试剂盒(Takara,日本)SYBR Green I作为荧光染料,Actin2作为内参基因,用Bio-rad IQ5荧光定量PCR仪进行扩增,结果数据用2-△△t法进行处理。
2 结果与分析
2.1 保守miRNA的靶基因及其功能分类
降解组测序后将miRNA序列与mRNA序列的配对预测结果和降解组密度文件的结果相结合进行靶基因的精确预测,共发现16个miRNA家族的95个miRNAs对应47个靶基因(P<0.1)(表1)。
根据其生物学功能,对测序检测到的95个miRNAs作用的47个靶基因进行Gene Ontology(GO)分类,发现处于细胞核和CCAAT结合因子复合体的靶基因占78.5%(图1-A),与 miRNA主要在基因转录后水平进行调控相一致;参与转录、DNA依赖、转录调控、细胞分化的靶基因占53.2%(图1-B),这些靶基因还与花器官发育、种子发育、细胞分裂、分生组织保持、根毛细胞生长、硫酸盐吸收、生长素信号转导等多种生物过程密切相关;在分子功能方面,与DNA结合、特殊序列DNA结合、金属离子DNA结合的靶基因占72.9%,同时参与与蛋白结合、蛋白活性、三磷酸鸟苷(GTP)结合、三磷酸鸟苷(GTP)、翻译延伸因子活性调控(图1-C)。
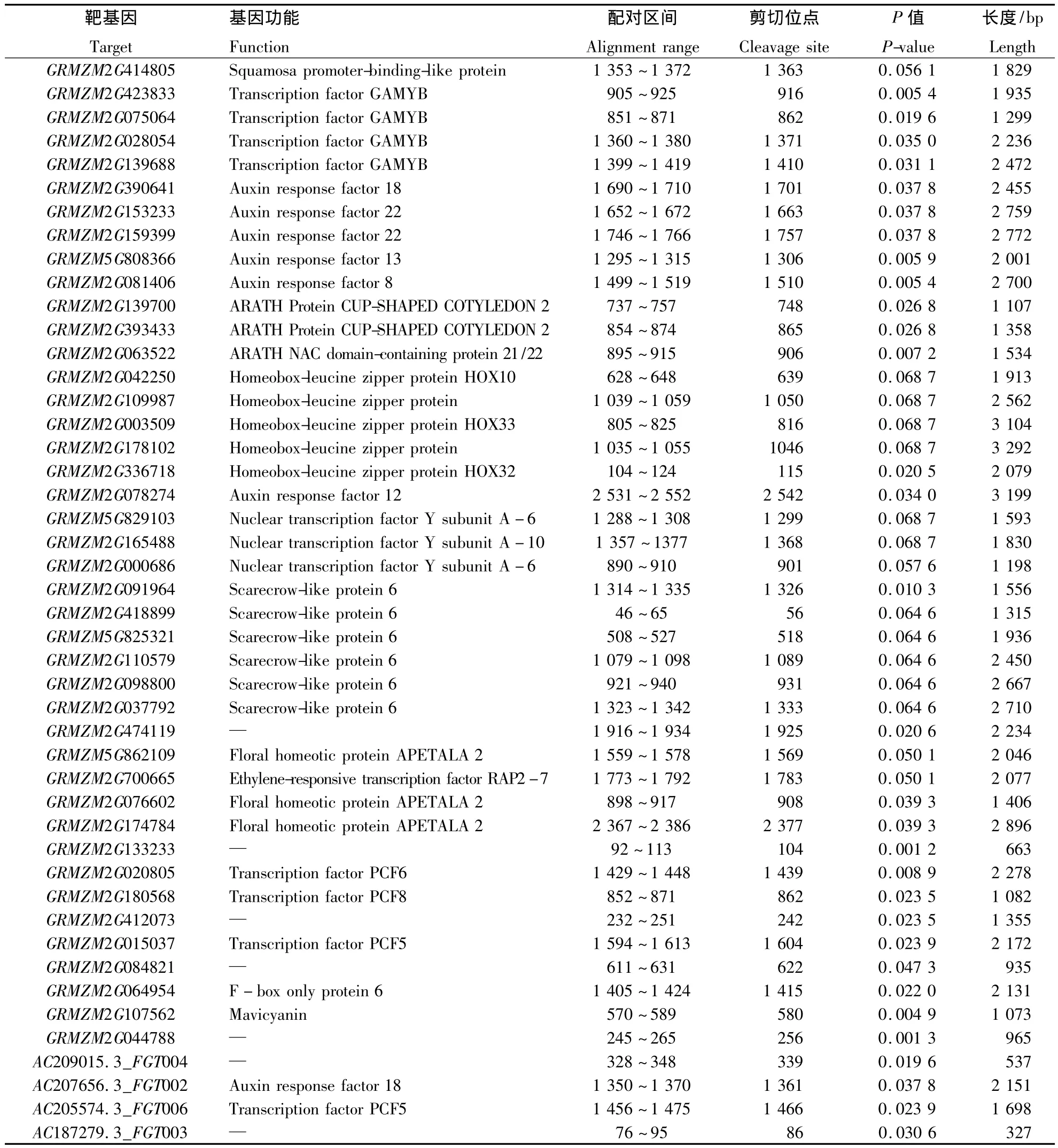
表1 昌7-2、郑58雌穗发育相关的miRNA靶基因Table 1 The target genes related to ear development of Chang 7-2 Zheng 58

图1 玉米雌穗发育相关miRNA相应靶基因GO统计Fig.1 The dereloping maize ear relevant miRNA target genes GO list
2.2 miRNA及靶基因的表达图谱
降解组测序数据分析表明,每个miRNA对应的转录本一般为多个,其中检测到miR159的转录本为7个,参与细胞分化、花器官发育、DNA结合等过程;miR160对应转录本为7个,与蛋白二聚反应活性、生长素介导的信号转导通路等过程相关;而miR167对应转录本仅1个,与蛋白二聚反应活性,生长素介导的信号转导通路等相关;miR169对应转录本为20个,与特异序列DNA结合,转录因子活性相关;miR172a,miR172e对应转录本各7个,参与细胞分化、种子发育、花器官发育、分生组织保持等过程;miR390有3个转录本,其功能未知(表2)。
选 中 miR159,miR160,miR167,miR169,miR172a,miR172e,miR390 等7 个 miRNAs及其靶基因构建其在昌7-2与郑58中的表达图谱(图2)。在昌7-2中miR159呈上升趋势,而T159没有明显变化;miR160,miR167,miR172a呈下降趋势,而T160先下降后上升,T167,T172a先上升后下降。在郑58中miR169呈上升趋势,T169呈下降趋势;miR159,miR167,miR390没有明显变化,而T159,T390呈上升趋势,T167先上升后下降;miR172a呈下降趋势,而T172a呈上升趋势。表明在雌穗发育过程中存在着复杂的miRNAs调控,miRNAs与其靶基因不存在简单的负线性调控关系,并且在不同自交系间也存在着较大的表达调控差异。

表2 7个miRNAs对应的靶基因Table 2 The target genens of the seven miRNAs
3 结论与讨论
降解组测序技术结合了高通量测序技术与生物信息学分析各自的优势,对细胞或组织中miRNA介导的降解的mRNA剪切片段进行深度测序分析,能够高效准确筛选出miRNA的靶基因,为研究miRNA以及其对应的靶基因的相互关系提供了有效的手段。采用降解组测序技术,检测到在玉米雌穗的发育过程中16个miRNA家族95个miRNAs对应的47个靶基因,多数存在于细胞核内,功能多为转录翻译的DNA结合蛋白,参与转录调控,与生长激素密切相关。这与在拟南芥及水稻中已经发现或者预测的结果相似[23-26]。
其中一些miRNA已被证明是涉及介导植物激素生长素响应信号转导途径,例如,在拟南芥中,miR159被发现特异剪切MYB(v-myb avian myeloblastosis viral oncogene homolog)家族转录因子MYB33和MYB65的转录本,以降低植物对脱落酸(abscisic acid,ABA)的敏感性[27],这可能有利于植物的生长发育,本研究测定的miR159靶基因多数也为转录调控因子MYB。拟南芥中miR160作用的靶基因为生长素响应因子基因(auxin response factors,ARF)ARF10和 ARF16,抑制植物的向性生长[28],在玉米自交系昌 7 -2,郑 58 中,miR160 靶标为 ARF8,ARF13,ARF18,ARF22,miR167 的靶标是ARF12,通过生长素作用因子的调控来调节雌穗生长发育。另外,miR162家族的靶标是基因中心,差异表达的miR162作用于类Dicker酶1(Dicer-Like1,DCL1),DCL1的同系物是 miRNA积累所需物[29]。

图2 7个miRNA及其靶基因的差异表达图谱Fig.2 Expression profiles of 7 miRNAs and the target genes
本研究中,测定的miRNA156,miR172在先前研究中显示在植物发育的整个阶段起作用,从最早的花诱导形成阶段到最终的性器官形成。miR172主要通过靶基因调控花器官的形成和花模式,miR172功能的缺失将延迟花的发育或造成花发育异常,相比之下,过表达的miR172将导致花期提前[30],而在玉米雌穗发育过程中miR172可能对花器官的形成也起到关键的调控,在玉米中miR156 a-l从玉米幼穗到成熟过渡阶段可能作用于几个Squamosa启动子结合蛋白基因(squamosa promoter binding protein like genes,SPL),可能是通过SPL 间接地激活miR172。miR172已被证明能够下调基因光泽15(Glossy15,GL15),促进幼穗阶段的维持[31]。同时miR172e可能会控制无限小穗1(Indeterminate Spikelet 1,IDS 1)和姊妹无限小穗1(Sister Indeterminate Spikelet 1,SID 1),通过翻译抑制和mRNA降解方式,对玉米小穗性别和茎尖细胞分化起决定作用。除了miR156和miR172,miR164靶基因编码无顶端分生组织(No Apical Meristem,NAM)蛋白,可能参与调节雌穗发育[32-35]。miR390通过切割反式作用干扰小 RNA(tasiRNA,TAS)产生多片段,这些片段在一系列RNA酶作用下,形成 TAS,靶向 ARF2,ARF3和ARF4,参与植物生长发育调控[36,37]。本研究通过混合样品测序分析,鉴定出与玉米雌穗发育相关miRNA的靶基因,为进一步研究miRNA及其靶基因在玉米雌穗发育过程中的调控作用奠定了基础。
[1] NAG A,JACk T.Sculpting the flower;the role of microRNAs in flower development[J].Plant Development,2010,91:349 -378.
[2] LIU Q,CHEN Y Q.Insights into the mechanism of plant development:interactions of miRNAs pathway with phytohormone response[J].Biochem Biophys Res Commun,2009,384(1):1 -5.
[3] LISCH D.How important are transposons for plant evolution?[J].Nature Reveiews Genetics,2013,14(1):49-61.
[4] VOINNET O.Post-transcriptional RNA silencing in plantmicrobe interactions:a touch of robustness and versatility[J].Curr Opin Plant Biol,2008,11(4):464 -470.
[5] SUNKAR R,CHINNUSAMY V,ZHU J,et al.Small RNAs as big players in plant abiotic stress responses and nutrient deprivation [J].Trends Plant Sci,2007,12(7):301-309.
[6] BOWMAN J L.Class III HD-Zip gene regulation,the golden fleece of ARGONAUTE activity?[J].Bio Essays,2004,26:938 -942.
[7] CHUCK G,CIGAN A M,SAETEURN K et al.The heterochromic maize mutant Corngrass1 results from overexpression of a tandem microRNA [J].Nature Genetics,2007,39:544 -549.
[8] CHUCK G,MEELEY R,IRISH E,et al.The maize tasselseed4 microRNA controls sex determination and meristem cell fate by targeting Tasselseed6/indeterminate spikelet1 [J].Nature Genetics,2007,39:1517-1521.
[9] MILLAR A A,GUBLER F.The Arabidopsis GAMYB-like genes,MYB33 and MYB65,are microRNA regulated genes that redundantly facilitate anther development[J].The Plant Cell,2005,17:705 -721.
[10] REN G H,WANG B J.Cloning,expression,and characterization of miR058 and its target PPO during the development of grapevine berry stone[J].Gene,2014,548:166-173.
[11] PENG T,SUN H Z,QIAO M M,et al.Differentially expressed microRNA cohorts in seed deve-lopment may contribute to poor grain filling of inferior spikelets in rice[J].BMC Plant Biology,2014,14:196.
[12] KURIHARA Y,WATANABE Y.Arabidopsis microRNA biogenesis through Dicer-like 1 protein functions[J].Proc Natl Acad Sci USA,2004,101(34):12753-12758.
[13] ADDO-QUAYE C,ESHOO T W,BARTEL D P,et al.Endogenous siRNA and miRNA targets identified by sequencing of the Arabidopsis degradome[J].Curr Biol,2008,18:758 -762.
[14] ADDO-QUAYE C,SNYDER J A,PARK Y B,et al.Sliced microRNA targets and precise loop-first processing of MIR319 hairpins revealed by analysis of the Physcomitrella patens degradome[J].RNA,2009,15:2112 -2121.
[15] LI Y F,ZHENG Y,ADDO-QUAYE C,et al.Transcriptome-wide identification of microRNA targets in rice[J].Plant J,2010,62:742 -759.
[16] SONG Q X,LIU Y F,HU X Y,et al.Identification of miRNAs and their target genes in developing soybean seeds by deep sequencing [J].Bmc Plant Biology,2011,11:5.
[17] PANTALEO V,SZITTYA G,MOXON S,et al.Identification of grapevine microRNAs and their targets using high-throughput sequencing and degradome analysis[J].Plant J,2010,62:960 -976.
[18] MAO W,LI Z,Xia X,et al.A combined approach of high-throughput sequencing and degradome analysis reveals tissue specific expression of microRNAs and their targetsin cucumber [J].PLoS One,2012,7(3):e33040.
[19] FENG J,LIU S,WANG M,et al.Identification of microRNAs and their targets in tomato infected with Cucumber mosaic virus based on deep sequencing[J].Planta,2014,240(6):1335 -1352.
[20] LIU N,TU L,TANG W,et al.Small RNA and degradome profiling reveals a role for miRNAs and their targets in the developing fibers of Gossypium barbadense[J].Plant J,2014,80(2):331 -344.
[21] NIU S,FAN G,XU E,et al.Transcriptome/degradome-wide discovery of microRNAs and transcript targets in two paulownia australis genotypes[J].PLoS One,2014,9(9):e106736.
[22] ZHAO M,TAI H,SUN S,et al.Cloning and characterization of maize miRNAs involved in responses to nitrogen deficiency[J].PLoS One,2012,7(1):e29669.
[23] GERMAN M A,PILLAY M,JEONG D H,et al.Global identification of microRNA-target RNA pairs by parallel analysis of RNA ends[J].Nat Biotechno,2008,l26:941-946.
[24] ADDO-QUAYEC,MILLERW,AXTELLMJ.CleaveLand:a pipeline for using degradome data to find cleaved small RNA targets[J].Bioinformatics,2009,25(1):130-131.
[25] ELBASHIR S M,LENDECKEL W,TUSCHL T.RNA interference is mediated by 21-and 22-nucleotide RNAs[J].Genes Dev,2001,15(2):188 -200.
[26] RHOADES,MATTHEW W,REINHART,et a1.Prediction of plant microRNA targets [J].Cell,2002,110(4):513-520.
[27] PALATNIK,JAVIER F,ALLEN.et al.Control of leaf morphogenesis by microRNAs [J].Nature,2003,425,257-263.
[28] CHEN X.A microRNA as a translational repressor of APETALA2 in Arabidopsis flower development[J].Science,2004,303:2022 -2025.
[29] XIE Z,KASSCHAU K D,CARRINGTON J C,et al.Negative feedback regulation of Dicer-like1 in Arabidopsis by microRNA degradation[J].Curent Biology,2003,13:784-789.
[30] AUKERMAN,SAKAI.Regulation of flowering time and floral organ identity by a microRNA and its APETALA2-like target genes [J].Plant Cell,2003,15(11):2730-2741.
[31] LAUTER N,KAMPANI A,CALSON S,et al.micro RNA172 down regulates glossy15 to promote vegetative phase change in maize[J].Proc Natl Acad Sci USA,2005,102(26):9412 -9417.
[32] MALLORY,ALLISON C,DUGAS,et al.MicroRNA regulation of NAC-domain targets is required for proper formation and separation of adjaeent embryonic,vegetative,and floral organs[J].Curt Biol,2004,14(12):1035-1046.
[33] ZHU Q H,SPRIGGS A,MATTHEW L,et al.A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains[J].Genome Res,2008,18(9):1456-1465.
[34] CHUCK G,CIGAN A M,SAETEURN K,et al.The heterochronic maize mutant Corngrass1 results from overexpression of a tandem microRNA [J].Nat Genet,2007,39(4):544 -549.
[35] CHUCK G,MEELEY R,HAKE S.Floral meristem initiation and meristem cell fate are regulated by the maize AP2 genes ids1 and sid1 [J].Development,2008,135(18):3013-3019.
[36] ALLEN E,XIE Z,GUSTAFSON A M,et al.MicroRNA-directed phasing during trans-acting siRNA biogenesis in plants[J].Cell,2005,121(2):207 -221.
[37] WILLIAMS L,CARLES C C,OSMONT K S,et al.A database analysis method identifies an endogenous transacting short-interfering RNA that targets the Arabidopsis ARF2,ARF3,and ARF4 genes[J].Proc Natl Acad Sci USA,2005,102(27):9703 -9708.
