草菇cat1基因序列分析及其低温胁迫下定量表达研究
2015-05-08颜素雅陈明杰
赵 妍,林 锋,2,+,姜 威,颜素雅,陈明杰,2,*
(1.上海市农业科学院食用菌研究所,上海市农业遗传育种重点开放实验室,农业部南方食用菌资源利用重点实验室,国家食用菌工程技术研究中心,上海 201403;2.上海海洋大学食品学院,上海 201306)
草菇cat1基因序列分析及其低温胁迫下定量表达研究
赵 妍1,林 锋1,2,+,姜 威1,颜素雅1,陈明杰1,2,*
(1.上海市农业科学院食用菌研究所,上海市农业遗传育种重点开放实验室,农业部南方食用菌资源利用重点实验室,国家食用菌工程技术研究中心,上海 201403;2.上海海洋大学食品学院,上海 201306)
为了探讨草菇过氧化氢酶cat1基因在低温胁迫下的变化情况,进一步了解草菇低温自溶的相关机制。本研究首先对草菇过氧化氢酶cat1基因的序列进行分析,并以低温敏感型菌株V23与耐低温型菌株VH3为实验材料,比较了低温胁迫下过氧化氢酶cat1基因在草菇不同菌株中的相对表达量差异。结果表明:草菇cat1基因的cDNA序列包含2274bp的开放阅读框,编码1个由757个氨基酸残基组成的蛋白质;对该基因蛋白质翻译产物Cat1进行的生物信息学分析表明,草菇与灰盖鬼伞、褐腐菌、双孢蘑菇、粉孢革菌、毛韧革菌等亲缘关系都很近,草菇的Cat1较为保守,且序列中存在着一个过氧化氢酶功能域;低温胁迫下草菇不同菌株的cat1基因实时荧光定量PCR结果显示,在整个低温处理期间,V23和VH3菌丝中的cat1基因相对表达量均整体呈下降趋势,但在相同低温处理时间下,VH3的cat1基因相对表达量始终高于V23。研究表明,草菇cat1基因的相对高表达可能与其耐低温能力有关,这将为解决草菇保鲜过程中的低温自溶问题提供一定的理论基础。
草菇,低温,过氧化氢酶cat1基因,实时荧光定量PCR,生物信息学
草菇[Volvariellavolvacea(Bull.)Singer]是一种原产自中国热带与亚热带地区的腐生性真菌[1],其味道鲜美、营养丰富,因而备受广大消费者喜爱。与此同时,草菇不仅能利用农业废弃物生产子实体,并且其栽培下脚料还能被制成有机肥,从而实现农业固废循环的高效利用[2]。虽然我国草菇产量已约占世界总产量的3/4,但目前仍远不能满足市场需求,因此草菇产业发展潜力巨大[3]。由于草菇喜在高温、高湿的环境下生长[4],常规低温冷藏0~4℃时,便会引起其菌丝体发生自溶死亡,子实体亦会渗水、褐变、产生异味[5]。草菇这种不耐低温储运的特性,严重制约了鲜草菇的生产和流通,因此对草菇低温应答机制的研究成为亟待解决的科学问题。
1 材料与方法
1.1 材料与仪器
草菇菌株V23、VH3 上海市农业科学院食用菌研究所菌种保藏中心提供。TaqDNA Polymerase in Storage Buffer B、pGEM®-T Easy Vector System I Promega公司;PrimeScriptTMRT reagent Kit with gDNA Eraser、SYBR®PremixExTaqTMⅡ TaKaRa公司;AxyPrepTMDNA Gel Extraction Kit 爱思进生物技术有限公司;Redzol试剂盒 北京赛百盛基因技术有限公司;TOP10感受态细胞 天根生化科技有限公司;质粒小量制备试剂盒 上海捷瑞生物工程有限公司;马铃薯葡萄糖肉汤 碧迪医疗器械有限公司;其它试剂均为市售分析纯。PDA培养基:马铃薯提取物200g,葡萄糖20g,琼脂20g,加蒸馏水定容至1L,121℃灭菌15min;马铃薯葡萄糖肉汤培养基按照说明书配制。
5810R型高速冷冻离心机 Eppendorf公司;DHP-9272型电热恒温培养箱 上海一恒科学仪器有限公司;制冰机 美国格兰特公司;超净工作台 苏州安泰空气技术有限公司;岛津紫外可见分光光度计UV-1800 日本SHIMADZU公司;EC3 Imaging System凝胶成像系统 SYNGENE公司;PCR扩增仪 TaKaRa公司;StepOne Plus荧光定量PCR仪 Applied Biosystems 公司。
1.2 实验方法
1.2.1 生物信息学分析 采用NCBI的ORF Finder程序预测cat1基因的开放阅读框(Open reading frame,ORF),并获得氨基酸序列。使用ExPASy ProtParam在线分析工具预测Cat1蛋白的理化性质、分子量、等电点。将cat1基因编码的氨基酸在http://smart.embl-heidelberg.de/网站上进行在线分析,获得该蛋白质Cat1所包含的功能域。同时,利用SWISS-Model预测和模拟草菇Cat1蛋白质的三级结构。
1.2.2 草菇菌丝的培养与低温处理 将草菇V23和VH3的菌丝转接到PDA固体培养基上,置于32℃恒温培养4d。向匀浆器中加入100mL马铃薯葡萄糖肉汤培养液,将长满菌丝的培养基加入匀浆器中,打碎后将匀浆转接入培养液中,置于32℃条件下,150r/min,培养5d。将培养好的草菇菌丝在0℃低温下分别处理0、2、4、6、8h,液氮冻存后备用。
1.2.3 实时荧光定量PCR分析
1.2.3.1 菌丝体总RNA的提取和cDNA的合成 参照Redzol试剂盒说明书提取草菇菌丝体总RNA,并用DEPC预处理的水溶解,然后进行凝胶电泳检测。测定RNA的浓度,依据PrimeScriptTMRT reagent Kit with gDNA Eraser说明书,去除基因组DNA,并反转录成cDNA。
1.2.3.2 目的片段的克隆与测序 在草菇全基因组序列信息(http://172.17.37.2)中进行Blast搜索,获得草菇的cat1基因序列,并设计特异性引物(表1),由上海捷瑞生物工程有限公司合成。以微管蛋白(Tubulin,TUB)基因作为内参基因,采用常规PCR扩增tub和cat1基因片段,并构建用于real-time PCR实验的标准品质粒,质粒由上海生工生物工程股份有限公司测序,具体操作参照汪虹与陈明杰(2007)的方法[12]。
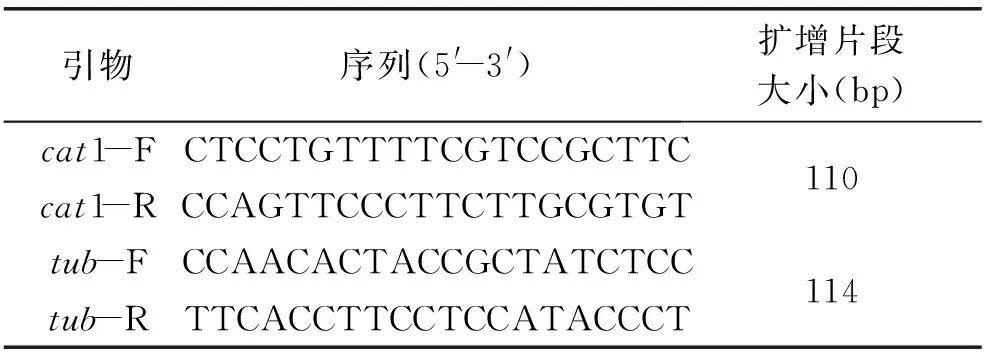
表1 实时荧光定量PCR扩增引物
1.2.3.3 Real-time PCR反应 将含有内参基因和目的基因的标准品质粒依次进行10倍稀释,得到5个梯度的标准品浓度,在StepOne Plus荧光定量PCR仪上进行扩增,构建标准曲线,并将所得数据按ΔΔCT法计算cat1基因的相对表达量[13-14]。Real-time PCR反应体系为20μL:2×SYBR®PremixExTaqTMⅡ 10μL,10μmol/L forward primer 0.4μL,10μmol/L reverse primer 0.4μL,50×Rox 0.4μL,cDNA模板2μL,ddH2O 6.8μL。反应条件为:95℃预变性20s,1个循环;95℃ 5s,60℃ 15s,72℃ 15s,该过程进行40个循环。每组样品设置3个平行样品孔,并设置无菌双蒸水代替模板作为阴性对照。
2 结果与分析
2.1 生物信息学分析
生物信息学分析结果表明:cat1基因的cDNA序列全长为2274bp,包含2274bp的ORF,编码1个由757个氨基酸残基组成的蛋白质(图1)。预测所编码蛋白Cat1的分子量为84.03ku,等电点为6.40,有酸性氨基酸88个,碱性氨基酸79个,理论半衰期大于20h,不稳定指数为29.88,属于稳定性蛋白质。

图1 cat1基因cDNA序列及编码的氨基酸序列注Fig.1 Nucleotide sequence and deduced amino acid sequence of cat1 gene注:*为终止密码子。
经Blast同源性分析发现,该蛋白质序列与多种真菌的CAT具有很高的同源性(图2),与灰盖鬼伞(Coprinopsiscinerea,XP_001833673.2)、褐腐菌(Postiaplacenta,XP_002475806.1)、双孢蘑菇(Agaricusbisporus,XP_006458554.1)、粉孢革菌(Coniophoraputeana,XP_007763262.1)、毛韧革菌(Stereumhirsutum,XP_007307584.1)的同源氨基酸序列相似性分别达到了81%、81%、80%、79%、79%,表明草菇的Cat1较为保守。

图2 草菇Cat1与其它真菌同源氨基酸序列比对Fig.2 Sequence alignment between Cat1 from Volvariella volvacea and other homologous sequences from different fungi注:*部分表示100%同源性,︰部分表示保守区,·部分表示同源性低。
在http://smart.embl-heidelberg.de/网站上,利用在线分析软件查找草菇Cat1的功能域,发现在该蛋白质序列中主要包含一个CAT功能域,其氨基酸序列为37~427(图3)。

图3 草菇Cat1的功能域Fig.3 Functional domain of Cat1 from Volvariella volvacea
利用SWISS-Model预测和模拟了草菇Cat1蛋白的三级结构(图4)。参考模板为粗糙脉孢菌(Neurosporacrassa)的catalase-1蛋白(PDB:1sy7.1.A),且在该区域中(N端第35~757个氨基酸)两者的氨基酸序列相似性达到了62.17%。

图4 SWISS-Model模拟草菇Cat1蛋白质三级结构Fig.4 Structure prediction of Cat1 from Volvariella volvacea by SWISS-Model
2.2 低温胁迫下草菇不同菌株中cat1基因表达变化分析
图5是草菇cat1基因扩增后的溶解曲线,在85℃左右时出现单一峰,表明cat1基因扩增具有特异性,其相对定量的结果可信。低温胁迫下草菇V23、VH3的cat1基因和tub基因经real-time PCR后得到CT值,并采用ΔΔCT法计算cat1基因的相对表达量(表2)。受低温胁迫后,V23和VH3菌丝中的cat1基因相对表达量均整体呈下降趋势(图6)。在低温处理2h时,两菌株的cat1基因相对表达量差异最大,低温敏感型菌株V23的表达量下降至0h的0.18倍,耐低温型菌株VH3的表达量下降至0h的0.84倍;在低温处理过程中,VH3菌株在4h时的cat1基因相对表达量最低,仅为0h的0.41倍,V23菌株的cat1基因在2h时达到最低值,随后其表达量虽然略有回升,但一直维持在较低的水平上。在整个低温胁迫期间,耐低温型菌株VH3的cat1基因相对表达量始终高于低温敏感型菌株V23。

图5 cat1基因扩增熔解曲线Fig.5 Melt curve of cat1 gene
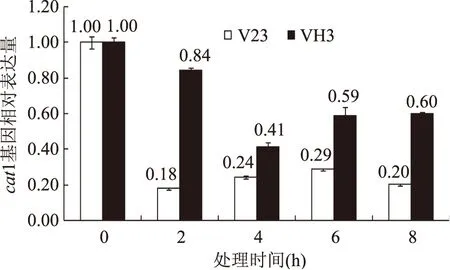
图6 低温胁迫下V23和VH3中cat1基因的相对表达量变化Fig.6 Relative expression of cat1 gene in V23 and VH3 under low temperature stress
3 结论与讨论
与其它活性氧种类相比,虽然H2O2毒性相对较小,但高浓度的H2O2则会引起过氧化胁迫,造成细胞程序性死亡[15]。与此同时,H2O2还能扩散到细胞其它部位,并且在金属离子的作用下,通过Fenton反应产生毒性更强的羟基自由基[16],羟基自由基几乎能够与包括DNA、蛋白质、脂质在内的所有生物大分子发生反应,给细胞带来损伤[7]。CAT作为直接催化H2O2分解的抗氧化酶,具有维持H2O2平衡,保证细胞正常生命活动的重要作用[17-18]。本研究首先对草菇cat1基因编码的蛋白质序列进行了生物信息学分析,发现在Cat1蛋白序列中含有一个catalase功能域,表明草菇Cat1具有催化H2O2分解的功能。对Cat1及其同源序列的多序列比对结果显示,草菇与灰盖鬼伞、褐腐菌、双孢蘑菇、粉孢革菌、毛韧革菌等同源性都很高(79%~81%),表明草菇的Cat1在进化过程中具有较高的保守性。随后选用低温敏感型菌株V23和耐低温型菌株VH3作为实验材料,其中V23是草菇生产上的主栽菌株,而VH3则是通过对V23的原生质体进行复合诱变得到的相对耐低温菌株[19],并利用real-time PCR技术研究了低温胁迫对草菇两菌株的cat1基因表达影响。实验结果显示,在整个低温处理期间,V23和VH3的cat1基因相对表达量总体呈下降趋势;在相同低温处理时间下,VH3的cat1基因下降幅度要比V23小,这可能是VH3更耐低温的原因之一。王松华等[20]的研究表明,草菇菌丝在低温处理2~8h时,CAT的活性随低温处理时间的延长而逐渐降低,这与本研究的cat1基因表达量变化趋势相似,说明低温胁迫可能通过影响cat1基因的转录表达进而影响酶活,最终使得低温下草菇的部分防御功能被破坏而引发自溶。通
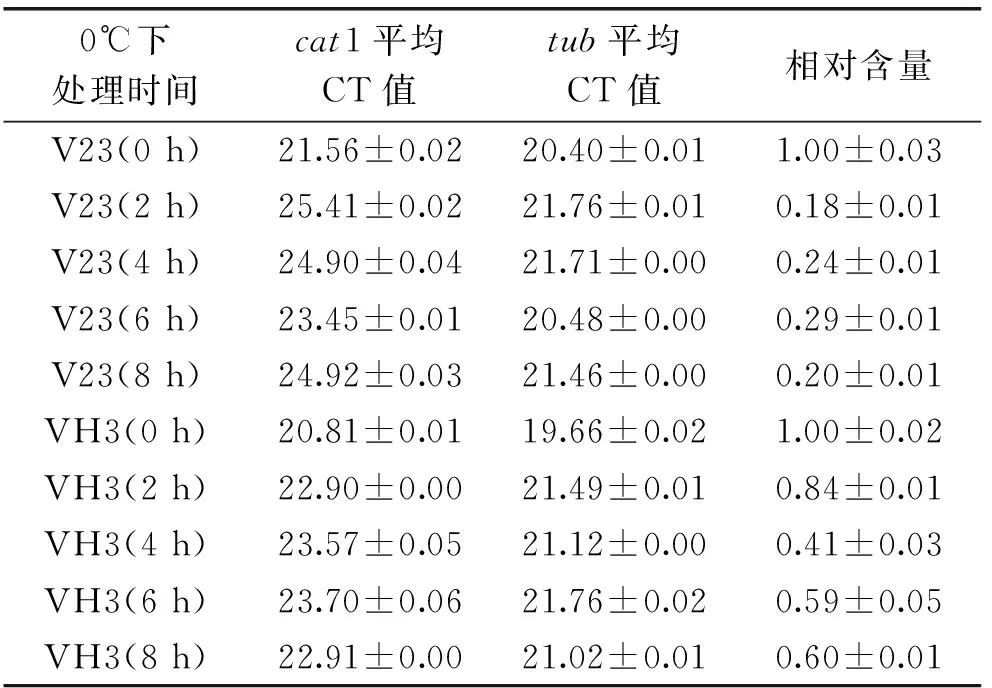
表2 低温胁迫下草菇V23和VH3菌丝中的cat1基因相对表达量
过对草菇cat1基因的序列分析及其低温下表达量变化的研究,将有助于深入了解草菇的低温应答机制,为后续筛选耐低温新菌株服务,从而更好地延长草菇的低温保鲜期。与此同时,由于本文只研究了cat1基因受低温胁迫的表达变化,而CAT仅是生物防御系统中的关键酶之一,因而后续需要对生物防御系统的其它关键酶进行研究,才能进一步了解草菇低温自溶的相关机制,为延长草菇低温保鲜时间提供理论依据。
[1]徐学锋,姬宏超,李巧玲,等. 草菇有性世代间rDNA序列变异分析及RAPD分析[J]. 食品工业科技,2012,33(10):198-201.
[2]林锋,李国贤,赵妍,等. 草菇育种方法及相关分子标记研究进展[J]. 热带作物学报,2014,35(3):609-615.
[3]邓优锦,朱坚,谢宝贵. 图说草菇栽培关键技术[M]. 北京:中国农业出版社,2011:1.
[4]杨淑云,刘朝贵,曹必好,等. 草菇采后生理及保鲜技术[J]. 生物学杂志,2008,19(4):36-38.
[5]段学武,庞学群,张昭其. 草菇低温储藏及有关生理变化研究[J]. 热带作物学报,2000,21(4):75-79.
[6]Gill SS,Tuteja N. Reactive oxygen species and antioxidant machinery in abiotic stress tolerance in crop plants[J]. Plant Physiol Bioch,2010,48(12):909-930.
[7]Jamieson DJ. Oxidative stress responses of the yeastSaccharomycescerevisiae[J]. Yeast,1998,14(16):1511-1527.
[8]Zhao YC,Li JL,Chen YR,et al. Response to oxidative stress ofCoriolusversicolorinduced by exogenous hydrogen peroxide and paraquat[J]. Ann Microbiol,2009,59(2):221-227.
[9]Kawasaki L,Wysong D,Diamond R,et al. Two divergent catalase genes are differentially regulated duringAspergillusnidulansdevelopment and oxidative stress[J]. J Bacteriol,1997,179(10):3284-3292.
[10]Izawa S,Inoue Y,Kimura A. Importance of catalase in the adaptive response to hydrogen peroxide:analysis of acatalasaemicSaccharomycescerevisiae[J]. Biochem J,1996,320(1):61-67.
[11]Talarczyk A,Krzymowska M,Borucki W,et al. Effect of yeast CTA1 gene expression on response of tobacco plants to tobacco mosaic virus infection[J]. Plant Physiol,2002,129:1032-1044.
[12]汪虹,陈明杰. 草菇冷诱导Cor3基因实时荧光定量PCR标准品质粒和标准曲线的构建[J]. 食用菌学报,2007,14(3):16-19.
[13]Bubner B,Baldwin IT. Use of real-time PCR for determining copy number and zygosity in transgenic plants[J]. Plant Cell Rep,2004,23(5):263-271.
[14]Livak KJ,Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T))method[J]. Methods,2001,25(4):402-408.
[15]Quan LJ,Zhang B,Shi WW,et al. Hydrogen peroxide in plants:a versatile molecule of the reactive oxygen species network[J]. J Integr Plant Biol,2008,50(1):2-18.
[16]Li Q,McNeil B M. Harvey L. Adaptive response to oxidative stress in the filamentous fungus Aspergillus niger B1-D[J]. Free Radical Bio Med,2008,44(3):394-402.
[17]Kato M,Shimizu S. Chlorophyll metabolism in higher plants. VII. Chlorophyll degradation in senescing tobacco leaves;phenolic-dependent peroxidative degradation[J]. Can J Botany,1987,65(4):729-735.
[18]Gocheva YG,Tosi S,Krumova ET,et al. Temperature downshift induces antioxidant response in fungi isolated from Antarctica[J]. Extremophiles,2009(13):273-281.
[19]韩业君,曹晖,陈明杰,等. 草菇耐低温菌株的诱变选育与鉴定[J]. 菌物学报,2004,23(3):417-422.
[20]王松华,张华,陈庆榆,等. 低温胁迫对草菇菌丝抗氧化酶表达的影响[J]. 热带作物学报,2009,30(5):594-597.
Sequence analysis ofcat1 and its relative expression inVolvariellavolvaceaunder low temperature stress
ZHAO Yan1,LIN Feng1,2,+,JIANG Wei1,YAN Su-ya1,CHEN Ming-jie1,2,*
(1.Institute of Edible Fungi,Shanghai Academy of Agricultural Sciences,Shanghai Key Laboratory of Agricultural Genetics and Breeding,Key Laboratory of Edible Fungi Resources and Utilization(South),Ministry of Agriculture,P. R. China,National Engineering Research Center of Edible Fungi,Shanghai 201403,China;2. College of Food Science & Technology,Shanghai Ocean University,Shanghai 201306,China)
In order to screen the relative expression ofcat1 gene during low temperature stress inVolvariellavolvaceafor getting a better understanding of its autolysis. The sequence ofcat1 gene and its relative expression in mycelia of twoVolvariellavolvaceastrains differing in cold tolerance,V23(cold-sensitive)and VH3(cold-tolerant),being exposed to low temperature stress were analyzed. Results showed thatcat1 gene included 2274bp open reading frame and consisted of 757 amino acid residues. Sequence alignment analysis indicated that Cat1 fromVolvariellavolvaceawas similar with the homologous sequence fromCoprinopsiscinerea,Postiaplacenta,Agaricusbisporus,ConiophoraputeanaorStereumhirsutum. Cat1 fromVolvariellavolvaceawas conservative with a catalase functional domain. The real time-PCR analysis showed that the relative expression ofcat1 gene decreased both in V23 and VH3 during low temperature process. However,the relative expression ofcat1 gene in VH3 was higher than that in V23 under the same low temperature treatment. All these results indicated that the less decline ofcat1 gene may be associated with the better adaptation of VH3 to low temperature stress,which would help to solve the problem of autolysis inVolvariellavolvacea.
Volvariellavolvacea;low temperature stress;cat1 gene;real-time PCR;bioinformatics
2014-06-30 +共同第一作者
赵妍(1981-),女,博士,助理研究员,研究方向:食用菌遗传育种与生理生化。 林锋(1989-),男,硕士研究生,研究方向:食用菌遗传育种与功能基因。
*通讯作者:陈明杰(1965-),男,博士,研究员,研究方向:食用菌遗传育种与生理生化。
国家自然科学基金项目(31301828);上海市科技兴农重点攻关项目应用基础类(2014第7-1-5号)。
TS201.3
A
1002-0306(2015)07-0196-05
10.13386/j.issn1002-0306.2015.07.033
