PCR-RDB技术在HCV基因分型中的应用研究
2015-03-22西安交通大学第二附属医院检验科西安710004
西安交通大学第二附属医院检验科(西安710004)
李步荣 张 彤 李丽华 高 宁 张 毅
PCR-RDB技术在HCV基因分型中的应用研究
西安交通大学第二附属医院检验科(西安710004)
李步荣 张 彤 李丽华 高 宁 张 毅
目的:评价PCR-RDB技术在丙型肝炎病毒(HCV)基因型检测中的临床实际应用价值。 方法:随机收集经实时荧光定量聚合酶链式反应(PCR)检测HCV RNA含量大于1×103IU/ml血清样本总计158份,其中无临床症状丙型肝炎病毒携带者90例,慢性丙型肝炎(CHC)患者48例,肝硬化(HS)患者16例,肝癌(LC)患者4例。全部血清样本应用PCR和反向斑点杂交技术(RDB)进行HCV基因分型检测,并对检测结果进行统计学分析。结果:所有标本HCV基因分型成功,其中1b型78例,2a型65份,3a型3例,3b型2例,6a型2例,1b和2a混合感染型8例。不同疾病组HCV不同基因型分布差异无统计学意义。结论:HCV基因型与疾病类型无相关性;PCR-RDB杂交技术适用于临床实验室开展HCV基因型检测。
丙型肝炎病毒(Hepatitis C virus,HCV)感染后只有少数感染者可以凭借自身免疫系统清除体内病毒,大部分感染者表现为持续感染,持续感染常导致慢性肝炎、肝硬化、肝癌甚至死亡。HCV基因型分布具有明显的地域差异性和人群差异性[1],不同基因型决定病毒某些生物学特性不尽相同,有报道不同基因型对干扰素的敏感性也有区别,引起的临床表现也不完全一致。近年随着分子生物学技术不断发展,出现了多种HCV基因分型方法。传统的基因测序方法虽然准确可靠,但费时费力,不利于临床实验室大范围广泛开展,应用聚合酶链式反应(Polymerase chain reaction,PCR)与反向斑点杂交(Reverse dot blot,RDB)技术相结合开展HCV基因型检测,其具有快速高效、高通量特点,花费相对低廉,便于及时掌握本地区HCV基因型分布特征和流行趋势,对临床诊断、治疗及预后的判断有重要指导意义,现报告如下。
对象与方法
1 研究对象 随机收集2013年7月至2014年12月就诊于西安交通大学第二附属医院消化科和感染科门诊,丙肝抗体检测阳性,HCV RNA定量检测结果高于1×103IU/ml患者血清样本共158份,其中男98例,年龄6~85岁,平均42.02±17.09岁;女60例,年龄12~69岁,平均37.82±18.99岁。所有患者诊断符合《中国丙型肝炎防治指南》制定的标准,并排除丙型肝炎以外的其它类型肝炎病毒感染。其中无临床症状丙型肝炎病毒携带者90例,慢性丙型肝炎(Chronic hepatitis C,CHC)患者48例,肝硬化(Hepatic sclerosis,HS)患者16例,肝癌(Liver cancer,LC)患者4例。所有患者早晨空腹静脉采血10 ml,室温静置2 h,4000 rpm离心5 min分离血清,立即检测或保存于-20℃冰箱备用。
2 主要试剂和仪器 丙型肝炎病毒基因分型试剂盒(中山大学达安基因股份有限公司);TC-412型TECHNE通用PCR 仪(英国Bibby Scientific Limited公司);YN-H16型分子杂交仪(深圳亚能生物技术有限公司);TGL-16g高速低温离心机(上海安亭科学仪器厂);THZ-82恒温振荡器(常州国华电器有限公司)。
3 HCV基因分型
3.1 RNA的提取:取灭菌1.5 ml离心管,加入50 μl RNA蛋白酶K,同时加入200 μl血清,再加入200 μl病毒裂解液,振荡混匀,高速离心10s,72℃放置10 min。小心加入250 μl无水乙醇,振荡混匀。将混合液转入离心柱,12000 rpm离心1 min,弃去离心液。离心柱中加入500 μl抑制物去除液,12000 rpm离心1min。离心柱中再加入500 μl去离子液,12000 rpm 离心1min,重复1次。将离心柱移入新的收集管,14000 rpm离心3 min去除残余的乙醇。将离心柱移入新的无菌离心管中,72℃放置2 min。加入预热的洗脱液50 μl,室温静置1min,14000 rpm离心1 min。
3.2 RT-PCR:在反应管中加入30 μl提取的病毒核酸,按下列条件进行扩增:50℃逆转录25 min,95℃预变性15 min (94℃ 30s;55℃ 40s;72℃ 45s)×45 cycles,72℃延伸7 min。
3.3 分子杂交:每个5 ml离心管中加入4±0.5 ml预热的杂交液Ⅰ,放入标记好的杂交膜条,将经98 ℃变性10min的扩增产物加入杂交液中,50 ℃杂交60 min。取50 ml灭菌离心管,加入25 ml预热的杂交液Ⅱ和30μl溶液Ⅰ,移入膜条,50 ℃振荡10 min。弃去杂交液Ⅱ,加入新的杂交液Ⅱ,室温振荡反应10min。将膜条移入新鲜配制的显色液,避光反应5~10min。将膜条转入去离子水中浸泡1~3min,肉眼直接判读结果,必要时可借助阅读仪扫描判读结果。
4 统计学方法 用GraphPad Prism 5.0进行统计学分析,计数资料采用频数和率表示。率的比较用χ2检验或Fisher’s 精确检验,P<0.05为差异有统计学意义。
结 果
1 HCV基因型检测结果 158份血清样本全部分型成功,其中1b型78例,2a型65例,3a型3例,3b型2例,6a型2例,1b和2a混合感染型8例(图1)。1b型和2a型为主要流行基因型,占比高达90.51%(143/158),1b型比2a型多8.23%。
2 不同疾病组HCV基因型检测结果 由于本研究对象肝癌疾病组标本只有4例,为了便于统计研究将肝硬化组和肝癌组合并分析,同时,将基因型3a、3b、6a合并进行统计分析,结果见附表。应用统计软件GraphPad Prism 5.0进行χ2检验,χ2=0.5893,P=0.9966,统计表明不同疾病组基因型分布差异无统计学意义(图2)。

图1 HCV基因型分布

附表 不同疾病组HCV基因型分布
注:不同疾病组比较χ2=0.5893,P=0.9966
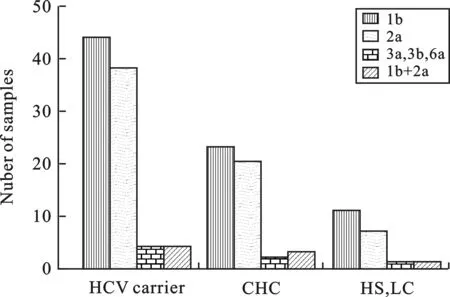
图2 不同疾病类型HCV基因型分布特征比较
讨 论
HCV基因型与亚型在世界范围内存在流行分布差异。HCV基因型与患者临床表现、干扰素治疗的敏感性、判断疾病预后高度相关[2-3]。HCV主要通过血液传播途径,少数通过密切接触途径传播。HCV基因组E1/E2/NS5为高可变区,常用作 HCV 的基因分型和进化分析的目标区。1993 年 Simmonds 等通过系统进化分析HCV非结构蛋白NS5区域基因序列,将HCV基因主要分为6个基因型,80多个亚型,被国际科研人员广泛接受。我国大部分地区汉族人群流行的HCV基因亚型主要是1b型,其次为2a型,不同区域不同人群不尽相同[4-5]。本次研究结果表明,西安地区HCV基因型分布存在多样性,以1b最为常见,其次为2a型。3a、3b及6a型检出率相对较低。HCV感染类型主要为单基因型感染,多基因感染是1b型和2a型混合感染,感染率5.06%(8/158),未见其它类型混合感染。统计学分析表明,不同疾病组HCV基因型分布差异无统计学意义。有报道HCV基因型和疾病的严重程度有关,但本研究显示基因型并不决定疾病类型,这还有可能和研究对象选择以及样本量的大小有关,还需要今后增大样本量进一步得出更科学合理的研究结论。
目前,HCV基因分型的方法有多种,全基因测序法是最为经典可靠的基因分型法。此方法耗时长、花费高、对实验室设备要求高,难以在较大范围开展。PCR-RDB技术是将型特异性寡核苷酸探针固定在硝酸纤维膜或尼龙膜特定检测区域,和带有生物素标记的RT-PCR扩增产物进行分子杂交,通过生物素、链霉素和辣根过氧化酶结合,在过氧化氢的催化下使四甲基联苯胺发生显色反应[6]。检测结果可直接用肉眼分辨,操作简便易行。本研究结果显示全部158份样本全部分型成功,间接说明该方法的广泛适用性。PCR-RDB反向杂交技术是一种高通量、高效率、低成本检测基因型方法,所需仪器设备要求不高,一般的分子诊断实验室都有条件开展。该方法要求样本HCV RNA含量在1×103IU/ml以上,对于低含量样本则存在敏感性不够的缺点,以后还需进一步提高。该方法对于新基因型病毒检测存在缺陷,对于无法确定基因型还需通过全基因测序确定基因型。
本研究对象主要来源于医院,尚不能够全面真实反映本地区HCV感染者基因型分布情况,有必要在今后的研究中,增加调查人群范围,提高研究样本总量,以便更好掌握本地区HCV基因型的分布及变化。PCR-RDB技术操作简便,其具有高通量特点,适合临床实验室广泛开展。我国HCV 感染率较高,通过HCV基因型检测,及时掌握HCV病毒的种系发育和进化特性,为 HCV 疫苗的设计研发提供必要依据,为临床制定合理的个体化治疗方案和判断预后提供科学的实验室依据。
[1] Khodabandehloo M,Roshani D. Prevalence of hepatitis C virus genotypes in Iranian patients: A systematic review and meta-analysis[J]. Hepat Mon,2014,14(12): 22915.
[2] Berak H,Laskus T,Kolakowska-Rzadzka A,etal. Peginterferon alfa-2a and peginterferon alfa-2b combined with ribavirin in patients with genotype 1 chronic hepatitis C: Results of a prospective single-centre study[J]. Adv Med Sci,2014,59(2): 261-265.
[3] Dixit VK,Dixit VK,Ghosh JK,etal. Clinical profile and response to treatment with pegylated interferon alpha 2b and ribavirin in chronic hepatitis c-a reappraisal from a tertiary care center in Northern India[J]. J Clin Exp Hepatol,2014,4(2): 101-105.
[4] 苏迎盈,刘慧鑫,汪 宁. 中国丙型肝炎病毒基因型分布[J]. 中华流行病学杂,2013,34(1): 80-84.
[5] 戎 霞,郑 英,熊华平,等. 广东地区临床患者和献血者HCV感染者HCV基因分型比较[J]. 中国输血杂志,2014,27(6): 593-596.
[6] Delvaux N,Da Costa VD,Da Costa MM,etal. Comparison of four methods of genotyping IL28B polymorphisms in chronic hepatitis C patients[J]. J Virol Methods,2015,220(8): 1-4.
(收稿:2015-07-10)
Application research of genotyping for hepatitis C virus by PCR-RDB Department of Clinical Laboratory,Second Affiliated Hospital,Xi’an Jiaotong University
( Xi’an 710004)
Li Burong Zhang Tong Li Lihua et al
Objective: To evaluate the clinical practical application value of polymerase chain reaction and reverse dot blot(PCR-RDB) method on genotyping HCV in different disease groups. Methods: We conducted a cohort study with 158 participants including 90 HCV carriers without clinical symptom,48 patients with chronic hepatitis C,16 patients with hepatic sclerosis and 5 patients with liver cancer. The HCV RNA levels of all sera samples were confirmed higher than 1×103IU/ml by real-time fluorescent quantitative polymerase chain reaction(PCR). HCV genotyping was carried out using type-specific primers by means of polymerase chain reaction and reverse dot blot(RDB). And the results were statistically analyzed with GraphPad Prism 5.0 software. Results: All 158 specimens were genotyped successfully. Among them,78 cases were genotype 1b,65 cases were genotype 2a,3 cases were genotype 3a,2 cases were genotype 3b,2 cases were genotype 6a,8 cases were mixed genotypes 1b and 2a. There was no significant difference among the different disease groups about the distribution of HCV genotypes. Conclusion: There is no relationship between the HCV genotypes and disease types. PCR-RDB was a suitable method for the HCV genotyping in clinical laboratories.
Hepacivirus Genotyping techniques Polymenrase chain reaction Fluoroimmunoassay
丙型肝炎病毒 基因分型技术 聚合酶链式反应 荧光免疫测定
R446.6
A
10.3969/j.issn.1000-7377.2015.05.047
