绵羊肺炎支原体延伸因子Tu基因的分子特性分析
2015-03-22尹正军
尹正军,岳 华,汤 承
(西南民族大学生命科学与技术学院,成都 610041)
绵羊肺炎支原体延伸因子Tu基因的分子特性分析
尹正军,岳 华,汤 承*
(西南民族大学生命科学与技术学院,成都 610041)
对绵羊肺炎支原体(Mycoplasmaovipneumoniae)参考菌株Y98株和11个临床分离株的tuf基因进行克隆、测序,生物信息学分析及构建进化树,分析绵羊肺炎支原体tuf基因的分子结构特性。结果表明:Y98株和11株分离株tuf基因编码区全长1 209 bp,编码402个氨基酸,氨基酸相似性为93.3%~100%,有较高的抗原指数和抗原表位,不含信号肽,平均GC含量为39.80%。Y98株与10个分离株延伸因子Tu存在1个跨膜区,含有6种不同的功能位点。而一分离株该蛋白质存在2个跨膜区,在271—274位点多出1个N-糖基化位点。12个菌株的tuf基因有117个单核苷酸突变位点,其中无义突变为41个,有义突变为76个,造成53个推导的氨基酸变化,主要发生在325—354位点靠近羧基端,有可能导致该蛋白质功能的改变。基于该基因的遗传进化与全基因组建立的进化关系一致,比16S rRNA基因更适合作为绵羊肺炎支原体进化关系的分子靶标。该基因种内保守同时也存在遗传多样性,作为分子分型的潜力值得进一步研究。
绵羊肺炎支原体;tuf基因;克隆;分子特性;遗传进化
绵羊肺炎支原体(Mycoplasmaovipneumoniae,Mo)是引起绵羊和山羊慢性非进行性肺炎的病原之一。Mo感染后使更多的呼吸道病原得以入侵,从而造成全球范围内养羊业的经济损失[1]。由于Mo无论在核酸还是蛋白质水平上都存在明显的异质性,给此病的诊断和防治带来了困难[2]。Mo基因组的释放为研究Mo基因功能提供了方便[3],但是目前对Mo毒力及免疫相关基因的了解不多,对重要结构基因的遗传多样性也知之甚少。
细菌的蛋白质合成可以分为三个步骤:起始、延伸、终止。延伸因子Tu(elongation factor Tu,EF-Tu)是由tuf基因编码的蛋白质,主要作用是负责肽链延伸,这是蛋白质合成过程中最主要的步骤。它是最丰富的细菌蛋白质[4-5],在肺炎支原体中也大量的存在,也是构成菌体膜蛋白成分之一[6]。它是一个GTP结合蛋白,不仅使tRNA移动到核糖体,而且监管和确保蛋白质完全翻译,Tu包括三种不同的结构域,每个域都有助于tRNA结合的位点[7]。此蛋白质还在蛋白质二硫化物的活性、类似伴侣性能及可能构成质膜成分等方面占有重要地位[8-10]。tuf基因功能保守,在真核生物、原核生物及古细菌中广泛存在。由于它的遗传稳定性和广泛分布性完全满足作为遗传进化分子靶标,已广泛用于真菌、细菌和支原体的生物分类鉴定和系统进化分析[11-13]。但目前还未见关于绵羊肺炎支原体tuf基因结构特征和遗传多样性的报道。本试验旨在克隆绵羊肺炎支原体tuf基因,分析其分子特征及遗传多样性,为进一步研究该基因的功能提供参考。
1 材料与方法
1.1 菌株
绵羊肺炎支原体参考株Y98株和11株临床分离株,编号为J15、J18、J24、J28、J43的来自J地,L3、L6、L9来自L地,Z32、Z33、Z34来自Z地,由西南民族大学动物医学实验室保存。
1.2 主要试剂
改良的Tht培养基;胶回收试剂盒购自AXYGEN公司;DL2000 Marker购自TIANGEN公司;TaqDNA聚合酶、MgCl2、dNTPs、pMD19-T载体、感受态DH5α购自TaKaRa公司;马血清购自郑州佰安生物工程有限公司;酵母浸出粉购自英国OXOID公司;PPLO肉汤购自法国BD公司;琼脂糖购自OXOID英国公司;蛋白酶K购自美国Sigma公司。
1.3 主要设备
CO2恒温培养箱(Thermostat,美国)、梯度PCR仪Px2(Thermo Hybaid 公司,美国)、凝胶成像系统Doc 2000(Bio-Rad公司,美国)、核酸电泳仪Powerpac universal TM(Bio-Rad公司,美国)、恒温水浴器(国华电器,中国江苏)、高速冷冻离心机(Eppendorf 公司,德国)、移液器(Eppendorf公司,德国)、纯水仪Milli-Q(Millipore公司,法国)等。
1.4 引物设计与合成
根据GenBank公布的绵羊肺炎支原体SC01株tuf基因序列用Primer 5设计特异性引物,tuf-F:5′-ATGGCAGTTGTTAAAACTGGTGC-3′,tuf-R:5′-TTATTTAATAATTTCAGTTACTG-3′,扩增片段大小为1 209 bp,引物由生工生物工程(上海)股份有限公司合成。
1.5 目的基因的扩增
对各支原体培养物均采用酚-氯仿萃取法来提取总DNA。DNA提取后,用核酸蛋白检测仪检测核酸,A260 nm/A280 nm比值均在1.8~2.0,置于-20 ℃保存。反应体系:10×Buffer 2.5 μL,dNTPs (各2.5 mmol·L-1)2 μL,MgCl2(25 mmol·L-1)1.5 μL,tuf-F(10 μmol·L-1)1 μL,tuf-R(10 μmol·L-1) 1 μL,TaqDNA聚合酶(5 U·μL-1) 0.125 μL,DNA模板1 μL,ddH2O 补足至25 μL。反应条件:95 ℃预变性5 min;94 ℃变性30 s,46 ℃退火30 s,72 ℃延伸90 s,共32个循环;72 ℃终延伸10 min,16 ℃结束反应。PCR产物于10 g·L-1的琼脂糖凝胶电泳检测。
1.6 目的基因的克隆和测序
按照上述反应体系和反应条件扩增125 μL PCR产物,1%琼脂糖凝胶电泳后,采用胶回收试剂盒对目的片段进行纯化回收,回收后再经1%琼脂糖凝胶电泳检测。检测正确后,将回收的DNA片段放置于-20 ℃保存,并按照pMD19-T载体推荐的连接体系进行连接,置于16 ℃过夜,然后转化到感受态DH5α中,涂于琼脂平板过夜培养,挑取阳性菌落,扩培,进行菌液PCR鉴定及测序。
1.7 分子特性分析
利用Lasergene 软件分析绵羊肺炎支原体的tuf基因核酸序列和氨基酸序列的同源性;利用ProtParam软件分析蛋白质的理化参数(http://www.expasy.ch/tools/ protparam.html);利用SignalP软件预测信号肽(http://www.cbs.dtu.dk/servic es/SignalP/);利用Tmpred软件预测蛋白质的跨膜区(http://www.ch.embnet.org/so ftware/TMPRED_form.html);利用ProtScale软件分析蛋白质的疏水性(www.expasy.org/cgi-bin/protscale.pl);利用HNN软件预测蛋白质的二级结构(http://npsa-pbil.ibcp);利用DNAStar软件预测蛋白质的抗原表位;利用PROSCAN软件分析蛋白质的功能结构域(http://npsa-pbil.ibcp.fr/);利用MEGA 4.1软件采用Neighbor-joining法、自举数据集为1 000次,构建系统进化树。
2 结 果
2.1 扩增、测序结果与序列分析
绵羊肺炎支原体临床分离株tuf基因的PCR扩增片段在1 200 bp左右出现一条特异性条带,该片段与预期目的片段大小相符(图1)。
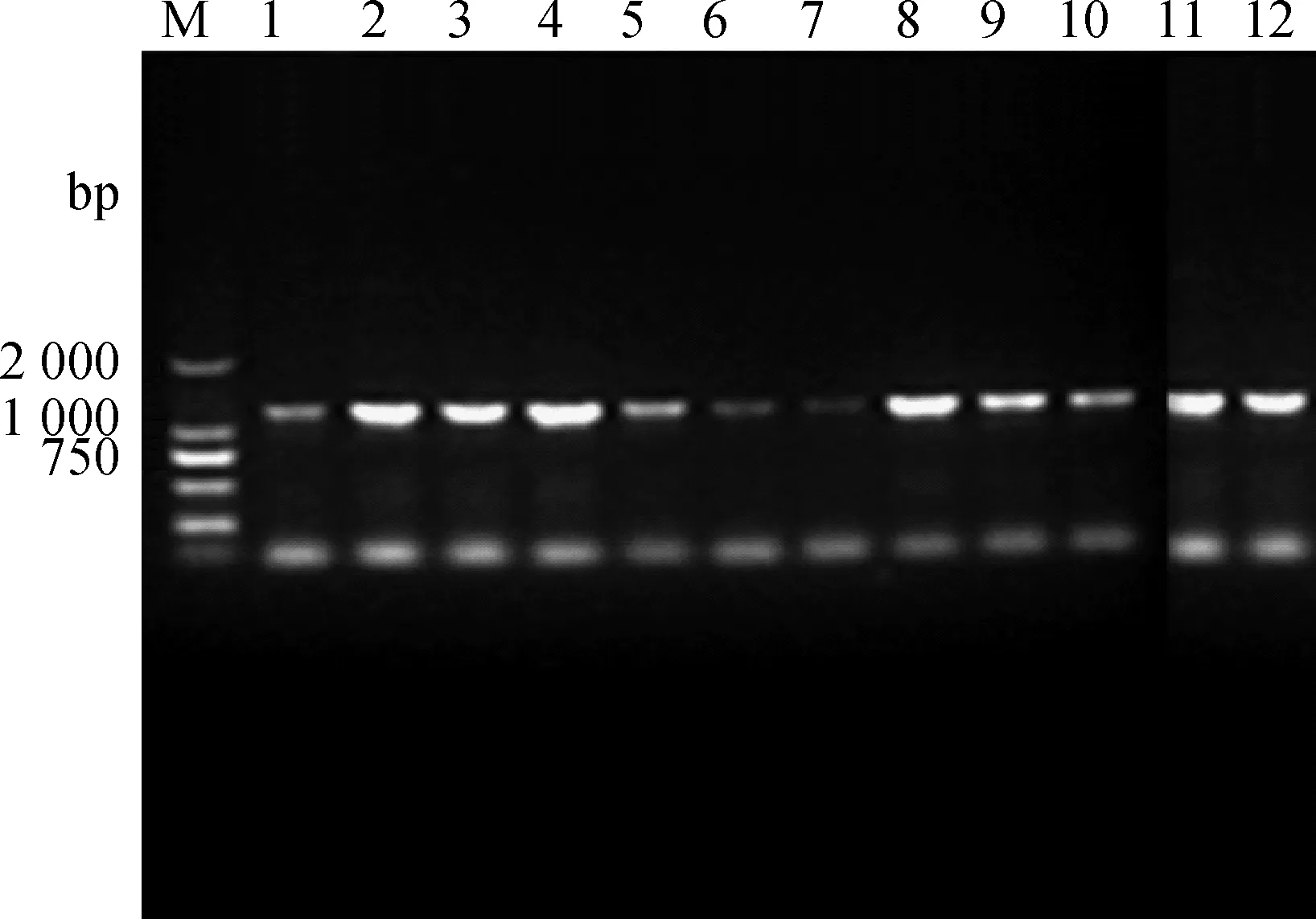
M.DL2000 DNA相对分子质量标准;1.Y98 株扩增片段;2~12.临床分离株扩增片段M.DL2000 DNA marker;1.Y98 PCR product;2-12.PCR products of 14 clinical isolates图1 tuf 基因扩增片段Fig.1 Amplification of tuf by PCR
对11株临床分离株和参考株Y98株tuf基因的编码区进行克隆测序。结果表明,Y98株tuf基因编码区长为1 209 bp,编码402个氨基酸,含有1个TGA密码子,在支原体中翻译为色氨酸。11株临床分离株tuf基因编码区长为1 209 bp,编码402个氨基酸,其中有2株分离株含有2个TGA密码子,其他分离株只含1个TGA密码子。
对11株临床分离株和参考株Y98进行生物信息学分析,结果表明,12株绵羊肺炎支原体tuf基因的GC平均含量为39.80%,核酸相似性为93.3%~100%,氨基酸相似性为93.3%~100%。此基因两端保守,但也存在变异位点,其中1—600 bp范围内突变率为2.5%,601—960 bp范围内突变率为6.94%,发生变异的位点主要在961—1 209 bp范围内突变率为30.9%。这12个菌株,有117个单核苷酸突变位点,其中无义突变为41个,有义突变为76个,推导53个氨基酸发生变化,氨基酸序列变化主要在靠近羧基端的325—354位点。
2.2 分离株绵羊肺炎支原体及其他支原体tuf基因序列和氨基酸序列比对与同源性分析
将绵羊肺炎支原体tuf基因序列与其他13种支原体的tuf基因序列及其编码的氨基酸序列进行同源性比对,可知该基因序列与其他13种支原体的tuf基因核酸序列相似性为58.8%~84.3%,与M.hyopneumoniae的相似性为84.3%。氨基酸序列相似性为62.0%~98.1%,与M.hyopneumoniae的相似性为98.1%。在138—309 bp和1 047—1 196 bp这两个区域内与其他支原体种间差异较大,但种内保守,为分子诊断提供了参考。
2.3 绵羊肺炎支原体延伸因子Tu蛋白质特性分析2.3.1 蛋白质基本特征分析 ProtParam软件在线分析表明,Y98株该蛋白质的相对分子质量为43 994.3 u,等电点为5.70,分子式为C1949H3140N532O597S13,半衰期为30 h,脂肪指数为88.03,不稳定系数为32.86,属于稳定蛋白质,该蛋白质总平均亲水性为-0.264,为亲水蛋白质。Y98株该蛋白质存在1个跨膜区,由98—115位之间的氨基酸组成。其中1株分离株含有2个跨膜区,其余分离株均为1个。2.3.2 信号肽预测分析 利用SignalP 4.1软件分析结果显示,Y98株和11株分离株均不含信号肽。2.3.3 蛋白质二级结构预测 利用HNN软件预测Y98株蛋白的二级结构,结果表明,该蛋白质的氨基酸序列中,α螺旋占28.86%,延伸链占25.37%,无规卷曲占45.77%,推断该蛋白质为混合型蛋白质;11株分离株也为混合型蛋白质。
2.3.4 蛋白质功能结构域预测 利用软件分析Y98株该蛋白质有6种功能位点,分别为: C-蛋白激酶磷酸化位点(43—45位、78—80位、181—183位、229—231位、236—238位、384—386位);酪蛋白激酶Ⅱ磷酸化位点(166—169位、205—208位、265—268位、384—387位、396—399位);酪氨酸激酶磷酸化位点(45—53位);N-豆蔻酰化位点(46—51位、66—71位、180—185位、184—189位、232—237位、298—303位、380—385位);ATP/GTP结合位点motif A(25—32位);GTP结合延伸因子信号(57—72位)。其中1株分离株在271—274位点多了一种N-糖基化功能位点,其余分离株与Y98株一样含有6种功能位点。
2.3.5 绵羊肺炎支原体tuf基因抗原表位分析 分析12个菌株氨基酸序列的抗原指数、亲水性、表面可能性。表明在氨基酸序列位点7—19、59—65、75—83、117—214、257—264、320—341区域,亲水性、抗原指数、表面可能性均较高,因此,这些区域都可能是B细胞表位(抗体表位)优势区域。如位点43—58、144—198、233—252、344—370、372—402区域表面可能性低,位于结构蛋白内部,但抗原指数却较高,因此这些可能是潜在的候选抗原位点,也可能成为免疫优势辅助性T淋巴细胞抗原位点。
2.4 基于tuf基因的遗传进化分析
基于tuf基因对14种支原体经行种间遗传进化关系分析,结果表明,绵羊肺炎支原体与猪肺炎支原体的遗传关系最近,与结膜炎支原体遗传关系次之,与其他常见能感染羊的支原体遗传关系较远(图2)与基于支原体全基因组(图3)的遗传关系一致。而基于16S rRNA的遗传关系(图4)则显示绵羊肺炎支原体与结膜炎支原体遗传关系最近,与其他支原体遗传关系较远。
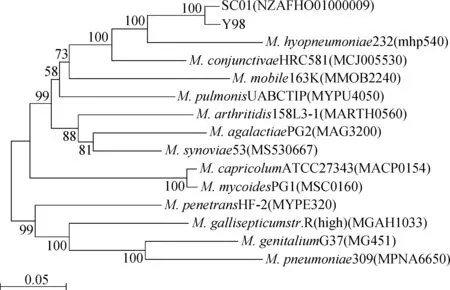
图2 支原体tuf基因邻近法进化树Fig.2 Neighbor-joining consensus tree for Mycoplasma tuf

图3 支原体全基因组邻近法进化树Fig.3 Neighbor-joining consensus tree for whole genome of Mycoplasma
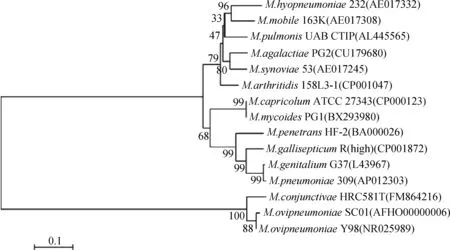
图4 支原体16S rRNA基因邻近法进化树Fig.4 Neighbor-joining consensus tree for Mycoplasma 16S rRNA
基于tuf基因对12株绵羊肺炎支原体进行种内遗传进化关系(图5),结果表明,11株分离株和Y98株单独各聚一支,11株临床分离株又分为2个亚支。有趣的是,来自Z地的3株分离株聚在一个亚支,来自J地的4株分离株聚在一个亚支,来自L地的分离株则不规则地分布在这2个亚支中。
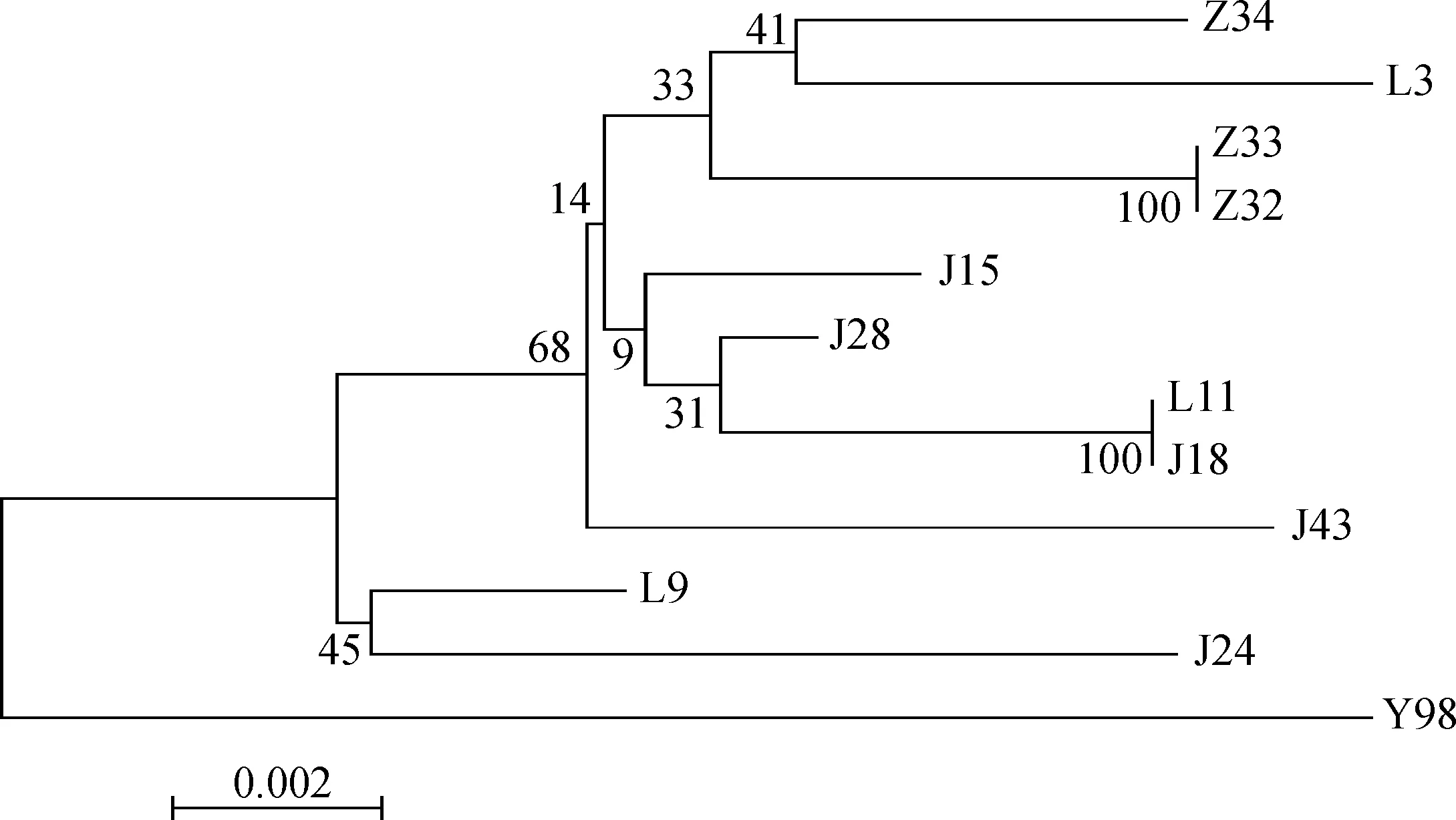
图5 绵羊肺炎支原体tuf基因邻近法进化树Fig.5 Neighbor-joining consensus tree for M.ovipneumonia tuf
3 讨 论
本研究主要对12个绵羊肺炎支原体菌株的tuf基因进行克隆和序列分析,发现有117个单核苷酸突变位点,其中无义突变为41个,有义突变为76个,可引起53个推导氨基酸发生变化,氨基酸序列突变主要发生在近羧基端325—354位点。其中1株分离株在271—274位点(NKSL)多出1个N-糖基化功能位点,在327—348位点多出1个跨膜结构域。有报道指出蛋白质的N-糖基化修饰是生物体调控蛋白质在组织和细胞中的定位、功能、活性、寿命和多样性的一种普遍翻译后方式,具有重要的意义[14]。但这1株和其他菌株在功能上究竟有无区别,还需进一步研究证实。
研究表明,tuf基因编码的蛋白质存在于细菌的表面,在肺炎支原体中亦如此[6,15]。支原体主要就是通过其特有的尖端结构与宿主呼吸道上皮细胞支架外的纤连蛋白结合进而黏附,是造成感染宿主的首要步骤。S.F.Dallo等[16]证实肺炎支原体延伸因子Tu和丙酮酸脱氢酶的E1β亚基复合物是作为纤连蛋白的结合蛋白。S.Balasubramanian等[17]发现肺炎支原体延伸因子Tu表面暴露的羧基区域是主要与纤连蛋白结合的区域,意味着延伸因子Tu羧基区域在宿主感染过程中起重要的作用。鉴于原核生物tuf基因的功能保守性,作者推测绵羊肺炎支原体tuf基因也可能在黏附过程中发挥作用,有待于进一步试验证实。
由于tuf基因在进化过程中相对保守,因此可作为遗传进化的靶标来分析不同支原体间的遗传进化关系。为此作者分别基于tuf、16S rRNA及全基因组基因对部分支原体的遗传进化关系进行了比较,结果表明,基于tuf基因绘制的进化树(图2)显示绵羊肺炎支原体和猪肺炎支原体亲缘关系最近,结膜炎支原体次之,与其他常见感染羊的支原体亲缘关系较远;其次,基于16S rRNA基因(图4) 绘制的进化树则显示绵羊肺炎支原体与结膜炎支原体亲缘关系较近,而与猪肺炎支原体亲缘关系有很大距离。这与基于tuf基因建立的进化关系相比有很大的不同。最后为了进一步确认遗传进化关系,进行了基于支原体全基因组的遗传进化分析(图3)以确定绵羊肺炎支原体与其他支原体的亲缘关系。在全基因组水平上,绵羊肺炎支原体与猪肺炎支原体亲缘关系最近,这与基于tuf基因显示的遗传进化关系一致。因此推断在绵羊肺炎支原体与其他支原体遗传进化关系中tuf基因比16S rRNA基因更适合作为分子靶标。
由于支原体基因组的GC含量一般都较低,且不同菌株之间存在很大的异质性而限制了分子检测方法的建立[2]。绵羊肺炎支原体tuf基因的平均GC含量达到了39.8%,相对于绵羊肺炎支原体全基因组28.85%的GC含量已经高了许多,相对较高的GC含量更有利于设计PCR检测引物。R.Söderlund等[18]利用该基因建立了检测猫支原体的荧光定量方法。作者的分析表明,绵羊肺炎支原体tuf基因在138—309 bp和1 047—1 196 bp区域内与其他感染羊的支原体差异较大,但种内保守,有作为设计检测绵羊肺炎支原体的分子靶点的潜力。绵羊肺炎支原体tuf基因虽然保守,但不同菌株在核苷酸序列和氨基酸序列都存在遗传多样性。由图5可见,参考株和11株分离株亲缘关系较远,而11株分离株又分成两大枝4个亚支,来自Z地的分离株都聚在一个亚支上,来自J地的5个分离株有4个也聚在一个亚支上,而来自L地的则分布在不同的亚支上。O.Makarova等[19]基于支原体tuf基因建立了鉴定支原体的DNA条码技术的方法;J.H.Shin等[20]以tuf基因为靶标建立了PCR限制性片段长度多态性分析方法用于非结核性分支杆菌的分子分型。以上报道证明在一些原核生物中tuf基因可用于分子分型。因此,进一步增加不同来源的菌株数量,评价tuf基因作为绵羊肺炎支原体分子分型的潜力,是一项值得研究的工作。
[1] PATEL H,MACKINTOSH D,AYLING R D,et al.A novel medium devoid of ruminant peptone for high yield growth ofMycoplasmaovipneumoniae[J].VetMicrobiol,2008,127(3-4): 309-314.
[2] PARHAM K,CHURCHWARD C P,MCAULIFFE L,et al.A high level of strain variation within theMycoplasmaovipneumoniaepopulation of the UK has implication for disease diagnosis and management [J].VetMicrobiol,2006,118(1-2):83-90.
[3] YANG F,TANG C,WANG Y,et al.Genome sequence ofMycoplasmaovipneumoniaestrain SC01 [J].JBacteriol,2011,193(18):5018.
[4] YOUNG C C,BERNLOHR R W.Elongation factor Tu is methylated in response to nutrient deprivation inEscherichiacoli[J].JBacteriol,1991,173(10):3096-3100.
[5] PROCELLA S F,BELLAND R J,JUDD R C.Identification of an EF-Tu protein that is periplasm-associated and processed in Neisseria gonorrhoeae [J].Microbiology,1996,142(9):2481-2489.
[6] REGULA J T,UEBERLE B,BOGUTH G,et al.Towards a two-dimensional proteome map ofMycoplasmapneumoniae[J].Electrophoresis,2000,21(17):3765-3780.
[7] EARGLE J,BLACK A A,SETHI A,et al.Dynamics of recognition between tRNA and elongation factor Tu [J].JMolBiol,2008,377(5):1382-1405.
[8] KRAB I M,PARMEQQIANI A.EF-Tu,a GTPase odyssey [J].BiochimBiophysActa,1998,1443(1-2):1-22.
[9] RICHARME G.Protein-disulfide isomerase activity of elongation factor EF-Tu [J].BiochemBiophysResCommun,1998,252(1):156-161.
[10] MAYER F.Cytoskeletons in prokaryotes [J].CellBiolInt,2003,27(5):429-438.
[11] KRISTENSEN R,GAUTHIER G,BERDAL K G,et al.DNA microarray to detect and identify trichotchecene- and moniliformin-producing Fusarium species [J].JApplMicrobiol,2007,102(4):1060-1070.
[12] PARADIS S,BOISSINOT M,PAQUETTE N,et al.Phylogeny of the Enterobacteriaceae based on genes encoding elongation factor Tu and F-ATPase beta-subunit [J].IntJSystEvolMicrobiol,2005,55(5):2013-2025.
[13] KAMLA V,HENRICH B,HADDING U.Phylogeny based on elongation factor Tu reflects the phenotypic features of mycoplasmas better than that based on 16S rRNA [J].Gene,1996,171(1):83-87.
[14] 周 蕾,顾建新.N-糖基化位点鉴定方法和非经典N-糖基化序列[J].生命科学,2011,23(6):605-611. ZHOU L,GU J X.N-glycosylation sites analysis and nonconsensus N-glycosylated sequences[J].ChineseBulletinofLifeSciences,2011,23(6):605-611.(in Chinese)
[15] MARQUES M A,CHITALE S,BRENNAN P J,et al.Mapping and identification of the major cell wall-associated components ofMycobacteriunleprae[J].InfectImmun,1998,66(6):2625-2631.
[16] DALLO S F,KANNAN T R,BLAYLOCK M W,et al.Elongation factor Tu and E1 beta subunit of pyruvate dehydrogenase complex act as fibronectin binding proteins inMycoplasmapneumoniae[J].MolMicrobiol,2002,46(4):1041-1051.
[17] BALASUBRAMANIAN S,KANNAN T R,BASEMAN J B.The surface-exposed carboxyl region ofMycoplasmapneumoniaeelongation factor Tu interacts with fibronectin [J].InfectImmun,2008,76(7):3116-3123.
[18] SÖDERLUND R,BÖLSKE G,HOLST B S,et al.Development and evaluation of a real-time polymerase chain reaction method for the detection ofMycoplasmafelis[J].JVetDiagnInvest, 2011,23(5):890-893.
[19] MAKAROVA O,CONTALDO N,PALTRINIERI S,et al.DNA barcoding for identification of ‘Candidatus Phytoplasmas’ using a fragment of the elongation factor Tu gene [J].PLoSOne,2012,7(12):e52092.
[20] SHIN J H,CHO E J,LEE J Y,et al.Novel diagnostic algorithm usingtufgene amplification and restriction fragment length polymorphism is promising tool for identification of nontuberculous mycobacteria [J].JMicrobiolBiotechnol,2009,19(3):323-330.
(编辑 白永平)
Molecular Characterization of the Elongation FactorTuGene inMycoplasmaovipneumoniae
YIN Zheng-jun,YUE Hua,TANG Cheng*
(CollegeofLifeScienceandTechnology,SouthwestUniversityforNationalities,Chengdu610041,China)
This study aimed at analyzing the molecular characterization of thetufgene inMycoplasmaovipneumoniae.Thetufgenes of the reference Y98 strain and 11 clinical isolates were cloned and sequenced,and then were used for bioinformatics analysis.A phylogenetic tree was constructed base on thetufgenes.The results showed that the open-reading frames (ORFs) oftufgenes in all the strains were 1 209 bp,which encoded 402 amino acids with 93.3%-100% amino acid identity and possessed higher antigenic index and higher lymphocyte epitopes.The average GC content of the protein coded bytufgene was 39.80%.The Tuf proteins of reference Y98 strain and 10 clinical isolates contained one strong transmembrane region and were absence of signal peptide,and contained six different functional sites.While another isolate contained two transmembrane regions,with a additional N-glycosylation site in the site of 271-274.The genes from 12 strains were identified 117 single nucleotide polymorphisms (SNPs),consisting of 41 synonymous polymorphisms and 76 non-synonymous polymorphisms leading to 53 amino acid changes,mainly in 325-354 region near the carboxyl region,which maybe leading to change the function of this protein.The phylogenetic relationship ofM.ovipneumoniaebased on whole genome and that based on thetufgene were consistent.So,thetufgene is a better target than 16S rRNA gene for the phylogenetic analysis forM.ovipneumoniae.Despite of its conservation,thetufgene still showed genetic diversity within the strains,making it potential for molecular typing.
Mycoplasmaovipneumoniae;elongation factorTugene;cloning;molecular characterization;phylogenetic
10.11843/j.issn.0366-6964.2015.02.016
2014-05-22
“十二五”高技术研究发展计划(863计划)(2012AA101304)
尹正军(1989-),男,江苏丹阳人,硕士,主要从事临床病原微生物快速检测技术的研究,E-mail:yinzhengjun1989@163.com
*通信作者:汤 承,E-mail:tangcheng101@163.com
S852.62
A
0366-6964(2015)02-0288-07
