一类细胞凋亡诱导剂的结构特征研究
2015-03-20张静晓付新梅潘艳秋
张静晓,李 燕,付新梅,潘艳秋*,杨 凌
(1.大连理工大学 化工学院,辽宁 大连 116024;2.大连理工大学 精细化工国家重点实验室,辽宁 大连 116024;3.中国科学院大连化学物理研究所 药物资源开发研究组,辽宁 大连 116023)
0 引 言
细胞凋亡(apoptosis)是程序性细胞死亡的一种形式,通过清除多余的衰老细胞维持体内细胞在数量、形态和功能上的平衡.细胞凋亡调控异常会导致个体不能正常发育、严重畸形或者不能存活,与许多疾病的发生密切相关.如在癌症的研究过程中,人们发现癌症的发生不仅与癌细胞的生长速度有关,而且与癌细胞的死亡速率密切相关,因此,细胞凋亡的异常调控被认为是癌症发生和发展的重要影响因素之一.在癌症治疗中,用细胞凋亡诱导剂加快癌细胞的凋亡速度,特异性地杀死癌细胞,将会达到治疗癌症的目的.
细胞凋亡可由两条途径介导:一条是死亡受体介导的信号激活细胞内的caspases(半胱氨酸天冬氨酸特异性蛋白酶),另一条是通过线粒体释放的凋亡酶激活因子激活caspases.这些活化的caspases可降解细胞内重要的蛋白,从而引起细胞死亡,因此,caspase在介导细胞凋亡的过程中起着非常重要的作用.Caspase-3是caspase家庭中最关键的凋亡执行分子之一,它可以剪切许多关键的凋亡靶蛋白酶,使细胞不可逆地走向死亡,研究具有caspase-3激活作用的细胞凋亡诱导剂是癌症研究的热点.
近二十多年中,已经发现了许多类具有药理学作用的细胞凋亡诱导剂,如黄酮类及其衍生物(如汉黄芩素[1]、紫杉醇[2]等)、蒽醌衍生物(如EMO、PHY[3]等)、多环类化合物(如N-苯基烟酰胺类[4]、藤黄酸[5]、JET-522[6]等)、烃类(如二烯丙基二硫[7]、姜酮酚[8]等)、多肽类(如RGD 多肽类[9]、polyoxypeptins A 和B[10])等,其中,N-苯基烟酰胺类、汉黄芩素、JET-522、EMO 和藤黄酸等诱导剂都被证明是通过激活caspase-3来达到诱导癌细胞凋亡的目的的.Cai的研究小组通过高通量筛选实验筛选出一类具有caspase-3激活作用的细胞凋亡诱导剂,包括N-苯基烟酰胺类[4]、4-苯胺基-2-(2-吡啶基)嘧啶类[11-12]、4-芳氨 基 喹唑啉 类[13-14]、N-苯基-1H-吡唑并[3,4-b]喹啉-4-胺类[15]等.这几类诱导剂通过作用在caspase-3活化上游某个靶点,诱导T47D 乳腺癌细胞的凋亡.
传统的药物研究方法研发周期长,非常消耗人力、物力和财力,计算机辅助药物设计通过定量地分析小分子结构和其物理化学特征与生物活性之间的关系,归纳出分子结构特点和生物活性之间的规律,从而能更好地指导开发具有高药理作用的化合物,大大缩短了研发周期,降低了人力和财力的消耗.本文以153个来自Cai的研究小组的细胞凋亡诱导剂为研究对象进行药物设计.由于这类诱导剂分子的具体作用靶点尚不清楚,采用间接药物设计方法,包括比较分子力场法(CoMFA)和比较分子相似性指数法(CoMSIA),建立诱导剂的分子结构特征与其生物活性之间的三维定量构效关系(3D-QSAR)模型,为设计和开发高活性的新型细胞凋亡诱导剂提供理论指导.
1 材料与方法
1.1 数据集
数据集由9类具有caspase-3激活作用的细胞凋亡诱导剂组成,如图1所示,包括N-苯基烟酰 胺[4](M001)、4-苯胺基-2-(2-吡 啶基)嘧啶类[11-12](M010)、4-芳氨基喹唑啉类[13-14](M034)、N-苯基-1H-吡唑并 [3,4-b]喹啉-4-胺类(M041)[15]、(4-萘基)苯基甲酮类(M054)[16]、N-甲基-N-苯基-1-萘胺类(M060)[16]、4-苯胺基-N-甲基噻吩并[3,2-d]嘧啶类(M097)[17]、4-苯胺基-N-甲基噻吩并[2,3-d]嘧啶类(M105)[17]、N-芳基-9-氧-9H-芴基-1-甲酰胺类(M115)[18-19],共153个化合物小分子.化合物的结构和其对应的活性数据(EC50)均来自Cai的研究小组.建模过程中,EC50值被转化为pEC50(-logEC50),作为定量构效关系研究的因变量.遵循训练集和验证集的分子比例为4∶1的原则,随机选择119个化合物作为训练集,其余34个化合物作为验证集来验证所建模型的可靠性.图2为20个代表性化合物的分子结构及其活性数据.
1.2 构象优化和分子叠合
3D-QSAR模型的建立和分析在SYBYL 6.9软件包(Tripos公司)中进行.利用该软件中的Sketch molecule模块构建小分子化合物初始的三维结构.小分子能量优化的参数设置为Tripos力场、Gasteiger-Huckel电荷、Powell共轭能量梯度法,能量收敛限定为2 090J/(mol·nm)[20].为了构建具有较好预测能力的QSAR 模型,基于配体分子的公共骨架对所有的化合物进行了叠合,在叠合过程中,以拥有最高pEC50值的M071 号分子(pEC50=8.70)作为模板分子.如图3(a)所示,蓝色加粗部分是分子叠合过程所选的公共子架,所有分子叠合的效果如图3(b)所示.由图可见,数据集中的所有分子叠合良好,为构建QSAR模型奠定了基础.

图1 9类细胞凋亡诱导剂的代表性分子结构Fig.1 Representative molecular structures of nine series of apoptosis inducers
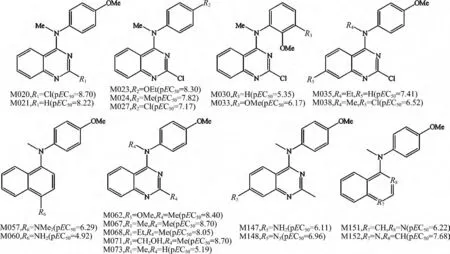
图2 代表性细胞凋亡诱导剂的分子结构及其活性信息Fig.2 Molecular structures and activities of representative apoptosis inducers
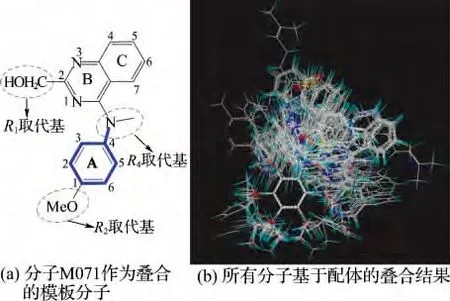
图3 数据集中所有分子的叠合图Fig.3 Molecular alignment of all molecules in the dataset
1.3 CoMFA和CoMSIA分析
骨采用CoMFA 和CoMSIA 两种分析模型研究小分子诱导剂与其活性之间的定量构效关系.在CoMFA 分析中,叠合后所有的小分子被放入一个步长为0.2nm 的三维网格中,以一个带正电荷的sp3碳原子为探针,分别对每个网格点上 位 阻 场(steric field)和 静 电 场(electrostatic field)的大小及分布进行了探测,计算过程中的能量阈值设定为144kJ/mol,其他参数采用系统默认值.CoMSIA 分析是CoMFA 分析的一种延伸,除了具有CoMFA 分析中的位阻场和静电场外,还引入了疏水场(hydrophobic field)、氢键供体场(hydrogen-bond donor field) 和 受 体 场(hydrogen-bond acceptor field).在CoMSIA 分析中,网格步长设置为0.5nm,其他参数的设置和CoMFA 分析中一致.分子j上的原子i在网格点q上CoMSIA相似性指数(AF)可由下面公式[21]计算得到:
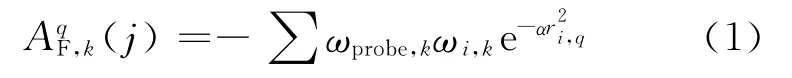
式中:k为位阻场、静电场、疏水场、氢键供体场和受体场的描述符,ωprobe,k为探针离子的分子场特征(原子半径为0.1nm、电荷为+1、疏水性为+1、氢键受体和供体的强度都为+1),ωi,k为原子i的分子场特征k的实际数值,ri,q为探针离子在网格点q上与原子i之间的距离.
1.4 PLS分析
为了构建具有统计显著性的3D-QSAR 模型,以CoMFA 和CoMSIA 中的各个分子力场为自变量,以pEC50为因变量进行了PLS 分析.在PLS分析中,先用交叉验证抽一法(leave-oneout)计算验证系数Q2,得到最佳主成分数Nc,然后采用非交叉验证法计算获得QSAR 模型,并得到非交叉验证相关系数R2ncv.相关系数R2pred由下式计算得到:

式中:Yactual为验证集中化合物的实验活性,Ymean为训练集中化合物的平均活性,Ypred为验证集中化合物的预测活性.
2 结果与讨论
2.1 CoMFA和CoMSIA模型
在QSAR 建模过程中,从153个诱导剂中随机选取119个化合物作为模型的训练集,其余34个化合物作为验证集.通过PLS分析,最终得到了较优的CoMFA 和CoMSIA 模型(如表1所示),最优的CoMFA 模型仅由位阻场描述符组成.最终7个成分的交叉验证系数Q2=0.32,非交叉验证系数R2ncv=0.88,标准差ese为0.31.使用验证集来检验该模型时,验证集预测值相关系数R2pred为0.75,验证集的最小预测偏差ep为0.74.在统计分析中,如果模型的Q2<0.4,表明此模型的预测能力小于均值预测能力.故最优CoMFA 模型的预测能力并不理想.
最优的CoMSIA 模型由位阻场、静电场、疏水场、氢键供体场和受体场5种力场的描述符构建而成.模型的统计结果显示,交叉验证系数Q2为0.51(Q2>0.4),最佳组分数Nc为7,标准差为0.29,表明此模型具有良好的内部预测能力.较高的非交叉验证系数(R2ncv=0.89)表明该模型中的预测值和实验值自洽性良好.用验证集来检验该模型,结果为R2pred=0.82,ep=0.63,表明此模型良好的外部预测能力.在构建CoMSIA 模型的5个力场中,静电场和氢键受体场的贡献率分别为25%和29%,明显高于其他3个力场的贡献率,表明氢键受体场和静电场对细胞凋亡诱导剂的生物活性影响比较大.
图4是最优CoMSIA 模型中所有小分子诱导剂实验和预测活性值之间线性拟合图.由图可见,数据集中所有分子均匀地分布在趋势线的两侧,分子的预测值和实验值非常接近,表明CoMSIA模型良好的预测能力.

表1 CoMFA 和CoMSIA 模型的统计结果Tab.1 Statistical results of the CoMFA and CoMSIA models
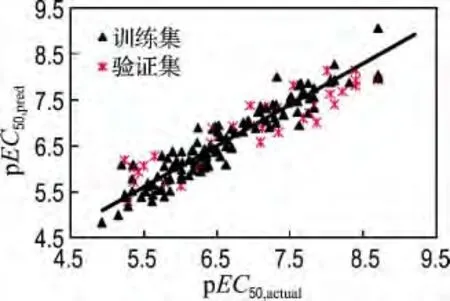
图4 最优CoMSIA 模型中小分子诱导剂的实验和预测活性值之间的线性拟合Fig.4 The predicted versus actual pEC50values of the training and test sets based on the optimal CoMSIA model
2.2 三维等值线图
为了进一步地分析各个力场对诱导剂分子活性的影响,以M071 号分子为模板分析最优CoMSIA 模型的StDev*Coeff三维等值线图,结果如图5所示.由图可见,这些等值线图能非常直观地反映诱导剂小分子的各个不同力场对其生物活性的影响.
图5(a)是最优CoMSIA 模型的位阻场等值线图,其中绿色云团表示这一区域中大的取代基有利于诱导剂分子的生物活性,黄色区域代表大的取代基不利于分子的活性.对应图2结构式由图5(a)可见,在R1和R4取代基附近有一个较大的黄色云团,而在R4取代基以及B 环和C 环后方有一个大的绿色云团,表明R1取代基位置上连接一个较小的取代基有利于提高小分子的诱导活性,而R4取代基处连接一个中等大小的取代基有利于提高小分子的生物活性.结果与诱导剂分子的实验结果是一致的,例如,M067 号分子(pEC50=8.70)在R1取代基处有一个相对较小的甲基取代基,活性高于在R1处取代基为乙基的M068号分子(pEC50=8.05);在R4处为甲基取代基的M020号分子(pEC50=8.70),活性明显高于M035号分子(R4位取代基为乙基,pEC50=7.41);R4取代基非常小时也不利于分子的活性,如M073号分子(pEC50=5.19)的R4位取代基为H,活性明显小于R4位取代基为甲基的M067号分子.同理,R2取代基处出现了一个小的绿色云团,表明此位置上一个较大的取代基有利于分子的活性.
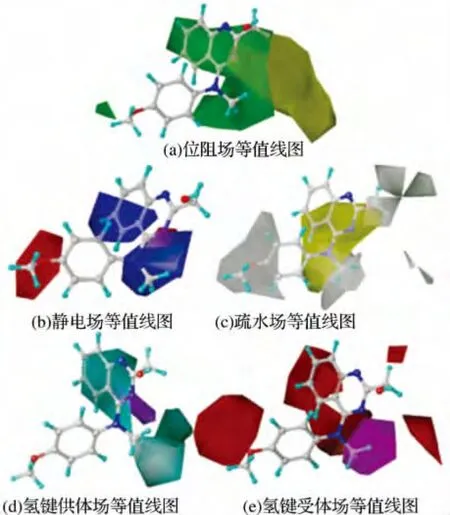
图5 最优CoMSIA 模型的等值线图Fig.5 Optimal CoMSIA model contour plots
最优CoMSIA 模型的静电场等值线图如图5(b)所示,在蓝色区域引入正电性的基团或在红色区域引入负电性的基团有利于提高诱导剂小分子的生物活性.对应图2结构式由图5(b)可见,在R2取代基处有一个较大的红色云团,表明在R2处引入负电性的基团有利于诱导剂的活性.譬如M020和M023(pEC50=8.30)号分子由于在R2位置引入了具有负电性取代基(分别是OMe和OEt取代基),而具有较高的活性.同样的现象也发生在B 环的1号位上,此处的红色小云团表明在此位置引入负电性的取代基有利于分子的诱导活性.在A 环的3号位上有一个较大的蓝色区域,表明诱导剂分子在该区域是正电性的基团能提高其生物活性.结果与实验值也是一致的,如M030(pEC50=5.35)和M033(pEC50=6.17)号分子在此3号位上含有OMe取代基,分子活性较低.同理,C环的7号位上有一个蓝色云团,表明在此区域引入正电性的基团有利于分子的生物活性.
图5(c)是最优CoMSIA 模型的疏水场等值线图.图中的黄色和白色云团分别代表该区域中疏水性和亲水性的取代基能提高分子的生物活性.对应图2结构式由图5(c)可见,R2取代基有一个较大的白色云团,表明该位置上亲水性的基团有益于分子生物活性,可以通过分子实验活性来证明,如M020、M023、M024(pEC50=7.82)和M027(pEC50=7.17)号分子具有共同的分子骨架,仅在R2位上的取代基不同,分别是OMe、OEt、Me和Cl,而其活性则是逐渐降低的.同理R1取代基上两个白色的云团也显示了该区域中引入亲水性基团是有利于诱导剂分子活性的.而围绕R4取代基和B环有一块大的黄色云团和两块小的白色云团,表明在这一区域内取代基的疏水性对诱导剂分子的活性影响比较大.
最优CoMSIA 模型氢键供体场的等值线图如图5(d)所示.图中青色云团代表该区域中氢键供体基团将有利于提高诱导剂分子的活性,紫色云团表示氢键供体基团在该区域不利于分子的生物活性.对应图2结构式由图5(d)可见,围绕着R4取代基有两个青色区域,表明在这一区域具有氢键供体作用的取代基能提高分子的生物活性,可以用实验数值来证明,如 M062(pEC50=8.40)、M067和M071号分子在该区域都含有氢键供体基团(Me),生物活性明显高于其他诱导剂的活性.同理C 环周围的青色区域表明该区域氢键供体取代基有利于分子的生物活性.另外,在B环的1号位上出现了紫色区域,表明该区域中氢键供体基团不利于小分子的诱导活性.M021 号分子(pEC50=8.22)在B 环1号位上具有氢键受体取代基(—N—),活性大于在该位上取代基为—C—的M152号分子(pEC50=7.68).
图5(e)为最优CoMSIA 模型的氢键受体场等值线图,其中紫色云团代表氢键受体场在这一区域有利于分子活性,红色云团代表氢键受体场在这一区域不利于分子活性.对应图2结构式由图5(e)可见,R4和R1取代基周围发现了红色和紫色云团,结果与氢键供体场的结果一致.此外,在R2取代基处出现一个红色的云团,表明在此区域氢键受体基团不利于诱导剂分子的活性.M020和M023号分子在该区域处存在氢键供体基团(OMe和OEt),分子的活性明显高于其他诱导剂分子的生物活性.
通过对最优CoMSIA 模型的5个力场等值线图进行分析,确定了此类化合物作为细胞凋亡诱导剂的分子结构特点.以M071号分子为模板,分析得出了各取代基处的力场作用特征,如图6所示.
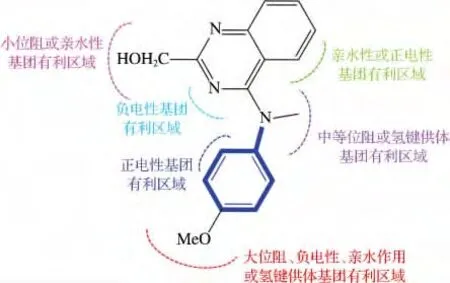
图6 各取代基处的力场作用特征Fig.6 Interaction features of compound at each substituent
2.3 诱导剂分子结构比较分析
目前,具有caspase-3激活作用的细胞凋亡诱导剂主要包括多环类、黄酮类和烃类等,代表性的化合物如图7所示.其中,Cai的研究小组通过T47D 乳腺癌细胞caspase-3活化检测法筛选出了一大批多环类的细胞凋亡诱导剂[22],如N-苯基烟酰胺[4]、藤黄酸[5]、2-氨基-3-氰基-7-二甲氨基-4-芳基-1H-苯并吡喃[23]、2-氯-N-(4-甲氧基苯基)-N-甲基喹唑啉-4-胺(M020)[13]等;来自大蒜的二烯丙基二硫(DADS)能通过激活caspase-3 诱导人类白血病HL-60细胞的凋亡[7];姜酮酚([6]-paradol)及其结构类似物也是通过激活caspase-3途径引起口腔鳞状细胞癌KB的凋亡[8].
由图7可见,具有caspase-3激活作用的凋亡诱导剂呈现出结构多样性,通过作用在caspase级联反应上游某个已知的或是未知的作用靶点来激活caspase-3,最终诱导细胞凋亡.由于这类分子结构多样,作用靶点不明,很难对其进行综合的比较和分析.因此,本文用间接的药物设计方法对153个具有公共骨架的细胞凋亡诱导剂进行结构和活性的比较分析,有助于指导开发更高效的细胞凋亡诱导剂.
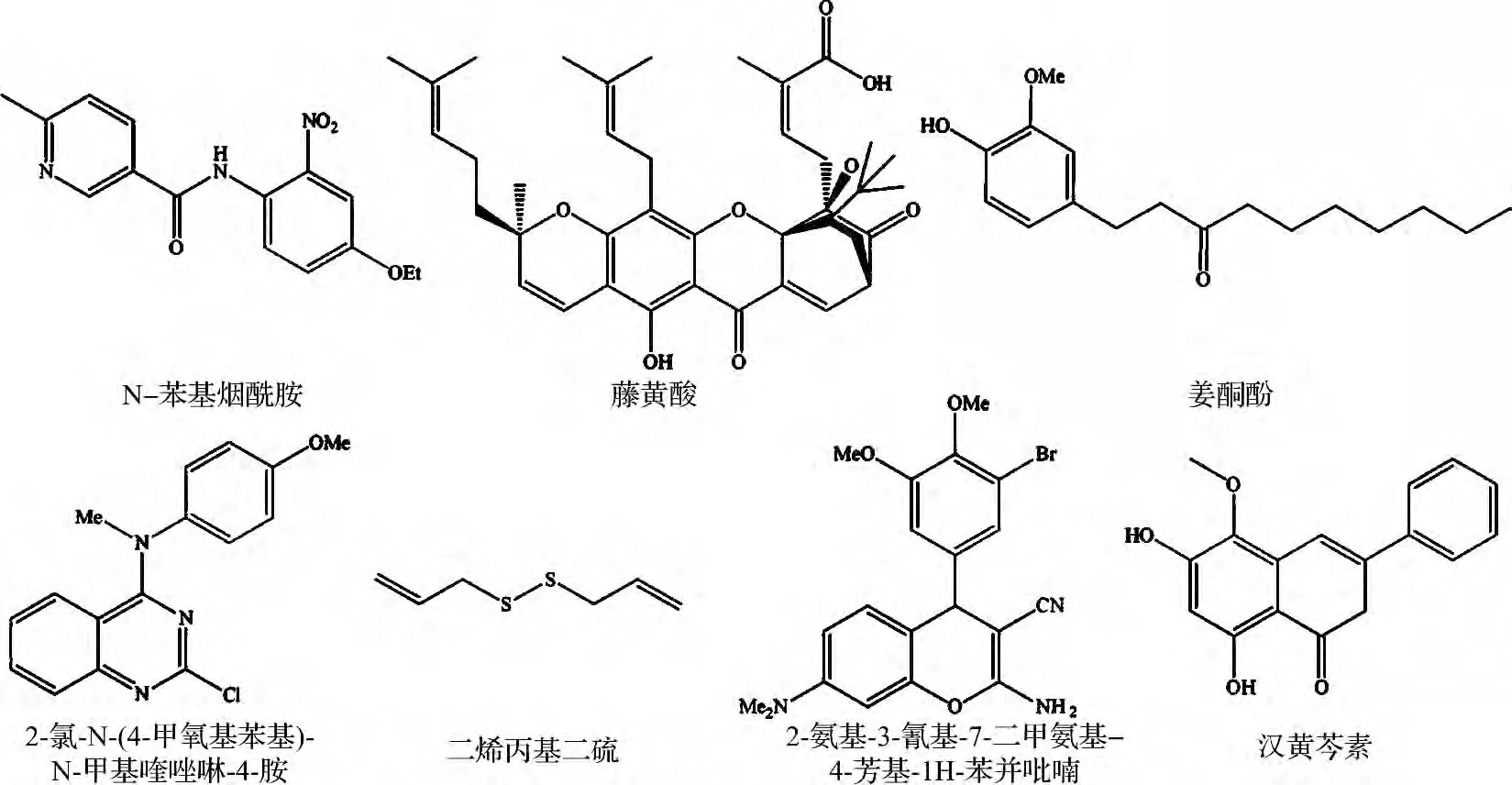
图7 Caspase-3激活作用的细胞凋亡诱导剂Fig.7 Apoptosis inducers through activation of caspase-3
3 结 论
采用3D-QSAR中的CoMFA 和CoMSIA 两种方法,对153个具有caspase-3激活作用的细胞凋亡诱导剂进行三维定量构效关系研究.结果显示,CoMSIA 模型(Q2=0.51,R2ncv=0.89 和R2pred=0.82)具有良好的可靠性和预测能力.通过对该模型中5个力场等值线图的分析,确定了此类化合物作为细胞凋亡诱导剂的结构特点.具体结论如下:
(1)诱导剂分子中的R1、R2和R4取代基,以及B环的1号位、A 环的3号位和C 环的7号位是提高生物活性的关键位点.
(2)R2位取代基提高诱导剂分子活性的因素有位阻大、负电性大、有亲水性和具有氢键供体作用.
(3)在R4取代基处引入位阻中等大小和(或)有氢键供体作用的基团,以及R1位置引入位阻小和(或)亲水的取代基时,诱导剂小分子的生物活性会增加.
(4)A 环的3号位和(或)C 环的7号位引入正电性基团,以及B 环的1号位引入负电性取代基,都将有利于增加诱导剂分子的活性.
[1] Lee Woan-ruoh,Shen Shing-chuan,Lin Hui-yi,et al.Wogonin and fisetin induce apoptosis in human promyeloleukemic cells,accompanied by a decrease of reactive oxygen species,and activation of caspase 3 and Ca2+-dependent endonuclease [J].Biochemical Pharmacology,2002,63(2):225-236.
[2] Wang T H,Wang H S,Soong Y K.Paclitaxelinduced cell death:where the cell cycle and apoptosis come together[J].Cancer,2000,88(11):2619-2628.
[3] Chen Yen-chou,Shen Shing-chuan,Lee Woanruoh,etal.Emodin induces apoptosis in human promyeloleukemic HL-60 cells accompanied by activation of caspase 3cascade but independent of reactive oxygen species production[J].Biochemical Pharmacology,2002,64(12):1713-1724.
[4] Cai S X,Nguyen B,Jia S J,etal.Discovery of substituted N-phenyl nicotinamides as potent inducers of apoptosis using a cell-and caspase-based high throughput screening assay [J].Journal of Medicinal Chemistry,2003,46(12):2474-2481.
[5] Zhang H Z,Kasibhatla S,Wang Y,etal.Discovery,characterization and SAR of gambogic acid as a potent apoptosis inducer by a HTS assay[J].Bioorganic & Medicinal Chemistry,2004,12(2):309-317.
[6] LI Hong-liang,ZHANG Hai-wei,CHEN Dan-dan,etal.JTE-522,a selective COX-2 inhibitor,inhibits cell proliferation and induces apoptosis in RL95-2 cells [J].Acta Pharmacologica Sinica,2002,23(7):631-637.
[7] Kwon K B,Yoo S J,Ryu D G,etal.Induction of apoptosis by diallyl disulfide through activation of caspase-3in human leukemia HL-60 cells [J].Biochemical Pharmacology,2002,63(1):41-47.
[8] Keum Young-sam,Kim Jin,Lee Keun-hyung,et al.Induction of apoptosis and caspase-3activation by chemopreventive [6]-paradol and structurally related compounds in KB cells[J].Cancer Letters,2002,177(1):41-47.
[9] Buckley C D,Pilling D,Henriquez N V,etal.RGD peptides induce apoptosis by direct caspase-3 activation[J].Nature,1999,397(6719):534-539.
[10] Umezawa K,Nakazawa K,Ikeda Y,etal.Polyoxypeptins A and B produced bystreptomyces:Apoptosis-inducing cyclic depsipeptides containing the novel amino acid (2S,3R)-3-hydroxy-3-methylproline[J].Journal of Organic Chemistry,1999,64(9):3034-3038.
[11] Sirisoma N,Kasibhatla S,Nguyen B,etal.Discovery of substituted 4-anilino-2-(2-pyridyl)pyrimidines as a new series of apoptosis inducers using a cell-and caspase-based high throughput screening assay.Part 1: Structure-activity relationships of the 4-anilino group[J].Bioorganic& Medicinal Chemistry,2006,14(23):7761-7773.
[12] Sirisoma N,Pervin A,Nguyen B,etal.Discovery of substituted 4-anilino-2-arylpyrimidines as a new series of apoptosis inducers using a cell-and caspase-based high throughput screening assay.2.Structure-activity relationships of the 2-aryl group[J].Bioorganic & Medicinal Chemistry Letters,2009,19(8):2305-2309.
[13] Sirisoma N,Kasibhatla S,Pervin A,etal.Discovery of 2-chloro-N-(4-methoxyphenyl)-Nmethylquinazolin-4-amine (EP128265,MPI-0441138)as a potent inducer of apoptosis with high in vivo activity[J].Journal of Medicinal Chemistry,2008,51(15):4771-4779.
[14] Sirisoma N,Pervin A,Zhang H,etal.Discovery of N-(4-methoxyphenyl)-N,2-dimethylquinazolin-4-amine,apotent apoptosis inducer and efficacious anticancer agent with high blood brain barrier penetration[J].Journal of Medicinal Chemistry,2009,52(8):2341-2351.
[15] Zhang H Z,Claassen G,Crogran-Grundy C,etal.Discovery and structure-activity relationship of Nphenyl-1H-pyrazolo[3,4-b]quinolin-4-amines as a new series of potent apoptosis inducers [J].Bioorganic & Medicinal Chemistry,2008,16(1):222-231.
[16] Jiang S,Crogan-Grundy C,Drewe J,etal.Discovery of(naphthalen-4-yl)(phenyl)methanones and N-methyl-N-phenylnaphthalen-1-amines as new apoptosis inducers using a cell-and caspase-based HTS assay[J].Bioorganic & Medicinal Chemistry Letters,2008,18(21):5725-5728.
[17] Kemnitzer W,Sirisoma N,Jiang S,etal.Discovery of N-aryl-9-oxo-9H-fluorene-1-carboxamides as a new series of apoptosis inducers using a cell-and caspase-based high-throughput screening assay.2.Structure-activity relationships of the 9-oxo-9Hfluorene ring [J].Bioorganic & Medicinal Chemistry Letters,2010,20(3):1288-1292.
[18] Kemnitzer W,Sirisoma N,Nguyen B,etal.Discovery of N-aryl-9-oxo-9H-fluorene-1-carboxamides as a new series of apoptosis inducers using a cell-and caspase-based high-throughput screening assay.1.Structure-activity relationships of the carboxamide group [J].Bioorganic & Medicinal Chemistry Letters,2009,19(11):3045-3049.
[19] Kemnitzer W,Sirisoma N,May C,etal.Discovery of 4-anilino-N-methylthieno[3,2-d]pyrimidines and 4-anilino-N-methylthieno[2,3-d]pyrimidines as potent apoptosis inducers [J].Bioorganic &Medicinal Chemistry Letters,2009,19(13):3536-3540.
[20] Grant J A,Williams R L,Scheraga H A.Ab initio self-consistent field and potential-dependent partial equalization of orbital electronegativity calculations of hydration properties of N-acetyl-N′-methylalanineamide[J].Biopolymers,1990,30(9-10):929-949.
[21] ZHANG Bai-dong,LI Yan,ZHANG Hui-xiao,et al.3D-QSAR and molecular docking studies on derivatives of MK-0457,GSK1070916and SNS-314 as inhibitors against Aurora B kinase [J].International Journal of Molecular Sciences,2010,11(11):4326-4347.
[22] Sirisoma N,Pervin A,Zhang H,etal.Discovery of N-methyl-4-(4-methoxyanilino)quinazolines as potent apoptosis inducers.Structure-activity relationship of the quinazoline ring[J].Bioorganic& Medicinal Chemistry Letters,2010,20(7):2330-2334.
[23] Kemnitzer W,Drewe J,Jiang S,etal.Discovery of 4-aryl-4H-chromenes as a new series of apoptosis inducers using a cell-and caspase-based highthroughput screening assay.1.Structure-activity relationships of the 4-aryl group [J].Journal of Medicinal Chemistry,2004,47(25):6299-6310.
