中国沿海不同地区泥蚶(Tegillarca granosa)的遗传多样性分析*
2015-03-08刘春芳王海艳
刘春芳 李 翠 张 振 王海艳①
(1. 中国科学院海洋研究所 青岛 266071; 2. 中国科学院大学 北京 100049)
泥蚶Tegillarca granosa (Linnaeus, 1758)隶属于软体动物门(Mollusca)、双壳纲(Bivalvia)、蚶目(Arcoida)、蚶科(Arcidae), 俗称血蚶、银蚶、花蚶、粒蚶、血螺、瓦垄哈, 是一种营穴居生活的底栖动物,广泛分布于我国沿海, 在山东、浙江、福建和广东均有养殖(李凤兰, 1983; 徐凤山等, 2008)。泥蚶的营养与药用价值高(雷焕宗等, 2004; 温扬敏等, 2009; 柯佳颖等, 2010), 是我国四大传统养殖贝类之一。
目前关于泥蚶不同地理群体形态变化差异与判别分析(张永普等, 2004)、生化遗传(贾守菊等, 2003;李松伟等, 2011)以及养殖(尤仲杰等, 2002; 吴洪喜等,2004)的研究有大量报道。王日昕等(2005)利用同工酶分析的研究认为, 我国沿海泥蚶以东海和南海的分界线为界, 可以分为两个类群, 两个类群内部遗传分化不明显, 而类群间的遗传距离较大, 而郑文娟等(2009)对泥蚶线粒体细胞色素氧化酶亚基I (cytochrome oxidase subunit I, COI)基因的研究则在此基础上提出南北类群的分化达到了亚种甚至种的水平。Ni等(2012)对中国东海和南海两个海区泥蚶 COI和 ITS1两个标记的研究, 提出了中国海区两个泥蚶的进化显著单元(evolutionarily significant units, ESUs), 其中一个ESU广泛分布中国沿海, 而另一ESU则分布在南海海域, 后者濒临消失。
线粒体DNA (mtDNA)是一种共价闭环双链DNA分子, 由于其相对于核基因具有遵循严格的母系遗传、几乎没有重组、结构简单、进化速度快以及在不同的区域进化速度存在差异等特点, 逐渐成为种群遗传学和分子系统发育研究的重要标记(Avise,2000)。线粒体 COI基因具有进化速度较快, 有相对保守区等特点, 可作为种属系统进化研究的良好标记(Martínez-Navarro et al, 2005), 目前被广泛应用于物种鉴定、分子系统发育分析(Matsumoto, 2003; Cho et al, 2007)。目前关于中国沿海泥蚶群体的遗传分化研究仍不够全面, 本研究利用线粒体 COI基因分析中国沿海泥蚶7个群体遗传多样性, 以期为泥蚶的分类、遗传多样性研究及合理、有效地利用和保护泥蚶种质资源提供数据参考。
1 材料与方法
1.1 样品的采集
样品分别采集于海南海口市、广西防城港市、广西北海市、广东湛江市、福建漳州市、山东荣成市和山东威海市。样品采集后立即用75%酒精固定, 保存备用。
1.2 DNA的提取、扩增及测序
取泥蚶闭壳肌肌肉约100mg充分剪碎, 用TIANamp海洋动物DNA提取试剂盒(北京天根生物有限公司)提取全基因组。线粒体COI基因扩增引物LCO1490和HCO2198 (Folmer et al, 1994)序列分别为:5′-GGTCAACAAATCATAAAGATATTGG-3′, 和5′-AAAC TTCAGGGTGACCAAAAAATCA-3′, 由上海桑尼生物技术公司合成。PCR反应体系为: 总体积25μL, 其中2μL 10× PCR缓冲液, 1.5mmol/L Mg2+, 200μmol/L dNTPs, 1U Taq DNA 聚合酶, 正反向引物各0.4μmol/L,2μL模板DNA溶液(10—100ng/μL)。PCR反应程序为:94°C预变性4min; 94°C 40s, 48°C 1min, 72°C 1min, 30个循环; 然后72°C延伸10min。PCR扩增产物送上海桑尼生物技术公司测序。
1.3 数据分析
序列结果利用Mega 5.1软件进行同源排序比对,同时辅以人工校对。采用Kimura 2-paramter (K2P)(Kimura, 1980)距离矩阵计算群体内和群体间遗传距离, 并结合非加权配对算数平均法(Unweighted Pair Group Method with Arithmetic Means, UPGMA)构建分子系统树。
采用DnaSP 5.1软件计算单倍型多样性指数(Haplotype diversity, Hd)、核苷酸多样性(Nucleotide diversity,Pi)、平均核苷酸差异数(K)、不同群体序列的碱基组成、变异位点, 并通过Mega 5.1软件基于K2P方法利用26个单倍型序列构建NJ树和UPGMA树。
用Arlequin 3.1软件计算群体间遗传分化系数(F-statistics, Fst)及其显著性(重复次数为1000), 并用分子变异分析(AMOVA)方法估算遗传变异在群体间和群体内的分布。
2 实验结果
2.1 线粒体COI基因片段的序列特征以及单倍型分析
经序列比对与分析后, 得到长度为592bp的泥蚶COI基因片段, 其中共有16个单突变位点, 87个简约信息位点。在所有群体的序列中, A+T含量(表 1)平均为60.83%。
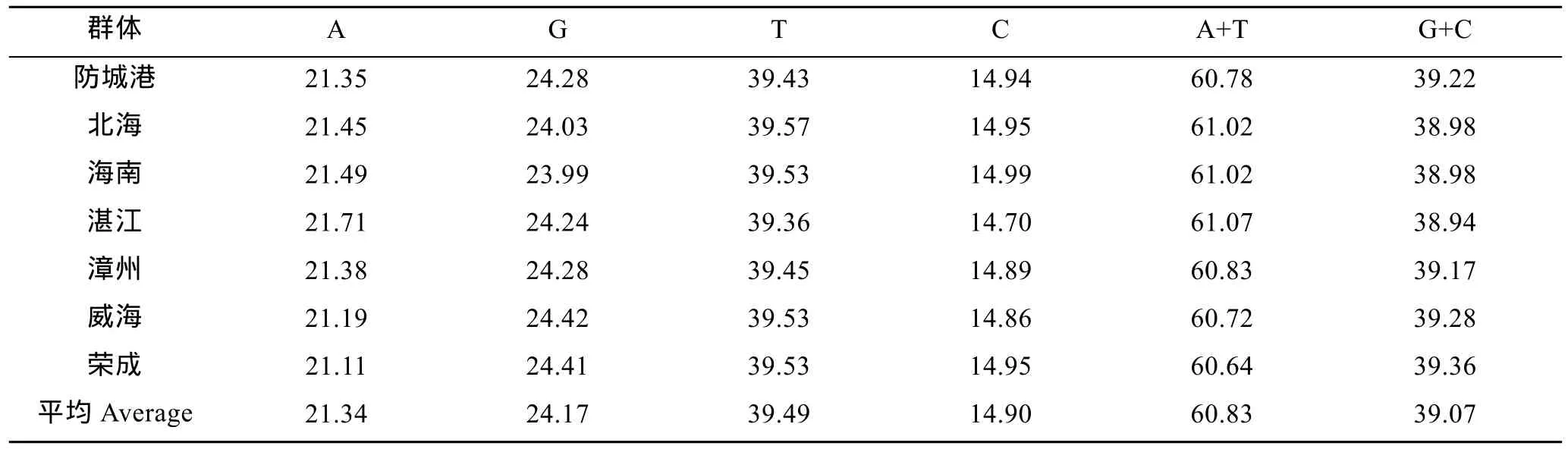
表1 7个泥蚶群体COI基因片段序列碱基组成(%)Tab.1 Percentage of base composition of partial COI gene sequences in the seven T. granosa populations
多态性遗传参数统计(表 2)显示, 62个个体共检出26个单倍型, 群体间共享单倍型4个, 占单倍型总数的15.4%, 其余22个单倍型均为某个群体所特有。单倍型Hap5为2个群体(防城港、威海)共享, 单倍型Hap8为4个群体(防城港、北海、漳州、威海)共享, 单倍型 Hap13为 2个群体(海南、北海)共享, 单倍型Hap14为 5个群体(防城港、北海、漳州、威海、荣成)共享。26个单倍型中, 共享单倍型Hap14的个体总数最多, 占分析个体总数的35.5%。
2.2 群体遗传多样性与遗传结构
群体内和平均遗传多样性参数见表 3, 南方类群(防城港、北海、海南和湛江)的遗传多样性参数普遍较高: Hd为0.914—1.000, 多态位点比例为1.18—15.7,Pi为0.01182—0.07095, K值为7.0000—42.00); 而北方类群(荣成和威海)的遗传多样性参数则较低: Hd为0.532和1.000, 多态位点比例为0.507和0.338, Pi为0.00124和0.00338, K值为0.73684,和2.000, 漳州群体遗传参数居中(Pi和K值较荣成群体略低)。
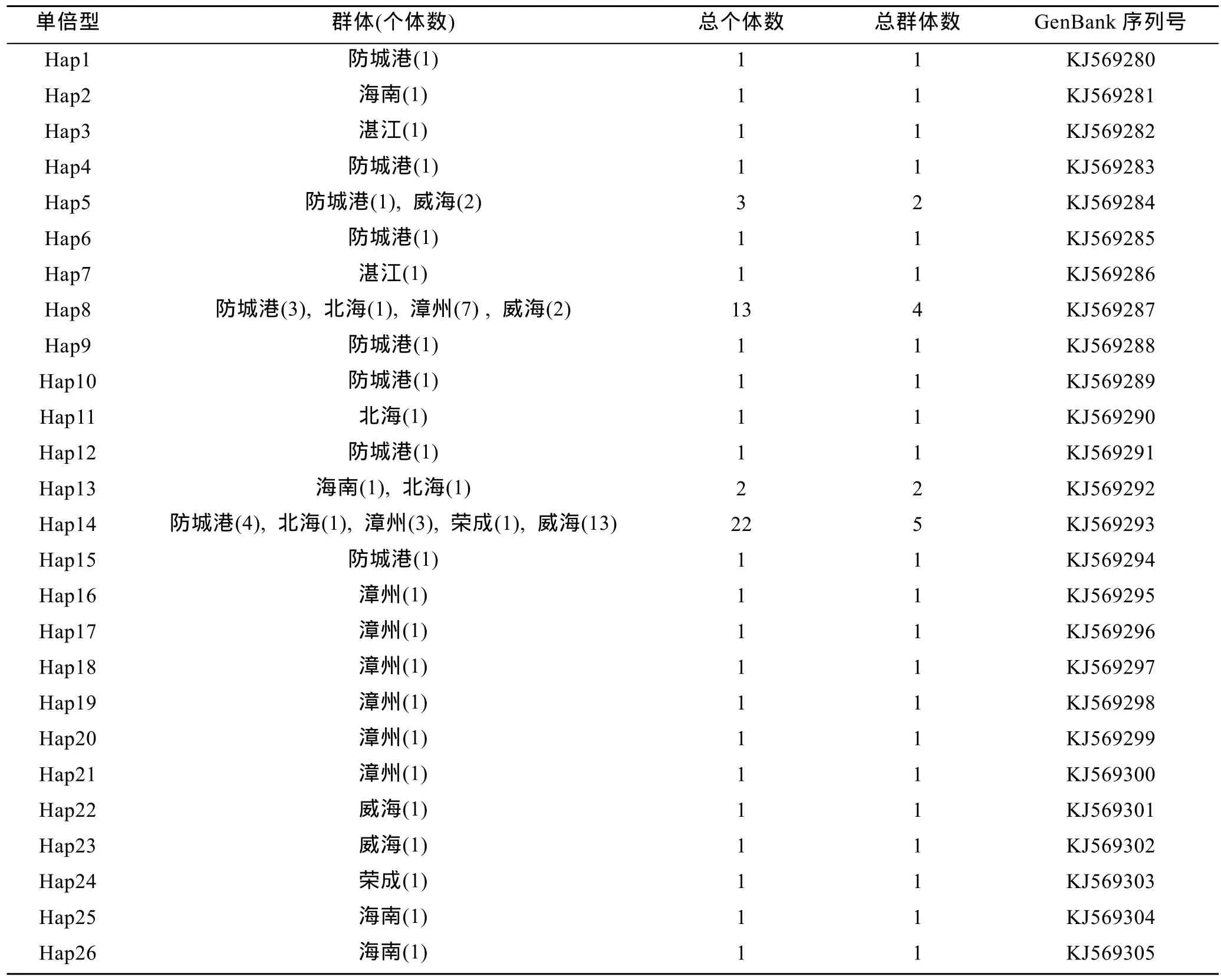
表2 泥蚶26个独立单倍型在7个群体中的分布Tab.2 Distribution of 26 unique haplotypes of COI sequences across the seven T. granosa populations
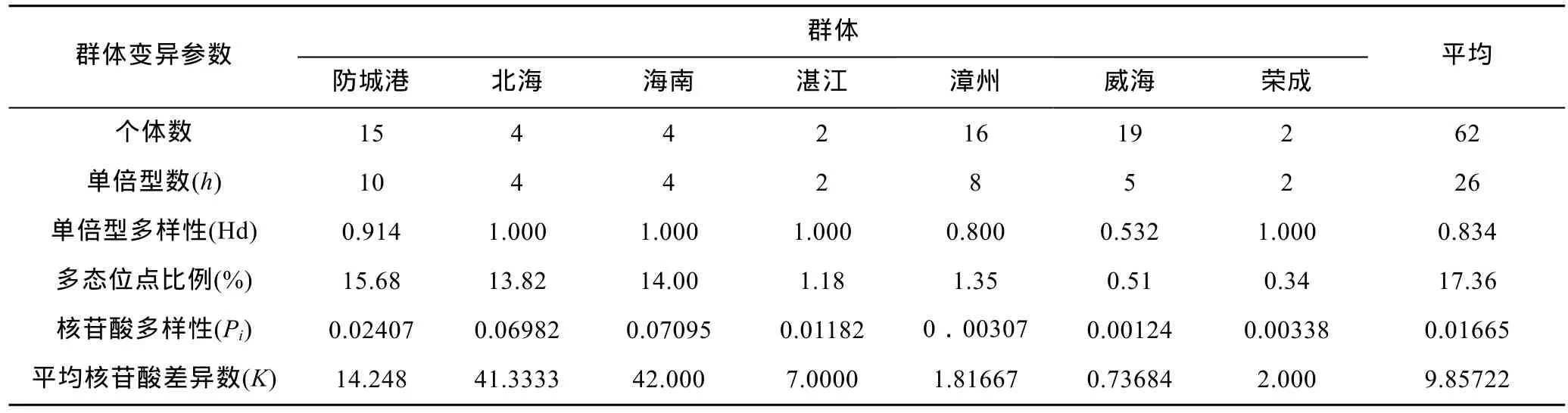
表3 基于COI基因片段序列的7个泥蚶群体的遗传多样性参数Tab.3 The genetic diversity parameters of partial COI gene in the seven populations of T. granosa
根据K2P方法, 利用Mega 5.1计算群体内、群体间遗传距离以及群体分化系数Fst见表 4。威海泥蚶群体内遗传距离最低, 海南群体内遗传距离最高; 荣成群体和威海群体之间的遗传距离最近, 漳州群体与两者之间的遗传距离次之; 北海群体、海南群体与其他群体的遗传距离普遍较高, 而两者群体内遗传距离最高; 防城港泥蚶群体内遗传距离大于与其他群体(北海和海南两个群体除外)之间的遗传距离; 湛江与漳州、威海、荣成的遗传距离均小于与北海和海南两个群体的遗传距离。而在群体间遗传距离关系在UPGMA聚类树中威海和荣成群体聚成一个小支, 之后依次和漳州、湛江、防城港和北海、海南聚在一起(图 1)。
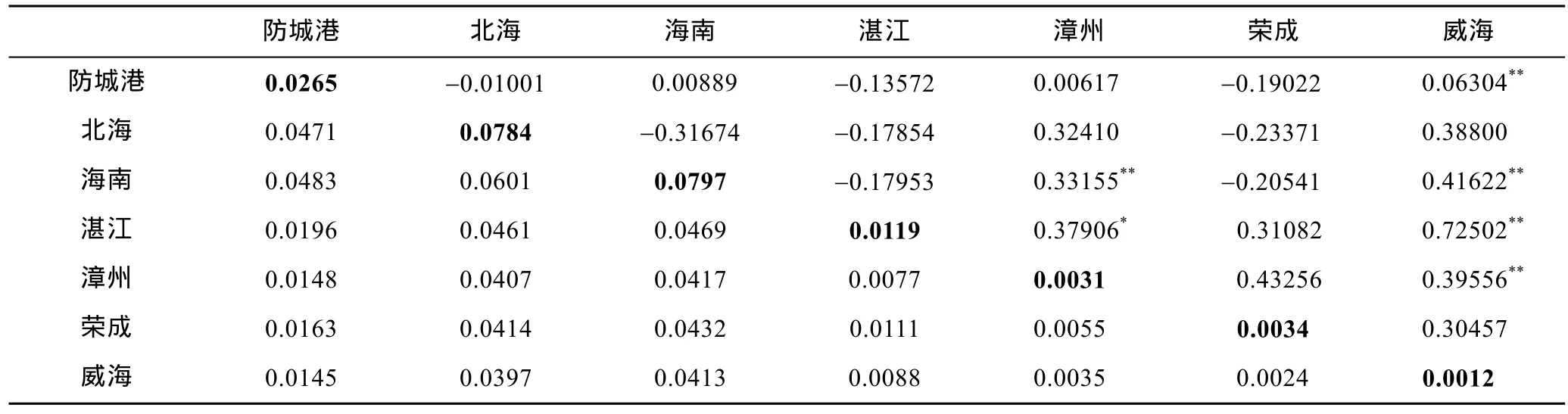
表4 基于COI基因片段序列的7个泥蚶群体间遗传距离(对角线下)、群体内遗传距离(粗体显示)和群体间分化系数(对角线上)Tab.4 Genetic distances between populations (below diagonal), genetic distances within populations (in bold values) and pair-wise Fst (above the diagonal) of the seven populations of T. granosa based on partial COI gene
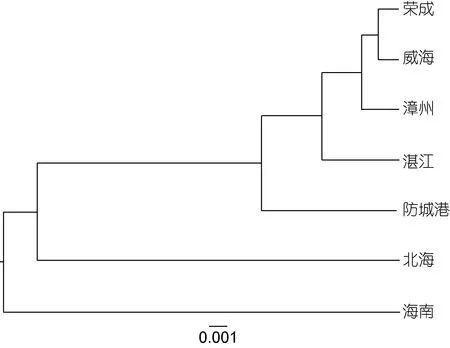
图1 基于泥蚶7个群体遗传距离构建的UPGMA聚类图Fig.1 UPGMA dendrogram based on genetic distances of seven populations of T. granosa
利用Tamura & Nei (Tamura et al., 1993)计算方法,得到群体间遗传分化系数Fst(表4)及其显著性(未列出)。群体遗传学认为, 可以根据Fst值将群体间分化程度分为4个等级: 较弱(0—0.05), 中等(0.05—0.15),较大(0.15—0.25)和极大(>0.25)。湛江群体与威海群体遗传分化最高, 漳州群体除与防城港群体无明显分化, 同其他群体分化极大。防城港群体与威海群体存在中等分化, 同其他群体间的遗传分化较弱。
基于Tamura & Nei计算方法的AMOVA分析(表5)表明, 泥蚶群体遗传分化的 90.51%来自群体内部,仅有9.49%来自群体间。说明, 泥蚶群体遗传分化主要表现为群体内分化, 群体内部遗传多态性较高。采用邻接法(NJ)和非加权配对算术平均法(UPGMA)构建的泥蚶7个群体26个单倍型的分子系统树见图2,3。在两种树中, 所有的26个单倍型被分成两个分支,Hap2, Hap11和Hap12聚在一支, 其余单倍型聚成一大支, 大支在NJ树和UPGMA树中有变化。

表5 基于COI基因片段序列的7个泥蚶群体遗传变异的AMOVA分析Tab.5 Hierarchical AMOVA results based on the partial COI sequences of the seven populations of T. granosa
3 讨论
3.1 泥蚶COI基因片段核苷酸组成及单倍型分析
在调查的7个泥蚶群体的序列中, A+T的平均含量为60.83%, 均大于G+C平均含量(表1), 其他贝类线粒体DNA的碱基组成也具有这种特点(Chen et al,2009; 李咏梅等, 2009; 郭宝英等, 2012)。Grant等(1998)通过对线粒体DNA序列的遗传变异分析, 将不同核苷酸多样性和单倍型多样性间的组合分成了四种类型: 第一种类型是较低的单倍型多样性(Hd<0.5)与较低的核苷酸多样性(Pi<0.005), 第二种类型是高Hd值与低Pi值, 第三种类型是低Hd值与高Pi值, 其余为第四种类型。据此可将北海、海南两个群体归为第四种类型, 而其余群体则为第二种类型。Grant等(1998)认为第四种类型是由于一个大而稳定的种群经过长时间演化所产生或两个不同种群二次接触所造成, 而第二种类型由瓶颈效应后伴随种群的迅速成长与突变的积累形成。泥蚶群体的这种特征可能是由于现阶段演化力量(如不同品系泥蚶的引入造成的基因渗透)、生境的不连续性、种群数量变动的不稳定性等因素的影响。
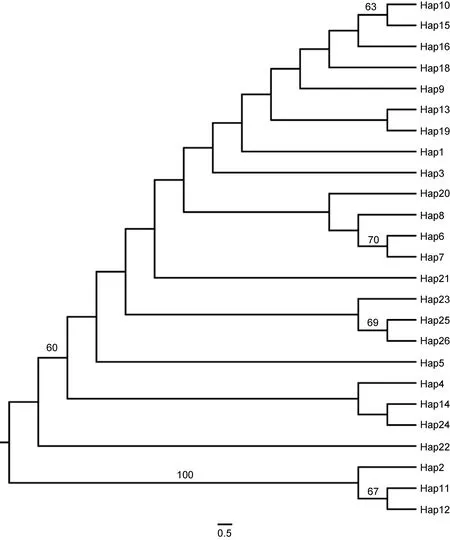
图2 基于泥蚶26个COI基因单倍型构建的NJ树Fig.2 The NJ dendrogram based on the sequences of 26 haplotypes of T. granosa
3.2 泥蚶群体的遗传多样性
王日昕等(2005)和郑文娟等(2009)认为中国沿海的泥蚶可以分为福建以南和福建以北(包括福建)两大类群, 后者发现福建南北群体间的遗传距离在0.1521—0.1540, 远大于类群内部群体间的遗传距离, 且南北两类群之间的遗传距离(0.1529)远大于类群内部遗传距离(南方类群内部遗传距离为0.0006, 北方类群内部的遗传距离为0.0016), 两类群的分化程度达到了亚种甚至种的水平。
Ni等(2012)根据对中国东部海域和南海泥蚶类群的COI和ITS1的研究, 提出了两个ESUs, 其中一个ESU广泛分布中国沿海, 而另一个则分布在汕头以南海区。在本研究以泥蚶26个单倍型构建的系统发育树中, 南方类群中的单倍型Hap2(海南)、Hap11(北海)和Hap12(防城港)形成稳定的一个小支, 聚在了发育树的根部, 这和Ni等(2012)提出的一个ESU相似, 即,均分布在中国南海区域。
而在本研究中, 南方类群(防城港, 北海, 海南和湛江)遗传多样性参数普遍高于北方类群(荣成, 威海), 而漳州泥蚶群体的遗传多样性参数居中, 表现出明显的地区差异(表3)。南方类群(防城港、北海、海南、湛江)群体间遗传距离(0.0196—0.0601)、南方群体与北方群体(荣成、威海)的遗传距离(0.0088—0.0432), 均大于北方群体间的遗传距离(0.0024—0.0055)。根据遗传学知识(见 2.2部分), 荣成与防城港、海南、北海分化较弱, 而与威海、湛江、漳州均有极大分化; 福建漳州群体与南方类群(防城港, 北海, 湛江, 海南)以及北方类群(威海, 荣成)的遗传分化程度均较高(表 5); 南方类群群体间分化较弱, 北方类群群体间有明显分化, 后者可能是荣成样本量偏少造成。而对各群体遗传分化参数Fst的AMOVA分析, 发现泥蚶的遗传分化主要来自于群体内(贡献率达90.52)。因此, 泥蚶群体由北向南遗传多样性增高, 而群体内遗传分化系数也呈现升高的趋势。这可能与 1980年以后每年从北方(主要为山东地区)大量引入南方泥蚶苗种(郑家声等, 1995), 以及北部湾地区复杂的生活环境有关。

图3 基于泥蚶26个COI基因单倍型构建的UPGMA树Fig.3 The UPGMA dendrogram based on the sequences of 26 haplotypes of T. granosa
王日昕, 李太武, 吕振明等, 2005. 泥蚶(Tegillarca granosa)不同地理居群同工酶变异及遗传分化的研究. 海洋与湖沼,36(3): 227—234
尤仲杰, 王一农, 陈 坚, 2002. 乐清湾塘养泥蚶的生长. 水产学报, 26(5): 440—447
李凤兰, 1983. 中国近海蚶科的研究, Ⅱ粗饰蚶亚科. 见: 中国贝类学会. 贝类学文集-第一辑. 北京: 科学出版社,40—41
李松伟, 李 晔, 李成华等, 2011. 泥蚶(Tegillarca granosa)精氨酸激酶的基因克隆与表达. 海洋与湖沼, 42(1):119—123
李咏梅, 陈秀荔, 彭 敏等, 2009. 基于线粒体COI基因序列探讨广西钦州湾牡蛎的遗传分化. 西北农林科技大学学报(自然科学版), 37(3): 60—65
吴洪喜, 柴雪良, 2004. 浙江乐清湾泥蚶的繁殖习性和生长特性. 动物学杂志, 39(3): 47—50
张永普, 林志华, 应雪萍, 2004. 不同地理种群泥蚶的形态差异与判别分析. 水产学报, 28(3): 339—342
郑文娟, 朱世华, 沈锡权等, 2009. 基于线粒体 COI基因序列探讨泥蚶的遗传分化. 动物学研究, 30(1): 17—23
郑家声, 王梅林, 王志勇等, 1995. 泥蚶的性腺发育和生殖周期. 青岛海洋大学学报, 25(4): 503—510
柯佳颖, 陈寅山, 戴聪杰, 2010. 泥蚶血清及血细胞免疫功能研究. 水生态学杂志, 3(3): 80—84
贾守菊, 张永普, 陈艳乐等, 2003. 不同种群泥蚶的 5种同工酶研究. 东海海洋, 21(3): 34—41
徐凤山, 张素萍, 2008. 中国海产双壳类图志. 北京: 科学出版社, 39
郭宝英, 祁鹏志, 李继姬等, 2012. 紫贻贝(Mytilus edulis)养殖群体的遗传多样性及遗传结构分析. 海洋与湖沼, 43(1):52—56
温扬敏, 高如承, 2009. 泥蚶的营养与药用价值. 经济动物学报, 13(3): 168—170, 182
雷焕宗, 林植华, 张永普, 2004. 不同种群泥蚶肉游离氨基酸及无机元素的含量和组成. 河南科学, 22(2): 203—205
Avise J C, 2000. Phylogeography: the History and Formation of Species. Oxford: Harvard University Press, 1—361
Chen A H, Li Z X, Feng G N, 2009. Phylogenetic relationships of the genus Meretrix (Mollusca: Veneridae) based on mitochondrial COI gene sequences. Zoological Research,30(3): 233—239
Cho E S, Jung C G, Sohn S G et al, 2007. Population genetic structure of the ark shell Scapharca broughtonii Schrenck from Korea, China, and Russia based on COI gene sequences. Marine Biotechnology, 9(2): 203—216
Folmer O, Black M, Hoeh W et al, 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology, 3(5): 294—299
Grant W S, Bowen B W, 1998. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation. Journal of Heredity, 89(5): 415—426
Kimura M, 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. Journal of Molecular Evolution, 16(2):111—120
Martínez-Navarro E M, Galián J, Serrano J, 2005. Phylogeny and molecular evolution of the tribe Harpalini (Coleoptera,Carabidae) inferred from mitochondrial cytochrome-oxidase I. Molecular Phylogenetics and Evolution, 35(1): 127—146
Matsumoto M, 2003. Phylogenetic analysis of the subclass Pteriomorphia (Bivalvia) from mtDNA COI sequences.Molecular Phylogenetics and Evolution, 27(3): 429—440
Ni G, Li Q, Kong L et al, 2012. Phylogeography of the bivalve Tegillarca granosa in coastal China: implications for management and conservation. Marine Ecology Progress Series, 452: 119—130
Tamura K, Nei M, 1993. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Molecular Biology and Evolution,10(3): 512—526
