三疣梭子蟹几丁质酶基因克隆鉴定及在低盐胁迫和蜕皮周期中的表达分析*
2015-03-08吕建建高保全
张 凤 吕建建 刘 萍① 高保全 李 健 陈 萍
(1. 中国水产科学研究院黄海水产研究所 青岛 266071; 2. 上海海洋大学水产与生命学院 上海 201306)
甲壳类几丁质酶基因(Chitinase)属于 18家族糖苷键水解酶多家族基因(Zhanget al,2014),几丁质酶结构域典型: 第 18家族几丁质酶催化结构域、富含S/T的连接区和结合几丁质的结构域,其基本功能可以特异降解几丁质。甲壳动物在蜕皮过程中必须通过Chitinase消化旧的几丁质外骨骼。因此,Chitinase是甲壳动物生长不可或缺的酶。除此之外,Chitinase在甲壳动物中还具有其它重要的生理功能,包括消化几丁质类食物以及参与免疫防御等(Arakaneet al,2010; Zhanget al,2014)。相比昆虫而言(Zhuet al,2008; Arakaneet al,2010; Zhanget al,2011),由于缺少基因组背景,甲壳类Chitinase研究开展较晚,然而由于其重要性,近几年已成研究热点。目前在中国对虾(Fenneropenaeus chinensis)、凡纳滨对虾(Litopenaeus vannamei)、脊尾白虾(Exopalaemon carinicauda)、日本仿长额虾(Pandalopsis japonica)以及日本沼虾(Macrobrachium nipponense)中均已开展Chitinase基因克隆、分类及功能研究。研究表明: 甲壳类几丁质酶基因至少分六类(陈少波等,2004),可能在甲壳类蜕皮及消化中发挥重要作用(Huanget al,2010; Proespraiwonget al,2010; Zhanget al,2010;Rochaet al,2012; Zhanget al,2014)。
通过筛选本实验室构建的三疣梭子蟹(Portunus trituberculatus)高通量转录组文库,发掘到一个三疣梭子蟹Chitinase基因,命名为PtCht。该基因在不同盐度胁迫下三疣梭子蟹鳃组织中显著差异表达(Lvet al,2013),且通过聚类分析发现其不属于任何一个已知甲壳类Chitinase分类,反而与昆虫Chitinase基因聚为一类,暗示其可能为甲壳类中新的Chitinase成员。本研究根据转录组文库中PtCht基因部分序列,通过RACE技术克隆该基因cDNA全长; 通过实时定量 PCR研究该基因在三疣梭子蟹各组织中的表达分布以及在不同蜕皮时期中表达规律; 同时研究其在三疣梭子蟹低盐胁迫进程中鳃和肝胰腺组织中的表达变化。研究结果将丰富甲壳类Chitinase研究内容,为进一步研究甲壳类Chitinase的功能提供了重要信息。
1 材料与方法
1.1 材料
实验材料来自于本实验养殖在昌邑市海丰水产养殖有限责任公司的健康三疣梭子蟹,体质量(5.78±1.11)g。暂养于 20m3的蟹池中,每池 200 只,共6池,暂养3d。自然海水养殖(盐度33),水温控制在24—26°C左右,pH 8.7,保持供氧充足,每天更换1/3的水量,暂养期间每天18:00喂食蓝蛤。
1.2 总RNA的提取及cDNA第一链的合成
Trizol试剂分别提取各组织的总RNA,用核酸定量仪(Biodropsis,BO-1000)与1.0%琼脂糖凝胶电泳检测总 RNA的质量及完整性; 分别取相同量的第六对鳃和肝胰腺的总 RNA 混匀,进行 3′和 5′RACE 的cDNA第一链的合成,具体方法参照 SMARTTMRACE Amplification Kit说明书。
1.3 全长cDNA的克隆及测序
根据从三疣梭子蟹 cDNA文库中获得的Chitinase的EST序列,利用Primer Premier 5.0软件设计 3′RACE 和 5′RACE 特异性引物。3′和 5′末端扩增使用 Advantage 2 PCR Kit进行。用特异性引物Cht-F和通用引物UPM进行3′末端扩增,用特异性引物Cht-R和通用引物UPM进行5′末端扩增(表1),方法同段亚飞等(2013)。PCR 程序设为: 94°C 30 s,65°C 1 min,72°C 3 min,30 个循环。
1.4 序列分析
利用Vecror NTI 11.0软件对测得的序列去除冗余序列并进行序列拼接,开放阅读框的预测和氨基酸的翻译用软件DNAStar的EditSeq完成(徐文斐等,2014)。同源性比对、蛋白质理化性质预测、蛋白质功能结构域分析、蛋白信号肽分析、PtCht与其它物种的Cht氨基酸序列多序列比对、构建系统进化树的具体方法见段亚飞等(2013)。
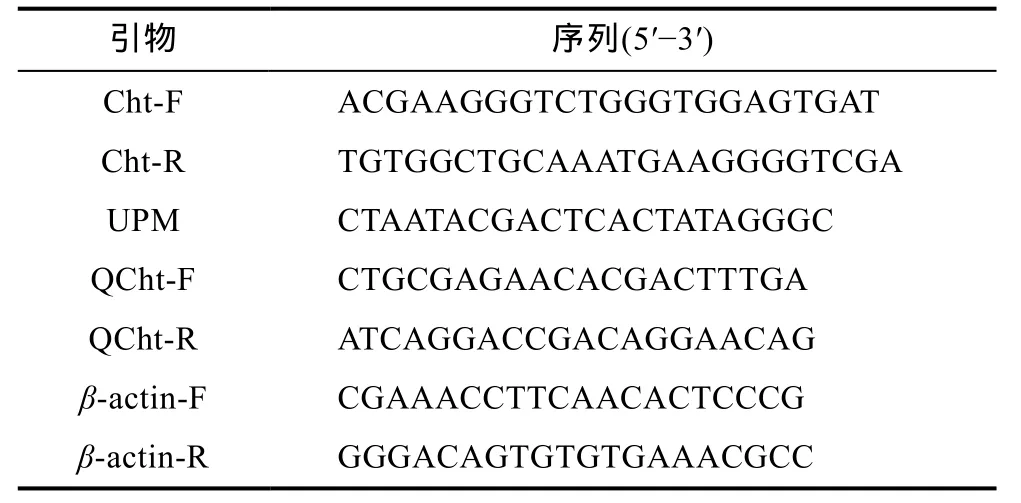
表1 三疣梭子蟹Chitinase克隆和mRNA相对表达分析所用引物序列Tab.1 Sequences of primers used for PtCht cloning and relative mRNA expression analysis
1.5 蜕皮周期实验
将三疣梭子蟹放在20m3蟹池自然海水中暂养3d,分2个池子,每池150只,按照朱冬发(沈洁等,2011)的方法,用解剖剪剪下游泳足趾节末端置于干净载玻片上,于Olympus CX21FS显微镜下观察,分别挑出处于蜕皮间期、前期、后期的三疣梭子蟹各3只。分别取第1对鳃、第6对鳃、肠、胃、表皮、肌肉、肝胰腺、心脏、眼柄、血细胞,于液氮中冻存,用于RNA的提取。
1.6 盐度胁迫实验
随机挑取暂养 3d的三疣梭子蟹分为 2组,对照组(自然海水33)和低盐组(盐度11),每组设3个平行,每个平行100只,于3m3蟹池中进行实验。低盐组盐度配制方法为往自然海水中注入淡水,混匀,使其盐度降至11,用YSI盐度仪进行盐度校准,具体方法见隋延鸣等(2012)。各组分别于胁迫0、3、6、12、24、48和72h取鳃和肝胰腺,每个时间点分别取3只三疣梭子蟹,于液氮中冻存,用于后续试验RNA的提取。
1.7 PtCht基因mRNA Real-time PCR定量检测
Trizol试剂分别提取对照组和实验组三疣梭子蟹所取组织的总RNA,使用PrimeScript RT reagent Kit反转录合成cDNA。
根据已知的三疣梭子蟹管家基因 β-actin和拼接获得的PtCht基因cDNA全长序列,分别设计一对正反特异引物(β-actin-F和β-actin-R、QCht-F 和 QCht-R)(表 1-1),对不同时间中盐度胁迫的三疣梭子蟹鳃和肝胰腺中PtCht基因的相对表达量进行检测。使用SYBR Premix Ex TaqⅡ试剂在Applied Biosystems 7500 Real Time PCR仪上利用Real-time PCR方法对胁迫进程中以及各个组织中PtCht基因的表达情况进行分析。采用 2–ΔΔCt方法计算PtCht基因的相对表达量,使用SPSS 19.0软件进行单因素方差分析。
2 结果与分析
2.1 总RNA提取
利用 Trizol试剂提取获得三疣梭子蟹所取各个组织的总 RNA,经核酸定量仪检测,其 OD260/OD280均在1.9—2.0之间,总RNA有较高的纯度; 1.0%琼脂糖凝胶电泳检测后,呈现 3条清晰的条带,分别是28S RNA、18S RNA和5S rRNA,表明总RNA完整性较好,可用于进行后续实验。
2.2 PtCht基因的全长cDNA克隆
以三疣梭子蟹鳃、肝胰腺和肌肉组织的总 RNA混合物为模板,反转录得到 3′RACE 和 5′RACE 的cDNA第一链。以特异引物Cht-F和Cht-R分别与通用引物UPM配对,进行3′和5′RACE扩增,分别获得大小为857bp和630bp的cDNA片段。将两片段与已知表达序列标签(EST)拼接,得到三疣梭子蟹几丁质酶 Chitinase基因 cDNA全长序列,命名为PtCht,GenBank登录号: KM100753。该基因cDNA序列全长为3694bp,其中,开放阅读框(ORF)3039bp,5′端非编码区(5′-UTR)200bp,3′非编码区(3′-UTR)455bp。3'端存在加尾信号AATTAAA和多聚腺苷酸Poly A尾(图 1)。
2.3 PtCht基因序列分析及结构域预测
氨基酸序列分析可知,PtCht基因编码一个由1012个氨基酸组成的的蛋白质,分子量为 113.4kDa,理论等电点为6.289。氨基酸组成分析表明,PtCht蛋白带有负电的氨基酸残基为127个(Asp和Glu),带有正电的氨基酸残基为116个(Arg和Lys),疏水性氨基酸残基为316个(Ala,Ile,Leu,Phe,Trp和Val),极性氨基酸残基为253个(Asn,Cys,Gln,Ser,Thr和Tyr),不稳定系数为 36.25,亲水性总平均数为–0.458,属于稳定蛋白。结构域分析表明,其N端含有21个氨基酸组成的信号肽,InterProScan软件分析表明,PtCht序列存在两个几丁质酶第 18家族活性位点228FDGLDLDWE236,664FDGLDIDWE672。在 113—457氨基酸和549—894氨基酸处分别存在两个几丁质酶第 18家族催化结构域,在 946—1004氨基酸处存在含6-Cys的Type-2几丁质结合结构域(ChtBD2)。
2.4 PtCht基因的同源性分析
BLAST同源性分析三疣梭子蟹PtCht的氨基酸序列,得知三疣梭子蟹PtCht基因与亚洲玉米螟(Ostrinia furnacalis)Cht的同源性最高,为74%。与其它物种如翅膀带黑白斑的果蝇(Drosophila grimshawi)、冈比亚按蚊(Anopheles gambiastr.)、嗜凤梨果蝇(Drosophila ananassae)、黑翅果蝇(Drosophila persimilis)和甜菜夜蛾(Spodoptera exigua)的同源性分别为67%、73%、72%、72%和72%。通过与亚洲玉米螟(Ostrinia furnacalis)、小菜蛾(Plutella xylostella)和家蚕(Bombyx mori)等动物的 Cht氨基酸序列比对发现,几丁质酶第 18家族的催化结构域保守基序MotifⅠ、Ⅱ、Ⅲ、Ⅳ和含6个Cys的几丁质结合结构域ChtBD2在几个物种中都存在(图2)。
利用MEGA 5.0软件进行系统进化分析表明,第18家族Ⅲ型几丁质酶主要聚为两大类群: 昆虫类和甲壳动物,三疣梭子蟹(P. trituberculatus)与昆虫类黑腹果蝇(Drosophila melanogaster)、冈比亚按蚊(Anopheles gambiastr.)紧密聚为一支,之后的聚类顺序依次为与赤拟谷盗(Tribolium castaneum)、亚洲玉米螟(Ostrinia furnacalis)、小菜蛾(Plutella xylostella)、甜菜夜蛾(Spodoptera exigua)等(图 3)。
2.5 PtCht基因组织表达分析
2.5.1PtCht基因的组织表达分布 利用实时荧光定量 PCR分析了三疣梭子蟹PtCht基因在不同组织中的表达分布特征,结果表明,PtCht基因在第 1对鳃、第6对鳃、眼柄、肝胰腺、血细胞、心脏、肌肉、胃和肠中均有表达。在所有实验组织中,表达量最高的是表皮,依次是眼柄、胃、第6对鳃、第1对鳃、肠、肌肉、和心脏,而在肝胰腺和血细胞中的表达量非常低(图4)。
2.5.2PtCht基因在不同蜕皮周期中的表达分析三疣梭子蟹不同蜕皮周期中PtCht基因在表皮和眼柄中的相对表达量如图5所示。与间期相比,在表皮中,前期PtCht基因的相对表达量出现显著下调,后期PtCht基因的相对表达量出现显著上调; 与间期相比,在眼柄中,前期和后期PtCht基因的相对表达量呈整体上调趋势。
2.5.3PtCht基因在盐度胁迫中的表达分析 三疣梭子蟹盐度胁迫后PtCht基因在第六对鳃中的相对表达量如图 6所示。与对照组相比,低盐胁迫组中,PtCht基因的相对表达量于低盐胁迫 3h后出现显著下调,6h出现轻微的上调后,于12h达到最小值,相对表达量为对照组的0.30倍(P<0.05); 随后,PtCht的相对表达量开始显著上调,于24h达到最大值,相对表达量分别为对照组的2.27倍(P<0.05),随后相对表达量于48—72h再次出现下调现象(图6)。

图1 三疣梭子蟹PtCht基因cDNA全长核苷酸序列和其推导出的氨基酸序列Fig.1 Nucleotide sequence and deduced amino acid sequence of PtCht gene起始密码子(ATG)、加尾信号(AATTAAA)和终止密码子(TGA)用细线方框标出; 粗线方框内为几丁质酶第18家族保守基序; 信号肽以细下划线标出; ChtBD2结构域用粗下划线标出

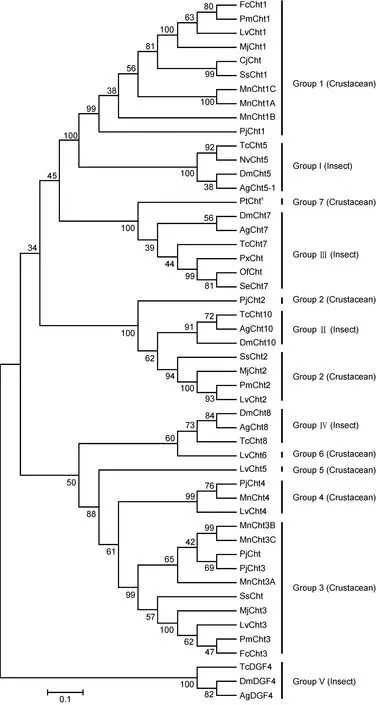
图3 三疣梭子蟹几丁质酶蛋白PtCht的系统进化树Fig.3 Phylogenetic analysis on chitinase protein of P. trituberculatus三疣梭子蟹几丁质酶蛋白PtCht; 各物种几丁质酶蛋白Cht GenBank登录号: 日本沼虾Cht1A (KF466274.1); 日本沼虾Cht1B(KF466275.1); 日本沼虾Cht1C (AHL24866.1); 日本沼虾Cht3A (KF466276.1); 日本沼虾Cht3B (KF466277.1); 日本沼虾Cht3C(KF466278.1); 日本沼虾Cht4 (KF466279.1); 中国对虾Cht1 (ABB85237.1); 中国对虾Cht3 (DQ000159.1); 斑节对虾Cht1 (AAD40313.1);斑节对虾Cht2 (ADG22164.1); 斑节对虾Cht3 (ADG22163.1); 锯缘青蟹Cht (ABY85409.1); 锯缘青蟹Cht1 (ACG60512.1); 锯缘青蟹Cht2 (ACZ53950.1); 日本蟳Cht (AFF59213.1); 日本囊对虾Cht1 (BAA12287); 日本囊对虾Cht2 (BAA14014); 日本囊对虾Cht3(BAA22854); 凡纳滨对虾Cht1 (EU883591.1); 凡纳滨对虾Cht2 (EU861222.1); 凡纳滨对虾Cht3 (AAN74647.1); 凡纳滨对虾Cht4(FJ888480.1); 凡纳滨对虾Cht5 (FJ888481.1); 凡纳滨对虾Cht6 (GQ916594.1); 日本仿长额虾Cht (AFC60660.1); 日本仿长额虾Cht1(JF694836.1); 日本仿长额虾Cht2 (JN982965.1); 日本仿长额虾Cht3 (JF694838.1); 日本仿长额虾Cht4 (JF694837.1); 黑腹果蝇Cht5(NP_650314.1); 黑腹果蝇Cht7 (NP_647768.2); 黑腹果蝇Cht8 (NP_611542.1); 黑腹果蝇Cht10 (EAA46011.1); 赤拟谷盗 Cht5(NP_001034524.1); 赤拟谷盗Cht7 (NP_001036035.1); 赤拟谷盗Cht8 (NP_001036067.1); 赤拟谷盗Cht10 (NP_001038091.1); 冈比亚按蚊Cht5-1 (HQ456129); 冈比亚按蚊Cht7 (XP_308858.4); 冈比亚按蚊Cht8 (XP_316448.2); 冈比亚按蚊Cht10 (XP_317398.3); 丽蝇蛹集金小蜂Cht5(NP_001155084.1); 亚洲玉米螟Cht (AGX32025.1); 小菜蛾Cht (AFI55112.1); 甜菜夜蛾Cht7 (AFM38213.1)
Real-time PCR检测了三疣梭子蟹盐度胁迫后不同时间点肝胰腺中PtCht基因的相对表达情况。与对照组相比,低盐胁迫后,PtCht基因的相对表达量于3 h出现下调现象,并于 12 h达到最小值,差异显著(P<0.05); 随后于12—48 h出现显著上调,并于48 h达到最大值,相对表达量为对照组的4.63倍(P<0.05),然后于48—72 h出现下调现象(图7)。
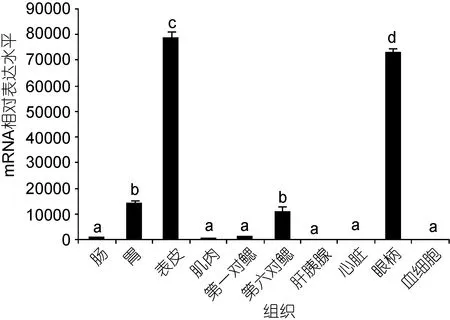
图4 三疣梭子蟹PtCht基因在不同组织中的表达分布Fig.4 Distribution of PtCht gene expression in different tissues of P. trituberculatus不同组织表达量均以与血细胞相比较的倍数表示,不同英文字母表示差异显著(P<0.05)
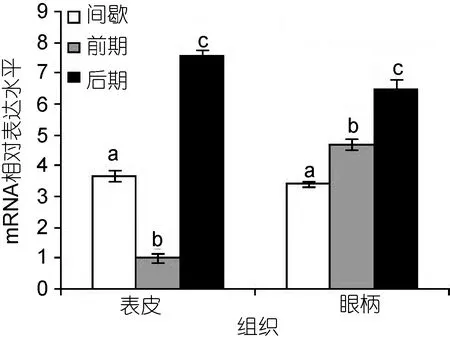
图5 不同蜕皮周期三疣梭子蟹表皮和眼柄中PtCht基因的表达情况Fig.5 Expression of PtCht gene in P. trituberculatus cuticle and eyestalk tissues during different molting cycle不同组织各蜕皮周期表达量均以与表皮前期相比较的倍数表示,同组织不同英文字母表示差异显著(P<0.05)
3 讨论
几丁质酶在甲壳动物的蜕皮过程中起着重要的生理作用,消化旧的外骨骼,将其变成可溶性物质,部分被机体重吸收,合成新的外骨骼(Huanget al,2010)。本实验克隆出一个全长为3694bp的几丁质酶基因PtCht。结构预测发现PtCht存在典型的几丁质酶结构,表明其具有几丁质分解活性,确为第 18家族几丁质酶家族成员(陈少波等,2004)。进化树分析表明其于已知的任何一种甲壳类几丁质酶成员均聚不到一类,反而与昆虫几丁质酶基因聚为一支,PtCht含有两个催化结构域,昆虫Ⅲ型几丁质酶也有两个催化结构域(Zhuet al,2008),广泛存在于昆虫、节肢动物中的Ⅲ型几丁质酶都归于第18 家族(Bormannet al,1999),暗示PtCht可能与昆虫一些几丁质酶基因同源。
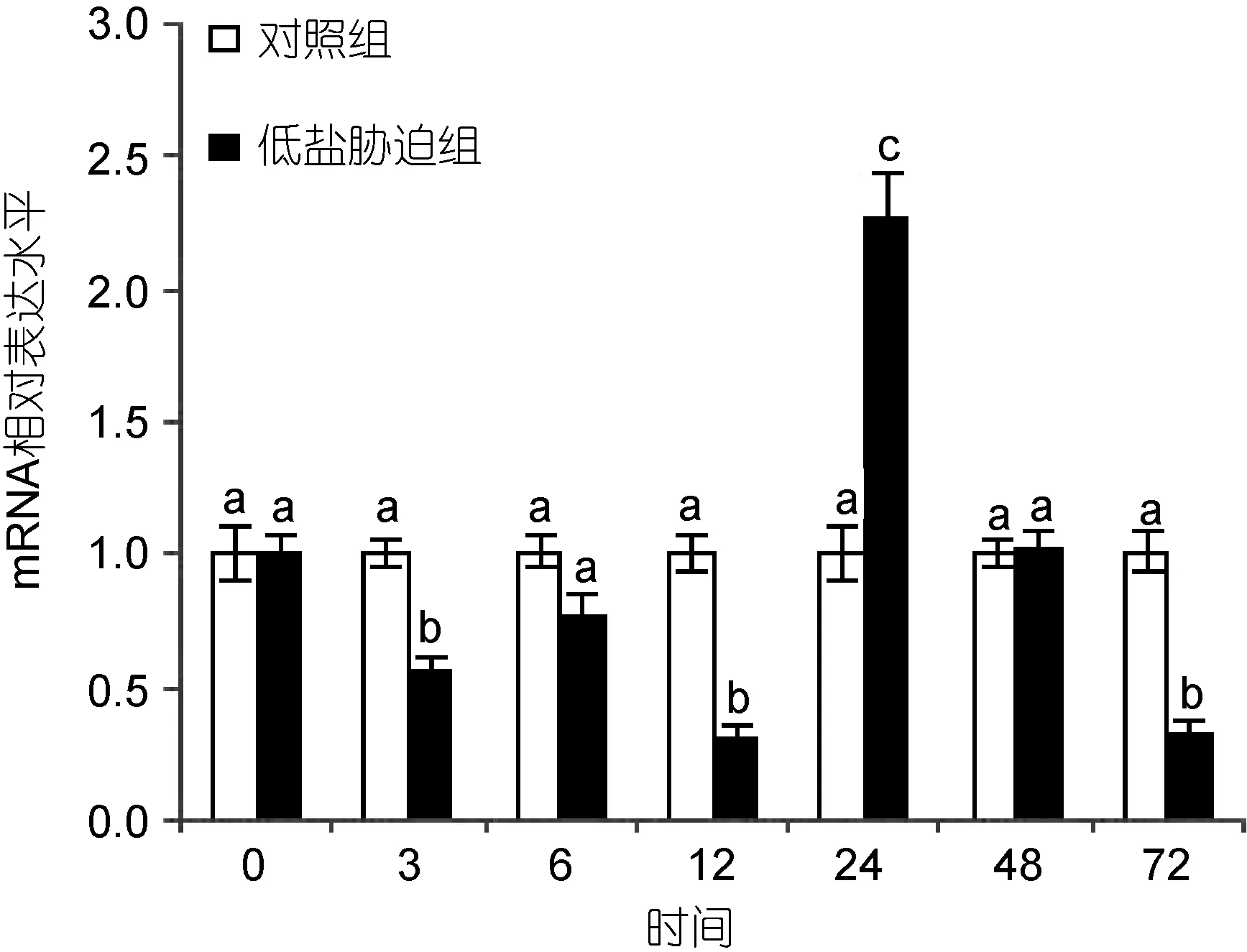
图6 低盐胁迫下三疣梭子蟹第六对鳃中PtCht基因的表达情况Fig.6 Expression of PtCht gene in P. trituberculatus gill tissue under low salinity stress低盐胁迫后不同时间点表达量均以与各时间点对照相比较的倍数表示,不同英文字母表示差异显著(P<0.05)
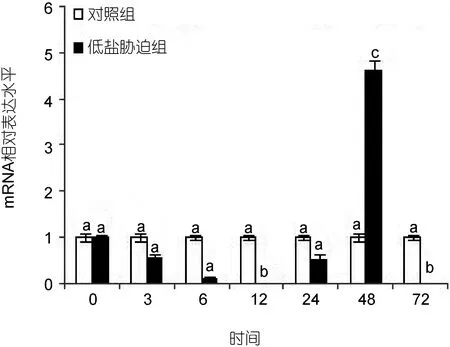
图7 低盐胁迫下PtCht基因在三疣梭子蟹肝胰腺中的表达变化Fig.7 Expression of PtCht in P. trituberculatus hepatopancreas tissue under low salinity stress低盐胁迫后不同时间点表达量均以与各时间点对照相比较的倍数表示,不同英文字母表示差异显著(P<0.05)
甲壳动物个体的生长发育伴随有周期性的蜕皮过程。眼柄-窦腺复合体为甲壳类重要的神经内分泌器官,是甲壳类蜕皮调控的重要器官,而甲壳类蜕皮首先需要降解旧的几丁质表皮,之后合成新的几丁质表皮,显而易见,眼柄和表皮是调控甲壳类蜕皮的关键组织。研究发现,甲壳动物几丁质酶在甲壳动物蜕皮周期中发挥作用,但基因不同,其在蜕皮过程中起的作用也可能不同(吕黎等,2011)。日本对虾(Penaeus japonicus)PjCht2(Watanabeet al,1996;Watanabeet al,1997)、斑节对虾(Penaeus monodon)PmCht1(Tanet al,2000)和PmCht2(Zou,2009)、中国对虾(Fenneropenaeus chinensis) (Priyaet al,2009)FcCht1和FcCht3、日本沼虾(Macrobrachium nipponense)(Zhanget al,2014)MnCht1A、MnCht1B和MnCht3B等几丁质酶基因的表达在甲壳动物不同蜕皮阶段中表现出一定的波动变化,这种变化和血淋巴中的蜕皮激素含量存在相关性(吕黎等,2011)。
本实验组织表达分布结果表明,PtCht基因在所研究组织中均有表达,在表皮和眼柄中表达量较高,说明该基因的主要功能很可能与三疣梭子蟹蜕皮相关。与间期相比,在表皮中,前期PtCht基因的相对表达量出现显著下调,后期PtCht基因的相对表达量出现显著上调,表达量达到最大; 与间期相比,在眼柄中,发现PtCht在蜕皮前期表达上调,到后期表达量达到最大,表明几丁质酶 mRNA的表达可能受到了蜕皮激素的调节而参与蜕皮过程。这与昆虫第十八家族几丁质酶 groupⅠ基因的时空表达特性基本一致,东亚飞蝗(Locusta migratoria)LmCht1也是在表皮中特异表达,并在昆虫发育后期表达量越来越高; 东亚飞蝗LmCht6在表皮中表达量也较高,随着昆虫发育表达量越来越高,到成虫阶段,表达量降低; RNAi结果表明LmCht6在东亚飞蝗蜕皮过程中发挥着非常重要的作用(李大琪等,2011)。作为昆虫Ⅲ型几丁质酶成员赤拟谷盗TcCht7和黑腹果蝇DmCht7可能在组织分化中起作用(Renet al,2005),纯化的壁虱Ⅲ型几丁质酶存在于新旧表皮之间,在蜕皮生理中发挥着一定的作用(Youet al,2003)。
盐度变化可以开启甲壳动物个体本身的渗透压调节机制,在一定阈值内,盐度的下降会导致甲壳动物蜕皮发育周期的缩短、蜕皮率增高,从而促进甲壳动物的生长(Brayet al,1994; Muet al,2005; 杨其彬等,2008; 王冲等,2010)。鳃又是三疣梭子蟹的重要器官,可以在水中进行气体交换,调节渗透压和调节离子平衡(韩晓琳等,2014)。比较本实验室转录组研究结果发现,该基因在不同盐度胁迫下三疣梭子蟹鳃中表达具有显著差异(Lvet al,2013),这些结果均暗示第六对鳃可能参与三疣梭子蟹渗透压调节进程。低盐胁迫下PtCht基因的表达在第六对鳃中总体呈先下调后稍微回升再下调到最低,再回升最后又下调的表达规律,在肝胰腺中呈现先下调后回升再下调的表达规律。表明PtCht基因参与了三疣梭子蟹低盐胁迫应答,在其透压调节进程中起到了一定的作用,提高了三疣梭子蟹抵抗盐度胁迫的能力,但是其具体的调节机制还有待于进一步的探索。
4 结论
首次克隆出了三疣梭子蟹PtCht基因cDNA序列全长,分析PtCht基因在三疣梭子蟹蜕皮周期和低盐胁迫进程中的表达规律,得知PtCht基因在三疣梭子蟹蜕皮发育和渗透压调节中都发挥了一定的作用,可初步认为PtCht参与了三疣梭子蟹的盐度适应性调节过程,进一步推测PtCht可能是盐度影响三疣梭子蟹蜕皮的重要调控因子之一,为深入研究几丁质酶在三疣梭子蟹和其它甲壳动物蜕皮机制中的功能提供了重要信息。
王 冲,姜令绪,王仁杰等,2010. 盐度骤变和渐变对三疣梭子蟹幼蟹发育和摄食的影响. 水产科学,29(9): 510—514
吕 黎,宁黔冀,2011. 甲壳动物几丁质酶基因结构与功能的研究进展. 生理科学进展,42(6): 457—459
李大琪,杜建中,张建琴等,2011. 东亚飞蝗几丁质酶家族基因的表达特性与功能研究. 中国农业科学,44(3):485—492
杨其彬,叶 乐,温为庚等,2008. 盐度对斑节对虾蜕壳、存活、生长和饲料转化率的影响. 南方水产,4(1): 16—21
沈 洁,朱冬发,胡则辉等,2011. 三疣梭子蟹蜕皮周期的分期. 水产学报,35(10): 1481—1487
陈少波,吴根福,2004. 几丁质酶研究进展. 科技通报,20(3):258—262
段亚飞,刘 萍,李吉涛等,2013. 脊尾白虾组织蛋白酶 L基因的克隆及其表达分析. 动物学研究,34(1): 39—46
徐文斐,刘 萍,李吉涛等,2014. 脊尾白虾(Exopalaemon carinicauda)vasa基因cDNA克隆及其在卵巢发育中的表达分析. 海洋与湖沼,45(3): 574—582
隋延鸣,高保全,刘 萍等,2012. 三疣梭子蟹“黄选 1号”盐度耐受性及适宜生长盐度分析. 大连海洋大学学报,27(5):398—401
韩晓琳,高保全,王好锋等,2014. 低盐胁迫对三疣梭子蟹鳃和肝胰腺显微结构及家系存活的影响. 渔业科学进展,35(1): 104—110
Arakane Y,Muthukrishnan S,2010. Insect chitinase and chitinase-like proteins. Cellular and Molecular Life Sciences,67(2): 201—216
Bormann C,Baier D,Hörr Iet al,1999. Characterization of a novel,antifungal chitin-binding protein fromStreptomyces tendaeTu901 that interferes with growth polarity. Journal of Bacteriology,181(24): 7421—7429
Bray W A,Lawrence A L,Leung-Turgillo J R,1994. The effect of salinity on growth and survival ofPenaeus vannamei,with observations on the interaction of IHHN virus and salinity. Aquaculture,122(2—3): 133—146
Huang Q S,Yan J H,Tang J Yet al,2010. Cloning and tissue expressions of seven chitinase family genes inLitopenaeus vannamei. Fish & Shellfish Immunology,29(1): 75—81
Lv J,Liu P,Wang Yet al,2013. Transcriptome analysis ofPortunus trituberculatusin response to salinity stress provides insights into the molecular basis of osmoregulation.PLoS One,8(12): e82155
Mu Y C,Wang F,Dong S Let al,2005. The effects of salinity fluctuation in different ranges on intermolt period and growth of juvenileFenneropenaeus chinensis. Acta Oceanologica Sinica,27(2): 122—126
Priya T A J,Li F H,Zhang J Qet al,2009. Molecular characterization and effect of RNA interference of retinoid X receptor (RXR) on E75 and chitinase gene expression in Chinese shrimpFenneropenaeus chinensis. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology,153(1): 121—129
Proespraiwong P,Tassanakajon A,Rimphanitchayakit V,2010.Chitinases from the black tiger shrimpPenaeus monodon:phylogenetics,expression and activities. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology,156(2): 86—96
Ren N,Zhu C M,Lee Het al,2005. Gene expression during Drosophila wing morphogenesis and differentiation.Genetics,171(2): 625—638
Rocha J,Garcia-Carreño F L,Muhlia-Almazán Aet al,2012.Cuticular chitin synthase and chitinase mRNA of whiteleg shrimpLitopenaeus vannameiduring the molting cycle.Aquaculture,330—333: 111—115
Tan S H,Degnan B M,Lehnert S A,2000. The Penaeus monodon chitinase 1 gene is differentially expressed in the hepatopancreas during the molt cycle. Marine Biotechnology,2(2): 126—135
Watanabe T,Kono M,1997. Isolation of a cDNA encoding a chitinase family protein from cuticular tissues of the Kuruma prawnPenaeus japonicus. Zoological Science,14(1): 65—68
Watanabe T,Kono M,Aida Ket al,1996. Isolation of cDNA encoding a putative chitinase precursor in the kuruma prawnPenaeus japonicus. Molecular Marine Biology and Biotechnology,5(4): 299—303
You M,Xuan X,Tsuji Net al,2003. Identification and molecular characterization of a chitinase from the hard tickHaemaphysalis longicornis. The Journal of Biological Chemistry,278: 8556—8563
Zhang J Q,Sun Y Y,Li F Het al,2010. Molecular characterization and expression analysis of chitinase(Fcchi-3) from Chinese shrimp,Fenneropenaeus chinensis.Molecular Biology Reports,37(4): 1913—1921
Zhang J,Zhang X,Arakane Yet al,2011. Comparative genomic analysis of chitinase and chitinase-like genes in the African malaria mosquito (Anopheles gambiae). PloS One,6(5):e19899
Zhang S Y,Jiang S F,Xiong Y Wet al,2014. Six chitinases from oriental river prawnMacrobrachium nipponense: cDNA characterization,classification and mRNA expression during post-embryonic development and moulting cycle.Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,167: 30—40
Zhu Q S,Arakane Y,Banerjee Det al,2008. Domain organization and phylogenetic analysis of the chitinase-like family of proteins in three species of insects. Insect Biochemistry and Molecular Biology,38(4): 452—466
Zou E M,2009. Effects of hypoxia and sedimentary naphthalene on the activity ofN-acetyl-β-glucosaminidase in the epidermis of the brown shrimp,Penaeus aztecus. Bulletin of Environment Contamination and Toxicology,82(5): 579—582
