杨树AP2/ERF转录因子家族生物信息学分析1)
2015-03-07赵金玲姚文静王升级姜廷波周博如
赵金玲 姚文静 王升级 姜廷波 周博如
(林木遗传育种国家重点实验室(东北林业大学),哈尔滨,150040)
责任编辑:潘 华。
杨树(Populus trichocarpa)既是广泛应用的绿化树种,也是生产纸浆材和一些木材产品的用材树种[1]。杨树的的基因组测序于2006年完成并公布。杨树以其生长速度快、遗传转化容易,使其成为研究植物抗旱、耐盐、抗低温等非生物胁迫的木本模式植物。在胁迫条件下,许多功能基因和调控基因被诱导表达,各种抗逆相关蛋白大量积累[2-3]。过量表达渗透复合物合成酶基因、转运蛋白基因、活性氧清除剂基因可明显提高转基因植物的抗旱性[4]。转录因子与其下游基因顺式结合元件结合,调控目标基因的表达,在应答环境胁迫中起着关键作用[5-8]。在拟南芥和水稻基因组各含有约1 300~1 600 的基因编码的转录因子。植物中转录因子按照其结构域的不同可分为MIKC、C2H2、WRKY、bZIP、MYB、SBP、HB、GATA、EIL、NAC 和AP2/ERF 等若干个家族。研究表明,过量表达应答逆境胁迫的转录因子基因,可以促进其下游抗逆相关基因的表达,提高转基因植物的抗胁迫能力[9-12]。AP2/ERF 基因家族是植物所特有的一类转录因子家族之一,含有一个或两个保守的AP2/ERF 结构域[13-14]。可以通过结合脱水应答元件DRE 和CRT 以应答干旱、低温、高盐胁迫[13],通过与GCC-box 及诱导元件结合应答胁迫[15]。当前,有关木本植物AP2/ERF 基因家族成员的功能分析研究还较少,本文利用生物信息学的方法分析杨树AP2/ERF 基因家族成员系统进化树、保守结构域、蛋白质理化性质、以及蛋白质二级和三级结构等,为进一步研究杨树转录因子功能提供参考。
1 材料与方法
1.1 数据库中杨树AP2/ERF 转录因子家族的搜索
从杨树基因组数据库(http://planttfdb.cbi.pku.edu.cn/)中得到210 条杨树AP2/ERF 转录因子家族蛋白的氨基酸序列,从拟南芥基因组数据库(https://www.arabidopsis.org/)中得到146 条AP2/ERF 转录因子家族的氨基酸序列。
1.2 分析方法
利用ClustalW 对210 个杨树AP2/ERF 转录因子家族家族蛋白和146 个拟南芥AP2/ERF 转录因子家族家族蛋白的氨基酸序列进行序列比对,默认参数设置。然后利用MEGA 6.0 构建邻接树(NJ),设置Bootstrap 为1000 次重复。
利用MEME 数据库(http://meme-suite.org/)提供的在线检索服务对杨树AP2/ERF 转录因子家族蛋白氨基酸序列的保守结构域进行分析。
根据杨树基因组数据库数据,确定210 个AP2/ERF 转录因子家族基因在19 条杨树染色体上的分布情况。
用ExPaSy 提供的在线Protparam 软件(http://web.expasy.org/protparam/)进行氨基酸数目、分子量、理论等电点、脂肪族氨基酸数和蛋白质疏水性分析。
利用DNAMAN6.0 软件预测210 条杨树AP2/ERF 转录因子家族蛋白的二级结构,并利用Swiss-Model 程序对15 个亚族中随机取的15 条蛋白进行三级结构同源建模。
2 结果与分析
2.1 杨树AP2/ERF 转录因子家族蛋白的分类
根据AP2/ERF 超家族蛋白氨基酸序列的相似性和AP2/ERF 结构域数量,将210 条杨树AP2/ERF 转录因子家族蛋白和146 条AP2/ERF 转录因子家族蛋白分为AP2、ERF、DREB 和RAV 亚家族等4 个亚家族及其他(Potri.002G246100)(表1)。AP2 亚家族蛋白含有两个重复的AP2/ERF 结构域,在调节植物生长发育过程中起着十分重要的作用[16-18];ERF 亚家族和DREB 亚家族蛋白含有一个单一的AP2/ERF 结构域[19],这两个亚家族的主要区别在AP2/ERF 结构域的第14 位和第19 位氨基酸残基,ERF 亚家族是丙氨酸和天冬氨酸,而DREB 亚家族第14 位和第19 位氨基酸分别是缬氨酸和谷氨酸[19]。DREB 亚家族成员可以识别干旱和冷诱导响应元件(DRE/CRT,A/GCCGAC),在植物抵抗非生物胁迫过程中起着非常重要的作用[20-21]。ERF 亚家族成员可以识别GCC 盒(AGCCGCC),在植物抵抗生物胁迫过程中发挥重要的作用[22]。RAV 亚家族蛋白含有一个AP2/ERF 域和一个B3 域,在乙烯响应[23]、油菜素内酯响应[24]、生物和非生物胁迫响应[25]过程中发挥着重要作用。
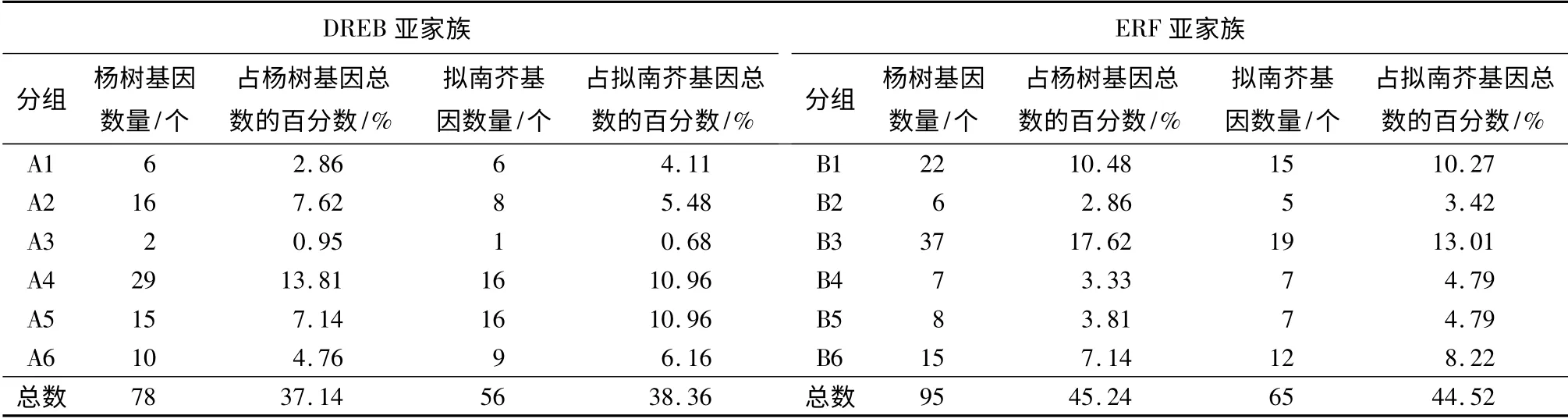
表1 杨树和拟南芥AP2/ERF 转录因子家族蛋白基本概况
2.2 杨树AP2/ERF 转录因子家族蛋白系统发生树构建
利用ClustalW 对210 个杨树AP2/ERF 转录因子家族蛋白和146 个拟南芥AP2/ERF 转录因子家族蛋白的氨基酸序列进行序列比对(如图1)。参照拟南芥分类方法将210 个杨树AP2/ ERF 转录因子家族蛋白分为DREB、ERF、AP2、RAV 等4 个亚家族,有一个蛋白单独分为一类。其中,DREB 亚家族蛋白可进一步分为6 个亚类分别为A1、A2、A3、A4、A5、A6;ERF 家族蛋白进一步也可分为6 个亚类分别为B1、B2、B3、B4、B5、B6。该结果与拟南芥AP2/ERF 家族蛋白的分类情况类似[19],说明杨树与拟南芥的AP2/ERF 转录因子家族蛋白的氨基酸序列之间虽有差异但也存在较高的保守性。
2.3 AP2/ERF 转录因子家族蛋白保守结构域
高度保守的AP2/ERF 结构域是AP2/ERF 家族蛋白的重要特征。AP2/ERF 家族蛋白的其他结构域在转录调控中也起很重要的作用。为确定不同结构域中氨基酸的保守程度,利用MEME 数据库提供的在线检索服务对所有杨树AP2/ERF 蛋白的氨基酸序列进行分析,结果表明在AP2/ERF 结构域在AP2/ERF 家族蛋白中保守程度最高,而其他结构域的保守程度依不同的亚类有所不同。在相同的亚类中除AP2/ERF 结构域保守性最高外,还存在一个或几个相对保守的其他结构域,这些相对保守结构域成为AP2/ERF 家族蛋白进一步分为不同亚族和和不同亚类的重要依据。在AP2/ERF 家族蛋白的相同亚族或相同亚类中,由于具有相同或相似的一个或多个结构域,使其在顺势作用元件结合、蛋白质与蛋白质之间相互作用中可能表现出类似的功能。

图1 杨树与拟南芥AP2/ERF 转录因子系统发生树
2.4 杨树AP2/ERF 转录因子家族基因物理定位
杨树AP2/ERF 基因在19 条染色体组上均有分布,但呈不均匀分布。位于1 号染色体上的AP2/ERF 基因最多,共有27 个基因;位于9 号染色体上的AP2/ERF 基因最少,只有2 个基因。不同亚家族AP2/ERF 基因在杨树染色体上也呈不均匀分布,ERF 亚家族基因多数在1 号染色体上分布最多,共有16 个基因;而在6、16、18 号染色体上没有分布。DREB 亚家族基因在6 号染色体上分布最多,共有14 个基因,在第11 号染色体上没有分布。AP2 亚家族基因的分布比较分散,但在第4、9、11、12、13、15、19 号染色体上没有分布。RAV 基因仅存在于3、6、8、10、18 号等5 条染色体上。单独基因Potri.002G246100 分布在第2 号染色体上。不同基因分布的染色体不同,其表达水平也可能不同。
2.5 杨树AP2/ERF 转录因子家族蛋白质理化性质
不同AP2/ERF 家族蛋白的氨基酸组成成分及理化性质不同,组成蛋白质的氨基酸数目在不同亚族之间差异较大。其中,AP2 亚族的平均氨基酸数目最多为504 个,DREB 亚族的平均氨基酸数目最少为248 个。总体看毛果杨AP2/ERF 家族蛋白质分子中富有含酸性氨基酸,蛋白的等电点大多数在酸性范围内,只有RAV 亚族的理论等电点大多在碱性范围内。脂肪系数即脂肪族氨基酸指数,该指数可以作为标定蛋白质热稳定性的一个指标,即脂肪族氨基酸指数高者蛋白质热稳定性高。总体来说AP2/ERF 家族的蛋白质热稳定性较高,RAV 亚家族的平均热稳定性最高,ERF-B4 组的平均热稳定性最低。不同亚族氨基酸序列间的疏水性(GRAVY值)虽存在差异,但均为负值,表明均属于亲水性蛋白(表2)。
2.6 AP2/ERF 转录因子家族蛋白二级结构和三级结构预测分析
蛋白质二级结构指它的多肽链中有规则重复的构象,二级结构主要有α-螺旋、β-折叠、β-转角和无规则卷曲。利用DNAMAN6.0 软件预测210 条杨树AP2/ERF 家族蛋白的二级结构,结果显示全部AP2/ERF 家族蛋白二级结构都以随机卷曲为主(图2)。
蛋白质的三级结构是指球状蛋白质的多肽链在二级结构的基础上相互配置而形成特定的构象。本研究利用Swiss-Model 程序对15 个亚族中随机取的15 条蛋白进行三级结构同源建模,结果显示同属ERF 亚族和DREB 亚族蛋白的三级结构总体相似,差异不明显。而AP2、RAV 和其他等3 个亚族蛋白的三级结构则各具特点,差异较大(图3)。主要表现在有些蛋白质的α-螺旋、β-折叠和无规则卷曲的长度都不同,可能会造成空间角度的不同,从而使它们的功能也有所不同。

表2 杨树AP2/ERF 家族蛋白理化性质
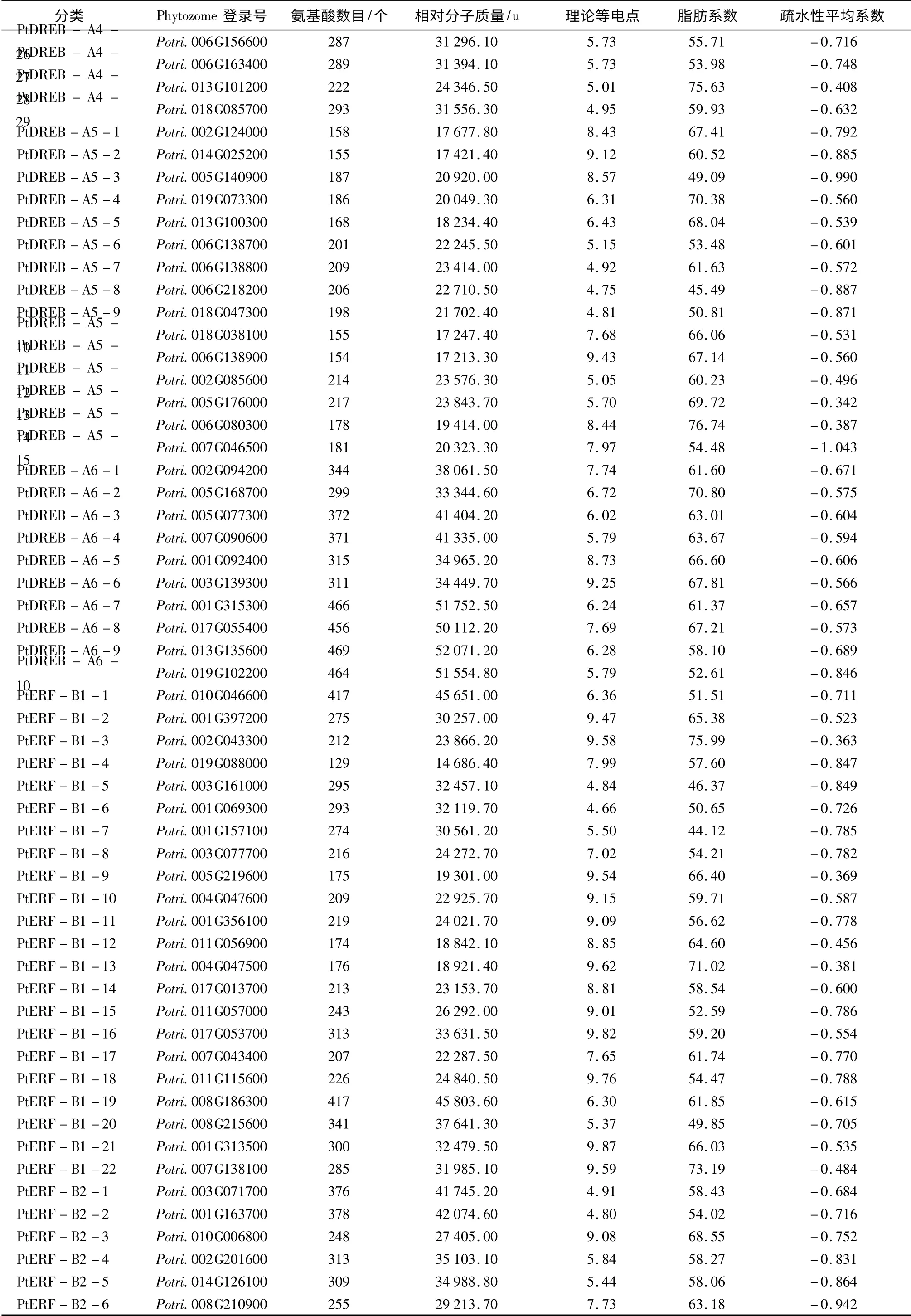
续(表2)

续(表2)
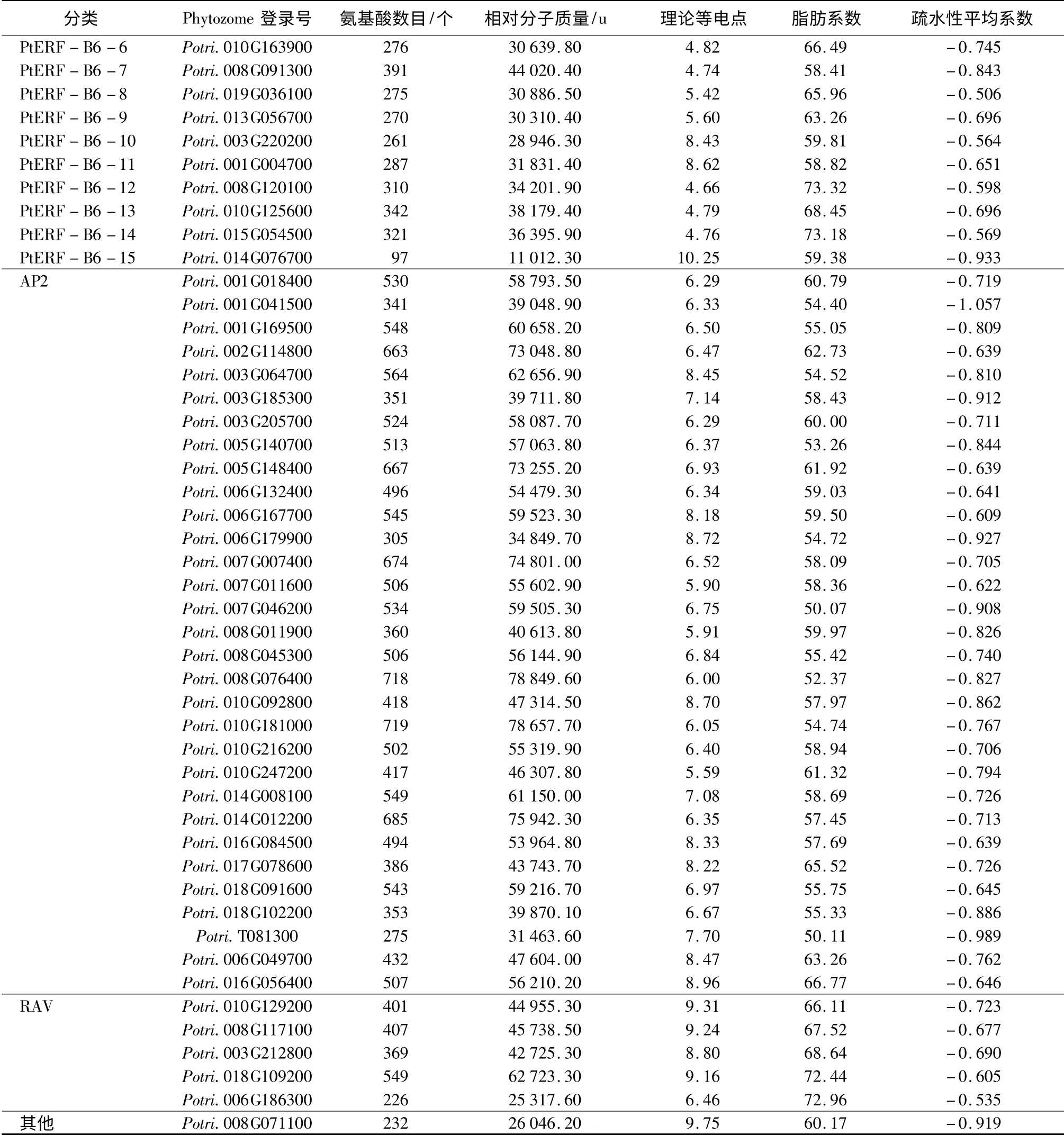
续(表2)
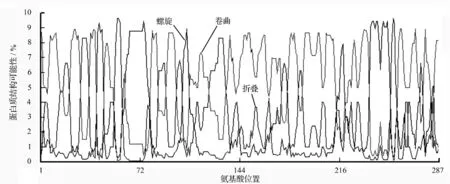
图2 杨树AP2/ERF 转录因子家族蛋白质二级结构预测
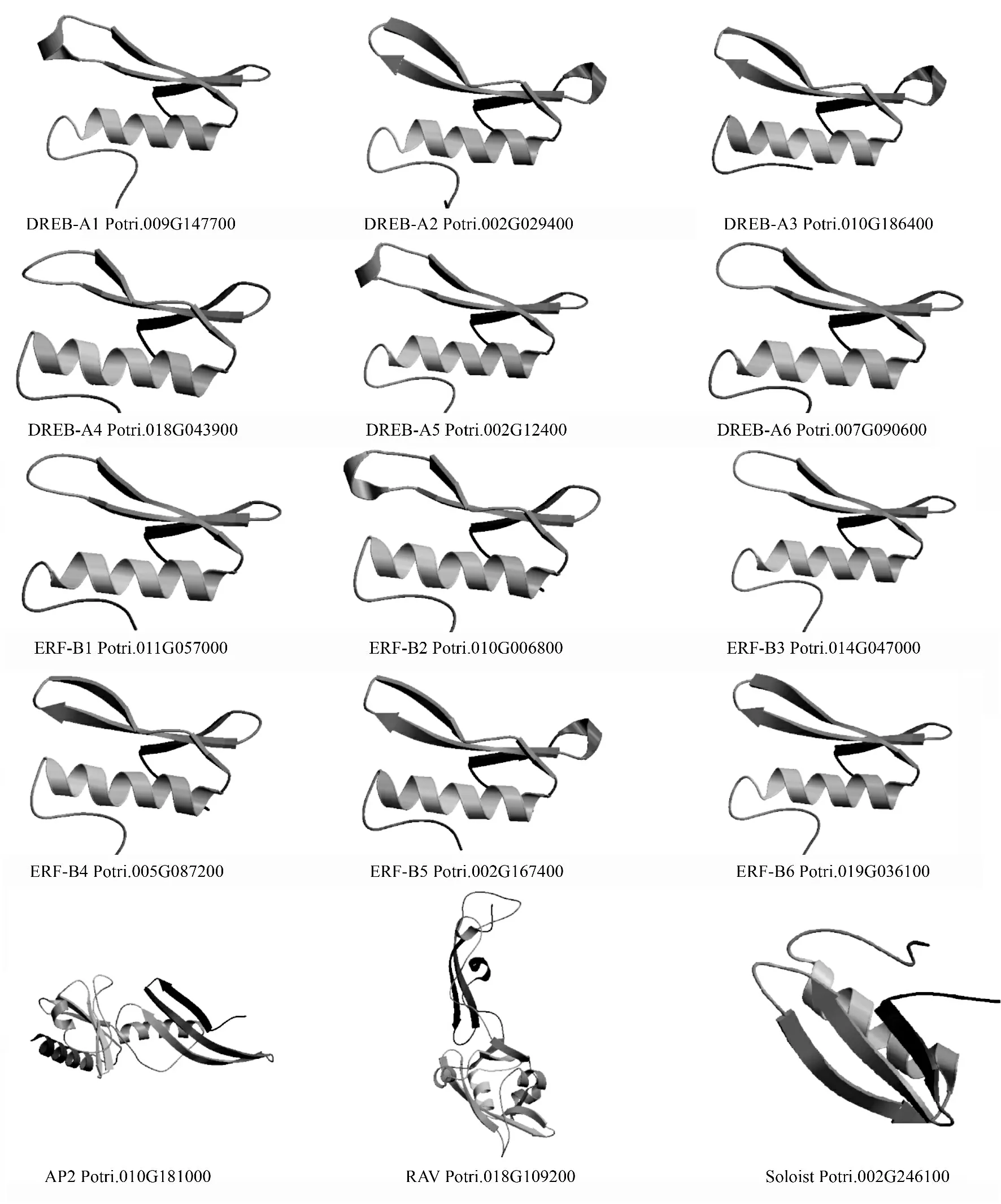
图3 AP2/ERF 蛋白质三级结构预测
3 结论与讨论
转录因子在植物的生长发育及应答外界环境变化过程中起着重要的调控作用,是调节各种生理活动的关键环节。近年来,人们从植物中分离出许多调控干旱、高盐、低温、激素、病原反应及发育相关的转录因子,有些转录因子过量表达可以增强植物对逆境胁迫的抵抗能力和适应能力[26-27]。AP2/ERF转录因子家族是植物特有的一类庞大的基因家族,家族成员之间的功能存在冗余,如何从已经测序的物种中筛选和鉴定出人们需要的抗逆基因具有一定的难度[28]。
杨树和拟南芥的AP2/ERF 转录因子家族蛋白的氨基酸序列之间虽有差异但也存在较高的保守性。依据含有AP2/ERF 结构域的数量及序列相似性均可以分为5 个亚家族,分别是AP2、ERF、DREB和RAV 及其他。210 个杨树AP2/ERF 转录因子均具有1~2 个AP2/ERF 结构域,其中的Gly-4、Arg-6、Glu-16、Trp-28、Leu-29、Gly-30 和Ala-38高度保守[13]。在对AP2/ERF 转录因子家族的AP2/ERF 结构域高度保守,其他结构域的保守程度依不同的亚类有所不同。但所有杨树AP2/ERF 转录因子家族蛋白均含有结构域2、3、4、5 或6 中的至少一个结构域,这些主要结构域可能在调节AP2/ERF 蛋白质的表达中发挥关键作用。比较AP2/ERF 转录因子家族蛋白进行理化性质,发现杨树AP2/ERF 家族成员蛋白质分子量介于11.01~53.12 ku,理论等电点分布介于4.62~10.25,多数蛋白表现偏酸性,平均值达到6.68。在对其他理化性质分析时发现疏水性虽存在差异,但均为负值,表明均属于亲水性蛋白。二级结构预测表明,杨树AP2/ERF 转录因子家族蛋白二级结构都以随机卷曲为主,在对15 个亚族中随机取的15 条蛋白进行三级结构同源建模,发现同一亚家族中各个组的蛋白质三级结构差异不明显,而不同亚家族之间的差异较大,由于空间角度的不同,可能使不同亚家族的成员之间具有不同的功能。蛋白质二级、三级结构等预测表明,亚类之间的AP2/ERF 转录因子家族蛋白的结构极为相似的,表明其有可能起着相近的生物学功能,但其在杨树中的具体功能还需要通过基因克隆及表达分析来验证。
AP2/ERF 转录因子家族基因中蕴含着许多抗逆关键基因,本研究以拟南芥AP2/ERF 转录因子家族基因为参考,通过生物信息学方法分析杨树AP2/ERF 基因家族的结构特征,有利于人们对该转录因子家族的了解,为进一步筛选和利用优良抗逆AP2/ERF 转录因子基因提供分类依据,为进一步研究该家族基因的功能提供参考。
[1] Gail Taylor.Populus:arabidopsis for forestry do we need a model tree[J].Annals of Botany,2002,90(6):681-689.
[2] Motoaki Seki,Mari Narusaka,Junko Ishida,et al.Monitoring the expression profiles of 7000 Arabidopsis genes under drought,cold and high-salinity stresses using a full-length cDNA microarray[J].Plant Journal,2002,31(3):279-292.
[3] Bhatnagar Mathur Pooja,Vadez V,Sharma Kiran K.Transgenic approaches for abiotic stress tolerance in plants:retrospect and prospects[J].Plant Cell Reports,2008,27(3):411-424.
[4] Taishi Umezawa,Masanori Okamoto,Tetsuo Kushiro,et al.CYP707A3,a major ABA 8’-hydroxylase involved indehydration and rehydration response in Arabidopsis thaliana[J].The Plant Journal,2006,46(2):171-182.
[5] Yanagisawa Shuichi.Dof domain proteins:plant-specific transcription factors associated with diverse phenomena unique to plants[J].Plant and Cell Physiology,2004,45(4):386-391.
[6] Junya Mizoi,Kazuo Shinozaki,Kazuko Yamaguchi Shinozaki.AP2/ERF family transcription factors in plant abiotic stress responses[J].Biochimica Et Biophysica Acta,2012,1819(2):86-96.
[7] Kazuo Nakashima,Hironori Takasaki,Junya Mizoi,et al.NAC transcription factors in plant abiotic stress responses[J].Biochimica Et Biophysica Acta,2012,1819(2):97-103.
[8] Rushton Deena L,Tripathi Prateek,Rabara Roel C,et al.WRKY transcription factors:key components in abscisic acid signalling[J].Plant Biotechnol J,2012,10(1):2-11.
[9] Kasuga M,Liu Q,Miura S,et al.Improving plant drought,salt,and freezing tolerance by gene transfer of a single stress-inducible transcription factor[J].Nature Biotechnology,1999,17(3):287-291.
[10] Liu Q,Kasuga M,Sakuma Y,et al.Two transcription factors,DREB1 and DREB2,with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought-and low-temperature-responsive gene expression,respectively,in Arabidopsis[J].The Plant Cell,1998,10(8):1391-1406.
[11] Gilmour S J,Sebolt A M,Salazar M P,et al.Overexpression of the Arabidopsis CBF3 transcriptional activator mimics multiple biochemical changes associated with cold acclimation[J].Plant Physiology,2000,124(4):1854-1865.
[12] Sakuma Yoh,Maruyama Kyonoshin,Osakabe Yuriko,et al.Functional analysis of an Arabidopsis transcription factor,DREB2A,involved in drought-responsive gene expression[J].The Plant Cell,2006,18(5):1292-1309.
[13] Nakano Toshitsugu,Suzuki Kaoru,Fujimura Tatsuhito,et al.Genome-Wide analysis of the ERF gene family in arabidopsis and rice[J].Plant Physiology,2006,140(2):411-432.
[14] Velasco Riccardo,Zharkikh Andrey,Affourtit Jason,et al.The genome of the domesticated apple (Malus × domestica Borkh.)[J].Nature Genetics,2010,42(10):833-839.
[15] Lorenzo Oscar,Piqueras Raquel,Sánchez-Serrano Jose J,et al.Ethylene response factor integrates signals from ethylene and jasmonate pathways in plant defense[J].The Plant Cell,2003,15(1):165-178.
[16] Elliott R C,Betzner A S,Huttner E,et al.Aintegumenta,an APETALA2-like gene of Arabidopsis with pleiotropic roles in ovule development and floral organ growth[J].The Plant Cell,1996,8(2):155-168.
[17] Chuck G,Meeley R B,Hake S.The control of maize spikelet meristem fate by the APETALA2-like gene indeterminate spikelet1[J].Genes & Development,1998,12(8):1145-1154.
[18] Boutilier Kim,Offringa Remko,Sharma Vijay K,et al.Ectopic expression of baby boom triggers a conversion from vegetative to embryonic growth[J].The Plant Cell,2002,14(8):1737-1749.
[19] Yoh Sakuma,Qiang Liu,Joseph G Dubouzet,et al.DNA-Binding specificity of the ERF/AP2 domain of arabidopsis DREBs,transcription factors involved in dehydration and cold-inducible gene expression[J].Biochemical and Biophysical Research Communications,2002,290(3):998-1009.
[20] Yamaguchi Shinozaki K,Shinozaki K.A novel cis-acting element in an Arabidopsis gene is involved in responsiveness to drought,low-temperature,or high-salt stress[J].The Plant Cell,1994,6(2):251-264.
[21] Thomashow Michael F.Plant cold acclimation:freezing tolerance genes and regulatory mechanisms[J].Annual Review of Plant Physiology and Plant Molecular Biology,2004,50:571-599.
[22] Hao D,Ohme Takagi M,Sarai A.Unique mode of GCC box recognition by the DNA-binding domain of ethylene-responsive element-binding factor (ERF domain)in plant[J].The Journal of Biological Chemistry,1998,273:26857-26861.
[23] Alonso José M,Stepanova Anna N,Leisse Thomas J,et al.Genome-wide insertional mutagenesis of Arabidopsis thaliana[J].Science,2003,301:653-657.
[24] Hu Yuxin,Wang Yongxong,Liu Xinfang,et al.Arabidopsis RAV1 is down-regulated by brassinosteroid and may act as a negative regulator during plant development[J].Cell Research,2004,14(1):8-15.
[25] Kee Hoon Sohn,Sung Chul Lee,Ho Won Jung,et al.Expression and functional roles of the pepper pathogen-induced transcription factor RAV1 in bacterial disease resistance,and drought and salt stress tolerance[J].Plant Molecular Biology,2006,61(6):897-915.
[26] 刘翠芳,邹杰,陈信波.DREB 转录因子与植物非生物胁迫抗性研究进展[J].生物技术通报,2010(10):26-30,48.
[27] 庄静,彭日荷,高峰,等.沪油15 中两个AP2/ERF-B1 亚族转录因子的克隆与分析[J].核农学报,2009,23(3):435-441,453.
[28] Johan Memelink.Regulation of gene expression by jasmonate hormones[J].Phytochemistry,2009,70(13):1560-1570.
